文章信息
- 房旭, 刘维贤
- FANG Xu, LIU Weixian
- 环状RNA hsa_circ_0009043在口腔鳞状细胞癌中的表达及临床意义
- Expression and clinical significance of circular RNA hsa_circ_0009043 in oral squamous
- 中国医科大学学报, 2022, 51(7): 633-637
- Journal of China Medical University, 2022, 51(7): 633-637
-
文章历史
- 收稿日期:2022-03-01
- 网络出版时间:2022-06-21 13:42
口腔鳞状细胞癌(oral squamous cell carcinoma,OSCC)占口腔癌的95%以上[1],具有很强的复发、侵袭、转移能力[2]。尽管近年来在诊断和治疗方面取得了一些进展,但OSCC患者的5年生存率仍低于50%[3],未能早期诊断是导致高死亡率的重要原因。因此,探索OSCC的发生机制是指导OSCC诊断和治疗的重点。
环状RNA(circular RNA,circRNA)是一种内源性非编码RNA,由前体mRNA反向剪接产生,具有可区分的单链,共价闭环,缺乏5’-3’极性等特点[4]。这种独特的结构使circRNA广泛分布于细胞质中,并对外切酶更具抵抗力,比线性RNA更稳定[5]。研究[6-7]表明,circRNA作为微RNA(microRNA,miRNA)分子的海绵具有巨大的潜力,以互补的碱基配对方式抑制miRNA活性,从而促进下游靶基因的翻译和稳定性。这种circRNA/miRNA/mRNA信号轴已被证明可在各种生理和病理条件下发挥多种功能[8-10]。已有研究[11]通过基因筛选发现,与子宫内膜癌旁组织相比,子宫内膜癌中hsa_circ_0009043表达显著下调,但它在OSCC中的表达情况尚不明确。
本研究拟探讨hsa_circ_0009043在OSCC组织中的表达情况,并结合相关病理因素分析其在OSCC发生、发展中的临床意义。
1 材料与方法 1.1 材料 1.1.1 组织标本收集2018年6月至2020年12月于中国医科大学附属盛京医院口腔颌面科接受手术治疗的48例OSCC患者的肿瘤及癌旁组织。所有患者术前均未接受过放化疗等治疗,且均经病理学检查确诊为OSCC。所有组织离体后立即置于液氮中,冷冻保存备用。所有患者均签署知情同意书。本研究获得中国医科大学附属盛京医院伦理委员会批准。
1.1.2 细胞OSCC细胞系SCC-9、SCC-15、CAL-27购自上海西宝生物科技有限公司。正常人口腔角质细胞(human oral keratinocytes,HOK)购自上海酶联科技有限公司。
1.2 方法 1.2.1 实时定量聚合酶链式反应(quantitative real-time polymerase chain reaction,qRT-PCR)用TRIzol试剂盒获得组织和细胞的总RNA。采用RNA反转录试剂盒将RNA反转录为cDNA。采用Light Cycler 480 ⅡReal-Time PCR系统进行qRT-PCR检测。GAPDH作为内参照。GAPDH正向引物序列为5’-CAGGAG GCATTGCTGATGAT-3’,反向引物序列为5’-GAAG GCTGGGGCTCATTT-3’;hsa_circ_0009043正向引物序列为5’-TGGTGGACAACATCCCCAAG-3’,反向引物序列为5’-ACTTGATTTTGCTTCATGGCAGT-3’。
1.2.2 hsa_circ_0009043功能富集分析及circRNA/miRNA/mRNA网络构建hsa_circ_0009043功能富集分析及circRNA/miRNA/mRNA网络构建:对hsa_circ_0009043进行基因本体(Gene Ontology,GO)和京都基因与基因组百科全书(Kyoto Encyclopedia of Genes and Genomes,KEGG)富集通路分析,P < 0.05表示富集结果显著。利用Mireap软件、Miranda(http://www.microrna.org/microrna/home.do)和TargetScan(http://www.targetscan.org)数据库预测下游miRNA,并利用miRTarBase数据库(http://mirtarbase.mbc.nctu.edu.tw/index.html)预测miRNA靶标mRNA,进而构建circRNA/miRNA/mRNA网络。
1.3 统计学分析采用SPSS 23.0和GraphPad Prism 7.0软件进行统计分析。采用t检验比较OSCC组织与配对的癌旁组织中hsa_circ_0009043表达水平的差异。通过非参数检验评估hsa_circ_0009043表达与临床病理特征之间的关系。通过受试者操作特征(receiver operating characteristic,ROC)曲线评估hsa_circ_0009043的诊断价值。每个实验重复3次。P < 0.05为差异有统计学意义。
2 结果 2.1 hsa_circ_0009043在组织和细胞中的表达情况qRT-PCR结果显示,OSCC细胞系SCC-9和CAL-27中hsa_circ_0009043表达水平较正常HOK显著降低(P < 0.01;P < 0.001),见图 1A;OSCC组织中hsa_circ_0009043表达水平[5.117(3.722~6.933)]较癌旁组织[3.849(2.945~5.088)]显著降低(P < 0.001),见图 1B。
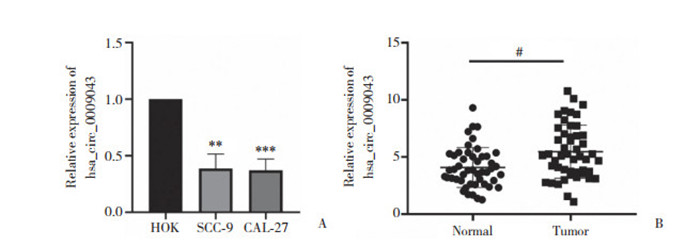
|
| A, compared with HOK cells, hsa_circ_0009043 expression was decreased in OSCC cell lines; B, the expression level of hsa_circ_0009043 in OSCC tissues was lower than that in adjacent tissues. ** P < 0.001, *** P < 0.001 vs HOK. # P < 0.001. 图 1 hsa_circ_0009043在OSCC组织及细胞中低表达 Fig.1 Decreased expression of hsa_circ_0009043 in OSCC tissues and OSCC cells |
2.2 hsa_circ_0009043表达水平与临床病理特征的相关性
如表 1所示,临床病理特征分析结果显示,hsa_circ_0009043的表达水平与肿瘤大小和淋巴结转移相关(P = 0.01,P = 0.037),与是否吸烟、饮酒等则无相关性。
| Characteristics | n | circRNA expression | P |
| Sex | 0.482 | ||
| Male | 38 | 0.485 2(0.142 3-1.166 0) | |
| Female | 10 | 0.242 9(0.090 2-0.626 9) | |
| Age | 0.555 | ||
| ≥ 60 years | 30 | 0.381 5(0.142 3-1.226 8) | |
| <60 years | 18 | 0.492 6(0.073 0-0.829 4) | |
| Alcohol abuse | 0.775 | ||
| Yes | 23 | 0.483 0(0.148 0-0.880 7) | |
| No | 25 | 0.298 0(0.088 6-0.991 2) | |
| Smoking | 0.562 | ||
| Yes | 28 | 0.485 2(0.130 8-0.863 6) | |
| No | 20 | 0.321 7(0.106 5-1.055 5) | |
| Tumor size | 0.010 | ||
| <4 cm | 25 | 0.664 3(0.292 3-1.719 8) | |
| ≥4 cm | 23 | 0.265 5(0.075 7-0.502 3) | |
| TNM stage | 0.089 | ||
| Ⅰ-Ⅱ | 11 | 0.664 3(0.298 0-1.892 1) | |
| Ⅲ-Ⅳ | 37 | 0.345 5(0.089 6-0.807 6) | |
| Differentiation | 0.161 | ||
| Poor/moderate | 43 | 0.417 5(0.125 1-0.812 3) | |
| Well | 5 | 0.880 7(0.254 9-2.776 7) | |
| Lymph node metastasis | 0.037 | ||
| Yes | 31 | 0.280 0(0.075 7-0.502 3) | |
| No | 17 | 0.812 3(0.548 4-1.739 7) |
2.3 hsa_circ_0009043对OSCC的诊断价值
ROC曲线评估结果显示,hsa_circ_0009043的ROC曲线下面积(area under curve,AUC)为0.6769(P < 0.001,95%CI:0.570 0~0783 7),见图 2。
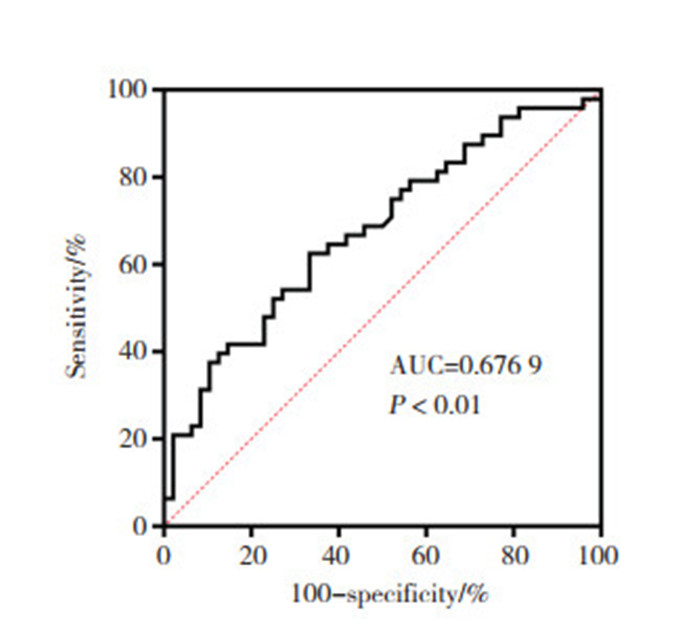
|
| 图 2 hsa_circ_0009043诊断OSCC的ROC曲线 Fig.2 The ROC curve of OSCC diagnosis by hsa_circ_0009043 |
2.4 circRNA/miRNA/mRNA相互作用网络预测
为了探索hsa_circ_0009043的潜在功能,对目标基因进行了GO及KEGG富集分析。GO富集分析发现,在分子功能、细胞组分及生物学过程中富集最强烈的分别是磷脂酰肌醇3-磷酸酶活性、被膜小泡及血管生成,见图 3A。KEGG富集分析的前10个条目显示hsa_circ_0009043可能与多种肿瘤存在相关性,如肝癌、非小细胞肺癌等,见图 3B。为了进一步确定hsa_circ_0009043的分子机制,构建了circRNA/miRNA/mRNA相互作用网络。结果显示,hsa_circ_0009043可能与hsa-miR-148b-3p、hsa-miR-148a-3p、hsa-miR-7-5p和hsa-miR-152-3p相互作用,并间接调控61个靶基因,见图 3C。
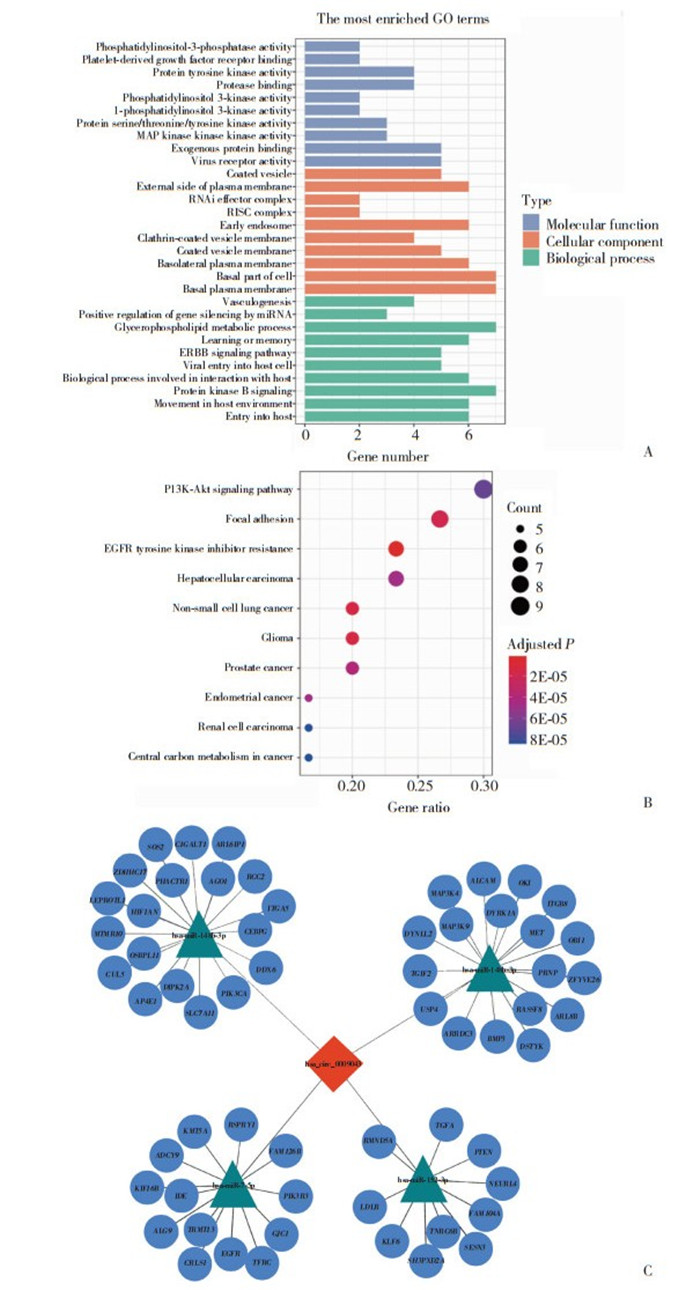
|
| A, GO analysis; B, KEGG pathway enrichment analysis; C, hsa_circ_0009043/miRNA/mRNA regulatory network. 图 3 hsa_circ_0009043的潜在功能分析 Fig.3 Potential functional analysis of hsa_circ_0009043 |
3 讨论
circRNA是一种可以调节基因表达的非编码RNA[12],与多种肿瘤的发生发展机制密切相关[13-15]。有研究[16]发现,circRNA的差异表达与肿瘤的远处转移、TNM分期等临床病理特征显著相关。因此,circRNA的出现为探索肿瘤发病机制和寻找癌症的潜在生物标志物提供了新的方向。与其他恶性肿瘤一样,OSCC的发生发展也是涉及编码和非编码基因的复杂生物学过程[17]。识别临床相关的OSCC特异性circRNA,并检测其功能及对OSCC的调控作用是了解该疾病的关键。
本研究首次发现在OSCC组织和细胞系中,hsa_circ_0009043表达显著下调。对临床病理相关资料的分析发现,hsa_circ_0009043的表达水平与肿瘤大小和淋巴结转移相关。ROC曲线结果显示,hsa_circ_0009043的表达与OSCC诊断具有相关性。提示hsa_circ_0009043是一种对OSCC的早期诊断和治疗具有一定价值的新型生物标志物。
本研究首次发现在OSCC组织和细胞系中,hsa_circ_0009043表达显著下调。对临床病理相关资料的分析发现,hsa_circ_0009043的表达水平与肿瘤大小和淋巴结转移相关。ROC曲线结果显示,hsa_circ_0009043的表达与OSCC诊断具有相关性。提示hsa_circ_0009043是一种对OSCC的早期诊断和治疗具有一定价值的新型生物标志物。
为了探索hsa_circ_0009043与OSCC发生、发展的关系,本研究还进行了GO、KEGG富集分析,结果显示,EGFR tyrosine kinase inhibitors与hsa_circ_0009043相关性最强,与已有研究[18]结果一致。以上结果均证实了hsa_circ_0009043在OSCC发生发展中的重要作用。
研究[19-20]表明,竞争性内源RNA(competing endo- genous RNA,ceRNA)在肿瘤细胞的扩张、侵袭和转移中发挥着重要作用。circRNA可作为ceRNA参与调控肿瘤的ceRNA网络[21-22]。本研究中,利用Target Scan、miRTarBase等数据库,构建了hsa_circ_0009043/miRNA/mRNA网络以预测hsa_circ_0009043可能发挥的作用机制,发现hsa_circ_0009043可能与hsa-miR-148b-3p、hsa-miR-148a-3p、hsa-miR-7-5p和hsa-miR-152-3p相互作用,并间接调控61个靶基因。说明hsa_circ_0009043可通过miRNA的海绵效应促进癌症的进展。
综上所述,本研究首次发现hsa_circ_0009043在OSCC细胞系及组织中表达水平显著下降,并与OSCC的临床病理特征及诊断相关。同时,通过生物信息学分析,对hsa_circ_0009043参与OSCC的可能的分子机制进行了预测。因此,hsa_circ_0009043具有成为OSCC生物标志物的潜力,但其具体作用机制仍有待进一步研究。
| [1] |
SIEGEL RL, MILLER KD, JEMAL A. Cancer statistics, 2017[J]. CA A Cancer J Clin, 2017, 67(1): 7-30. DOI:10.3322/caac.21387 |
| [2] |
OOSTING SF, HADDAD RI. Best practice in systemic therapy for head and neck squamous cell carcinoma[J]. Front Oncol, 2019, 9: 815. DOI:10.3389/fonc.2019.00815 |
| [3] |
GLAZER CA, CHANG SS, HA PK, et al. Applying the molecular biology and epigenetics of head and neck cancer in everyday clinical practice[J]. Oral Oncol, 2009, 45(4-5): 440-446. DOI:10.1016/j.oraloncology.2008.05.013 |
| [4] |
KRISTENSEN LS, ANDERSEN MS, STAGSTED LVW, et al. The biogenesis, biology and characterization of circular RNAs[J]. Nat Rev Genet, 2019, 20(11): 675-691. DOI:10.1038/s41576-019-0158-7 |
| [5] |
CHEN G, CUI JW, WANG L, et al. Genome-wide identification of circular RNAs in Arabidopsis thaliana[J]. Front Plant Sci, 2017, 8: 1678. DOI:10.3389/fpls.2017.01678 |
| [6] |
CAO MJ, ZHANG LS, WANG JH, et al. Identifying circRNA-associa- ted-CeRNA networks in retinal neovascularization in mice[J]. Int J Med Sci, 2019, 16(10): 1356-1365. DOI:10.7150/ijms.35149 |
| [7] |
CHEN LL. The expanding regulatory mechanisms and cellular functions of circular RNAs[J]. Nat Rev Mol Cell Biol, 2020, 21(8): 475-490. DOI:10.1038/s41580-020-0243-y |
| [8] |
SU Y, LV XR, YIN W, et al. CircRNA Cdr1as functions as a competitive endogenous RNA to promote hepatocellular carcinoma progression[J]. Aging, 2019, 11(19): 8183-8203. DOI:10.18632/aging.102312 |
| [9] |
PENG P, ZHANG B, HUANG JY, et al. Identification of a circRNA-miRNA-mRNA network to explore the effects of circRNAs on pathogenesis and treatment of spinal cord injury[J]. Life Sci, 2020, 257: 118039. DOI:10.1016/j.lfs.2020.118039 |
| [10] |
GUO ZY, ZHAO LY, JI SH, et al. CircRNA-23525 regulates osteogenic differentiation of adipose-derived mesenchymal stem cells via miR-30a-3p[J]. Cell Tissue Res, 2021, 383(2): 795-807. DOI:10.1007/s00441-020-03305-7 |
| [11] |
YE F, TANG QL, MA F, et al. Analysis of the circular RNA transcriptome in the grade 3 endometrial cancer[J]. Cancer Manag Res, 2019, 11: 6215-6227. DOI:10.2147/CMAR.S197343 |
| [12] |
VO JN, CIESLIK M, ZHANG YJ, et al. The landscape of circular RNA in cancer[J]. Cell, 2019, 176(4): 869-881.e13. DOI:10.1016/j.cell.2018.12.021 |
| [13] |
ZHANG X, WANG S, WANG HX, et al. Circular RNA circNRIP1 acts as a microRNA-149-5p sponge to promote gastric cancer progression via the AKT1/mTOR pathway[J]. Mol Cancer, 2019, 18(1): 20. DOI:10.1186/s12943-018-0935-5 |
| [14] |
CHEN SJ, HUANG V, XU X, et al. Widespread and functional RNA circularization in localized prostate cancer[J]. Cell, 2019, 176(4): 831-843.e22. DOI:10.1016/j.cell.2019.01.025 |
| [15] |
FAN LY, CAO Q, LIU J, et al. Circular RNA profiling and its potential for esophageal squamous cell cancer diagnosis and prognosis[J]. Mol Cancer, 2019, 18(1): 16. DOI:10.1186/s12943-018-0936-4 |
| [16] |
WU P, MO YZ, PENG M, et al. Emerging role of tumor-related functional peptides encoded by lncRNA and circRNA[J]. Mol Cancer, 2020, 19(1): 22. DOI:10.1186/s12943-020-1147-3 |
| [17] |
CHAKRABORTY D, NATARAJAN C, MUKHERJEE A. Advances in oral cancer detection[J]. Adv Clin Chem, 2019, 91: 181-200. DOI:10.1016/bs.acc.2019.03.006 |
| [18] |
CAI JY, SUN M, GE X, et al. EGFR tyrosine kinase inhibitors differentially affect autophagy in head and neck squamous cell carcinoma[J]. Biochem Biophys Res Commun, 2017, 486(4): 1027-1033. DOI:10.1016/j.bbrc.2017.03.157 |
| [19] |
LIU Y, KHAN S, LI L, et al. Molecular mechanisms of thyroid cancer: a competing endogenous RNA(CeRNA) point of view[J]. Biomed Pharmacother, 2022, 146: 112251. DOI:10.1016/j.biopha.2021.112251 |
| [20] |
MORENO-GARCÍA L, LÓPEZ-ROYO T, CALVO AC, et al. Competing endogenous RNA networks as biomarkers in neurodegenerative diseases[J]. Int J Mol Sci, 2020, 21(24): 9582. DOI:10.3390/ijms21249582 |
| [21] |
CHAN JJ, TAY Y. Noncoding RNA: RNA regulatory networks in cancer[J]. Int J Mol Sci, 2018, 19(5): 1310. DOI:10.3390/ijms19051310 |
| [22] |
JIANG WP, ZHANG CX, ZHANG XM, et al. CircRNA HIPK3 promotes the progression of oral squamous cell carcinoma through upregulation of the NUPR1/PI3K/AKT pathway by sponging miR-637[J]. Ann Transl Med, 2021, 9(10): 860. DOI:10.21037/atm-21-1908 |
 2022, Vol. 51
2022, Vol. 51




