文章信息
- 管超, 李梦玲, 蒋礼青, 李思敏, 张宇冲, 张博, 李智, 赵明芳
- GUAN Chao, LI Mengling, JIANG Liqing, LI Simin, ZHANG Yuchong, ZHANG Bo, LI Zhi, ZHAO Mingfang
- 构建CT影像组学模型预测肺鳞状细胞癌肿瘤突变负荷
- Predicting tumor mutational burden in squamous cell lung carcinoma by constructing a computed tomography radiomics model
- 中国医科大学学报, 2022, 51(6): 481-485
- Journal of China Medical University, 2022, 51(6): 481-485
-
文章历史
- 收稿日期:2021-09-28
- 网络出版时间:2022-06-02 9:19
2. 中国医科大学附属盛京医院肿瘤科,沈阳 110004;
3. 中国医科大学附属第一医院临床流行病学与循证医学教研室,沈阳 110001
2. Department of Oncology, Shengjing Hospital of China Medical University, Shenyang 110004, China;
3. Department of Clinical Epidemiology and Evidence-based Medicine, The First Hospital of China Medical University, Shenyang 11000, China
肺癌是全球范围内发病率和死亡率最高的恶性肿瘤。作为非小细胞肺癌(non-small cell lung cancer,NSCLC) 的主要亚型之一,肺鳞状细胞癌(简称鳞癌)约占肺癌的25%~30%[1-2]。因肺鳞癌独特的临床病理和生物学特征,部分患者可获益于免疫治疗。但免疫治疗并非对所有患者有效[3],因此,迫切需要寻找到免疫治疗的生物标志物。目前,最常用的2种生物标志物为程序性死亡配体1 (programmed cell death-ligand 1,PD-L1) 和肿瘤突变负荷(tumor mutational burden,TMB) [4-5]。有研究[6]表明,TMB在预测免疫检查点抑制剂(immune checkpoint inhibitors,ICIs) 治疗方面优于PD-L1。但由于侵入性活检及全外显子组测序(whole exome sequencing,WES)的高成本等因素,阻碍了TMB检测的大规模推广。
放射组学作为一个快速崛起的领域,已在预测肺癌的诊断、预后和最佳治疗方面显示出重要作用[7]。有研究[8-9]显示,以CT为基础的影像组学在体细胞突变[如NSCLC的表皮生长因子受体(epithelial growth factor receptor,EGFR) 和间变性大细胞淋巴瘤激酶(anaplastic lymphoma kinase,ALK) 突变]中存在潜在的预测价值。此外,有研究[10]显示影像组学可以预测早期肺腺癌患者的TMB状态。
本研究旨在探讨从CT图像中提取的放射组学特征预测肺鳞癌患者TMB状态的价值,并基于放射组学数据开发TMB的预测模型。
1 材料与方法 1.1 研究对象共纳入35例初治肺鳞癌患者。纳入标准:(1) 病理结果为肺鳞癌;(2) 在癌症影像数据库(the cancer imaging archive,TCIA) 中可下载胸部CT原发病灶全部影像。从TCIA数据库下载TCGA-LUSC数据集中37例患者的全部CT影像,其中1例因病灶边缘过于模糊无法勾画靶病灶,另1例因存在多个原发病灶而被排除。
1.2 图像分割与特征提取本研究以开源医学图像处理软件(3D Slicer,版本4.11.0,http://www.slicer.org) 为分析平台,从而实现图像分割及影像特征提取。将下载的普通CT图像加载到3D Slicer软件,调整肺窗(窗口宽度:1 400,窗位:-500),以实现图像统一化处理。由2名具有9年工作经验的放射科医生沿CT横轴位病灶最大层面轮廓进行三维半自动分割,并手动逐层修改以勾画感兴趣区(region of interest,ROI),尽量避开血管、气管、神经等正常组织。再由1名具有15年以上工作经验的高级放射科医生核实并确认ROI。
采用3D-Slicer软件中的Pyradiomics包提取851个ROI的放射组学特征。所有分割数据都用1 mm×1 mm×1 mm的体素重采样进行标准化,以减少图像采集异质性的影响。
1.3 TMB分组从TCGA数据库下载肺鳞癌SNV数据集,用R软件计算下载数据中每个样品的TMB值,以数据集的TMB中位数为界,≤中位数的样品为低TMB组,> 中位数的样品为高TMB组。
1.4 统计学分析采用SPSS 25.0软件和R4.0.2软件对影像组学数据进行处理。采用LASSO回归筛选影像学特征。采用logistic回归对得到的影像学特征建立模型。所得受试者操作特征(receiver operating characteristic,ROC) 曲线和曲线下面积(area under the curve,AUC) 用于评价所建模型的诊断效能。临床决策曲线(decision curve analysis,DCA) 用于评估预测模型的临床实用性。
2 结果 2.1 图像分割与特征提取本研究采用3D-Slicer软件对35例初治肺鳞癌患者CT影像的原发病灶进行三维图像立体分割,主要方法为沿CT横轴位病灶最大层面轮廓边缘进行三维半自动分割,并逐层手动修改,从而获得原发病灶的三维立体图像即放射组学的ROI。
采用3D-Slicer软件中的Pyradiomics包对获得的35个ROI进行影像组学特征提取,共获得851个ROI的影像组学特征,其中包括shape、firstorder、glcm、gldm、glrlm、glszm和ngtdm 7种类型。
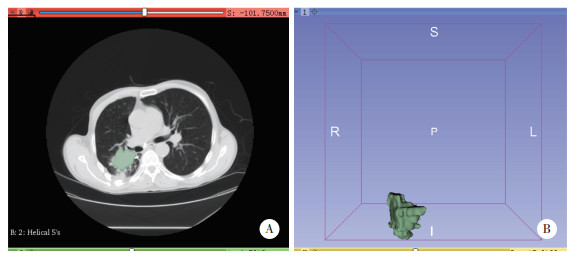
|
| A, medical image; B, segmentation. 图 1 原发灶的三维立体图像 Fig.1 Three-dimensional image of the primary foci |
2.2 TMB分组
从TCGA数据库共下载491例肺鳞癌SNV数据集,采用R软件(maftools包) 计算每个样品的TMB值,从而得出TMB的中位数为3.64/MB (图 2)。然后与从TCIA下载的35例肺鳞癌样本进行匹配,得到35例TCIA肺鳞癌样本的TMB分组情况。
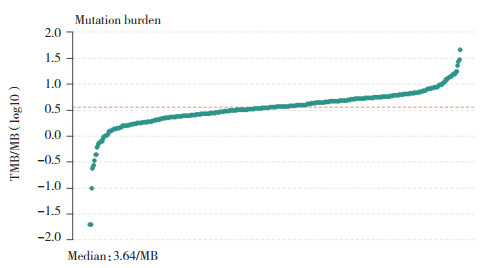
|
| 图 2 TMB分组情况 Fig.2 TMB grouping |
2.3 筛选与TMB相关的影像学特征
采用LASSO回归对851个特征进行降维处理,筛选出与TMB相关的影像学特征。根据最优λ值(图 3),最终筛选出9个相关的影像学特征,分别为wavelet-HHH_ glcm_Correlation,wavelet-HHL_glcm_Imc1,wavelet-LLH_glcm_MCC,wavelet-HHL_glszm_LowGrayLevelZoneEmphasis,wavelet-HHH_glszm_ZoneVariance,wavelet-HHL_gldm_LargeDependenceHighGrayLevelEmphasis,wavelet-LLH_ngtdm_Busyness,wavelet-HLL_firstorder_MeanAbsoluteDeviation和wavelet-LLL_glcm_MCC。
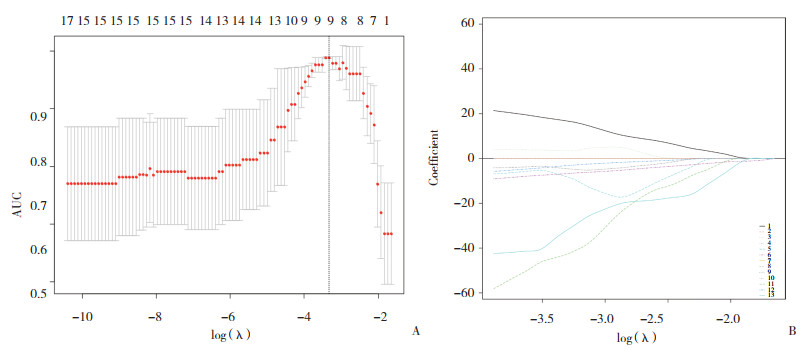
|
| A, the area under the curve for the least absolute shrinkage and selection operator coefficient profiles; B, least absolute shrinkage and selection operator coefficient profilesof variables. 1, wavelet-LLLglcmMCC; 2, wavelet-LLHngtdmBusyness; 3, wavelet-LLHglszmSmallAreaLowGrayLevelEmphasis; 4, wavelet-LLHgldmSmallDependenceHighGrayLevelEmphasis; 5, wavelet-LLHgldmDependenceNonUniformityNormalized; 6, wavelet-LLHglcmMCC; 7, wavelet-LHLgldmLargeDependenceHighGrayLevelEmphasis; 8, wavelet-HLLglcmClusterShade; 9, wavelet-HLHglcmCorrelation; 10, wavelet-HHLgldmLargeDependenceLowGrayLevelEmphasis; 11, wavelet-HHLglcmImc1;12, wavelet-HHHglszmSizeZoneNonUniformityNormalized; 13, wavelet-HHHglcmCorrelation. 图 3 三折交叉验证图 Fig.3 Tri-fold cross-validation diagram |
2.4 预测模型的构建及其诊断效能
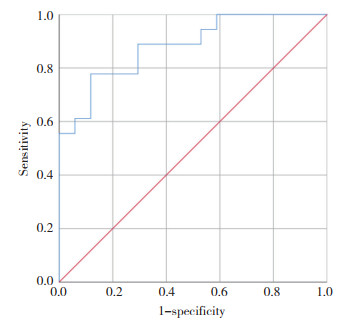
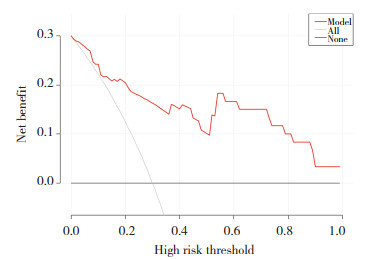
将筛选的9个影像学特征与TMB同时纳入logistic回归方程,进行逐步回归,最后筛选得到预测模型的3个影像学特征,分别为wavelet-HHH_glcm_Correlation、wavelet-HHL_gldm_Large Dependence High Gray Level Emphasis和wavelet-LLL_glcm_MCC (表 1)。绘制3个影像学特征与TMB的nomogram图(图 4)。绘制3个影像学特征预测的ROC曲线(图 5),AUC可达0.882,说明此模型预测TMB精度良好。利用logistic回归模型进行建模,绘制DCA对临床预测模型进行评价。DCA显示,当阈值取0.10~1.00时,影像组学模型预测效能良好,见图 6。
| Imaging characteristics | Estimate | SE | Z | P |
| (Intercept) | -27.333 395 | 10.257 296 | -2.66 | 0.007 7 |
| wavelet.HHH_glcm_Correlation | -76.339 093 | 32.723 208 | -2.33 | 0.019 7 |
| wavelet.HHL_gldm_LargeDependenceHighGrayLevelEmphasis | 0.000 198 | 0.000 121 | 1.63 | 0.103 1 |
| wavelet.LLL_glcm_MCC | 30.346 919 | 11.609 889 | 2.61 | 0.009 0 |
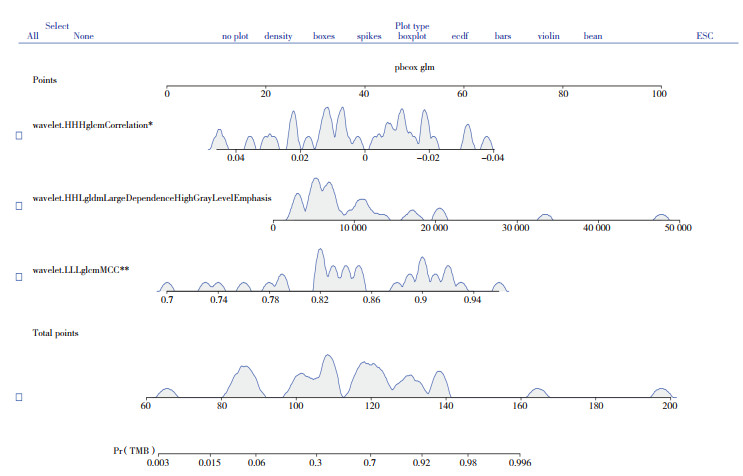
|
| 图 4 预测TMB的影像组学nomogram图 Fig.4 Imaging nomogram of predicted TMB |
|
| 图 5 影像学组学预测模型的ROC曲线 Fig.5 ROC curves for the imaging histological features of prediction models |
|
| 图 6 影像组预测模型的DCA Fig.6 DCA of the image group prediction model |
3 讨论
NSCLC患者经过ICIs治疗后,仅有一小部分人群临床获益。除了PD-L1、MSI等预测ICIs疗效的生物标志物,近年来,越来越多的数据证实TMB越高的肿瘤抗原负荷越高,更有可能从ICIs治疗中获益,因此,TMB受到的临床关注度逐渐升高。
TMB是一种广泛用于ICIs治疗独立的疗效预测生物标志物。TMB首次在2014年黑色素瘤的研究[11]中被证实与CTLA-4抗体的疗效存在相关性。2017年发现TMB对27种肿瘤的免疫治疗研究[12-13]有显著的疗效预测作用,奠定了其作为预测泛癌种治疗探索的基础。
尽管TMB具有前景的预测价值,但TMB检测仍面临着一些挑战,包括肺癌患者动态获取活检组织的可行性差,重症患者无法获取活检组织,以及WES检查的高成本等。本研究基于CT影像组学预测TMB,可以弥补传统TMB检测方法的不足。
放射组学研究因其获取全面图像信息方面的优势而在广泛肿瘤研究领域迅速发展[14]。在NSCLC中,影像学组在预测病理和分子分型[15-16]、器官特异性转移[17-18]、复发和总生存[19-20]等方面表现出潜力。因此,假设放射组学特征可用于通过预测高TMB的概率来选择临床上最需要的患者群体进行TMB检测。此外,对于那些无法接受昂贵WES测试的人群来说,放射组学可以作为TMB检测的替代或辅助诊断。
本研究中,TMB预测模型通过列线图可视化,可以方便地计算肺鳞癌患者高TMB的可能性。该研究有可能帮助临床医生筛选出TMB检测的最佳候选人,即最有可能具有高TMB的患者。另一方面,本研究的模型也可以作为TMB的替代品,用于无法负担昂贵的TMB检测或没有可获取的组织样本进行检测的患者。与传统的TMB检测方法相比,本研究放射组学TMB预测模型为完全非侵入性。此外,由于大多数肺鳞癌患者在初始诊断之前或初始诊断时已经接受了胸部CT扫描,因此,基于影像组学的TMB预测模型几乎不需要额外的检查。
本研究存在一定的局限性。首先,本研究数据来自TCIA数据库,样本量相对较小,因此不可避免的会存在与回顾性研究相关的缺点;其次,由于目前对肺鳞癌中TMB的最佳临界值没有达成共识,选择了通过R包(maftools包) 计算得出的中位TMB来定义TMB的高、低组;第三,影像组学预测肺鳞癌患者TMB能力背后的生物学解释尚不清楚,还需要进一步深入研究。最后,本研究缺乏外部数据对预测模型进行验证。
本研究结果显示,从胸部CT中提取的影像组学特征与TMB之间存在内在联系。因此建立了一个基于放射组学特征的TMB预测模型,通过本研究证明了采用放射组学预测TMB的可行性。并且,基于影像组学特征建立的预测模型对TMB状态有较高的预测价值,有待进一步研究探索。
| [1] |
HOUSTON KA, HENLEY SJ, LI J, WHITE MC, RICHARDS TB. Patterns in lung cancer incidence rates and trends by histologic type in the United States, 2004-2009[J]. Lung Cancer, 2014, 86(1): 22-28. DOI:10.1016/j.lungcan.2014.08.001 |
| [2] |
SMITH BD, SMITH GL, HURRIA A, et al. Future of cancer incidence in the United States: burdens upon an aging, changing nation[J]. J Clin Oncol, 2009, 27(17): 2758-2765. DOI:10.1200/jco.2008.20.8983 |
| [3] |
KENNEDY LB, SALAMA AKS. A review of cancer immunotherapy toxicity[J]. CA Cancer J Clin, 2020, 70(2): 86-104. DOI:10.3322/caac.21596 |
| [4] |
FUMET JD, TRUNTZER C, YARCHOAN M, et al. Tumour mutational burden as a biomarker for immunotherapy: current data and emerging concepts[J]. Eur J Cancer, 2020, 131: 40-50. DOI:10.1016/j.ejca.2020.02.038 |
| [5] |
PATEL SP, KURZROCK R. PD-L1 expression as a predictive biomarker in cancer immunotherapy[J]. Mol Cancer Ther, 2015, 14(4): 847-856. DOI:10.1158/1535-7163.mct-14-0983 |
| [6] |
BOCHNER BH. Re: atezolizumab in patients with locally advanced and metastatic urothelial carcinoma who have progressed following treatment with platinum-based chemotherapy: a single-arm, multicenter, phase 2 trial[J]. Eur Urol, 2017, 71(2): 299-300. DOI:10.1016/j.eururo.2016.10.012 |
| [7] |
THAWANI R, MCLANE M, BEIG N, et al. Radiomics and radioge- nomics in lung cancer: a review for the clinician[J]. Lung Cancer, 2018, 115: 34-41. DOI:10.1016/j.lungcan.2017.10.015 |
| [8] |
YAMAMOTO S, KORN RL, OKLU R, et al. ALK molecular phenotype in non-small cell lung cancer: ct radiogenomic characterization[J]. Radiology, 2014, 272(2): 568-576. DOI:10.1148/radiol.14140789 |
| [9] |
RIZZO S, PETRELLA F, BUSCARINO V, et al. CT radiogenomic characterization of EGFR, K-RAS, and ALK mutations in non-small cell lung cancer[J]. Eur Radiol, 2016, 26(1): 32-42. DOI:10.1007/s00330-015-3814-0 |
| [10] |
WANG XX, KONG C. Decoding tumor mutation burden and driver mutations in early stage lung adenocarcinoma using CT-based radiomics signature[J]. Thorac Cancer, 2019, 10(10): 1904-1912. DOI:10.1111/1759-7714.13163 |
| [11] |
SNYDER A, MAKAROV V, MERGHOUB T, et al. Genetic basis for clinical response to CTLA-4 blockade in melanoma[J]. N Engl J Med, 2014, 371(23): 2189-2199. DOI:10.1056/nejmoa1406498 |
| [12] |
RIZVI NA, HELLMANN MD, SNYDER A, et al. Cancer immunology. Mutational landscape determines sensitivity to PD-1 blockade in non-small cell lung cancer[J]. Science, 2015, 348(6230): 124-128. DOI:10.1126/science.aaa1348 |
| [13] |
YARCHOAN M, HOPKINS A, JAFFEE EM. Tumor mutational burden and response rate to PD-1 inhibition[J]. N Engl J Med, 2017, 377(25): 2500-2501. DOI:10.1056/nejmc1713444 |
| [14] |
AVANZO M, STANCANELLO J, EL NAQA I. Beyond imaging: the promise of radiomics[J]. Phys Med, 2017, 38: 122-139. DOI:10.1016/j.ejmp.2017.05.071 |
| [15] |
ROSSI G, BARABINO E, FEDELI A, et al. Radiomic detection of EGFR mutations in NSCLC[J]. Cancer Res, 2021, 81(3): 724-731. DOI:10.1158/0008-5472.CAN-20-0999 |
| [16] |
KOYASU S, NISHIO M, ISODA H, et al. Usefulness of gradient tree boosting for predicting histological subtype and EGFR mutation status of non-small cell lung cancer on 18F FDG-PET/CT[J]. Ann Nucl Med, 2020, 34(1): 49-57. DOI:10.1007/s12149-019-01414-0 |
| [17] |
CHEN A, LU L, PU X, et al. CT-based radiomics model for predicting brain metastasis in category T1 lung adenocarcinoma[J]. AJR Am J Roentgenol, 2019, 213(1): 134-139. DOI:10.2214/ajr.18.20591 |
| [18] |
SUN F, CHEN Y, CHEN X, et al. CT-based radiomics for predicting brain metastases as the first failure in patients with curatively resected locally advanced non-small cell lung cancer[J]. Eur J Radiol, 2021, 134: 109411. DOI:10.1016/j.ejrad.2020.109411 |
| [19] |
MU W, JIANG L, SHI Y, et al. Non-invasive measurement of PD-L1 status and prediction of immunotherapy response using deep learning of PET/CT images[J]. J Immunother Cancer, 2021, 9(6): e002118. DOI:10.1136/jitc-2020-002118 |
| [20] |
BLANC-DURAND P, CAMPEDEL L, MULE S, et al. Prognostic value of anthropometric measures extracted from whole-body CT using deep learning in patients with non-small-cell lung cancer[J]. Eur Radiol, 2020, 30(6): 3528-3537. DOI:10.1007/s00330-019-06630-w |
 2022, Vol. 51
2022, Vol. 51




