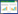文章信息
- 颜南, 王润新, 王正东, 刘洪, 张珉, 张忠
- YAN Nan, WANG Runxin, WANG Zhengdong, LIU Hong, ZHANG Min, ZHANG Zhong
- 胃癌GEO芯片结合TCGA数据差异基因筛选、功能及通路研究
- Screening and integrative analysis of GEO chips and TCGA data for differentially expressed genes in gastric cancer
- 中国医科大学学报, 2022, 51(4): 329-335
- Journal of China Medical University, 2022, 51(4): 329-335
-
文章历史
- 收稿日期:2021-01-14
- 网络出版时间:2022-04-29 11:06
2. 沈阳医学院2015级本科生, 沈阳 110034;
3. 沈阳医学院解剖学教研室, 沈阳 110034;
4. 沈阳医学院计算机教研室, 沈阳 110034;
5. 沈阳医学院病理学教研室, 沈阳 110034
2. 2015 grade undergraduate school, Shenyang Medical College, Shenyang 110034, China;
3. Department of Anatomy, Shenyang Medical College, Shenyang 110034, China;
4. Department of Computer Science, Shenyang Medical College, Shenyang 110034, China;
5. Department of Pathology, Shenyang Medical College, Shenyang 110034, China
2015年我国流行病学调查结果显示,胃癌发生率为29.31/10万,居癌症死亡原因第三位[1],晚期胃/胃食管交界处胃癌患者中位生存期通常 < 1年[2]。因此,早期筛查和靶向治疗对于胃癌的早期发现、诊断及治疗至关重要。胃癌的筛查方法包括胃镜、X线钡餐检查,血清幽门螺杆菌抗体检测,胃萎缩(胃癌癌前病变)标志物,如血清生长激素释放肽或血清胃蛋白酶原检测等[3]。内窥镜或手术病理活检是诊断胃癌的金标准,胃癌相关的血清肿瘤标志物也可作为胃癌辅助诊断依据,但其灵敏度及特异度均不佳[4]。虽然HER2、EGFR、VEGFR等靶基因可用于胃癌靶向治疗,但因胃癌具有细胞分化程度低、增殖速度快、侵袭性强等性质,以上靶向治疗均未能获得良好的预期效果[5]。
大多数的疾病都会发生基因翻译及转录方面的特异改变,尤其癌症这种遗传或表观遗传改变的疾病,基因表达的改变对癌症的发生发展进程有重要影响。基因芯片技术和生物学分析方法的结合是探究胃癌等疾病发生、发展分子机制的有效方法之一[6]。因此,本研究拟利用生物信息学方法筛选影响胃癌发生、发展过程中的核心基因,以探索胃癌发病的分子生物学机制。
1 材料与方法 1.1 生物信息数据库及分析工具本研究采用的数据库包括基因表达数据库(Gene Expression Omnibus,GEO)(https://www.ncbi.nlm.nih.gov/geo/GEO),癌症基因组图谱(The Cancer Genome Atlas,TCGA)(https://portal.gdc.cancer.gov),韦恩图(Biological venn,Bio venn)数据库(http://bioinformatics.psb.ugent.be),注释、可视化和集成发现数据库(Database for Annotation,Visualization and Integrated Discovery,DAVID)(http://www.david.niaid.nih.gov),京都基因与基因组百科全书(Kyoto Encyclopedia of Genes and Genomes,KEGG)(https://www.genome.jp/kegg),基因蛋白质相互作用检索(search tool for the retrival of interacting genes/proteins,STRING)数据库(http://string.db.org),Kaplan-Meier plotter数据库(http://kmplot.com/analysis)。
1.2 生物信息学分析方法在GEO数据库中,限定选项筛选获得数据集。利用GEO2R对数据集进行差异表达分析。保存GEO2R的分析结果,导入R studio,使用R包plot函数进行火山图绘制,P < 0.05时,logFC<0为下调基因,用蓝色表示,logFC>0为上调基因,用红色表示。用GraghPad Prism8.0绘制热图,将两芯片共有基因数据logFC导入Prism表格中,最小值用绿色表示,基准值用黄色表示,最大值用红色表示。进入Bio venn在线制图软件,制作差异基因的韦恩图。在TCGA数据库中下载有关胃癌的病理数据,导入R中提取注释信息和分组信息,使用R语言DESeq2包进行基因的差异表达分析,筛选差异表达基因(P < 0.05,logFC<-1为下调基因,logFC>1为上调基因)。通过DAVID在线分析对共有差异基因进行基因本体(gene ontology,GO)功能注释及KEGG信号通路富集分析。通过STRING在线分析工具获得共有差异蛋白之间的互作图。利用Kaplan-Meier数据库对互作基因进行生存分析,绘制基因与胃癌患者的生存曲线可视化图。
2 结果 2.1 GEO胃癌数据GEO数据库筛选2个微列阵数据集GSE79973、GSE55696,数据均为GPL570芯片平台。GSE79973包括10个胃癌样本和10个正常样本;GSE55696包括19个低级别上皮内瘤变(low-grade intraepithelial neoplasia,LGIN)、20个高级别上皮内瘤变(high-grade intraepithelial neoplasia,HGIN)、19个早期胃癌(early gastric cancer,EGC)和19个慢性胃炎组织样本。TCGA-STAD数据集包括343个胃癌样本和30个胃炎样本。
2.2 差异表达基因通过GEO2R在GSE55696中筛选出2 145个差异基因,包括822个下调基因和1 323个上调基因,在GSE79973中筛选出551个差异基因,获得128个下调基因和383个上调基因。TCGA数据库胃癌病理数据经R语言DESeq2包进行筛选差异表达基因标准化处理后,共获得14 053个差异表达基因。两芯片及TCGA筛选出胃癌差异基因总体表达情况见图 1。
|
| 图 1 两芯片及TCGA筛选胃癌差异基因表达火山图 Fig.1 Two microarrays and TCGA were used to screen out the volcanogram of differentially expressed genes in gastric cancer |
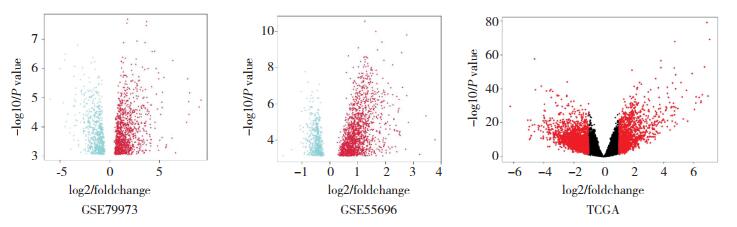
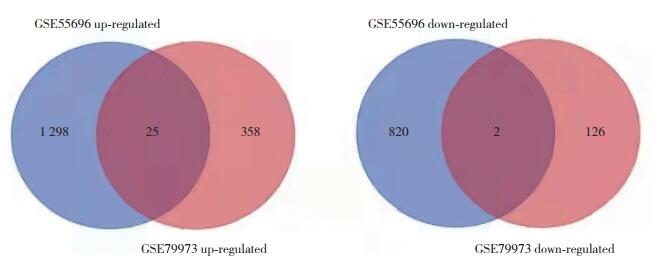
韦恩图获得27个共有差异基因,见图 2。其中,17个基因(上调基因IRX3、FCRL5、FAM3D、GHRL、TRPA1、SLC5A9、LRRC31、IRX2、APOA1、SLC51A、HMGCS2、ACE2、MT1M、PCK1、ALDOB,下调基因FNDC1、NKX3-2)是和TCGA-STAD的共同差异表达基因。胃癌组织中,25个基因(HMGCS2、NPY6R、GHRL、IRX3、TRPA1、ACE2、CSHL1、CFAP74、KNG1、FCRL5、SLC2A2、FAM3D、MEP1B、MT1M、LRRC31、PDILT、PCK1、SLC5A9、TMPRSS15、CPA6、IRX2、CHIA、ALDOB、APOA1、SLC51A)表达上调,2个基因(FNDC1、NKX3-2)表达下调。根据两芯片数据共有的27个基因logFC值作为基因表达情况制作热图,进一步验证了上述结果。见图 3。
|
| 图 2 2组数据差异基因韦恩图 Fig.2 The venn diagram of difference gene between the two groups |
|
| 图 3 两芯片数据集基因表达热图 Fig.3 The heat maps of gene expression in two chip datasets |
2.3 GO功能分析和KEGG通路分析结果
GO功能分析显示,27个共有差异表达基因主要存在于细胞外空隙、分泌颗粒内腔,具有金属羧肽酶活性,参与亨利恒等循环、葡萄糖的跨膜转运、胰岛素分泌的负调节和胆固醇生物合成等生物过程,见表 1。
| Category | Term | n(%) | P |
| GOTERM_CC_DIRECT | Extracellular region | 8(29.6) | 4.8e-3 |
| GOTERM_BP_DIRECT | Specification of loop of Henle identity | 2(7.4) | 5.2e-3 |
| GOTERM_CC_DIRECT | Extracellular space | 7(25.9) | 8.4e-3 |
| GOTERM_CC_DIRECT | Secretory granule lumen | 2(7.4) | 1.6e-2 |
| GOTERM_BP_DIRECT | Glucose transmembrane transport | 2(7.4) | 2.5e-2 |
| GOTERM_MF_DIRECT | Metallocarboxypeptidase activity | 2(7.4) | 3.5e-2 |
| GOTERM_BP_DIRECT | Negative regulation of insulin secretion | 2(7.4) | 4.0e-2 |
| GOTERM_BP_DIRECT | Cholesterol biosynthetic process | 2(7.4) | 4.9e-2 |
| GOTERM_BP_DIRECT | Gluconeogenesis | 2(7.4) | 5.6e-2 |
| GOTERM_MF_DIRECT | Zinc ion binding | 5(18.5) | 6.2e-2 |
| GOTERM_CC_DIRECT | Brush border | 2(7.4) | 7.9e-2 |
| GOTERM_BP_DIRECT | Digestion | 2(7.4) | 7.9e-2 |
| GOTERM_BP_DIRECT | Response to estrogen | 2(7.4) | 8.2e-2 |
| GOTERM_BP_DIRECT | Glucose metabolic process | 2(7.4) | 8.4e-2 |
| GOTERM_BP_DIRECT | Negative regulation of inflammatory response | 2(7.4) | 9.9e-2 |
KEGG通路分析显示,27个差异表达基因出现在过氧化物酶体增殖物激活受体(peroxisome proliferators-activated receptor,PPAR)信号通路、炎症介质对瞬时受体电位(transient receptor potential,TRP)通道的调节、胆固醇代谢、碳水化合物的消化吸收、刺激神经组织中的交互、缺氧诱导因子-1(hypoxia inducible factor-1,HIF-1)信号通路、胆汁分泌、胰岛素的耐受性、酮体的合成和降解、补体系统、矿物质吸收、鞘脂类信号通路、磷酸戊糖途径、维生素的消化吸收、肾素-血管紧张素系统等39个信号通路。由于有3个基因(HMGCS2、Apo-AI、PEPCK)显著富集于hsa03320 PPAR信号通路,见图 4。因此,在影响胃癌发生的相关诸多主要信号通路中筛选出PPAR信号通路。
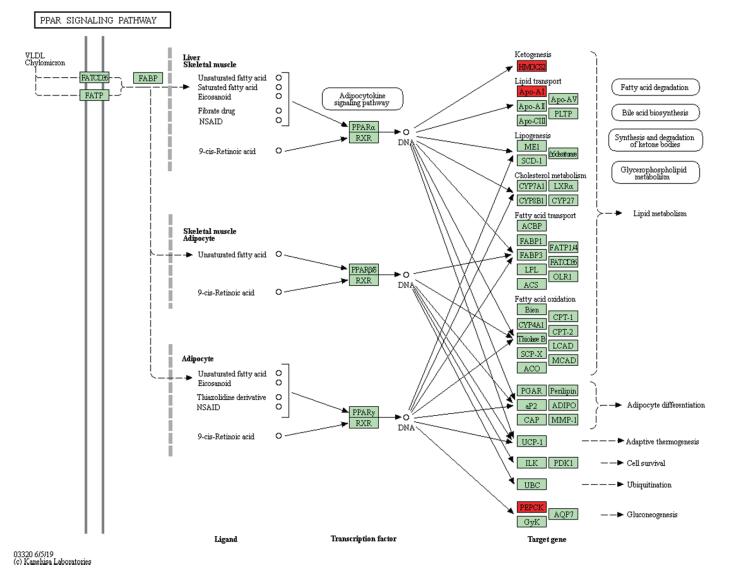
|
| 图 4 PPAR信号通路图 Fig.4 The diagram of the PPAR signal pathway |
2.4 蛋白质-蛋白质相互作用(protein-protein interaction,PPI)网络分析
将27个共同差异基因导入STRING数据库进行PPI分析,在得到的PPI网络中,发现7个关键基因(SLC2A2、HMGCS2、ALDOB、PCK1、APOA1、KNG1、ACE2)相互密切作用,见图 5。其中,PCK1、ACE2、APOA1、HMGCS2、ALDOB是与TCGA-STAD共有差异表达基因。
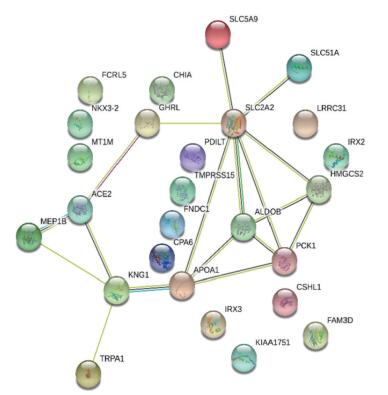
|
| 图 5 共有差异基因蛋白质-蛋白质相互作用图 Fig.5 The protein-protein interaction map of the shared differential gene |
2.5 核心基因与生存率的关系
为了获得差异表达基因表达水平改变对患者生存率的影响,应用Kaplan-Meier数据库在线分析了7个关键基因和患者总体生存率之间的关系。结果显示,SLC2A2、HMGCS2、APOA1和KNG1高表达的胃癌患者生存率较低,与正常人群的总体生存率比较,差异有统计学意义(P < 0.05)。其中,HMGCS2、APOA1属于TCGA-STAD共有差异表达基因。见图 6。
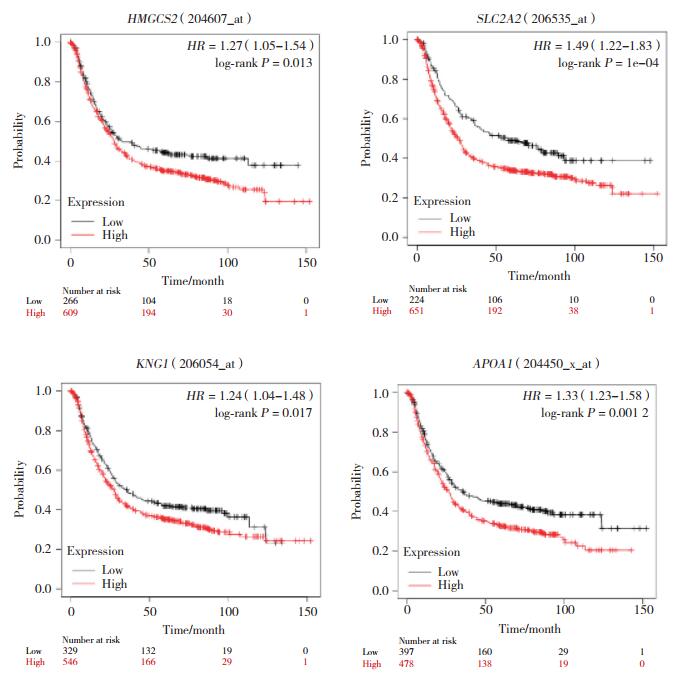
|
| 图 6 差异基因生存曲线图 Fig.6 The survival curve of differential gene |
3 讨论
胃癌的发生是一个多因素参与、多步骤渐进的过程,涉及遗传因素、癌前状态、饮食因素等。研究显示,p53、NGAL、TBL1XR1、FZD7、FAT4、NDRG1、BRD4、LKB1、CHFR、BUBI、MAD2以及部分微RNA(micro RNA,miRNA)在胃癌细胞凋亡、增殖、转移、侵袭、血管生成等机制中发挥重要作用。
本研究从GEO基因表达数据库中筛选数据集,并利用GEO2R分析胃癌组织和正常组织中显著差异表达基因,共获得27个共有差异基因。其中,3个基因(HMGCS2、APOA1、PEPCK)显著富集于PPAR信号通路,主要影响肿瘤的微环境,并诱导胃癌发展。PPAR作为核激素受体超家族成员,与细胞核中的类维生素AX受体(retinoid X receptor,RXR)形成异源二聚体后,与靶基因的PPAR反应元件结合发挥作用[7]。在膀胱、胶质瘤、肝脏、肾脏和胃癌、食道癌中,PPAR信号失调集中参与多种代谢过程相关的共同下游通路[8]。用特定的COX-2抑制剂治疗过表达COX-2胃细胞株MKN45可导致PPARα表达时间和剂量依赖性的抑制[9]。胃癌细胞系MGC803中,PPAR-γ呈高表达,PPAR-γ在免疫系统、糖脂代谢、脂肪形成等生物过程中起重要作用,与肥胖、高血压、帕金森病、癌症等发展有关。抑制PPAR-γ功能可能是治疗和预防胃癌的一种新方法[10]。GO和KEGG结果提示,胆固醇代谢途径在癌症发生过程中可发挥一定作用,另有研究[11]表明胆固醇稳态基因可以调节肿瘤发育。
本研究中,通过Kaplan-Meier plotter分析发现,7个互作差异基因中,SLC2A2、HMGCS2、APOA1、KNG1基因高表达的胃癌患者预后明显较差。SLC2A2(Glut2)是溶质载体家族2成员,为满足肿瘤细胞高代谢需求,肿瘤细胞通过溶质载体[SLC2A,葡萄糖转运体(glucose transporters,GLUT)]高速运输葡萄糖,尤其在细胞缺氧、缺血状态下表达水平会明显增高。研究[12]发现,HIF-1、Ras、c-Myc、PI3K-Akt及p53途径都对GLUT有调节作用。肿瘤细胞葡萄糖代谢速率增加说明其在恶性增殖过程中需要葡萄糖代谢的支持,研究提示在多种恶性肿瘤中葡萄糖转运蛋白尤其是Glut1的表达增加。研究[13]表明,SLC2A2与糖代谢异常疾病、肝癌有关,SLC2A2可能是影响肝细胞癌的一个重要因素。HMGCS2是生酮限速酶,研究[14]显示HMGCS2与肿瘤血管生成有关,还可能与大鼠非酒精性脂肪肝、结肠癌有关,在结直肠癌和口腔鳞状细胞癌患者中,HMGCS2表达水平与临床预后不良有关[15]。提示HMGCS2可能是未来治疗晚期癌症的一个重要靶点。APOA1是正常人体组织中表达的蛋白质,研究[16]显示其表达与胃癌的浸润深度、淋巴结分期及分型呈明显负相关。KNG1具有抗血管生成和抑制内皮细胞增殖的作用,晚期结肠直肠腺瘤患者KNG1明显差异表达[17],可作为早期结直肠癌的标志物[18]。APOA1和HMGCS2属于TCGA-STAD数据集中差异表达的基因,并同属于显著富集于PPAR信号通路且与TCGA-STAD有交集的共有差异表达基因,且位于PPI网络相互作用最紧密的7个基因构成的模块中,因此,认为APOA1和HMGCS2在胃癌发生、发展的分子机制中具有重要的意义。
综上所述,本研究发现PPAR信号通路及SLC2A2、HMGCS2、APOA1、KNG1基因均可影响胃癌的发生、发展。本研究结果为胃癌的易感基因鉴定、分子化的靶向治疗、预后评价提供了新的研究方向。
| [1] |
王少明, 郑荣寿, 张思维, 等. 2015年中国胃癌流行特征分析[J]. 中华流行病学杂志, 2019, 40(12): 1517-1521. DOI:10.3760/cma.j.issn.0254-6450.2019.12.003 |
| [2] |
BOKU N, RYU MH, KATO K, et al. Safety and efficacy of nivolumab in combination with S-1/capecitabine plus oxaliplatin in patients with previously untreated, unresectable, advanced, or recurrent gastric/gastroesophageal junction cancer: interim results of a randomized, phaseⅡtrial (ATTRACTION-4)[J]. Ann Oncol, 2019, 30(2): 250-258. DOI:10.1093/annonc/mdy540 |
| [3] |
YAMAGUCHI Y, NAGATA Y, HIRATSUKA R, et al. Gastric cancer screening by combined assay for serum anti-Helicobacter pylori IgG antibody and serum pepsinogen levels: the ABC method[J]. Digestion, 2016, 93(1): 13-18. DOI:10.1159/000441742 |
| [4] |
HUANG ZB, ZHOU X, XU J, et al. Prognostic value of preoperative serum tumor markers in gastric cancer[J]. World J Clin Oncol, 2014, 5(2): 170-176. DOI:10.5306/wjco.v5.i2.170 |
| [5] |
BRAY F, FERLAY J, SOERJOMATARAM I, et al. Global cancer statistics 2018:globocan estimates of incidence and mortality worldwide for 36 cancers in 185 countries[J]. CA Cancer J Clin, 2018, 68(6): 394-424. DOI:10.3322/caac.21492 |
| [6] |
BEST MG, WESSELING P, WURDINGER T. Tumor-educated platelets as a noninvasive biomarker source for cancer detection and progression monitoring[J]. Cancer Res, 2018, 78(13): 3407-3412. DOI:10.1158/0008-5472.can-18-0887 |
| [7] |
CAVE MC, CLAIR HB, HARDESTY JE, et al. Nuclear receptors and nonalcoholic fatty liver disease[J]. Biochim Biophys Acta, 2016, 1859(9): 1083-1099. DOI:10.1016/j.bbagrm.2016.03.002 |
| [8] |
CHANG WH, LAI AG. The Pan-cancer mutational landscape of the PPAR pathway reveals universal patterns of dysregulated metabolism and interactions with tumor immunity and hypoxia[J]. Ann N Y Acad Sci, 2019, 1448(1): 65-82. DOI:10.1111/nyas.14170 |
| [9] |
GENG LJ, ZHOU W, LIU B, et al. DHA induces apoptosis of human malignant breast cancer tissues by the TLR-4/PPAR-α pathways[J]. Oncol Lett, 2018, 15(3): 2967-2977. DOI:10.3892/ol.2017.7702 |
| [10] |
MA XM, YU H, HUAI N. Peroxisome proliferator-activated receptor-gamma is essential in the pathogenesis of gastric carcinoma[J]. World J Gastroenterol, 2009, 15(31): 3874-3883. DOI:10.3748/wjg.15.3874 |
| [11] |
GUO F, HONG W, YANG M, et al. Upregulation of 24(R/S), 25-epoxycholesterol and 27-hydroxycholesterol suppresses the proliferation and migration of gastric cancer cells[J]. Biochem Biophys Res Commun, 2018, 504(4): 892-898. DOI:10.1016/j.bbrc.2018.09.058 |
| [12] |
KURAOKA K, MATSUMURA S, SANADA Y, et al. A single nucleotide polymorphism in the extracellular domain of TRAIL receptor DR4 at nucleotide 626 in gastric cancer patients in Japan[J]. Oncol Rep, 2005, 14(2): 465-470. DOI:10.3892/or.14.2.465 |
| [13] |
KIM YH, JEONG DC, PAK K, et al. SLC2A2(GLUT2) as a novel prognostic factor for hepatocellular carcinoma[J]. Oncotarget, 2017, 8(40): 68381-68392. DOI:10.18632/oncotarget.20266 |
| [14] |
ZOU K, HU Y, LI M, et al. Potential role of HMGCS2 in tumor angiogenesis in colorectal cancer and its potential use as a diagnostic marker[J]. Can J Gastroenterol Hepatol, 2019, 2019: 8348967. DOI:10.1155/2019/8348967 |
| [15] |
CHEN SW, CHOU CT, CHANG CC, et al. HMGCS2 enhances invasion and metastasis via direct interaction with PPARα to activate Src signaling in colorectal cancer and oral cancer[J]. Oncotarget, 2017, 8(14): 22460-22476. DOI:10.18632/oncotarget.13006 |
| [16] |
CHONG PK, LEE H, ZHOU J, et al. Reduced plasma APOA1 level is associated with gastric tumor growth in MKN45 mouse xenograft model[J]. J Proteomics, 2010, 73(8): 1632-1640. DOI:10.1016/j.jprot.2010.04.005 |
| [17] |
YU J, HUANG Y, LIN C, et al. Identification of kininogen 1 as a serum protein marker of colorectal adenoma in patients with a family history of colorectal cancer[J]. J Cancer, 2018, 9(3): 540-547. DOI:10.7150/jca.22405 |
| [18] |
QUESADA-CALVO F, MASSOT C, BERTRAND V, et al. OLFM4, KNG1 and Sec24C identified by proteomics and immunohistochemistry as potential markers of early colorectal cancer stages[J]. Clin Proteomics, 2017, 14: 9. DOI:10.1186/s12014-017-9143-3 |
 2022, Vol. 51
2022, Vol. 51