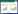文章信息
- 佟春晓, 雷则鸣
- TONG Chunxiao, LEI Zeming
- 多芯片联合分析揭示肌腱粘连的分子机制
- Multi-chip joint analysis reveals molecular mechanisms of tendon adhesions
- 中国医科大学学报, 2022, 51(10): 913-918
- Journal of China Medical University, 2022, 51(10): 913-918
-
文章历史
- 收稿日期:2022-07-16
- 网络出版时间:2022-10-01 7:30
2. 沈阳医学院附属中心医院手外五科, 沈阳 110024;
3. 中国医科大学附属盛京医院骨外科, 沈阳 110004
2. Department of the Fifth Ward of Hand Surgery, Central Hospital Affiliated to Shenyang Medical College, Shenyang 110024, China;
3. Department of Orthopaedic Surgery, Shengjing Hospital of China Medical University, Shenyang 110004, China
肌腱粘连是常见且棘手的并发症,可导致肢体活动严重受限,甚至功能丧失。目前肌腱粘连的发生机制仍未完全阐明,主要学说认为是由肌腱内源性和外源性愈合不平衡引起[1]。内源性愈合主要是腱细胞经增殖及迁移致肌腱的愈合。外源性愈合是肌腱损伤断面形成肉芽组织,腱外膜的成纤维细胞增殖侵袭,由周围组织向肌腱断端生长,腱周纤维化,胶原蛋白和纤维蛋白沉积,形成肌腱与周围组织的瘢痕组织。外源性愈合占据主导时会导致肌腱粘连,减弱外源性愈合及炎症参与,促进内源性愈合,是当前解决肌腱粘连问题和提高肌腱术后或损伤后腱愈合预后的方法。鉴于目前对天然生态位和动态肌腱愈合过程的了解有限,需要进一步研究肌腱损伤、愈合、移除过程中的肌腱生物学变化。本研究利用生物信息学技术对基因表达数据库(Gene Expression Omnibus,GEO)中含有肌腱病和转化生长因子β(transforming growth factor-β,TGF-β)处理的成纤维细胞的表达谱芯片进行联合分析,挖掘肌腱粘连中的关键基因、重要通路、调控网络及生物代谢过程,为探索肌腱粘连的分子机制提供依据。
1 材料与方法 1.1 数据来源基因芯片数据集GSE26051和GSE1724均来源于GEO数据库。GSE26051芯片基于GPL570平台(Affymetrix Human Genome U133 Plus 2.0 Array),GSE1724芯片基于GPL8300平台(Affymetrix Human Genome U95 Version 2 Array)。GSE26051研究对象为23例慢性肌腱病患者肌腱病变组织和肌腱非病变组织。在GSE1724数据集中选取研究对象为特发性肺纤维化患者的肺成纤维细胞非处理组和TGF-β处理组,各3例,比较基因表达谱。联合分析2个数据集的基因芯片数据。
1.2 差异表达基因(different expression genes,DEGs)的筛选采用R语言“limma”包筛选组间DEGs。对样本质检表达量的差异倍数(fold change,FC)取对数,用于评价基因的表达水平,若P < 0.05且|logFC| > 1,认为有统计学意义。采用Pheatmap软件包绘制热图,分析DEGs在样本中的表达。
1.3 韦恩图绘制利用韦恩图绘制工具(https://bioinformatics.psb.ugent.be/webtools/Venn/)制作韦恩图,将GSE26051和GSE1724数据集筛选出的DEGs取交集。
1.4 DEGs的京都基因和基因组数据库(Kyoto Encyclopedia of Genes and Genomes,KEGG)通路富集和基因本体(gene ontology,GO)功能富集分析为了探索DEGs的主要功能和途径,通过R语言“Bioconductor”包对DEGs进行GO富集和KEGG通路富集,选出P < 0.05的结果;最后通过“ggplot2”包对富集结果分别进行可视化。
1.5 预测DEGs可能结合的微RNA(microRNA,mi-RNA)利用R语言结合miRDB数据库(https://mirdb.org)、ENCORI数据库(https://starbase.sysu.edu.cn)、miRTarBase数据库(https://mirtarbase.mbc.nctu.edu.tw)、miRWalk数据库(https://mirwalk.umm.uni-heidelberg.de)、RNA22数据库(https://cm.jefferson.edu/rna22)、RNAInter数据库(https://www.rnainter.org)、TargetMiner数据库(https://www.isical.ac.in/~bioinfo_miu/targetminer20.htm)、TargetScan数据库(https://www.targetscan.org),联合预测DEGs可能结合的miRNA,结果用R语言“UpSetR”展示。
1.6 蛋白质-蛋白质相互作用(protein-protein interaction,PPI)网络分析将DEGs导入STRING数据库(https://string-db.org/)进行PPI分析,并设定筛选阈值为0.4,获得共有差异蛋白之间的互作图。
2 结果 2.1 DEGs的筛选对GSE26051芯片数据集(肌腱病患者病灶与非病灶,各23例)及GSE1724芯片数据集(特发性肺纤维化患者的肺成纤维细胞TGF-β处理组与成纤维细胞对照组各3例)进行分析,根据P < 0.05且|logFC| > 1筛选组间DEGs。按照|logFC|从大到小排列,GSE26051和GSE1724筛选出的前十位DEGs分别为TRIM54、BE156417、LRP1B、JSRP1、BF514585、ACADL、BF680284、ESRRG、DOK7、BG541187以及ESM1、IL11、NGF、HBEGF、SOCS1、PCYT2、MEOX1、IER3、TXNIP、NEDD9。
2.2 火山图和聚类图绘制分析差异表达谱根据芯片筛选出的DEGs绘制火山图和热图。火山图(图 1)反映总体基因的表达情况,红色和绿色区域代表P < 0.05且|logFC| > 1的基因。根据基因表达水平,将检测的基因和标本进行聚类分析,采用Pheatmap软件包绘制聚类分析热图,红色代表基因表达量相对高,绿色代表基因表达量相对低。由聚类分析热图发现2组样本中基因的表达存在差异。
|
| A, GSE1724; B, GSE26051. 图 1 GSE1724和GSE26051芯片中DEGs的火山图 Fig.1 Volcano map of differentially expressed genes in the GSE1724 and GSE26051 gene microarray |
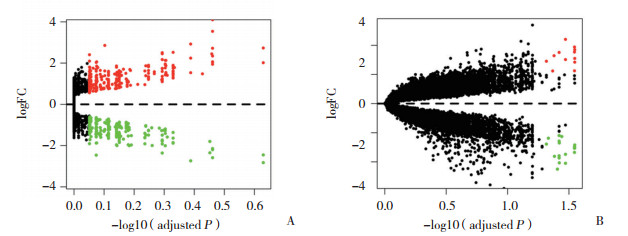
2.3 韦恩图
利用韦恩图绘制工具绘制韦恩图(图 2),将GSE26051和GSE1724数据集分别筛选出的45个和526个DEGs取交集,共筛选出2个基因(EPB41、ETV6)。
|
| 图 2 GSE1724与GSE26051数据集DEGs的韦恩图 Fig.2 Mapping of the differential expression of gene between GSE1724 and GSE26051 data sets |
2.4 DEGs的miRNA预测
利用miRDB数据库、ENCORI数据库、miRTarBase数据库、miRWalk数据库、RNA22数据库、RNAInter数据库、TargetMiner数据库、TargetScan数据库预测筛选出的差异基因(EPB41、ETV6)可能结合的miRNA,结果用R语言“UpSetR”包可视化。结果显示,EPB41、ETV6分别最可能与hsa-miR-30e-5p、hsa-miR-129-5p结合,EPB41、ETV6联合在以上数据库筛选出最可能与hsa-miR-181b-5p、hsa-miR-181c-5p、hsa-miR-181d-5p结合。
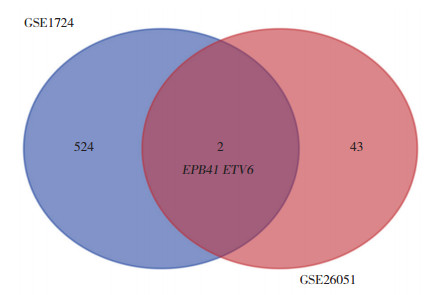
2.5 GO功能富集和KEGG通路富集分析由于GSE26051筛选出的差异基因数目较少,不对其富集。通过R软件对GSE1724筛选出的526个DEGs进行富集分析,以了解其生物学功能,结果显示,有13个GO项目和12个KEGG项目,对富集结果进行可视化处理。其中与肌腱粘连密切相关的GO分析主要富集在转录激活与抑制、G蛋白耦联受体、生长因子活性、受体配体活性等(图 3A);与肌腱粘连高度相关的KEGG通路有PI3K/AKT、MAPK、钙通道等通路(图 3B)。
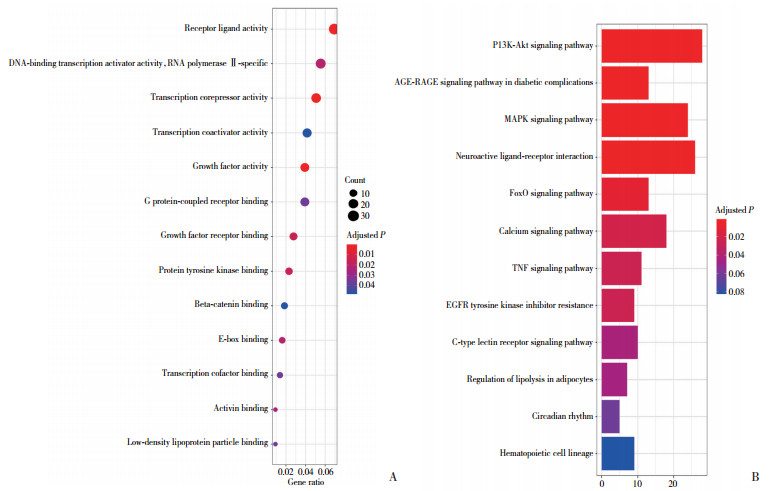
|
| A, bubble plot of GO enrichment analysis of GSE1724; B, KEGG pathway enrichment analysis of GSE1724 DEGs. 图 3 GSE1724筛选出的差异基因功能富集 Fig.3 Functional and signaling pathway analysis of GSE1724 DEGs |
2.6 PPI网络
将EPB41、ETV6导入STRING数据库,在获得的PPI网络中,DLG1、CASK、GYPC、SF3A1、SF3A3、SPTAN1是已被验证过的与EPB41关系密切且相互作用的蛋白(图 4A);BRCA1、SUMO1、MDC1、TP53BP1、SIN3A、NCOR1、ETV7、SOCS1是已被验证过的与ETV6关系密切且相互作用的蛋白(图 4B)。
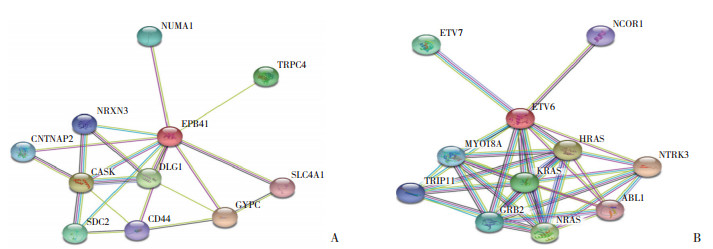
|
| A, EPB41; B, ETV6. 图 4 差异基因的PPI网络 Fig.4 The protein-protein interaction map of the differential gene |
3 讨论
如何改善及预防术后粘连是外科医生关注的重要问题。急性或慢性肌腱损伤后,如治疗效果不理想,愈合时间长,最终会导致粘连。肌腱粘连被认为是继发于炎症过程的纤维化组织反应,持续的炎症会加重纤维化[2]。目前研究较多的TGF-β被认为与肌腱粘连的纤维化最相关[3]。肌腱损伤后,巨噬细胞被募集到愈合部位,释放TGF-β,与成纤维细胞表面TGF-β受体结合,促进成纤维细胞增殖及分化,胶原合成增加,最终导致粘连形成[4-5]。而阻断TGF-β途径被认为是避免肌腱粘连形成且不干预肌腱愈合强度的有效方法[6-7]。除TGF-β外,其他通路也有被研究,如核转录因子-κB(nuclear factor-kappa B,NF-κB)通路及丝裂原活化蛋白激酶(mitogen-activated protein kinase,MAPK)通路也被认为与肌腱粘连的形成相关。NF-κB途径主要参与肌腱修复过程中炎症的初始阶段[8]。MAPK途径则参与外源性成纤维细胞增殖、炎症反应及调节胶原合成,抑制该通路或蛋白磷酸化可阻止肌腱粘连的发生[9]。
通过纳米颗粒、水凝胶、电纺纤维膜、羊膜等[1]物理屏障搭载药物治疗的方式是肌腱粘连主要的治疗策略。从分子水平上寻找有效的药物靶点目前仍是研究难题。本研究检索人类肌腱粘连疾病的相关数据集无结果,考虑肌腱粘连为肌腱损伤未愈导致的疾病,故选用人类慢性肌腱损伤病灶组织芯片数据集GSE26051。而TGF-β与腱外膜中的成纤维细胞功能的关系与肌腱粘连的发生发展高度相关,且肌腱粘连[1]与特发性肺纤维化[10]的标志都是丰富的胶原沉积和成纤维细胞增殖,故搜索到数据集GSE1724,试图从TGF-β及成纤维细胞的角度探讨慢性肌腱损伤导致的肌腱粘连疾病背后的分子机制。通过生物信息学分析,筛选出2个差异基因,即EPB41和ETV6。ETV6属于ETS转录因子家族,作为转录抑制剂,在造血及癌症的恶性转化等方面发挥功能[11]。EPB41通过结构域与各种跨膜蛋白结合,介导肌动蛋白-膜蛋白的连接,在细胞黏附、细胞迁移、细胞膜架构等生理过程中起重要作用[12]。研究[13]表明,在纤维连接蛋白上培养的EPB41-/-角质形成细胞不能形成肌动蛋白应力纤维,不产生局灶性粘连。本研究从TGF-β处理的成纤维细胞及肌腱病灶数据集中筛选出的EPB41是否可能通过影响成纤维细胞的增殖、迁移、侵袭水平而改变肌腱粘连的结局,有待进一步研究。
miRNA在肌腱粘连的分子机制中起关键作用,或可搭载载体治疗肌腱粘连。如骨髓巨噬细胞可分泌直接靶向Smad7的外体miR-21-5p,从而激活肌腱细胞中的纤维生成,参与腱周纤维化[14]。有研究[15]通过将环氧合酶(COX-1和COX-2)工程化的miRNA质粒嵌入透明质酸水凝胶中,在屈肌肌腱愈合的早期阶段,通过炎症反应下调肌腱组织中环氧合酶的表达,减少肌腱粘连。本研究通过预测DEGs可能结合的miRNA,拟构建较完整的miRNA-mRNA网络。为了预测DEGs可能结合的miRNA,分别将EPB41和ETV6的6个miRNA数据库的预测结果取交集,结果聚焦于hsa-miR-181家族。研究[16]表明,miR-181b可介导表皮生长因子受体依赖的血管细胞黏附分子-1表达,介导单核细胞通过整合素α4β1与多形性胶质母细胞瘤之间的黏附。miR-181家族参与包括MYC、TGF-β、Wnt、STAT3、NOTCH、NF-κB、PI3K、MAPK等多条信号通路,可调节细胞增殖、凋亡、代谢、上皮-间质转化、迁移、侵袭、细胞周期、自噬等细胞功能[17]。miR-181b通过影响TGF-β通路在细胞外基质重建功能方面发挥作用[18]。miR-181家族是否能靶作用于EPB41,影响TGF-β通路的表达,从而影响肌腱愈合阶段的细胞外基质重建,则有待后续深入研究。在功能富集方面,GO分析主要富集涉及转录激活与抑制、生长因子活性等,KEGG通路富集涉及PI3K/AKT、MAPK、钙通道、神经激活配体受体等通路,这些是否与本研究的miRNA-mRNA网络、PPI网络相关,还需进一步挖掘。
综上所述,本研究通过分析基因芯片数据集,筛选肌腱粘连病变中的重要基因和通路,构建潜在的miRNA-mRNA、PPI网络,为阐明肌腱粘连的分子机制提供了科学依据。
| [1] |
ZHANG Q, YANG YH, YILDIRIMER L, et al. Advanced technology-driven therapeutic interventions for prevention of tendon adhesion: design, intrinsic and extrinsic factor considerations[J]. Acta Biomater, 2021, 124: 15-32. DOI:10.1016/j.actbio.2021.01.027 |
| [2] |
LUI PPY, ZHANG X, YAO SY, et al. Roles of oxidative stress in acute tendon injury and degenerative tendinopathy-a target for intervention[J]. Int J Mol Sci, 2022, 23(7): 3571. DOI:10.3390/ijms23073571 |
| [3] |
TRELLA KJ, LI J, STYLIANOU E, et al. Genome-wide analysis identifies differential promoter methylation of Leprel2, Foxf1, Mmp25, Igfbp6, and Peg12 in murine tendinopathy[J]. J Orthop Res, 2017, 35(5): 947-955. DOI:10.1002/jor.23393 |
| [4] |
CHANG J, THUNDER R, MOST D, et al. Studies in flexor tendon wound healing: neutralizing antibody to TGF-beta1 increases postope-rative range of motion[J]. Plast Reconstr Surg, 2000, 105(1): 148-155. DOI:10.1097/00006534-200001000-00025 |
| [5] |
HAYS PL, KAWAMURA S, DENG XH, et al. The role of macrophages in early healing of a tendon graft in a bone tunnel[J]. J Bone Joint Surg Am, 2008, 90(3): 565-579. DOI:10.2106/JBJS.F.00531 |
| [6] |
LOISELLE AE, YUKATA K, GEARY MB, et al. Development of antisense oligonucleotide (ASO) technology against Tgf-β signaling to prevent scarring during flexor tendon repair[J]. J Orthop Res, 2015, 33(6): 859-866. DOI:10.1002/jor.22890 |
| [7] |
CHAN KM, FU SC, WONG YP, et al. Expression of transforming growth factor beta isoforms and their roles in tendon healing[J]. Wound Repair Regen, 2008, 16(3): 399-407. DOI:10.1111/j.1524-475X.2008.00379.x |
| [8] |
CHEN S, JIANG SC, ZHENG W, et al. RelA/p65 inhibition prevents tendon adhesion by modulating inflammation, cell proliferation, and apoptosis[J]. Cell Death Dis, 2017, 8(3): e2710. DOI:10.1038/cddis.2017.135 |
| [9] |
JIANG SC, ZHAO X, CHEN S, et al. Down-regulating ERK1/2 and SMAD2/3 phosphorylation by physical barrier of celecoxib-loaded electrospun fibrous membranes prevents tendon adhesions[J]. Biomaterials, 2014, 35(37): 9920-9929. DOI:10.1016/j.biomaterials.2014.08.028 |
| [10] |
STAHL M, SCHUPP J, JÄGER B, et al. Lung collagens perpetuate pulmonary fibrosis via CD204 and M2 macrophage activation[J]. PLoS One, 2013, 8(11): e81382. DOI:10.1371/journal.pone.0081382 |
| [11] |
HOCK H, SHIMAMURA A. ETV6 in hematopoiesis and leukemia predisposition[J]. Semin Hematol, 2017, 54(2): 98-104. DOI:10.1053/j.seminhematol.2017.04.005 |
| [12] |
RANGEL L, LOSPITAO E, RUIZ-SÁENZ A, et al. Alternative polyadenylation in a family of paralogous EPB41 genes generates protein 4.1 diversity[J]. RNA Biol, 2017, 14(2): 236-244. DOI:10.1080/15476286.2016.1270003 |
| [13] |
CHEN LX, HUGHES RA, BAINES AJ, et al. Protein 4.1R regulates cell adhesion, spreading, migration and motility of mouse keratinocytes by modulating surface expression of beta1 integrin[J]. J Cell Sci, 2011, 124(Pt 14): 2478-2487. DOI:10.1242/jcs.078170 |
| [14] |
CUI HM, HE Y, CHEN S, et al. Macrophage-derived miRNA-containing exosomes induce peritendinous fibrosis after tendon injury through the miR-21-5p/Smad7 pathway[J]. Mol Ther Nucleic Acids, 2019, 14: 114-130. DOI:10.1016/j.omtn.2018.11.006 |
| [15] |
ZHOU YL, YANG QQ, YAN YY, et al. Localized delivery of miRNAs targets cyclooxygenases and reduces flexor tendon adhesions[J]. Acta Biomater, 2018, 70: 237-248. DOI:10.1016/j.actbio.2018.01.047 |
| [16] |
LIU YS, LIN HY, LAI SW, et al. miR-181b modulates EGFR-dependent VCAM-1 expression and monocyte adhesion in glioblastoma[J]. Oncogene, 2017, 36(35): 5006-5022. DOI:10.1038/onc.2017.129 |
| [17] |
REZAEI T, AMINI M, HASHEMI ZS, et al. microRNA-181 serves as a dual-role regulator in the development of human cancers[J]. Free Radic Biol Med, 2020, 152: 432-454. DOI:10.1016/j.freeradbiomed.2019.12.043 |
| [18] |
HORI D, DUNKERLY-EYRING B, NOMURA Y, et al. miR-181b regulates vascular stiffness age dependently in part by regulating TGF-β signaling[J]. PLoS One, 2017, 12(3): e0174108. DOI:10.1371/journal.pone.0174108 |
 2022, Vol. 51
2022, Vol. 51