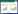文章信息
- 单雨, 卞华, 郭嘉, 陈爽, 卞博
- SHAN Yu, BIAN Hua, GUO Jia, CHEN Shuang, BIAN Bo
- 基于生物信息学的系统性硬化症潜在miRNA-mRNA调控网络的构建
- Construction of a potential miRNA-mRNA regulatory network in systemic sclerosis based on bioinformatics
- 中国医科大学学报, 2022, 51(10): 876-883, 895
- Journal of China Medical University, 2022, 51(10): 876-883, 895
-
文章历史
- 收稿日期:2021-11-18
- 网络出版时间:2022-10-01 7:52
2. 南阳理工学院张仲景国医国药学院, 河南 南阳 473004
2. Zhang Zhongjing College of Traditional Chinese Medicine, Nanyang Institute of Technology, Nanyang 473004, China
系统性硬化症(systemic sclerosis,SSc)是以皮肤和内脏纤维化、炎症和血管病变为特征的特发性全身性自身免疫性疾病,也是一种慢性炎性结缔组织疾病,累及皮肤、肌肉骨骼系统和多个内脏器官[1-2]。我国SSc发病率和患病率居自身免疫性疾病的第三位,仅次于类风湿性关节炎和系统性红斑狼疮[3]。我国成年人SSc的年发病率较高,为(190~250)/100万[4-5]。研究[6]显示,SSc的整体发病率不高,但却有较高的病死率。目前,SSc的发病机制还未明确,因此给临床治疗带来了极大的困难。
微RNA(microRNA,miRNA)是一类内源性小分子非编码RNA,由18~25个核苷酸组成[7]。miRNA通过直接降解mRNA或阻断蛋白质的翻译过程调控基因表达,从而参与细胞增殖、分化、凋亡等许多生物学过程[8-9]。miRNA通过靶向细胞外基质(extracellular matrix,ECM)蛋白、结缔组织生长因子(connective tissue growth factor,CTGF)、转化生长因子-β(transforming growth factor-β,TGF-β)通路、上皮-间质转化(epithelial-mesenchymal transition,EMT)或肌成纤维细胞增殖来增强或抑制纤维化[10]。miRNA相对稳定,可在组织和体液中检测到,因此有希望成为监测疾病进程和治疗反应的生物标志物[11]。研究[12]发现miRNA-181b-5p、miRNA-223-3p以及miRNA-21-5p在SSc中的表达显著增加。WUTTGE等[13]研究发现miR-20a-5p和miR-203a-3p可作为SSc相关的肺动脉高压发展的预测因子。bortolott等[14]发现包括miRNA-409在内的特定miRNA可作为SSc诊断、进展评估和抗纤维化治疗的生物标志物,这些都说明miRNA在SSc诊断和治疗中具有极大潜力。本研究基于生物信息学分析构建SSc潜在的miRNA-mRNA调控网络,希望能够提供有前景的SSc诊断和治疗靶点。
1 材料与方法 1.1 miRNA数据集获取搜索基因表达图谱(gene expression omnibus,GEO)数据库高通量基因表达数据库(https://www.ncbi.nlm.nih.gov/geo/),关键词选择“systemic sclerosis”或“scleroderma”;研究类型选择“array by non-coding RNA profiles”;种族选择“home sapiens”,筛选SSc血浆相关miRNA数据集,见表 1。选取GSE81293数据集进行后续分析。
| Accession | Platform | SSc | Normal | Gene/miRNA |
| GSE81293 | GPL16384 | 15 | 5 | miRNA |
| GSE146093 | GPL23459 | 48 | 16 | Gene |
1.2 差异表达miRNA(differential expression miRNA,DEM)筛选、DEM靶基因获取和靶基因上游转录因子的分析
GEO2R(https://www.ncbi.nlm.nih.gov/geo/geo2r/)是基于R语言的网络应用程序,可用于筛选SSc患者和正常健康者之间的DEM。采用Benjamini和Hochberg方法调整P值,纠正假阳性结果的发生,将调整后的P < 0.05和|log FC | > 1设置为阈值。利用TBtools工具包在样本中筛选出DEM。利用MiRNet(https://www.mirnet.ca/)获取DEM的潜在靶基因。利用FunRich(https://www.funrich.org/)分析靶基因的潜在上游转录因子,P < 0.05为差异有统计学意义。
1.3 靶基因基因本体(gene ontology,GO)和京都基因和基因组数据库(Kyoto encyclopedia of genes and genomes,KEGG)分析为研究DEM靶基因的生物学功能,利用DAVID在线注释、可视化和集成发现数据库(https://www.david.ncifcrf.gov/)进行GO和KEGG路径分析,P < 0.05为差异有统计学意义。
1.4 蛋白质-蛋白质相互作用(protein-protein interaction,PPI)网络的构建以及核心(Hub)基因的筛选利用STRING在线数据库(https://www.stringdb.org/)对DEM的靶基因进行PPI网络构建和分析,并在Cytoscape软件中进行可视化。利用Cytoscape的插件CytoHubba,通过几种拓扑算法,根据预先加载的PPI网络的特征对节点进行排序。采用最大Clique中心性(maximal clique centrality,MCC)方法,筛选出在PPI网络中排名前10位的Hub基因。
1.5 Hub基因表达的验证从GEO数据库下载GSE146093数据集的表达矩阵及其GPL570注释文件,根据注释文件找到表达矩阵中相关的Hub基因,验证并分析Hub基因的表达。显著Hub基因需要满足2个条件:(1)上调DEM的靶基因显著下调或者下调的DEM的靶基因显著上调;(2)P < 0.05为差异有统计学意义。
2 结果 2.1 DEM筛选结果使用GEO在线工具对GSE81293数据集进行分析,该数据集包含15个SSc样本和5个正常对照样本,以调整后P < 0.05和|logFC| > 1为阈值。筛选得到26个DEM,其中上调有let-7e-5p、miR-455-3p,下调有miR-4459、miR-4507、miR-4417、miR-3687、miR-1246、miR-4668-5p、miR-4530、miR-4299、miR-4516、miR-663b、miR-762、miR-665、miR-4463、miR-4656、miR-3663-3p、miR-4505、miR-4440、miR-4685-5p、miR-4281、miR-149-5p、miR-4793-3p、miR-27a-5p、miR-1469、miR-4508。见表 2、图 1。
| ID | t | B | logFC | P |
| hsa-miR-4459_st | -6.64 | 4.994 5 | -2.75 | 0.003 62 |
| hsa-miR-4507_st | -5.99 | 3.743 2 | -2.15 | 0.004 80 |
| hsa-let-7e_st | 5.50 | 2.774 5 | 1.02 | 0.010 39 |
| hsa-miR-4417_st | -5.37 | 2.491 6 | -3.55 | 0.011 31 |
| hsa-miR-3687_st | -5.07 | 1.876 0 | -4.35 | 0.015 54 |
| hsa-miR-1246_st | -5.03 | 1.791 4 | -4.42 | 0.015 54 |
| hsa-miR-4668-5p_st | -4.89 | 1.501 1 | -1.54 | 0.015 54 |
| hsa-miR-4530_st | -4.87 | 1.472 0 | -2.15 | 0.015 54 |
| hsa-miR-4299_st | -4.80 | 1.309 5 | -1.97 | 0.016 99 |
| hsa-miR-4516_st | -4.63 | 0.946 6 | -1.31 | 0.021 56 |
| hsa-miR-455-3p_st | 4.58 | 0.854 9 | 1.25 | 0.022 22 |
| hsa-miR-663b_st | -4.55 | 0.779 6 | -2.44 | 0.022 60 |
| hsa-miR-762_st | -4.45 | 0.578 8 | -1.45 | 0.025 19 |
| hsa-miR-665_st | -4.31 | 0.284 3 | -1.84 | 0.029 44 |
| hsa-miR-4463_st | -4.31 | 0.283 4 | -1.24 | 0.029 44 |
| hsa-miR-4656_st | -4.28 | 0.214 9 | -2.41 | 0.030 23 |
| hsa-miR-3663-3p_st | -4.24 | 0.135 4 | -2.61 | 0.030 23 |
| hsa-miR-4505_st | -4.13 | -0.117 4 | -2.80 | 0.034 36 |
| hsa-miR-4440_st | -4.12 | -0.125 7 | -2.12 | 0.034 36 |
| hsa-miR-4685-5p_st | -4.01 | -0.363 9 | -1.21 | 0.041 49 |
| hsa-miR-4281_st | -3.91 | -0.588 0 | -1.39 | 0.049 05 |
| hsa-miR-149-star_st | -3.88 | -0.636 9 | -1.49 | 0.049 05 |
| hsa-miR-4793-3p_st | -3.87 | -0.663 9 | -1.90 | 0.049 05 |
| hsa-miR-27a-star_st | -3.86 | -0.690 9 | -2.29 | 0.049 05 |
| hsa-miR-1469_st | -3.84 | -0.731 9 | -1.13 | 0.049 05 |
| hsa-miR-4508_st | -3.84 | -0.736 8 | -1.12 | 0.049 05 |
|
| 图 1 DEM热图 Fig.1 Heat map of DEM |
2.2 DEM靶基因的筛选结果
通过miRNet数据库共筛选出候选DEM的靶向基因5 450个,其中上调1 407个,下调4 043个。每个DEM的靶基因数量见表 3。
| DEM | Number | DEM | Number | |
| Down-regulated DEM | 4 043 | hsa-miR-4656 | 71 | |
| hsa-miR-4459 | 691 | hsa-miR-3663-3p | 56 | |
| hsa-miR-4507 | 66 | hsa-miR-4505 | 181 | |
| hsa-miR-4417 | 59 | hsa-miR-4440 | 6 | |
| hsa-miR-3687 | 13 | hsa-miR-4685-5p | 125 | |
| hsa-miR-1246 | 44 | hsa-miR-4281 | 35 | |
| hsa-miR-4668-5p | 402 | hsa-miR-149-5p | 397 | |
| hsa-miR-4530 | 134 | hsa-miR-4793-3p | 418 | |
| hsa-miR-4299 | 47 | hsa-miR-27a-5p | 45 | |
| hsa-miR-4516 | 205 | hsa-miR-1469 | 3 | |
| hsa-miR-663b | 60 | hsa-miR-4508 | 40 | |
| hsa-miR-762 | 123 | Up-regulated DEM | 1 407 | |
| hsa-miR-665 | 737 | hsa-let-7e | 623 | |
| hsa-miR-4463 | 85 | hsa-miR-455-3p | 784 |
2.3 靶基因转录因子的分析
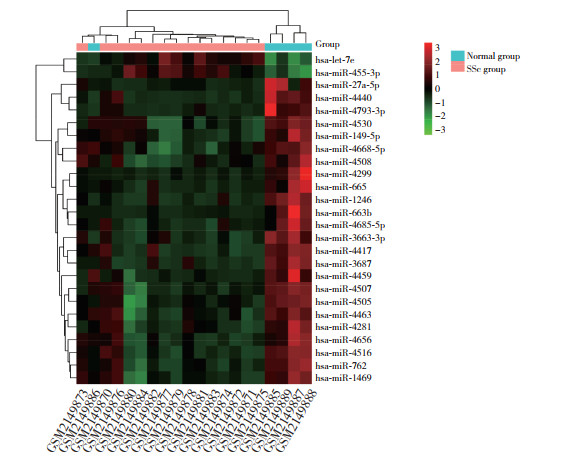
利用FunRich软件预测DEM靶基因的转录因子,结果显示,上调DEM的转录因子包括NFYA、SP4、NRF1、ELK1、SP1、YY1;下调DEM的转录因子包括CACO、KLF7、E2F1、YY1、MYC、SP1。见图 2。
|
| A, up-regulated; B, down-regulated. 图 2 DEM上调和下调的转录因子 Fig.2 Up-and down-regulated transcription factors in DEM |
2.4 靶基因GO和KEGG分析
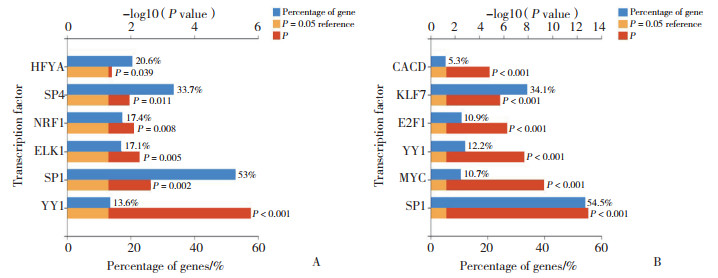
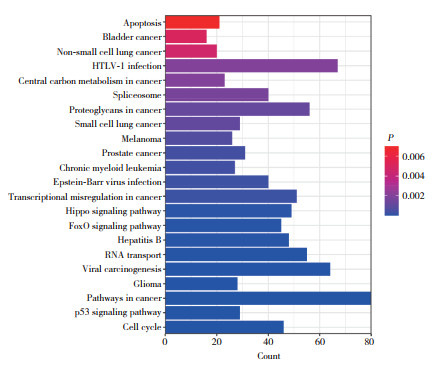
生物过程(biological process,BP)分析显示,DEM靶基因主要富集在DNA转录及其调控、细胞分裂和细胞间黏附方面;细胞组分(cellular component,CC)分析显示,靶基因主要富集于细胞膜、细胞溶质、核质以及蛋白质磺酰化等方面;分子功能(molecular function,MF)分析表明,靶基因在多聚腺嘌呤核糖核苷酸、DNA结合、转录因子结合、钙黏蛋白结合参与细胞黏附等方面显著富集。KEGG通路分析显示,DEM靶基因显著富集于P53信号通路、癌症通路、FOXO信号通路、神经胶质瘤、RNA转运、乙型肝炎、转录失调等。见图 3、4。
|
| 图 3 靶基因GO分析条形图 Fig.3 Bar chart of target gene GO analysis |
|
| 图 4 靶基因KEGG分析条形图 Fig.4 Bar chart of target gene KEGG analysis |
2.5 PPI和DEM-Hub基因网络的构建
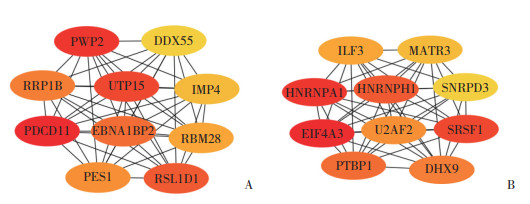
通过STRING数据库分别建立了上调和下调DEM靶基因的PPI网络。然后将上述的PPI网络上传到Cytoscape软件,并使用Cytohubba插件筛选Hub基因。上调和下调的前10位Hub基因见图 5。
|
| A, up-regulated; B, down-regulated. 图 5 DEM靶基因的PPI网络 Fig.5 Protein-protein interaction network of DEM target genes |
为了更好地研究DEM在SSc中的分子机制,利用Cytoscape软件构建了DEM-Hub基因网络。结果显示,miR-455-3p与6个Hub基因(DDX55、RBM28、EBNA1BP2、IMP4、PWP2、RRP1B)相互作用;let-7e-5p与4个Hub基因(UTP15、RSL1D1、PDCD11、PES1)相互作用;miR-149-5p与6个Hub基因(DHX9、SRSF1、ILF3、HNRNPH1、MATR3、HNRNPA1)相互作用;mir-4440与PTBP1相互作用。
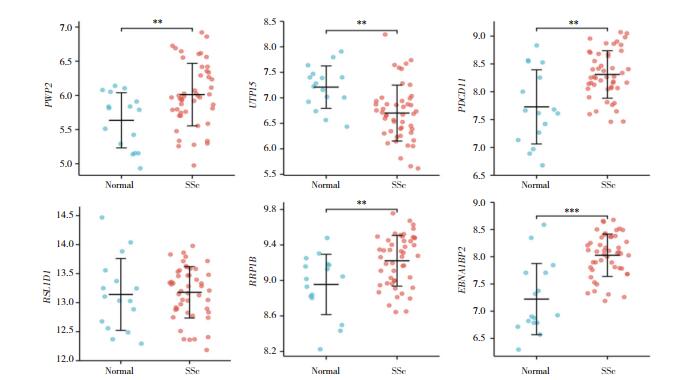
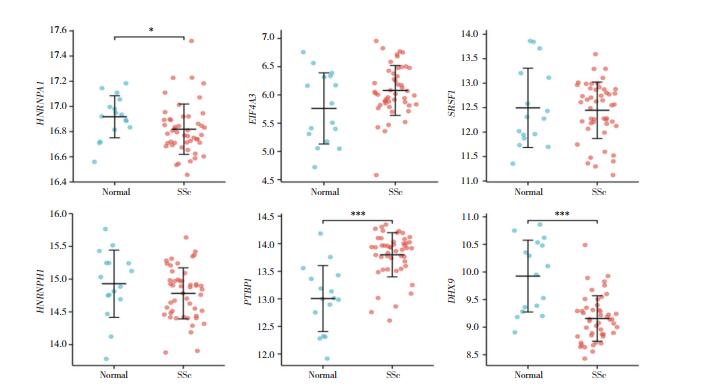
2.6 Hub基因表达的印证根据DEM-Hub基因网络,利用GSE146093数据集,印证前6个上调DEM靶基因(PWP2、UTP15、PDCD11、RSL1D1、RRP1B、EBNA1BP2)和下调DEM靶基因(HNRNPA1、EIF4A3、SRSFI、HNRNPH1、PTBP1、DHX9)的表达水平。对于上调的DEM,只有UTP15的表达呈现下降状态,而PWP2、PDCD11、RRP1B、EBNA1BP2的表达则相反(图 6)。下调的DEM中,PTBP1的表达显著增加,HNRNPA1、DHX9的表达反而下降(图 7)。因此认为let-7e-5p-UTP15和miR-149-5p-PTBP1是SSc潜在的调控途径,见表 4。
|
| ** P < 0.01;*** P < 0.001. 图 6 上调DEM中Hub基因的表达印证 Fig.6 Validation of the expression of Hub gene in up-regulated DEM |
|
| * P < 0.05;*** P < 0.001. 图 7 下调DEM中Hub基因的表达印证 Fig.7 Validation of the expression of Hub gene in down-regulated DEM |
| Gene | Difference | P |
| Hub gene of up-regulated DEM | ||
| PWP2 | 0.376 | 0.005 |
| UTP15 | -0.509 | 0.001 |
| PDCD11 | 0.528 | 0.004 |
| RSL1D1 | 0.038 | 0.791 |
| RRP1B | 0.267 | 0.003 |
| EBNA1BP2 | 0.805 | < 0.001 |
| Hub gene of down-regulated DEM | ||
| HNRNPA1 | -0.123 | 0.025 |
| EIF4A3 | 0.318 | 0.076 |
| SRSF1 | -0.049 | 0.793 |
| HNRNPH1 | -0.147 | 0.234 |
| PTBP1 | 0.802 | < 0.001 |
| DHX9 | -0.768 | < 0.001 |
3 讨论
随着微阵列技术的发展,对各种疾病进展中的数千种基因的变化有了更深的研究。本研究提取GSE81293数据集数据,识别和筛选SSc与正常人血浆的DEM。结果显示,共筛选出了2个上调的DEM(let-7e-5p、miR-455-3p)和24个下调的DEM(miR-4459、miR-4507、miR-4417、miR-3687、miR-1246、miR-4668-5p、miR-4530、miR-4299、miR-4516、miR-663b、miR-762、miR-665、miR-4463、miR-4656、miR-3663-3p、miR-4505、miR-4440、miR-4685-5p、miR-4281、miR-149-5p、miR-4793-3p、miR-27a-5p、miR-1469、miR-4508)。
研究[15]表明,miRNA表达可以被转录因子调控,本研究预测了SSc与正常人血浆的DEM的上游转录因子。其中,上调DEM的转录因子主要是SP1、SP4和NFYA,下调DEM的转录因子是SP1和KLF7,而且SP1在上行调控和下行调控中均占比最高。SP1是重要的转录因子,有研究[16-17]指出SP1可通过参与调控肿瘤细胞增殖相关转录因子FOXM1在卵巢癌中的表达,促进卵巢癌病情进展。KYPRIOTOU等[18]发现SP1和SP4转录因子的相互作用可以促进SSc成纤维细胞中Ⅰ型胶原的表达。NFYA在多种癌症中的表达增加,与不良预后相关;并且在氧化固醇结合蛋白的基础转录中起着重要的作用,可以通过P53/SRE BF2 /NFYA信号通路来下调氧化固醇结合蛋白2的表达[19]。KLF7是神经系统发育和脂肪形成的关键因子,JIANG等[20]研究表明KLF7的表达可诱导内皮细胞的氧化应激、炎症和细胞凋亡。
GO和KEGG分析显示,DEM主要富集于DNA转录及其调控、细胞分裂和细胞间黏附方面以及P53信号通路、FOXO信号通路。P53可通过调控多种细胞免疫相关信号通路来参与机体的免疫应答[21],通过上调Toll样受体增加促炎性细胞因子的产生[22],P53功能失调与SSc患者的成纤维细胞中的细胞凋亡途径受损相关,可能导致SSc患者皮肤的慢性纤维化的发生[23]。FOXO是介导细胞存活的经典通路,PI3K/Akt/FOXO的相互作用对机体的免疫功能产生影响,在机体细胞的增殖、凋亡等方面发挥重要作用[24]。
通过构建DEM-Hub基因网络,本研究发现大多数Hub基因与miR-455-3p、let-7e-5p、miR-149-5p联系密切。在排名前12位的Hub基因中,只有UTP15、PTBP1的表达与GSE146093数据集一致,可能是样本的来源不同所致。MOUILLESSEAUX等[25]发现UTP15的缺陷对细胞存活和血管系统的正常运行产生了影响,并且与P53信号通路联系密切,而GUI等[26]研究结果显示let-7e-5p可通过激活JAK1/ STAT3通路抑制促炎性细胞因子表达。本研究验证过程中发现,作为上调let-7e-5p的靶基因UTP15在SSc中下调表达,因此let-7e-5p-UTP15有可能作为SSc潜在的调控途径。PTBP1在人类几乎所有的细胞类型中都有表达,并且介导多种细胞过程,包括神经元细胞的生长和分化以及免疫细胞的激活,其功能受多种分子调控,包括miRNA、长链非编码RNA和RNA结合蛋白[27]。通过miR-4440-PTBP1途径调控PTBP1的表达从而抑制免疫细胞的激活,可能成为控制SSc发展的一种有效方法。
综上所述,基于GEO数据库和生物信息学分析,本研究构建了SSc血浆中潜在的miRNA-mRNA调控网络,发现了let-7e-5p-UTP15、miR-4440-PTBP1可作为SSc患者诊断和治疗的潜在生物标志物。本研究的不足之处:(1)仅研究了SSc与正常对照之间的miRNA和mRNA,在SSc的不同阶段结果可能会不同;(2)与生物标志物分析通常需要的样本量比较,本研究采用的数据集的样本量较小;(3)仅基于公共数据库预测miRNA-mRNA的相互作用结果存在局限性,还需进一步通过体内和体外实验验证。
| [1] |
RAMOS PS, SILVER RM, FEGHALI-BOSTWICK CA. Genetics of systemic sclerosis: recent advances[J]. Curr Opin Rheumatol, 2015, 27(6): 521-529. DOI:10.1097/BOR.0000000000000214 |
| [2] |
SKOPIS M, ELGHAWY AA, POCIURKO B, et al. Pyoderma gangrenosum associated with limited cutaneous systemic sclerosis: a rare case with literature review[J]. Clin Rheumatol, 2021, 40(3): 1141-1145. DOI:10.1007/s10067-020-05285-z |
| [3] |
杨书琦, 张慧芳, 宋书林, 等. 系统性硬化症发病机制的研究进展[J]. 医学综述, 2018, 24(1): 17-21. DOI:10.3969/j.issn.1006-2084.2018.01.004 |
| [4] |
SHAH AA, CASCIOLA-ROSEN L. Mechanistic and clinical insights at the scleroderma-cancer interface[J]. J Scleroderma Relat Disord, 2017, 2(3): 153-159. DOI:10.5301/jsrd.5000250 |
| [5] |
WANG HC, WANG XJ. The risk of conducting overgrafting of fat for treating facial atrophy induced by localized scleroderma[J]. Facial Plast Surg, 2021, 37(3): 412-414. DOI:10.1055/s-0040-1717059 |
| [6] |
SHANMUGAM VK, FRECH TM, STEEN VD, et al. Collaborative National Quality and Efficacy Registry (CONQUER) for scleroderma: outcomes from a multicenter US-based systemic sclerosis registry[J]. Clin Rheumatol, 2020, 39(1): 93-102. DOI:10.1007/s10067-019-04792-y |
| [7] |
OIKAWA S, SHIN J, AKAMA T, et al. Dicer-mediated miRNA processing is not involved in controlling muscle mass during muscle atrophy[J]. Sci Rep, 2021, 11(1): 19361. DOI:10.1038/s41598-021-98545-0 |
| [8] |
BARTEL DP. microRNAs: genomics, biogenesis, mechanism, and function[J]. Cell, 2004, 116(2): 281-297. DOI:10.1016/s0092-8674(04)00045-5 |
| [9] |
BARTEL DP. microRNAs: target recognition and regulatory functions[J]. Cell, 2009, 136(2): 215-233. DOI:10.1016/j.cell.2009.01.002 |
| [10] |
UTHAYOPAS K, DE SÁ AGC, ALAVI A, et al. TSMDA: Target and symptom-based computational model for miRNA-disease-association prediction[J]. Mol Ther Nucleic Acids, 2021, 26: 536-546. DOI:10.1016/j.omtn.2021.08.016 |
| [11] |
ZHANG QZ, WANG Y, ZHOU YT, et al. Potential biomarkers of miRNA in non-functional pituitary adenomas[J]. World J Surg Oncol, 2021, 19(1): 270. DOI:10.1186/s12957-021-02383-3 |
| [12] |
WOLSKA-GAWRON K, BARTOSIŃSKA J, RUSEK M, et al. Circulating miRNA-181b-5p, miRNA-223-3p, miRNA-210-3p, let 7i-5p, miRNA-21-5p and miRNA-29a-3p in patients with localized scleroderma as potential biomarkers[J]. Sci Rep, 2020, 10(1): 20218. DOI:10.1038/s41598-020-76995-2 |
| [13] |
WUTTGE DM, CARLSEN AL, TEKU G, et al. Specific autoantibody profiles and disease subgroups correlate with circulating micro-RNA in systemic sclerosis[J]. Rheumatology (Oxford), 2015, 54(11): 2100-2107. DOI:10.1093/rheumatology/kev234 |
| [14] |
BORTOLOTTI D, SOFFRITTI I, D'ACCOLTI M, et al. HHV-6A infection of endometrial epithelial cells affects miRNA expression and trophoblast cell attachment[J]. Reproductive Sci Thousand Oaks Calif, 2020, 27(3): 779-786. DOI:10.1007/s43032-019-00102-8 |
| [15] |
MEREDITH H, NHAM T. miRNA interplay: mechanisms and consequences in cancer[J]. Dis Model Mech, 2021, 14(4): dmm047662. DOI:10.1242/dmm.047662 |
| [16] |
MAHMOUD MM, BELAL NA, YOUSSIF A. Prediction of transcription factor binding sites of SP1 on human Chromosome1[J]. Appl Sci, 2021, 11(11): 5123. DOI:10.3390/app11115123 |
| [17] |
ZHAO YN, YAO DJ, LI Y, et al. Loss of polarity protein Par3 is mediated by transcription factor Sp1 in breast cancer[J]. Biochem Biophys Res Commun, 2021, 561: 172-179. DOI:10.1016/j.bbrc.2021.05.025 |
| [18] |
KYPRIOTOU M, BEAUCHEF G, CHADJICHRISTOS C, et al. Human collagen Krox up-regulates type I collagen expression in normal and Scleroderma fibroblasts through interaction with Sp1 and Sp3 transcription factors[J]. J Biol Chem, 2007, 282(44): 32000-32014. DOI:10.1074/jbc.M705197200 |
| [19] |
WANG Q, LIN CS, ZHANG C, et al. 25-hydroxycholesterol down-regulates oxysterol binding protein like 2(OSBPL2) via the p53/SREBF2/NFYA signaling pathway[J]. J Steroid Biochem Mol Biol, 2019, 187: 17-26. DOI:10.1016/j.jsbmb.2018.10.018 |
| [20] |
JIANG HQ, LV JR. microRNA-301a-3p increases oxidative stress, inflammation and apoptosis in ox-LDL-induced HUVECs by targeting KLF7[J]. Exp Ther Med, 2021, 21(6): 569. DOI:10.3892/etm.2021.10001 |
| [21] |
MEEK DW. Regulation of the p53 response and its relationship to cancer[J]. Biochem J, 2015, 469(3): 325-346. DOI:10.1042/BJ20150517 |
| [22] |
MORTAZ E, ADCOCK IM, ABEDINI A, et al. The role of pattern recognition receptors in lung sarcoidosis[J]. Eur J Pharmacol, 2017, 808: 44-48. DOI:10.1016/j.ejphar.2017.01.020 |
| [23] |
MAHMOUDI MB, ABED KHOJASTEH M, ALSAHEBFOSOUL F, et al. Expressions of p53 and PUMA in fibroblasts of systemic sclerosis patients are normal at transcription level[J]. J Cosmet Dermatol, 2018, 17(3): 549-554. DOI:10.1111/jocd.12420 |
| [24] |
CUI XB, WANG C, LI L, et al. Insulin decreases myocardial adiponectin receptor 1 expression via PI3K/Akt and FoxO1 pathway[J]. Cardiovasc Res, 2012, 93(1): 69-78. DOI:10.1093/cvr/cvr273 |
| [25] |
MOUILLESSEAUX K, CHEN JN. Mutation in UTP15 disrupts vascular patterning in a p53-dependent manner in zebrafish embryos[J]. PLoS One, 2011, 6(9): e25013. DOI:10.1371/journal.pone.0025013 |
| [26] |
GUI L, ZHANG QQ, CAI Y, et al. Front cover: effects of let-7e on LPS-stimulated THP-1 cells assessed by iTRAQ proteomic analysis[J]. Prot Clin Appl, 2018, 12(5): 1870040. DOI:10.1002/prca.201870040 |
| [27] |
CHENG Y, WANG N, ZHAO L, et al. Knockdown of NOVA1 inhibits inflammation and migration of asthmatic airway smooth muscle cells to regulate PTEN/Akt pathway by targeting PTBP1[J]. Mol Immunol, 2021, 138: 31-37. DOI:10.1016/j.molimm.2021.07.016 |
 2022, Vol. 51
2022, Vol. 51