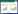文章信息
- 王佳敏, 初冬美, 张家铜, 员瑜, 赵滢, 孙威, 张天彪
- WANG Jiamin, CHU Dongmei, ZHANG Jiatong, YUAN Yu, ZHAO Ying, SUN Wei, ZHANG Tianbiao
- 基于GEO和TCGA联合分析SUFL1对胃癌总体生存率的影响
- Effect of SUFL1 on overall survival rate of gastric cancer based on Gene Expression Omnibus and The Cancer Genome Atlas
- 中国医科大学学报, 2022, 51(10): 865-869
- Journal of China Medical University, 2022, 51(10): 865-869
-
文章历史
- 收稿日期:2021-12-08
- 网络出版时间:2022-09-30 19:35
2. 中国医科大学临床一系, 沈阳 110122;
3. 中国医科大学附属盛京医院普通外科, 沈阳 110004
2. The First Division of Clinical Medicine, China Medical University, Shenyang 110122, China;
3. Department of General Surgery, Shengjing Hospital of China Medical University, Shenyang 110004, China
胃癌是世界范围内最常见的恶性肿瘤之一[1]。2020年新增胃癌病例超过100万例,其发病率排名第五,仅次于乳腺癌、肺癌、结直肠癌和前列腺癌。估计每年胃癌导致76.9万人死亡,其死亡率排名第四,仅次于肺癌、结直肠癌和肝癌[2]。由于胃癌的发病较其他癌症更隐蔽,症状不典型,且缺乏有效的早期生物标志物,确诊时往往处于中晚期,因此胃癌的防治极具挑战性[3-4]。目前的综合治疗方案主要包括手术、化疗和免疫治疗,但中晚期胃癌转移和复发往往导致最终治疗失败[4-5]。早期胃癌和进展期胃癌的预后有很大差异[3-6],因此迫切需要寻找有效的早期生物标志物和相关干预靶点来抑制胃癌的进展。
硫酸酯酶1(sulfatase 1,SUFL1)是近年发现的硫酸酯酶家族的新成员,位于细胞表面,通过调节细胞表面的硫酸肝素蛋白聚糖的硫酸化状态,参与细胞信号传导,从而发挥调节多种生理和病理过程的作用[7]。研究[8-11]证实,SUFL1在胃癌组织中的表达显著高于正常组织,但是SUFL1与胃癌总体生存率(overall survival,OS)的关系尚无研究报道。本研究通过分析胃癌组织中SUFL1基因的表达,进一步解析SUFL1基因表达对胃癌患者OS的影响,从而为胃癌的诊断和治疗提供新的思路。
1 材料与方法 1.1 数据来源基因表达谱数据(GSE19826、GSE79973)来自基因表达数据库(Gene Expression Omnibus,GEO)。所有纳入的数据集符合以下标准:(1)使用的组织样本来自人类胃癌组织或正常组织;(2)至少包含10个样本。作为目前最权威的癌症基因信息数据库,癌症基因组图谱(The Cancer Genome Atlas,TCGA)数据库覆盖33种癌症类型,含有超过30 000例肿瘤样本以及20 000个基因表达的信息。本研究利用R语言下载胃癌测序数据,其中包括373例原发性胃癌组织和32例正常对照组织。
1.2 差异表达RNA筛选应用R/Bioconductor软件中的Limma包对每个GEO数据集的GSE19826、GSE79973矩阵数据进行归一化和log2转换,并对每个微阵列中的差异表达基因(differential expression gene,DEG)进行筛选。利用RobustRankAggreg,对从2个数据集中识别出的DEG进行基因整合。DEG筛选条件:两样品(组)间倍数变化(fold change,FC)以2为底的对数(|log2FC|)≥1,误报率(false discovery rate,FDR) < 0.05。
从TCGA-STAD数据集中检索373例原发性胃癌组织和32例正常组织的3级数据(mRNA-seq数据、miRNA-seq数据和临床数据)。405例样本具有完整的mRNA-seq数据。用R软件的package edgeR对原始计数数据进行归一化处理,并鉴定正常组织与胃癌组织间的DEG,DEG筛选条件为FDR < 0.05,|log2FC| ≥1,在排除特异性TNM分期不完整、生存数据不完整的患者后,351例具有完整mRNA-seq数据和临床数据的接受胃切除术的患者最终纳入临床相关研究。
1.3 利用cBioPortal for Cancer Genomics进行生物信息学分析利用cBioPortal for Cancer Genomics检测TCGA-STAD的SUFL1基因改变。通过Kaplan-Meier生存曲线评估SUFL1基因突变与胃癌患者OS的关系。
1.4 统计学分析根据SUFL1表达的中位数,将胃癌样本分为SUFL1高表达组和低表达组。采用Kaplan-Meier生存曲线分析,比较SUFL1低表达组和高表达组的OS。确定最优OS阈值。采用Cox比例风险模型,识别与OS相关的独立指标,并计算风险比(hazard ratio,HR)和95%置信区间(confidence interval,CI)。根据OS的Cox多变量分析结果构建诺模图。应用受试者操作特征(receiver operating characteristic,ROC)曲线判断SUFL1对胃癌的诊断价值,并计算曲线下面积(area under the curve,AUC)。采用R语言4.0.4版进行上述统计分析。双尾P < 0.05为差异有统计学意义。
2 结果 2.1 DEG分析根据GEO中的胃癌芯片数据集GSE19826、GSE79973,利用R语言整理并绘制DEG的火山图和热图。
火山图结果(图 1)中,待分析样本相对于对照样本表达量上调基因用红色点表示,下调基因用蓝色点表示,无显著差异基因用灰色点表示。判断标准为|log2FC| ≥1,FDR < 0.05。
|
| A, GSE19826 differential analysis of volcano plot; B, GSE79973 differential analysis of volcano plot. 图 1 GEO数据集中DEG火山图 Fig.1 Volcano plot of DEGs in the GEO dataset |
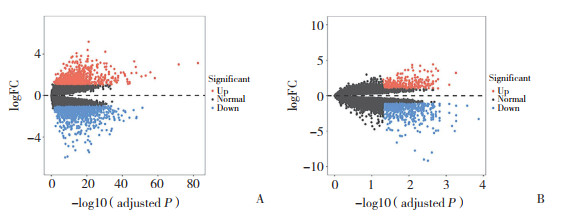
热图结果(图 2)中,表达上调的基因用红色表示,表达下调的基因用蓝色表示。分别选出排名前20的表达上调和表达下调的DEG,根据先前的研究[8]结果,初步选择SUFL1作为下一步研究分析的目的基因。接着应用TCGA数据库中的TCGA-STAD测序数据,进一步验证SUFL1在胃癌组织样本与正常组织样本中是否差异表达,并且绘制差异分布的散点图。结果显示,胃癌组织样本中SUFL1表达显著高于正常组织样本。
|
| A, GSE19826 and GSE79973 differential analysis heat map; B, scatter plot of SUFL1 gene differences in the TCGA-STAD dataset. 图 2 GEO中DEG热图和TCGA中DEG散点图 Fig.2 Heat map of DEGs in GEO and scatter plot of DEGs in TCGA |
2.2 cBioPortal分析SUFL1基因突变情况
使用cBioPortal在线分析网站检测TCGA-STAD的拷贝数改变,结果显示,胃癌中SUFL1基因突变率为10%,并且主要与扩增和SUFL1基因表达增加有关。见图 3。

|
| 图 3 SUFL1在胃癌中基因突变情况 Fig.3 Gene mutation of SUFL1 in gastric cancer |
2.3 SUFL1高表达独立预测胃癌OS较差
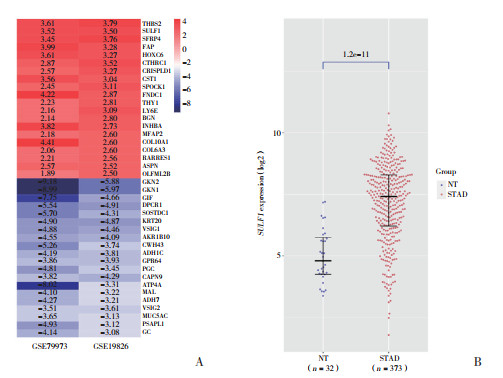
通过Kaplan-Meier生存曲线,评估SUFL1在胃癌中的预后价值。结果显示,SUFL1高表达的胃癌患者OS较差(P < 0.05)。见图 4。
|
| 图 4 胃癌OS的Kaplan-Meier生存曲线 Fig.4 Kaplan-Meier survival curve of OS in gastric cancer |
2.4 SUFL1表达与胃癌进展相关
根据最优OS阈值18.80将纳入研究的351例患者分为SUFL1低表达组和高表达组,探讨SUFL1表达的临床意义。利用R语言,对TCGA中的胃癌临床数据进行单因素和多因素Cox回归分析。单因素Cox回归分析结果显示,年龄、肿瘤分期、SUFL1基因表达的差异有统计学意义(P < 0.05)。进一步进行多因素Cox回归分析,结果发现SUFL1基因表达是胃癌患者OS的独立预后标志物。见表 1。利用R语言绘制可视化的森林图,见图 5。
| Item | Univariate analysis | Multivariate analysis | |||||
| HR | 95% CI | P | HR | 95% CI | P | ||
| Age | 1.578 0 | 1.122 4-2.219 3 | 0.008 0 | 1.665 0 | 1.170 4-2.368 6 | 0.004 6 | |
| Sex | 0.754 3 | 0.527 1-1.079 4 | 0.123 1 | ||||
| Grade | 0.545 8 | 0.195 3-1.525 8 | 0.248 3 | ||||
| History | 0.988 2 | 0.483 7-2.019 0 | 0.974 1 | ||||
| Stage | 1.972 0 | 1.374 5-2.828 0 | 0.000 2 | 1.987 8 | 1.385 5-2.851 9 | 0.000 2 | |
| SULF1 expression | 1.576 2 | 1.132 5-2.193 7 | 0.007 0 | 1.514 9 | 1.077 8-2.129 3 | 0.016 8 | |
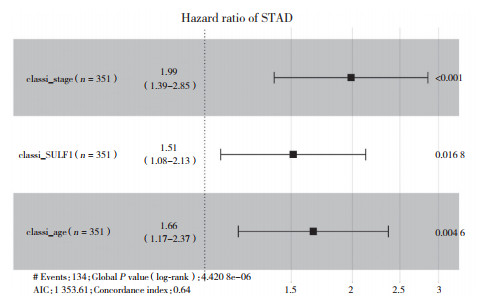
|
| 图 5 多因素Cox回归分析可视化森林图 Fig.5 Visual forest map by multivariate Cox regression analysis |
2.5 基于列线图验证SUFL1对胃癌预后的预测价值
为了进一步验证SUFL1对胃癌预后的预测价值,通过多因素分析确定SUFL1 mRNA表达是OS的独立指标。根据SUFL1 mRNA表达绘制列线图,见图 6。

|
| 图 6 SULF1基因表达列线图 Fig.6 Histogram of SULF1 gene expression |
2.6 ROC曲线分析
利用TCGA-STAD测序数据集绘制ROC曲线,ROC的AUC在0.5~1之间,且AUC在0.7~0.9时有一定准确性。当AUC > 0.5时,AUC值越接近于1,诊断效果越好。结果显示,SUFL1上调对胃癌具有较好的诊断价值(AUC=0.857,P < 0.000 1)。见图 7。
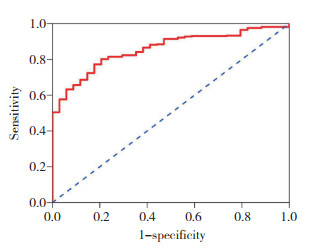
|
| 图 7 ROC曲线验证SULF1上调对胃癌的诊断价值 Fig.7 ROC curve verifying the diagnostic value of SULF1 up-regulation in gastric cancer |
3 讨论
近年来,随着遗传学和分子生物学的快速发展,胃癌的早期发现和更加个性化的治疗得到了推动,其中关键生物标志物的识别起到至关重要的作用。先前的研究[8]表明,SUFL1在胃癌中起到促癌作用,但是SUFL1与胃癌OS的关系尚无研究报道。
本研究初步观察到,与正常胃组织相比,胃癌组织中SUFL1 mRNA水平显著上调。cBioPortal网站分析结果显示,胃癌中存在SUFL1基因突变的情况。Kaplan-Meier生存曲线分析显示,SUFL1高表达的胃癌患者OS相对较差(P < 0.05)。此外,本研究评估了SUFL1上调在胃癌中的临床意义。结果表明,胃癌的OS与患者年龄、肿瘤分期、SUFL1基因表达有关;多因素Cox回归分析结果表明,SUFL1基因表达是胃癌患者OS的一个独立预后标志物,表明SUFL1可能参与胃癌的发生和进展。为了进一步验证SUFL1的预后价值,本研究根据SUFL1 mRNA表达构建列线图,结果显示SUFL1基因高表达与不良预后相关,从而推测SUFL1表达对胃癌的预后有重要意义。ROC曲线显示,SUFL1 mRNA表达水平对胃癌有较好的诊断价值,提示SUFL1可能是一种有助于胃癌分子和病理诊断的生物标志物。
综上所述,SUFL1在胃癌中呈高表达,且与不良预后相关,因此可作为胃癌诊断和治疗的潜在靶点。
| [1] |
GU ML, ZHOU XX, REN MT, et al. Blockage of ETS homologous factor inhibits the proliferation and invasion of gastric cancer cells through the c-Met pathway[J]. World J Gastroenterol, 2020, 26(47): 7497-7512. DOI:10.3748/wjg.v26.i47.7497 |
| [2] |
SUNG H, FERLAY J, SIEGEL RL, et al. Global cancer statistics 2020:GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries[J]. CA Cancer J Clin, 2021, 71(3): 209-249. DOI:10.3322/caac.21660 |
| [3] |
JOSHI SS, BADGWELL BD. Current treatment and recent progress in gastric cancer[J]. CA Cancer J Clin, 2021, 71(3): 264-279. DOI:10.3322/caac.21657 |
| [4] |
SMYTH EC, NILSSON M, GRABSCH HI, et al. Gastric cancer[J]. Lancet, 2020, 396(10251): 635-648. DOI:10.1016/S0140-6736(20)31288-5 |
| [5] |
SLOMSKI A. H pylori treatment may reduce long-term gastric cancer risk[J]. JAMA, 2019, 322(20): 1948. DOI:10.1001/jama.2019.18424 |
| [6] |
CHOI IJ, KOOK MC, KIM YI, et al. Helicobacter pylori therapy for the prevention of metachronous gastric cancer[J]. N Engl J Med, 2018, 378(12): 1085-1095. DOI:10.1056/NEJMoa1708423 |
| [7] |
刘春涛, 张澍田. SUFL1在消化系统恶性肿瘤中的作用研究进展[J]. 临床和实验医学杂志, 2016, 15(5): 510-513. DOI:10.3969/j.issn.1671-4695.2016.05.038 |
| [8] |
LI J, MO ML, CHEN Z, et al. HSulf-1 inhibits cell proliferation and invasion in human gastric cancer[J]. Cancer Sci, 2011, 102(10): 1815-1821. DOI:10.1111/j.1349-7006.2011.02024.x |
| [9] |
HUR K, HAN TS, JUNG EJ, et al. Up-regulated expression of sulfatases (SUFL1 and SULF2) as prognostic and metastasis predictive markers in human gastric cancer[J]. J Pathol, 2012, 228(1): 88-98. DOI:10.1002/path.4055 |
| [10] |
ABIATARI I, KLEEFF J, LI J, et al. Hsulf-1 regulates growth and invasion of pancreatic cancer cells[J]. J Clin Pathol, 2006, 59(10): 1052-1058. DOI:10.1136/jcp.2005.031716 |
| [11] |
HU Y, LI JJ, LUO HF, et al. Differential expression of COL1A1, COL1A2, COL6A3, and SUFL1 as prognostic biomarkers in gastric cancer[J]. Int J Gen Med, 2021, 14: 5835-5843. DOI:10.2147/IJGM.S321265 |
 2022, Vol. 51
2022, Vol. 51