文章信息
- 佟春晓, 陈海英, 孙曼妮, 孟涛
- TONG Chunxiao, CHEN Haiying, SUN Manni, MENG Tao
- 子痫前期患者胎盘组织中环状RNA差异表达的生物信息学分析
- Bioinformatics analysis-based identification of key circular RNAs in the placentas of patients with pre-eclampsia
- 中国医科大学学报, 2021, 50(8): 673-677
- Journal of China Medical University, 2021, 50(8): 673-677
-
文章历史
- 收稿日期:2021-05-11
- 网络出版时间:2021-07-28 9:17
子痫前期是以高血压和尿蛋白作为主要诊断的妊娠疾病,是导致围产期母胎死亡的主要原因之一,发病率约为3%~5%,可导致严重的多系统器官损害,威胁孕产妇生命。目前,该病缺乏真正有效的治疗手段,维持母胎稳定直至在适当时机终止妊娠,是目前该病的管理策略。研究[1]表明,子痫前期的发生和发展可能导致孕妇晚年的心血管功能受损,为了保障母胎安全、改善产妇预后,深度探索子痫前期的发病机制,并根据其发病机制寻找真正有效的分子预测和治疗手段至关重要。
环状RNA(circular RNA,circRNA)是反向剪接产生的非编码RNA,由于具有共价闭合的环结构,保护其免受核酸外切酶介导的降解,具有很高的稳定性。近年来,子痫前期circRNA相关研究集中在胎盘因子的表达变化及circRNA对胎盘滋养细胞功能的影响。考虑单个芯片数据集样本量较少,可信度不高,本研究对子痫前期患者与正常对照孕妇胎盘组织进行比较,对子痫前期患者胎盘circRNA芯片数据集进行整合和重新分析,得到更有意义的差异表达的circRNA,并预测可能与circRNA相互作用的微RNA(microRNA,miRNA),进行筛选、鉴定和功能预测,以深入探讨子痫前期发生和发展中改变的基因表达和生物代谢过程,为子痫前期预测和诊治提供新的思路。
1 材料与方法 1.1 circRNA芯片数据采集基因芯片数据集GSE96984和GSE102897均来源于美国国家生物技术信息中心的高通量基因表达数据库(Gene Expression Omnibus,GEO)。GSE96984和GSE102897的circRNA芯片均基于GPL22120平台(Agilent-078298 human ceRNA array V1.0 4X180K)。GSE102897根据其研究方案可知,研究对象为3例重度子痫前期患者和3例正常对照孕妇的胎盘组织。GSE96984根据其研究方案可知,研究对象为3例子痫前期患者和4例正常对照孕妇的胎盘组织。本研究通过生物信息学方法,联合分析2个数据集的基因芯片数据,芯片数据已通过Quantile算法归一化处理。
1.2 差异基因的筛选R语言limma包筛选组间差异表达基因(differentially expressed genes,DEGs)。对样本质检表达量的差异倍数(fold change,FC)取对数,用于评价基因的表达水平,若P < 0.05且|logFC| > 2,认为有统计学意义。采用Pheatmap软件包绘制热图,提示差异基因在样本中的表达。
1.3 维恩图绘制利用维恩图在线绘制工具(http://bioinformatics.psb.ugent.be/webtools/Venn/)制作维恩图,将GSE102897和GSE96984两个数据集筛选出的差异基因取交集。
1.4 miRNA预测通过两者联合分析共同筛选的基因,按|logFC|值进一步筛选,从大到小排列前10位circRNA的miRNA经过人类癌症特异circRNA数据库(cancer-specific circRNA database,CSCD)网站(http://gb.whu.edu.cn/CSCD/)预测。
2 结果 2.1 子痫前期患者胎盘组织中差异表达的circRNA通过P < 0.05且|logFC| > 2筛选后,按照|logFC|从大到小排列,筛选差异表达的前10个circRNA。与对照组相比,子痫前期患者胎盘组织中GSE96984芯片前10条circRNA均表达上调(表 1),GSE102897芯片前10条circRNA均表达下调(表 2)。
| No. | circRNA | logFC | Adjusted P | Expression |
| 1 | hsa_circ_0026392 | 7.018 692 98 | 0.038 629 33 | Upregulated |
| 2 | hsa_circ_0026430 | 6.638 316 51 | 0.034 635 36 | Upregulated |
| 3 | hsa_circ_0026410 | 6.555 353 21 | 0.037 453 97 | Upregulated |
| 4 | hsa_circ_0026427 | 6.532 289 99 | 0.043 436 97 | Upregulated |
| 5 | hsa_circ_0026418 | 6.208 223 04 | 0.033 768 89 | Upregulated |
| 6 | hsa_circ_0026420 | 6.097 557 38 | 0.046 233 38 | Upregulated |
| 7 | hsa_circ_0019217 | 6.087 003 98 | 0.019 472 37 | Upregulated |
| 8 | hsa_circ_0026416 | 5.719 477 56 | 0.044 437 76 | Upregulated |
| 9 | hsa_circ_0026552 | 5.668 794 46 | 0.002 316 60 | Upregulated |
| 10 | hsa_circ_0033074 | 5.448 206 01 | 0.001 427 81 | Upregulated |
| No. | circRNA | logFC | Adjusted P | Expression |
| 1 | hsa_circ_0055724 | -7.017 271 38 | 0.000 509 36 | Downregulated |
| 2 | hsa_circ_0049730 | -5.561 552 16 | 0.000 072 90 | Downregulated |
| 3 | hsa_circ_0001235 | -5.549 496 05 | 0.000 048 40 | Downregulated |
| 4 | hsa_circ_0063517 | -5.548 411 50 | 0.000 317 75 | Downregulated |
| 5 | hsa_circ_0063523 | -5.445 835 83 | 0.000 048 40 | Downregulated |
| 6 | hsa_circ_0066025 | -5.432 505 56 | 0.000 092 10 | Downregulated |
| 7 | hsa_circ_0062833 | -5.329 559 16 | 0.000 098 90 | Downregulated |
| 8 | hsa_circ_0066559 | -4.967 540 27 | 0.001 539 85 | Downregulated |
| 9 | hsa_circ_0080574 | -4.916 892 33 | 0.000 210 03 | Downregulated |
| 10 | hsa_circ_0080573 | -4.853 482 83 | 0.000 338 79 | Downregulated |
2.2 火山图和聚类图绘制分析差异表达谱
根据芯片筛选出差异表达的circRNA,绘制火山图和热图。红色和绿色区域代表P < 0.05且|logFC| > 2的circRNA,火山图反映总体基因的表达情况(图 1)。根据circRNA的表达水平,将检测的circRNA和标本进行聚类分析,红色代表circRNA表达量相对高,绿色代表circRNA表达量相对低,聚类分析热图表明2组样本中circRNA的表达存在差异(图 2)。
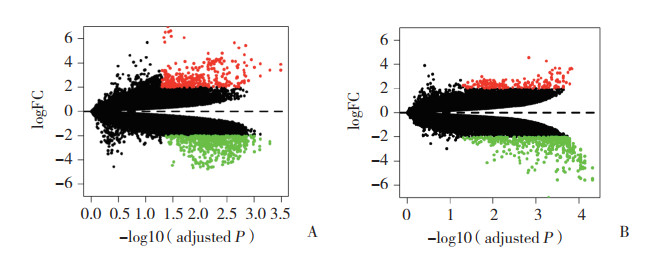
|
| 图 1 GSE96984和GSE102897芯片中差异表达circRNA的火山图 Fig.1 Volcano map of differentially expressed circRNAs in the GSE96984 and GSE102897 circRNA microarrays |
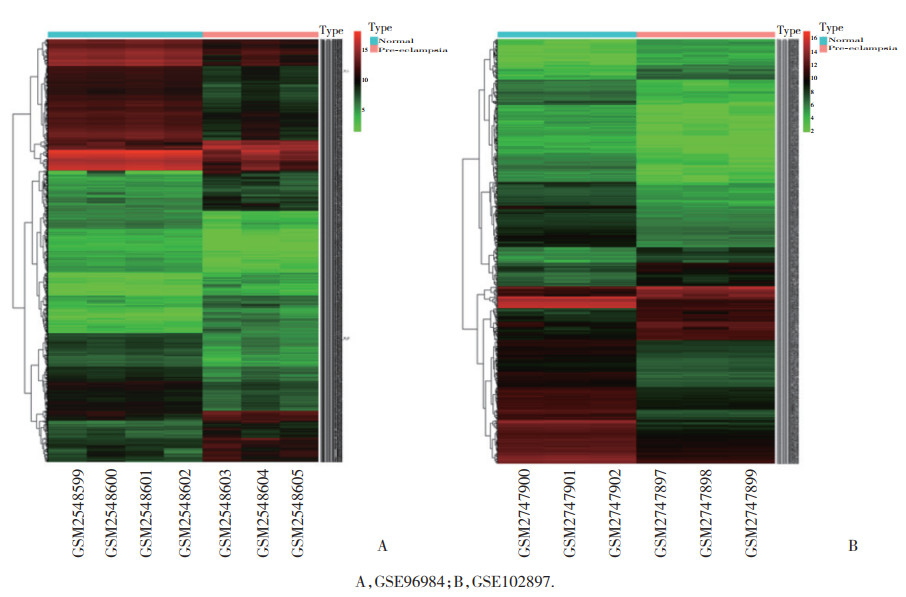
|
| 图 2 GSE96984和GSE102897芯片中差异表达circRNA的热图 Fig.2 Heat map of differentially expressed circRNAs in the GSE96984 and GSE102897 circRNA microarrays |
2.3 维恩图绘制
GSE102897数据集中通过P < 0.05且|logFC| > 2筛选出653个差异表达circRNA,GSE96984数据集中通过P < 0.05且|logFC| > 2筛选出997个差异表达circRNA,两者联合分析共同筛选出160个基因(图 3)。
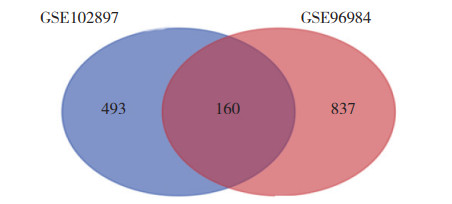
|
| 图 3 GSE96984与GSE102897数据集差异表达circRNA的维恩图 Fig.3 Venn diagram of the differential circRNA expression between the GSE96984 and GSE102897 datasets |
2.4 在子痫前期中具有重要意义的circRNA及其miRNA预测
通过两者联合分析共同筛选的基因,按照|logFC|值从大到小排列,进一步筛选得到的前10位circRNA,见表 3。前10位circRNA的miRNA经过CSCD网站预测,有4个circRNA定位在人类第7号染色体,预测的miRNA最高频率出现的为miR-1207-3p。见表 4。
| No. | circRNA | Expression |
| 1 | hsa_circ_0042013 | Downregulated |
| 2 | hsa_circ_0080573 | Downregulated |
| 3 | hsa_circ_0055724 | Downregulated |
| 4 | hsa_circ_0080570 | Downregulated |
| 5 | hsa_circ_0080571 | Downregulated |
| 6 | hsa_circ_0063517 | Downregulated |
| 7 | hsa_circ_0080574 | Downregulated |
| 8 | hsa_circ_0088586 | Downregulated |
| 9 | hsa_circ_0065183 | Downregulated |
| 10 | hsa_circ_0048192 | Downregulated |
| No. | circRNA | Expression | circRNA type | chromosome | miRNA-binding site |
| 1 | hsa_circ_0042013 | Downregulated | Exon | chr17 | miR-103-5p,miR-1245-5p/3142,miR-1249-5p/6797-5p |
| 2 | hsa_circ_0080573 | Downregulated | Exon | chr7 | miR-1193,miR-1207-3p,miR-1228-5p |
| 3 | hsa_circ_0055724 | Downregulated | Exon | chr2 | miR-10b-3p,miR-1252-3p,miR-1253 |
| 4 | hsa_circ_0080570 | Downregulated | Exon | chr7 | miR-1193,miR-1207-3p,miR-1228-5p |
| 5 | hsa_circ_0080571 | Downregulated | Exon | chr7 | miR-1193,miR-1207-3p,miR-1228-5p |
| 6 | hsa_circ_0063517 | Downregulated | Exon | chr22 | miR-1237-3p,miR-1248,miR-1273f |
| 7 | hsa_circ_0080574 | Downregulated | Exon,intron | chr7 | miR-1200,miR-1207-3p,miR-128-3p/216-3p/3681-3p |
| 8 | hsa_circ_0088586 | Downregulated | Exon | chr9 | miR-1224-5p,miR-1255-5p,miR-1289 |
| 9 | hsa_circ_0065183 | Downregulated | Exon,intron | chr3 | miR-100-3p,miR-1237-3p,miR-1248 |
| 10 | hsa_circ_0048192 | Downregulated | Exon | chr19 | miR-105-3p,miR-1207-5p/4763-3p,miR-122-3p |
| chr,chromosome. | |||||
3 讨论
近年来,人们逐渐在多个疾病领域中对circRNA展开大量研究。研究[2]表明,circRNA在神经发生过程中表达上调,在癌症和其他细胞增殖率高的疾病中下调,这可能由于细胞增殖后circRNA的表达水平被稀释。本研究通过生物信息学分析,找到10个在子痫前期患者胎盘组织中显著差异表达的circRNA,发现其在疾病组织中均表达下调。滋养细胞侵袭力下降是子痫前期的致病学说之一。在本研究中,与对照组相比,在子痫前期患者胎盘组织中有显著差异的circRNA,其表达均下调,与细胞增殖、侵袭水平变化的癌症的整体表达情况不一,可能由于子痫前期的发病多源性。circRNA的表达水平不仅影响滋养细胞的侵袭能力,还可能通过其他机制促进疾病的发生。由于目前关于circRNA表达水平的研究多为确定时间和空间的报告,缺乏实时动态的研究结果,所以目前circRNA芯片呈现出来的结果可能是疾病相关应激变化的结果。筛选出的10个circRNA里,有4个基因位于人类第7号染色体。内皮型一氧化氮合酶(endothelial nitric oxide synthase,eNOS)与子痫前期的关系已有很多研究报道。1997年就有学者提出第7号染色体上编码eNOS基因的7q36区域是家族妊娠期高血压的易感基因位点[3]。有研究[4]认为中国人群的eNOS基因多态性可能对子痫前期有保护作用,但也有研究[5]认为eNOS基因本身对子痫前期易感的证据较弱,对子痫前期易感的是7q36区域。内皮细胞型纤溶酶原激活物抑制因子1也位于染色体7q22.1,学者们对日本、意大利、巴西、奥地利等国家女性中其基因多态性与子痫前期的关系展开了大量研究,发现与子痫前期具有相关性[6]。这些研究说明,第7号染色体区域基因与子痫前期的关系值得深入研究,而本研究发现的位于第7号染色体区域的circRNA更值得进一步探索。
circRNA在子痫前期方面的研究内容包括血液标志物的预测作用和胎盘功能机制。子痫前期的机制研究主要集中在circRNA分子作为竞争性内源RNA(competing endogenous RNA,ceRNA)调控细胞表型。SHEN等[7]发现circTRNC18通过调节miR-762/Grhl2信号通路,从而抑制滋养细胞的迁移和上皮-间质转化,参与子痫前期的发生、发展。GAI等[8]探讨了hsa_circ_0007121在人类子痫前期胎盘组织中表达下调的潜在机制,发现hsa_circ_0007121靶作用于miR-182-5p/PGF轴,影响滋养细胞上皮-间质转化。高水平的circZDHHC20至少可以通过部分作为海绵体结合miR-144,从而上调GRHL2抑制滋养层细胞的增殖、迁移和侵袭,参与子痫前期发病机制的调控[9]。体内和体外细胞实验发现,circ_0063517通过ceRNA机制靶作用于miR-31-5p-ETBR轴,调节子痫前期内皮细胞血管生成作用等[10]。
本研究通过生物信息学分析预测circRNA作为ceRNA结合的miRNA,其中miR-1207-3p频率较高,值得注意。miR-1207-3p在胃癌组织中表达上调[11],编码于染色体8q24区域的非编码PVT1基因座(前列腺癌的易感位点),可抑制前列腺癌细胞增殖、迁移,并诱导细胞凋亡[12]。目前尚无在妊娠相关疾病中miR-1207-3p的研究,而其家族成员miR-1207-5p被认为与孕期低血清铁水平及低出生体重儿相关[13]。miR-1207-3p与子痫前期的关系还有待进一步研究。
综上所述,circRNA及其作为ceRNA发挥功能对子痫前期的发生、发展及预测起到关键作用。第7号染色体区域相关circRNA与子痫前期密切相关,值得进一步探索。
| [1] |
MOL BWJ, ROBERTS CT, THANGARATINAM S, et al. Pre-eclampsia[J]. Lancet, 2016, 387(10022): 999-1011. DOI:10.1016/S0140-6736(15)00070-7 |
| [2] |
KRISTENSEN LS, ANDERSEN MS, STAGSTED LVW, et al. The biogenesis, biology and characterization of circular RNAs[J]. Nat Rev Genet, 2019, 20(11): 675-691. DOI:10.1038/s41576-019-0158-7 |
| [3] |
ARNGRÍMSSON R, HAYWARD C, NADAUD S, et al. Evidence for a familial pregnancy-induced hypertension locus in the eNOS-gene region[J]. Am J Hum Genet, 1997, 61(2): 354-362. DOI:10.1086/514843 |
| [4] |
CHEN LK, HUANG CH, YEH HM, et al. Polymorphisms in the endothelial nitric oxide synthase gene may be protective against preeclampsia in a Chinese population[J]. Reprod Sci, 2007, 14(2): 175-181. DOI:10.1177/1933719107299570 |
| [5] |
HINGORANI AD. Endothelial nitric oxide synthase polymorphisms and hypertension[J]. Curr Hypertens Rep, 2003, 5(1): 19-25. DOI:10.1007/s11906-003-0006-0 |
| [6] |
D'ELIA AV, FABBRO D, DRIUL L, et al. Plasminogen activator inhibitor-1 gene polymorphisms in pre-eclampsia[J]. Semin Thromb Hemost, 2011, 37(2): 97-105. DOI:10.1055/s-0030-1270334 |
| [7] |
SHEN XY, ZHENG LL, HUANG J, et al. CircTRNC18 inhibits trophoblast cell migration and epithelial-mesenchymal transition by regulating miR-762/Grhl2 pathway in pre-eclampsia[J]. RNA Biol, 2019, 16(11): 1565-1573. DOI:10.1080/15476286.2019.1644591 |
| [8] |
GAI S, SUN L, WANG H, et al. Circular RNA hsa_circ_0007121 regulates proliferation, migration, invasion, and epithelial-mesenchymal transition of trophoblast cells by miR-182-5p/PGF axis in preeclampsia[J]. Open Med, 2020, 15(1): 1061-1071. DOI:10.1515/med-2020-0230 |
| [9] |
ZHOU B, ZHANG X, LI T, et al. CircZDHHC20 represses the proliferation, migration and invasion in trophoblast cells by miR-144/GRHL2 axis[J]. Cancer Cell Int, 2020, 20: 19. DOI:10.1186/s12935-020-1097-2 |
| [10] |
LI W, YU N, FAN L, et al. Circ_0063517 acts as ceRNA, targeting the miR-31-5p-ETBR axis to regulate angiogenesis of vascular endothelial cells in preeclampsia[J]. Life Sci, 2020, 244: 117306. DOI:10.1016/j.lfs.2020.117306 |
| [11] |
ANAUATE AC, LEAL MF, WISNIESKI F, et al. Analysis of 8q24.21 miRNA cluster expression and copy number variation in gastric cancer[J]. Future Med Chem, 2019, 11(9): 947-958. DOI:10.4155/fmc-2018-0477 |
| [12] |
DAS DK, NAIDOO M, ILBOUDO A, et al. miR-1207-3p regulates the androgen receptor in prostate cancer via FNDC1/fibronectin[J]. Exp Cell Res, 2016, 348(2): 190-200. DOI:10.1016/j.yexcr.2016.09.021 |
| [13] |
NAIDOO P, NAIDOO RN, RAMKARAN P, et al. The Tyr113His T/C rs1051740 and 'very slow' phenotype of the EPHX1 gene alters miR-26b-5p and miR-1207-5p expression in pregnancy[J]. Gene, 2017, 633: 71-81. DOI:10.1016/j.gene.2017.07.080 |
 2021, Vol. 50
2021, Vol. 50




