文章信息
- 王奕丹, 王一涵, 黄钰雯, 阴迪, 于运, 刘丽梅
- WANG Yidan, WANG Yihan, HUANG Yuwen, YIN Di, YU Yun, LIU Limei
- DNA甲基转移酶3b对乳腺癌细胞微小RNA表达的影响
- Effect of DNA methyltransferase 3b on microRNA expression in breast cancer cells
- 中国医科大学学报, 2021, 50(2): 116-123
- Journal of China Medical University, 2021, 50(2): 116-123
-
文章历史
- 收稿日期:2020-04-30
- 网络出版时间:2021-01-13 15:56
乳腺癌是女性最常见的恶性肿瘤之一,死亡率高,严重危害女性健康。乳腺癌的发生发展常受到多种基因的调控(原癌基因的激活和抑癌基因的失活),同时也受到一些表观遗传学因素[DNA甲基化和微小RNA(microRNA,miRNA)等]影响[1-3]。其中DNA甲基化的发生不仅可以影响肿瘤相关基因的表达变化,而且对位于CpG岛附近或位于CpG岛中的miRNA表达产生影响[4-6]。本研究探讨抑制DNA甲基化和siRNA干扰DNA甲基转移酶3b(DNA methyltransferase 3b,Dnmt3b)后乳腺癌细胞MCF-7中miRNA和肿瘤相关基因的表达情况,旨在为乳腺癌的诊断和治疗提供依据。
1 材料与方法 1.1 材料人乳腺癌细胞株MCF-7购自中国科学院上海细胞生物学研究所,DMEM培养液、胎牛血清和胰蛋白酶购于美国HyClone公司,转染试剂购于美国QIAGEN公司,RNA提取、反转录试剂盒和荧光定量PCR试剂盒购于日本TaKaRa BIOINC公司,GAPDH抗体、DNA甲基化抗体购于英国Abcam公司,二抗购于中国博士德公司,抗荧光淬灭剂、Hoechst33258购于德国Sigma公司。siRNA由上海吉玛公司合成,引物由上海生工生物有限公司合成。
1.2 方法 1.2.1 免疫荧光染色检测MCF-7细胞甲基化水平将盖玻片过酸处理高压灭菌后,放入24孔板中,接种MCF-7细胞,24 h后实验组分别加入5-氮杂-2’-脱氧胞苷(5-Aza-2’-deoxycytidine,5-Aza-CdR),终浓度分别为5-Aza-CdR 10、5、1 μmol/mL;对照组加入等体积PBS,每24 h加入1次5-Aza-CdR,同时更换培养液,连续处理4 d。4 d后取出盖玻片,PBS清洗3次,用5%多聚甲醛固定30 min,PBS清洗3次。用TritonX-100(0.2%)透化40 min,盐酸(4N)酸化30 min,弃去液体后,用Tris盐酸(pH8.0)中和20 min,PBS清洗3次。BSA(1%)4 ℃封闭过夜后,加入适量5-甲基胞嘧啶直接荧光抗体(1%BSA 1:50稀释),避光放置4 ℃过夜,PBS清洗3次,每次30 min。在适量Hoechst33258染色液(5 mg/L)中避光染色10 min,PBS避光清洗3次,每次5 min。将含抗荧光淬灭剂的封片剂DABCO滴于载玻片上封片,30 min内荧光倒置显微镜下观察,拍照并记录。
1.2.2 siRNA转染及细胞生长曲线绘制MCF-7细胞用含有10%胎牛血清和90%DMEM培养液,置于37 ℃、5%CO2中培养。实验分为空白对照组(Mock组)、阴性对照组(NC组)和干扰组(siRNA组)。针对Dnmt3b的siRNA-1511及其阴性对照序列如下:5’-CGCCUCAAGACAAAUUGCUTT-3 ’;5’-AGCAAUUUGUCUUGAGGCGTT-3’。转染前1 d将细胞按照1×105/mL接种于24孔板中,每孔加入1 mL培养液。过夜贴壁后,每孔更换为400 μL无血清培养液待用。用100 μL无血清培养液稀释80 ng siRNA,与3 μL转染试剂混匀,室温静止5~10 min,轻轻将转染复合物滴加至24孔板中,轻轻摇匀,转染6 h后荧光显微镜下观察转染情况,计算转染效率。48 h后提取RNA检测目的基因表达情况,96 h后检测miRNA与相关基因表达情况。转染前后连续5 d显微镜下计数细胞,绘制细胞生长曲线。
1.2.3 qRT-PCR检测siRNA干扰后细胞Dnmt3b mRNA的表达转染48 h后提取NC组、Mock组、siRNA组细胞RNA逆转录,获得的cDNA进行qRT-PCR扩增Dnmt3b mRNA,β-actin作为内参,采用2-ΔΔCt法进行数据分析,引物序列见表 1。
| Gene | Forward primer(5’-3’) | Reverse primer(5’-3’) |
| Dnmt3b | CCCATTCGAGTCCTGTCATT | GGTTCCAACAGCAATGGACT |
| WWOX | GCAGTGCATCCTGGAAATATGAT | CAGTTGTTGAAGTACATCCCTCC |
| TCF21 | TTCATCCACCTGTCTATTTGCAC | CCTCTGGTAGAAAGGAGAGAACA |
| BRCA1 | TATGGGCCCTTCACCAACAT | AAGCCATTGTCCTCTGTCCA |
| BRCA2 | CTCAGCGTTTGTGTATCGGG | CTCCCACCTCAGCTTCTCAA |
| Survivin | ACCTGAAAGCTTCCTCGACA | TTCCCAGACTCCACTCCAAC |
| MMP7 | AATGTTAAACTCCCGCGTCATAG | AAGCCTTTGACACTAATCGATCC |
| TIMP1 | GACCACCTTATACCAGCGTTATG | TTTCCAGCAATGAGAAACTCCTC |
| PKM2 | GAGCAGGATGTTGATATGGTGTT | CAGGATTTCATCAAACCTCCGAA |
| β-actin | CACCTTCACCGTTCCAGTTT | GATGAGATTGGCATGGCTTT |
1.2.4 Western blotting检测siRNA干扰后细胞Dnmt3b蛋白的表达
收集Mock组、NC组、siRNA组细胞,加入RAPI细胞裂解液,4℃裂解10 min,其间涡旋振荡2次,30 s/次,4℃离心15 min,取上清,BCA法测定蛋白浓度。调蛋白浓度一致,进行SDS聚丙烯酰胺凝胶电泳,半干法转膜。封闭后加入Dnmt3b单克隆抗体(1:100稀释),内参加入兔抗-GAPDH抗体(1:200稀释),慢摇1 h,用PBS-T洗膜3次,每次20 min。加入二抗(Dnmt3b 1:3 000稀释;GAPDH 1:2 000稀释),室温孵育40 min,PBS-T洗膜1次,PBS洗膜2次,每次10 min。在显影仪中滴加显影液显影,用Image J软件对蛋白表达条带进行光密度值的定量分析。
1.2.5 高通量测序检测miRNA的表达情况将细胞按1×105/mL接种于6孔板,每孔加入含10%胎牛血清的DMEM,3孔/组,分为实验组和对照组,实验组每孔加入终浓度5 μmol/mL的5-Aza-CdR,换液1次/24 h,并重新加入5-Aza-CdR。72 h后每孔加入1 mL Trizol将细胞完全裂解,miRNA高通量测序委托上海吉玛公司进行。
1.2.6 qRT-PCR检测mRNA和miRNA的表达采用Trizol法提取RNA,检测浓度与质量,进行逆转录,qRT-PCR扩增,退火温度及引物序列见表 1、表 2。以β-actin为内参,采用2-ΔΔCt法计算各基因相对表达量。
| miRNA | Sequences of the primers |
| miR-29c | CTTGTGGTCCTCTTTAGCCAGT |
| miR-125b | GCTCCCTGAGACCCTAACTTGTGA |
| miR-21 | GCGCTAGCTTATCAGACTGATGTTGA |
| miR-26b | GCGCTTCAAGTAATTCAGGATAGGT |
| miR-29b | GCGCTAGCACCATTTGAAATCAGTGTT |
| miR-155 | GGCTTAATGCTAATCGTGATAGGGGT |
| miR-506 | GCTAAGGCACCCTTCTGAGTAGA |
| miR-145 | GTCCAGTTTTCCCAGGAATCCCT |
| miR-200a | GCGCTAACACTGTCTGGTAACGATGT |
| miR-203a | GCGGTGAATGTTTAGGACCACTAG |
| miR-200b | CATCTTACTGGGCAGCATTGGA |
| miR-411 | GCTATGTAACACGGTCCACTAACC |
| miR-382 | CGCGAATCATTCACGGACAACACTT |
| miR-409 | GAATGTTGCTCGGTGAACCCCT |
| miR-493 | TGAAGGTCTACTGTGTGCCAGG |
| miR-127 | TCGGATCCGTCTGAGCTTGGCT |
| miR-520a | GCAAAGTGCTTCCCTTTGGACTGT |
| miR-654 | GCTATGTCTGCTGACCATCACCTT |
1.3 统计学分析
采用SPSS 16.0统计学软件进行统计处理。计量资料用x±s表示,组间比较采用t检验,P < 0.05为差异有统计学意义。
2 结果 2.1 5-Aza-CdR对细胞DNA甲基化水平的影响荧光显微镜观察MCF-7甲基化程度,结果可见,随着5-Aza-CdR浓度的升高,5-甲基胞嘧啶甲基化抗体染色逐渐减弱,表示其整体甲基化水平逐渐降低。与对照组(59.37±8.45)比较,1、5、10 μmol/mL组(分别为34.30±9.47、5.11±2.78、4.99±2.64)甲基化水平均有统计学差异(均P < 0.05),但5 μmol/mL组与10 μmol/mL组比较差异无统计学意义(P > 0.05),因此后续实验选用5 μmol/mL浓度5-Aza-CdR,见图 1。
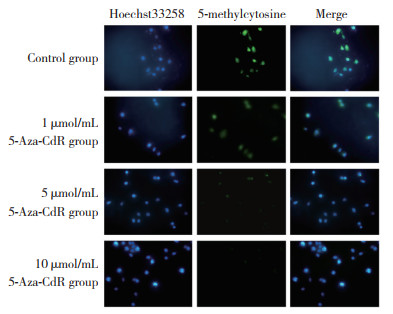
|
| 5-methylcytosine antibody was labeled green, and hoechst 33258 labeled cell nucleus was blue. 图 1 5-Aza-CdR处理后细胞DNA甲基化水平的改变×400 Fig.1 Changes in DNA methylation levels in cells after 5-Aza-2'-deoxycytidine (5-Aza-CdR) treatment ×400 |
2.2 siRNA转染后细胞生长及Dnmt3b的表达
Western blotting结果显示,siRNA干扰组中Dnmt3b的表达低于Mock组和NC组(P < 0.05),见图 2A、2B。qRT-PCR结果显示,与对照组比较,siRNA干扰组Dnmt3b的表达显著下降(P < 0.05),抑制达75%以上,见图 2C;细胞计数可见Mock组和NC组细胞生长速度相近,而与Mock组和NC组比较,siRNA干扰组细胞生长受到抑制(P < 0.05),见图 2D。说明siRNA转染后显著下调Dnmt3b的表达。
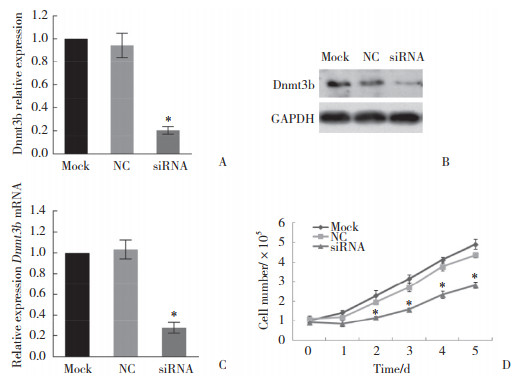
|
| A, B, the expression of Dnmt3b;C, the expression of Dnmt3b mRNA; D, cell growth curve. mock, mock group; NC, negative control group; siRNA, siRNA interference group.*P < 0.05 vs mock or NC groups. 图 2 siRNA干扰后细胞生长及Dnmt3b的表达 Fig.2 Cell growth and DNMT3b expression after siRNA interference |
2.3 5-Aza-CdR处理、Dnmt3b siRNA干扰后癌基因和抑癌基因mRNA的表达
与对照组比较,经5 μmol/mL 5-Aza-CdR处理后WWOX、MMP7表达显著下调,TCF21、TIMP1表达显著上调,差异均有统计学意义(P < 0.05),见表 3。与NC组比较,siRNA干扰组抑癌基因BRCA1、BRCA2及癌基因MMP7、survivin的表达均显著下调(均P < 0.05),见表 3。
| Group | WWOX mRNA | TCF21 mRNA | BRCA1 mRNA | BRCA2 mRNA | MMP7 mRNA | TIMP1 mRNA | PKM2 mRNA | survivin mRNA |
| Control | 1.00±0 | 1.00±0 | 1.00±0 | 1.00±0 | 1.00±0 | 1.00±0 | 1.00±0 | 1.00±0 |
| 5-Aza-CdR | 0.54±0.031) | 2.44±0.101) | 1.14±0.04 | 0.99±0.09 | 0.59±0.031) | 1.45±0.131) | 0.87±0.02 | 1.01±0.04 |
| Mock | 1.00±0 | 1.00±0 | 1.00±0 | 1.00±0 | 1.00±0 | 1.00±0 | 1.00±0 | 1.00±0 |
| NC | 0.69±0.15 | 0.21±0.05 | 0.96±0.04 | 1.07±0.03 | 1.30±0.45 | 0.55±0.01 | 0.30±0.04 | 1.18±0.04 |
| siRNA | 0.59±0.09 | 0.34±0.04 | 0.62±0.012) | 0.48±0.012) | 1.08±0.302) | 0.50±0.07 | 0.25±0.01 | 0.76±0.022) |
| 1)P < 0.05 vs control group;2) P < 0.05 vs NC group. | ||||||||
2.4 高通量测序检测5-Aza-CdR处理后miRNA的表达情况
高通量测序结果显示,1 319个miRNA表达存在差异,其中,67个miRNA表达有统计学差异(均P < 0.05),见图 3。20个miRNA实验组与对照组均有表达,见表 4;47个miRNA对照组中无表达,见表 5。
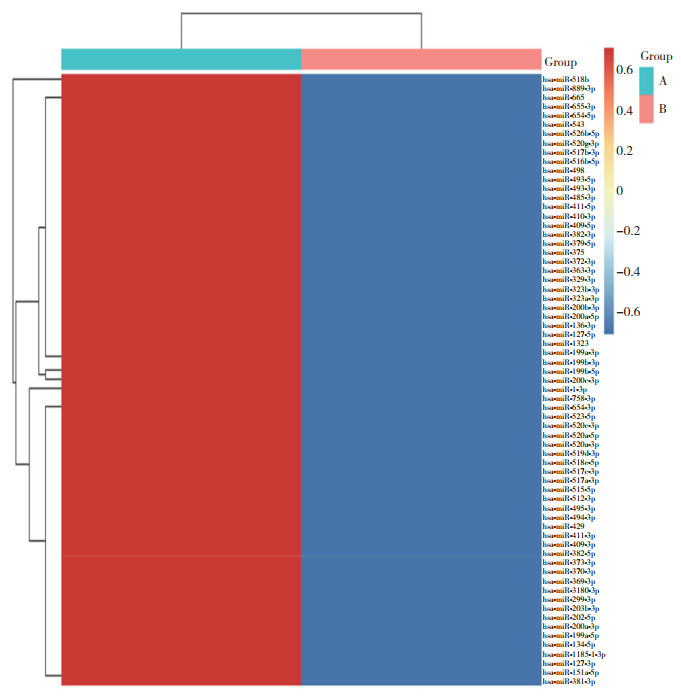
|
| A, 5-Aza-CdR treatment group; B, control group. 图 3 5-Aza-CdR处理后MCF-7 miRNA高通量测序差异表达 Fig.3 Identification of differentially expressed miRNAs after 5-Aza-2'-deoxycytidine (5-Aza-cdR) treatment |
| miRNA | Difference(log2) | P |
| hsa-miR-127-3p | 10.331 30 | 2.48E-16 |
| hsa-miR-1-3p | 1.857 62 | 0.006 858 |
| hsa-miR-151a-5p | 1.369 14 | 0.027 241 |
| hsa-miR-199a-3p | 3.738 95 | 0.002 406 |
| hsa-miR-199b-3p | 3.721 02 | 0.002 591 |
| hsa-miR-199b-5p | 2.044 34 | 0.047 962 |
| hsa-miR-200a-3p | 3.803 98 | 3.88E-06 |
| hsa-miR-200a-5p | 4.342 51 | 0.001 181 |
| hsa-miR-200b-3p | 4.239 78 | 8.23E-10 |
| hsa-miR-200c-3p | 1.786 00 | 0.030 173 |
| hsa-miR-203b-3p | 3.381 54 | 0.005 214 |
| hsa-miR-323a-3p | 6.231 99 | 0.003 510 |
| hsa-miR-375 | 6.120 20 | 3.40E-05 |
| hsa-miR-379-5p | 7.553 91 | 5.04E-07 |
| hsa-miR-381-3p | 7.721 02 | 2.22E-05 |
| hsa-miR-429 | 4.486 56 | 0.000 489 |
| hsa-miR-494-3p | 6.721 02 | 0.000 952 |
| hsa-miR-518b | 5.257 08 | 0.024 202 |
| hsa-miR-654-3p | 6.465 19 | 0.001 949 |
| hsa-miR-889-3p | 6.738 95 | 0.000 902 |
| miRNA | P |
| hsa-miR-1185-1-3p | 0.029 278 |
| hsa-miR-127-5p | 0.007 737 |
| hsa-miR-1323 | 0.000 131 |
| hsa-miR-134-5p | 0.002 422 |
| hsa-miR-136-3p | 0.011 924 |
| hsa-miR-199a-5p | 1.77E-05 |
| hsa-miR-202-5p | 0.047 847 |
| hsa-miR-299-3p | 0.047 847 |
| hsa-miR-3180-3p | 0.047 847 |
| hsa-miR-323b-3p | 0.032 912 |
| hsa-miR-329-3p | 0.042 233 |
| hsa-miR-363-3p | 0.014 315 |
| hsa-miR-369-3p | 0.006 553 |
| hsa-miR-370-3p | 3.74E-06 |
| hsa-miR-372-3p | 0.007 117 |
| hsa-miR-373-3p | 0.019 113 |
| hsa-miR-517b-3p | 0.002 798 |
| hsa-miR-517c-3p | 0.015 728 |
| hsa-miR-518e-5p | 0.047 847 |
| hsa-miR-519d-3p | 0.015 728 |
| hsa-miR-520a-3p | 0.000 163 |
| hsa-miR-520a-5p | 0.047 847 |
| hsa-miR-520c-3p | 0.029 278 |
| hsa-miR-520g-3p | 0.007 117 |
| hsa-miR-382-3p | 0.004 065 |
| hsa-miR-382-5p | 0.005 139 |
| hsa-miR-409-3p | 8.02E-06 |
| hsa-miR-409-5p | 0.010 909 |
| hsa-miR-410-3p | 0.042 233 |
| hsa-miR-411-3p | 0.029 278 |
| hsa-miR-411-5p | 0.001 709 |
| hsa-miR-485-3p | 0.037 202 |
| hsa-miR-493-3p | 0.002 798 |
| hsa-miR-493-5p | 5.16E-05 |
| hsa-miR-495-3p | 0.023 486 |
| hsa-miR-498 | 0.010 909 |
| hsa-miR-512-3p | 3.91E-07 |
| hsa-miR-515-5p | 0.013 054 |
| hsa-miR-516b-5p | 0.002 256 |
| hsa-miR-517a-3p | 0.001 829 |
| hsa-miR-523-5p | 0.047 847 |
| hsa-miR-526b-5p | 0.001 306 |
| hsa-miR-543 | 0.000 300 |
| hsa-miR-654-5p | 0.009 167 |
| hsa-miR-655-3p | 0.032 912 |
| hsa-miR-665 | 0.032 912 |
| hsa-miR-758-3p | 0.013 054 |
2.5 qRT-PCR检测5-Aza-CdR处理、Dnmt3b siRNA干扰后miRNA的表达情况
与对照组比较,经5 μmol/mL 5-Aza-Cdr处理后miR-200a、miR-411、miR-382、miR-409、miR-493、miR-127、miR-520a、miR-654表达量显著上调(P < 0.05),并且此8个miRNA的差异性表达与高通量测序结果一致。与NC组比较,siRNA干扰组中miR-29b、miR-506、miR-200a、miR-203b、miR-409、miR-520a、miR-654的表达显著上调,而miR-200b、miR-127的表达显著下调(均P < 0.05),见表 6。
| miRNA | Control group | 5-Aza-Cdr group | Mock group | NC group | siRNA group |
| miR-29c | 1.00±0 | 0.52±0.06 | 1.00±0 | 1.98±0.60 | 1.54±0.78 |
| miR-125b | 1.00±0 | 0.22±0.02 | 1.00±0 | 0.95±0.21 | 0.77±0.10 |
| miR-21 | 1.00±0 | 0.29±0.07 | 1.00±0 | 0.87±0.07 | 0.87±0.09 |
| miR-26b | 1.00±0 | 0.22±0.03 | 1.00±0 | 1.00±0.16 | 1.02±0.09 |
| miR-29b | 1.00±0 | 0.45±0.15 | 1.00±0 | 0.67±0.09 | 1.04±0.102) |
| miR-155 | 1.00±0 | 0.42±0.06 | 1.00±0 | 1.44±0.19 | 1.67±0.30 |
| miR-506 | 1.00±0 | 0.47±0.05 | 1.00±0 | 0.53±0.06 | 0.91±0.152) |
| miR-145 | 1.00±0 | 0.55±0.12 | 1.00±0 | 1.49±0.31 | 1.37±0.34 |
| miR-200a | 1.00±0 | 7.03±0.331) | 1.00±0 | 1.24±0.25 | 2.32±0.552) |
| miR-203b | 1.00±0 | 0.26±0.07 | 1.00±0 | 1.27±0.29 | 1.64±0.502) |
| miR-152 | 1.00±0 | 0.23±0.05 | 1.00±0 | 0.70±0.33 | 0.77±0.20 |
| miR-203a | 1.00±0 | 0.33±0.02 | 1.00±0 | 1.18±0.13 | 1.14±0.10 |
| miR-200b | 1.00±0 | 0.27±0.05 | 1.00±0 | 1.18±0.08 | 0.72±0.152) |
| miR-411 | 1.00±0 | 22.37±1.461) | 1.00±0 | 0.06±0.01 | 0.03±0.12 |
| miR-382 | 1.00±0 | 13.90±0.491) | 1.00±0 | 1.36±0.11 | 1.69±0.17 |
| miR-409 | 1.00±0 | 7.71±0.541) | 1.00±0 | 1.24±0.05 | 3.45±0.212) |
| miR-493 | 1.00±0 | 7.23±0.781) | 1.00±0 | 2.01±0.11 | 2.27±0.04 |
| miR-127 | 1.00±0 | 3.49±0.251) | 1.00±0 | 1.45±0.07 | 0.91±0.232) |
| miR-520a | 1.00±0 | 8.86±0.761) | 1.00±0 | 0.02±0.00 | 0.95±0.062) |
| miR-654 | 1.00±0 | 4.89±0.381) | 1.00±0 | 0.10±0.01 | 0.59±0.082) |
| 1)P < 0.05 vs control group;2)P < 0.05 vs NC group. | |||||
3 讨论
miRNA来源于细胞核内基因组的miRNA基因,部分miRNA位于CpG岛附近或位于CpG岛中,这些miRNA表达极易受到DNA异常甲基化的调控[7-9]。miRNA的异常表达与肿瘤的发生发展密切相关,尽管完整的miRNA表达调控机制目前尚不清楚,但已有研究提示肿瘤细胞中DNA甲基化异常会引起miRNA表达改变。HASHIMOTO等[10]研究显示胃癌组织和细胞中miR-181c基因CpG岛高甲基化,而miR-181c低表达,经5-Aza-CdR处理后miR-181c表达恢复,同时抑制了胃癌细胞增殖。在DNA甲基化过程中,DNA甲基转移酶(DNA methyltransferase,Dnmt)参与催化,其包含Dnmt1、Dnmt3a和Dnmt3b等,其中Dnmt3b参与CpG从头甲基化[11]。有研究[7]表明乳腺癌组织中Dnmt3b表达增加,并且与肿瘤分期及预后相关。本研究通过5-Aza-CdR抑制乳腺癌细胞整体甲基化水平,结果显示,miR-200a、miR-411、miR-382、miR-409、miR-493、miR-127、miR-520、miR-654的表达显著上调,而仅干扰Dnmt3b表达时,miR-29b、miR-506、miR-200a、miR-203b、miR-409、miR-502a、miR-654表达显著上调,miR-127、miR-200b表达显著下调,此结果与高通量测序结果基本一致。有研究[13-14]表明miR-29家族可调控Dnmt3a和Dnmt3b的表达。由此可见,Dnmt3b会受到miRNA的调控,也可以改变部分miRNA基因甲基化状态,从而影响它们的转录表达。
乳腺癌的发生发展与癌基因的表达增高和抑癌基因的沉默密切相关,Dnmt3b作为甲基化的催化剂可以影响部分基因表达的功能。已有研究表明,在癌组织中抑癌基因WWOX、TCF21、BRCA1、BRCA2[15-20]表达下降,而癌基因MMP7[21]、TIMP1[22]、PKM2[23]、survivin[24]表达增高。本研究中5-Aza-CdR可以使乳腺癌细胞中抑癌基因WWOX和癌基因MMP7表达降低,而上调抑癌基因TCF21和癌基因TIMP1,其中TCF21和MMP7的表达符合预期结果,而WWOX和TIMP1的结果分析原因可能是由于转录后调控影响这些基因的表达。对于siRNA干扰抑制Dnmt3b的表达中可见癌基因TIMP1、PKM2、survivin的表达显著降低,而抑癌基因WWOX、TCF21、BRCA1、BRCA2的表达也降低,分析原因可能是由于甲基化程度与组织特异性的关系存在多样化,虽然这些基因在前列腺癌、肺癌、大肠癌甚至乳腺癌中甲基化程度较高,但实验中所培养的细胞种类不同、肿瘤的分期进展不同、肿瘤的组织种类不同,其甲基化水平也不相同。另外本研究仅对Dnmt3b进行干扰抑制Dnmt3b的表达,推测Dnmt3b对WWOX、TCF21、BRCA1、BRCA2的表达影响较小,并不占主要调控位置,其他甲基转移酶是这些基因发生甲基化的主要因素,目前研究者对乳腺癌易感基因的甲基化研究还不够成熟,往往出现矛盾的结果,具体原因还有待进一步探讨。
综上所述,Dnmt3b可影响乳腺癌细胞相关基因WWOX、TCF21、BRCA1、BRCA2、MMP7、TIMP1、PKM2、survivin及miR-29b、miR-506、miR-200a、miR-200b、miR-203b、miR-409、miR-127、miR-520a的表达。DNA甲基化是一种可逆的表观遗传学修饰方式,逆转与肿瘤发生发展相关的miRNA基因甲基化水平将改变肿瘤细胞的生物学特性,为肿瘤的精准治疗提供新的方向[25]。本实验从Dnmt3b对miRNA及mRNA的影响作用出发,应用siRNA干扰的方法改变Dnmt3b的表达,检测乳腺癌相关miRNA及mRNA的表达情况,明确了Dnmt3b对miRNA及靶基因mRNA的相互调控机制,为乳腺癌的临床精确诊治提供了依据。
| [1] |
EDIRIWEERA MK, CHO SK. Targeting miRNAs by histone deacetylase inhibitors (HDACi):rationalizing epigenetics-based therapies for breast cancer[J]. Pharmacol Ther, 2020, 206: 107437. DOI:10.1016/j.pharmthera.2019.107437 |
| [2] |
CHEN X, ZHANG J, DAI X. DNA methylation profiles capturing breast cancer heterogeneity[J]. BMC Genomics, 2019, 20(1): 823. DOI:10.1186/s12864-019-6142-y |
| [3] |
LIU F, JIA JH, SUN LP, et al. lncRNA DSCAM-AS1 downregulates miR-216b to promote the migration and invasion of colorectal adenocarcinoma cells[J]. Oncotargets Ther, 2019, 12: 6789-6795. DOI:10.2147/ott.s213301 |
| [4] |
HE XX, KUANG SZ, LIAO JZ, et al. The regulation of microRNA expression by DNA methylation in hepatocellular carcinoma[J]. Mol BioSyst, 2015, 11(2): 532-539. DOI:10.1039/c4mb00563e |
| [5] |
黄文剑, 吴琍, 侯琳, 等. 乳癌组织miRNA-429基因表达及甲基化检测[J]. 青岛大学学报(医学版), 2018, 54(1): 26-29. DOI:10.11712/jms201801007 |
| [6] |
江海波. miRNA-204通过DNA甲基化调控星形细胞瘤的侵袭与迁移[D].芜湖: 皖南医学院, 2018.
|
| [7] |
OLTRA SS, PEÑA-CHILET M, VIDAL-TOMAS V, et al. Methylation deregulation of miRNA promoters identifies miR124-2 as a survival biomarker in breast cancer in very young women[J]. Sci Rep, 2018, 8(1): 14373. DOI:10.1038/s41598-018-32393-3 |
| [8] |
SUZUKI H, MARUYAMA R, YAMAMOTO E, et al. DNA methylation and microRNA dysregulation in cancer[J]. Mol Oncol, 2012, 6(6): 567-578. DOI:10.1016/j.molonc.2012.07.007 |
| [9] |
RODRIGUES MFSD, ESTEVES CM, XAVIER FCA, et al. Methylation status of homeobox genes in common human cancers[J]. Genomics, 2016, 108(5/6): 185-193. DOI:10.1016/j.ygeno.2016.11.001 |
| [10] |
HASHIMOTO Y, AKIYAMA Y, OTSUBO T, et al. Involvement of epigenetically silenced microRNA-181c in gastric carcinogenesis[J]. Carcinogenesis, 2010, 31(5): 777-784. DOI:10.1093/carcin/bgq013 |
| [11] |
JURKOWSKA RZ, JURKOWSKI TP, JELTSCH A. Structure and function of mammalian DNA methyltransferases[J]. Chembiochem, 2011, 12(2): 206-222. DOI:10.1002/cbic.201000195 |
| [12] |
GIRAULT I, TOZLU S, LIDEREAU R, et al. Expression analysis of DNA methyltransferases 1, 3A, and 3B in sporadic breast carcinomas[J]. Clin Cancer Res, 2003, 9(12): 4415-4422. |
| [13] |
FABBRI M, GARZON R, CIMMINO A, et al. MicroRNA-29 family reverts aberrant methylation in lung cancer by targeting DNA methyltransferases 3A and 3B[J]. PNAS, 2007, 104(40): 15805-15810. DOI:10.1073/pnas.0707628104 |
| [14] |
GARZON R, LIU SJ, FABBRI M, et al. MicroRNA-29b induces global DNA hypomethylation and tumor suppressor gene reexpression in acute myeloid leukemia by targeting directly DNMT3A and 3B and indirectly DNMT1[J]. Blood, 2009, 113(25): 6411-6418. DOI:10.1182/blood-2008-07-170589 |
| [15] |
张庆莉.年轻乳腺癌的临床病理特点及BRCA1、WWOX表达的研究[D].山东大学, 2009.
|
| [16] |
李金洁, 刁艳君, 李蕊, 等. 高通量测序技术检测乳腺癌患者BRCA1、BRCA2基因的意义[J]. 检验医学, 2019, 34(11): 1026-1031. DOI:10.3969/j.issn.1673-8640.2019.11.015 |
| [17] |
杨文娟, 杨爱平, 张群威, 等. BRCA1/BRCA2基因突变的乳腺癌患者特征分析[J]. 中国妇幼保健, 2019, 34(9): 2007-2009. DOI:10.7620/zgfybj.j.issn.1001-4411.2019.09.22 |
| [18] |
黄赛. WWOX与JNK在乳腺癌组织中表达与相关性研究[D].济南: 山东大学, 2018.
|
| [19] |
高学仁. TCF21基因多态性与中国汉族女性乳腺癌发病风险的关联研究及功能分析[D].南京: 东南大学, 2017.
|
| [20] |
RUAN XY, LIU HF, BOARDMAN L, et al. Genome-wide analysis of loss of heterozygosity in breast infiltrating ductal carcinoma distant normal tissue highlights arm specific enrichment and expansion across tumor stages[J]. PLoS One, 2014, 9(4): e95783. DOI:10.1371/journal.pone.0095783 |
| [21] |
SHAO Q, LUO X, YANG D, et al. Phospholipase Cδ1 suppresses cell migration and invasion of breast cancer cells by modulating KIF3A-mediated ERK1/2/β-catenin/MMP7 signalling[J]. Oncotarget, 2017, 8(17): 29056-29066. DOI:10.18632/oncotarget.16072 |
| [22] |
李鹏. CCNL1和TIMP1基因对人乳腺癌细胞增殖及相关基因表达的影响[D].长春: 吉林大学, 2012.
|
| [23] |
ZHAO Z, SONG ZJ, LIAO ZJ, et al. PKM2 promotes stemness of breast cancer cell by through Wnt/β-catenin pathway[J]. Tumor Biol, 2016, 37(3): 4223-4234. DOI:10.1007/s13277-015-4121-8 |
| [24] |
WANG W, ZHANG B, MANI AM, et al. Survivin inhibitors mitigate chemotherapeutic resistance in breast cancer cells by suppressing genotoxic nuclear factor-κB activation[J]. J Pharmacol Exp Ther, 2018, 366(1): 184-193. DOI:10.1124/jpet.118.249151 |
| [25] |
林帅, 吴诚义. 乳腺癌与抑癌基因甲基化及MicroRNA的研究进展[J]. 临床与实验病理学杂志, 2010, 26(1): 89-93. DOI:10.3969/j.issn.1001-7399.2010.01.022 |
 2021, Vol. 50
2021, Vol. 50




