文章信息
- 黄俊杰, 梁彬
- HUANG Junjie, LIANG Bin
- 基于TCGA和GEO数据库分析FOXD1在口腔鳞状细胞癌中的表达和临床意义
- FOXD1 expression in oral squamous cell carcinoma and its clinical significance via TCGA and GEO databases
- 中国医科大学学报, 2021, 50(11): 990-996
- Journal of China Medical University, 2021, 50(11): 990-996
-
文章历史
- 收稿日期:2021-01-15
- 网络出版时间:2021-11-04 8:59
2018年全球癌症统计学研究[1]显示,世界范围内口腔癌发病率占所有癌症的2%,死亡率占1.9%。其中口腔鳞状细胞癌(oral squamous cell carcinoma,OSCC)最常见,约占口腔恶性肿瘤的90%以上。尽管放疗、化疗以及靶向治疗技术等医疗手段在OSCC的治疗上取得很大进展,但是OSCC的5年生存率仍然仅为40%~50%,严重影响了患者的生活质量[2]。目前,OSCC的发展、侵袭和转移机制尚不完全明确,阐明OSCC的发病机制,寻找高效的治疗靶点,对OSCC的预防及治疗具有重要的指导意义。FOXD1属于叉头框(forkhead box,FOX)蛋白转录因子家族,研究[3]证明FOXD1参与多种肿瘤的发生发展过程。目前,FOXD1在OSCC发生发展中扮演的角色知之甚少,本研究基于癌症基因组图谱(the cancer genome atlas,TCGA)数据库和Gene Expression Omnibus(GEO)数据库的信息,通过分析FOXD1在OSCC中的表达特点,探讨FOXD1在OSCC预后中的价值,同时分析FOXD1与肿瘤微环境(tumor microenvironment,TME)的关系,为进一步明确OSCC的发病机制和治疗策略提供依据。
1 材料与方法 1.1 数据来源从TCGA数据库(https://portal.gdc.cancer.gov/)下载头颈部鳞状细胞癌的基因表达谱三级数据,并进行标准化处理,提取FOXD1基因表达信息。从cBioportal数据库(https://www.cbioportal.org/)下载患者的临床信息,包括年龄、性别、TNM分期、病理分期以及预后情况。剔除临床信息和生存资料不完整的样本,共314例OSCC样本和44例癌旁组织样本纳入研究,见表 1。
| Item | n | Percentage(%) |
| Age | ||
| ≤60 years | 146 | 46.5 |
| > 60 years | 168 | 53.5 |
| Sex | ||
| Male | 218 | 69.4 |
| Female | 96 | 30.6 |
| Clinical stage | ||
| Ⅰ | 11 | 3.5 |
| Ⅱ | 77 | 24.5 |
| Ⅲ | 67 | 21.3 |
| Ⅳ | 159 | 50.6 |
| N stage | ||
| N0 | 169 | 53.8 |
| N1 | 57 | 18.2 |
| N2 | 84 | 26.7 |
| N3 | 4 | 1.3 |
| T stage | ||
| T1 | 21 | 6.7 |
| T2 | 102 | 32.5 |
| T3 | 79 | 25.1 |
| T4 | 112 | 35.7 |
| Grade | ||
| G1 | 46 | 14.6 |
| G2 | 192 | 61.1 |
| G3 | 76 | 24.3 |
通过关键词“head and neck” “oral”和“carcinoma” “cancer” “tumor” “neoplasma”在GEO数据库(https://www.ncbi.nlm.nih.gov/geo/)中搜索相应的GEO数据集,下载芯片表达数据,提取FOXD1基因表达信息。共4个数据集纳入研究,见表 2。
| Datasets | Contributor | Disease type | Experimental platform | Number of cases(cancer/control) |
| GSE25099 | PENG C, et al.(2011) | OSCC | Affymetrix Human Exon 1.0 ST Array | 57/22 |
| GSE30784 | CHEN C, et al.(2011) | OSCC | Affymetrix Human Genome U133 Array | 167/45 |
| GSE31056 | REIS PP, et al.(2011) | OSCC | Affymetrix Human Genome U133 Array | 23/24 |
| GSE74530 | OGHUMU S, et al.(2017) | OSCC | Affymetrix Human Genome U133 Array | 6/6 |
1.2 FOXD1表达及其与临床特征、预后的关系
比较TCGA数据集中FOXD1在癌组织和癌旁组织/正常组织中的表达差异。GEO数据集作为外部验证数据集,分别分析4个GEO数据集中FOXD1在癌组织和癌旁组织/正常组织中的表达差异。分析FOXD1表达与临床参数的关系。根据FOXD1表达的中位数划分,将OSCC样本分为FOXD1高表达组和低表达组。
1.3 FOXD1与TME的关系采用Estimation of Stromal and Immune Cells in Malignant Tumor Tissues Using Expression Data(ESTIMATE)算法对下载的RNA表达数据进行基质评分和免疫评分,并依据FOXD1的中位数将样本分成FOXD1高表达组和低表达组,比较2组基质评分和免疫评分的差异。Cell-type Identification by Estimating Relative Subsets of known RNA Transcripts(CIBERSORT)是一种采用标准化基因表达数据来评估免疫细胞组成的生物信息学方法。采用CIBERSORT算法比较FOXD1高表达组和低表达组22种免疫细胞构成比。22种免疫细胞包括巨噬细胞(M0、M1、M2),7种T细胞亚型、B细胞(初始B细胞、记忆性B细胞)、肥大细胞(未活化、活化)、自然杀伤细胞(未活化、活化)、树突细胞(未活化、活化)、浆细胞、单核细胞、嗜酸性粒细胞和中性粒细胞。
1.4 基因集富集分析(gene set enrichment analysis,GSEA)采用GSEA工具,利用MSigDB数据库中参考数据集“c2.cp.kegg.v7.2.symbols.gmt gene sets”和“C5.go.bp.v7.2.symbols.gmt gene set”进行FOXD1基因相关生物学功能和信号通路富集分析。以错误发现率(false discovery rate,FDR) < 0.25和P < 0.05的基因集为显著富集基因集。
1.5 统计学分析采用SPSS 22.0软件进行统计学分析。计量资料2组比较采用t检验,多组比较采用单因素方差分析。采用Kapalan-Meier曲线和Log-rank检验方法比较FOXD1高表达组和低表达组的生存差异。采用单因素和多因素Cox回归分析方法评估FOXD1的预后价值。P < 0.05为差异有统计学意义。
2 结果 2.1 FOXD1在OSCC中的表达TCGA数据集中,OSCC样本FOXD1表达显著高于癌旁组织(P < 0.001,图 1A)。4个GEO数据集,GSE25099(P < 0.001),GSE31056(P < 0.001),GSE74530(P < 0.001)分析结果表明FOXD1在癌组织中的表达量均显著高于正常组织(图 1B~1D);GSE30784数据集中FOXD1表达显著高于良性增生组织(P < 0.001,图 1E)。
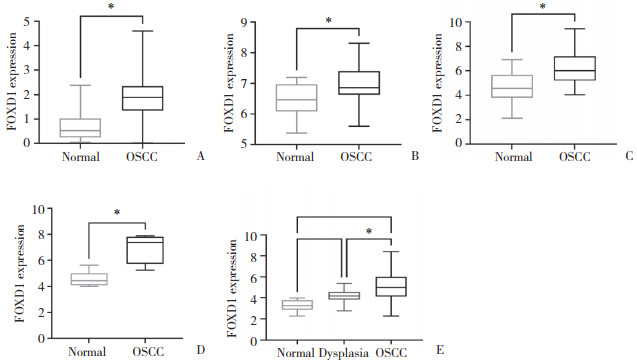
|
| A,TCGA dataset;B,GSE25099 dataset;C,GSE31056 dataset;D,GSE74530 dataset;E,GSE30784 dataset.*P < 0.001. 图 1 TCGA和GEO数据集中FOXD1在OSCC组织和正常组织中的表达 Fig.1 FOXD1 expression in OSCC and normal control group in TCGA and GEO datasets |
2.2 FOXD1表达与临床特征、预后的关系
TCGA数据集中,FOXD1表达与临床分期(P = 0.107)、病理分期(P = 0.496)、N分期(P = 0.150)和T分期(P = 0.102)不相关(图 2)。但FOXD1在临床分期Ⅱ期(P = 0.050)和Ⅲ期(P = 0.027)中的表达显著高于分期Ⅰ期。在N分期和T分期中,FOXD1表达随着N分期升级有逐渐增高的趋势。
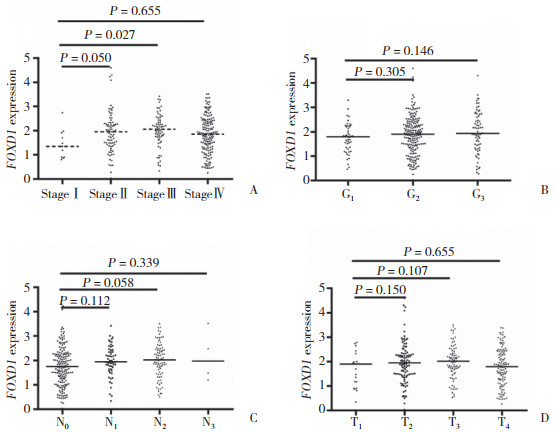
|
| A,clinical stage;B,pathological stage;C,N stage;D,T stage. 图 2 FOXD1表达与临床特征的关系 Fig.2 Correlation between FOXD1 expression and clinical parameters |
Kaplan-Meier生存曲线显示FOXD1表达状态对OSCC患者总体生存率和无病生存率的影响。结果显示,FOXD1高表达组的总体生存率(P = 0.039)和无病生存率(P = 0.041)显著低于FOXD1低表达组(图 3)。进一步分析FOXD1表达和OSCC的临床参数与预后的关系,如表 3所示,单因素Cox回归分析中,FOXD1表达与总体生存率(HR = 1.408,95%CI:1.017~1.949,P = 0.039)和无病生存率(HR = 1.360,95%CI:1.011~1.830,P = 0.042)相关。多因素Cox回归分析结果显示FOXD1表达仅与无病生存率(HR = 1.401,95%CI:1.038~1.891,P = 0.027)相关,可作为无病生存的独立预后指标。
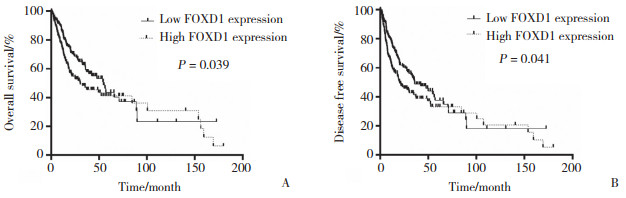
|
| A,overall survival;B,disease free survival. 图 3 OSCC患者的FOXD1表达与预后的Kaplan-Meier生存曲线 Fig.3 Kaplan-Meier curves showing effect of FOXD1 expression on prognosis |
| Item | Univariate analysis | Multivariate analysis | |||||
| HR | 95%CI | P | HR | 95%CI | P | ||
| Overall survival | |||||||
| Age | 1.296 | 0.936-1.795 | 0.118 | ||||
| Gender | 0.923 | 0.656-1.299 | 0.647 | ||||
| Metastasis | 2.607 | 0.363-18.731 | 0.341 | ||||
| Clinical stage | 1.187 | 0.992-1.420 | 0.061 | ||||
| N stage | 1.210 | 1.012-1.448 | 0.037 | 1.227 | 1.024-1.469 | 0.026 | |
| T stage | 1.152 | 0.975-1.361 | 0.096 | ||||
| Grade | 1.230 | 0.958-1.580 | 0.104 | ||||
| FOXD1 expression | 1.408 | 1.017-1.949 | 0.039 | ||||
| Disease-free survival | |||||||
| Age | 1.262 | 0.937-1.699 | 0.126 | ||||
| Gender | 0.963 | 0.704-1.317 | 0.813 | ||||
| Metastasis | 2.160 | 0.301-15.474 | 0.443 | ||||
| Clinical stage | 1.182 | 1.003-1.393 | 0.045 | 1.194 | 1.010-1.412 | 0.038 | |
| N stage | 1.122 | 0.950-1.324 | 0.176 | ||||
| T stage | 1.156 | 0.993-1.347 | 0.062 | ||||
| Grade | 1.148 | 0.914-1.442 | 0.235 | ||||
| FOXD1 expression | 1.360 | 1.011-1.830 | 0.042 | 1.401 | 1.038-1.891 | 0.027 | |
2.3 FOXD1与TME的关系
ESTIMATE分析结果显示,FOXD1高表达组的基质评分(P = 0.014)和免疫评分(P = 0.028)显著低于FOXD1低表达组(图 4A)。CIBERSORT分析结果显示FOXD1高表达组的M1型巨噬细胞(P = 0.037)和活化肥大细胞(P = 0.010)含量显著高于低表达组;而FOXD1高表达组未活化的树突状细胞(P = 0.040)和滤泡辅助性T细胞(P = 0.043)含量显著低于低表达组(图 4B)。
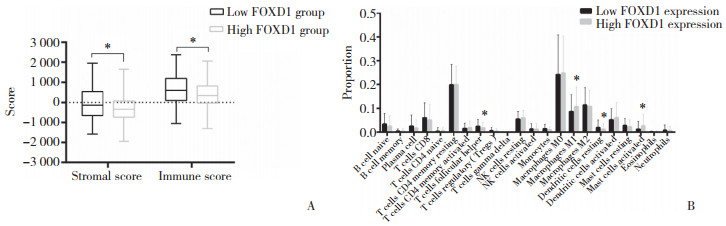
|
| A,comparison of stromal score and immune score between high FOXD1 expression and low FOXD1 expression groups;B,proportions of immune infiltrating cell subtypes between high FOXD1 expression and low FOXD1 expression groups. *P < 0.05 vs low FOXD1 expression group. 图 4 FOXD1表达与TME的关系 Fig.4 Relationship between FOXD1 expression and tumor microenvironment |
2.4 GSEA分析
GSEA分析结果显示,与FOXD1高表达表型相关的信号通路包括:花生四烯酸代谢、细胞色素P450代谢、甾类激素生物合成、细胞黏附分子和PPAR信号通路(图 5A);另外,FOXD1高表达表型与角质细胞分化、上皮细胞分化、补体活化、B细胞调控免疫和吞噬作用等信号通路相关(图 5B)。
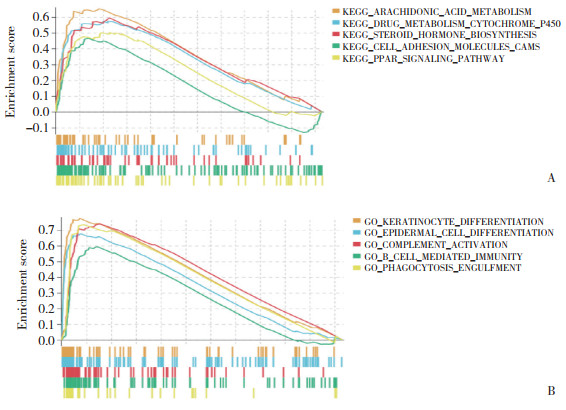
|
| A,KEGG pathways;B,biological process. 图 5 高表达FOXD1和低表达FOXD1的GESA分析 Fig.5 GSEA analysis of high and low FOXD1 expression phenotypes |
3 讨论
FOX家族成员可通过FOX结构域结合DNA来调控胚胎发育,调节免疫、调节糖类和脂类代谢,以及调控细胞增殖、凋亡、迁徙和转移等多种生物学过程[4]。FOX家族成员主要包括FOXA、FOXC、FOXM、FOXO和FOXP [5]。FOXD1作为FOX转录因子家族的新成员,位于染色体5q13.2。目前,研究[6-11]显示FOXD1参与多种肿瘤(肺癌、大肠癌、卵巢癌、乳腺癌,鼻咽癌、胃癌和前列腺癌)的发生发展过程。TCGA大数据分析显示,FOXD1在乳腺癌、胆管癌、结肠癌、食管癌、头颈部鳞状细胞癌、肝细胞癌、肺癌、直肠癌和胃癌中上调,而在肾透明细胞癌、甲状腺癌、子宫内膜癌中下调。一项有关卵巢癌的研究[8]显示卵巢癌细胞系中上调FOXD1可诱导卵巢癌细胞阻滞在G1期,从而抑制细胞增殖,且高表达的FOXD1与良好的预后相关。因此,FOXD1可能在肿瘤发展过程中呈现促癌和抑癌的两面性。
本研究基于TCGA和GEO数据库,发现FOXD1在OSCC组织中的表达显著上调。预后分析结果显示FOXD1高表达与不良预后相关,可作为评估预后的独立因素。CHEN等[12]研究结果显示在口腔癌的发展过程中,FOXD1可通过上调lncRNA CYTOR促进上皮间质转化和化疗抗性。LIN等[13]发现抑制FOXD1可显著增强OSCC患者放疗的有效性,且促进肿瘤免疫监视。通过对TCGA数据进行分析发现,FOXD1 mRNA水平可作为OSCC诊断和预后的潜在标志物[5]。本研究结果与这些研究结果一致,推测FOXD1可作为新的癌基因参与OSCC发生发展过程。
TME是肿瘤细胞生长的内环境,主要由血管生成细胞、免疫浸润细胞及癌相关的成纤维细胞三大基质细胞组成[14]。研究[15]发现,OSCC发生发展与TME的变化紧密相连。在OSCC的TME中,免疫细胞微环境、免疫抑制细胞微环境、间质微环境是一个相互联系、相互调节的整体,协同作用决定OSCC的发展及转归。本研究基于OSCC转录组学数据,采用ESTIMATE算法,预测肿瘤组织中浸润的基质细胞和免疫细胞的比例。结果显示OSCC样本中,FOXD1高表达组的基质评分和免疫评分均显著低于FOXD1低表达组(P < 0.05)。为了进一步探讨TME中免疫浸润细胞的变化趋势,本研究采用CIBERSORT算法对TME中22种免疫细胞亚型的组成进行评估。结果显示,M1型巨噬细胞、未活化树突状细胞、活化肥大细胞和滤泡辅助性T细胞在FOXD1高表达组和低表达组存在显著差异。因此推测FOXD1与TME的变化密切相关,且影响TME中免疫浸润细胞的组成。
本研究采用生物信息学方法对FOXD1影响的生物学过程和信号通路进行预测,结果显示,FOXD1主要富集在细胞黏附途径、花生四烯酸、类固醇激素生物合成以及PPAR信号通路。高FOXD1表达表型主要与上皮细胞、角化细胞分化以及免疫细胞调控相关。
综上所述,本研究通过对生物信息数据库的挖掘,发现FOXD1表达在OSCC组织中呈现上调的趋势,且与OSCC患者的不良预后相关。FOXD1表达影响TME中基质细胞和免疫浸润细胞的变化。FOXD1可能作为潜在的癌基因参与OSCC的发生发展过程,通过调控物质代谢和免疫反应调控OSCC的进展。
| [1] |
BRAY F, FERLAY J, SOERJOMATARAM I, et al. Global cancer statistics 2018:globocan estimates of incidence and mortality worldwide for 36 cancers in 185 countries[J]. CA: a Cancer J Clin, 2018, 68(6): 394-424. DOI:10.3322/caac.21492 |
| [2] |
RIVERA C, OLIVEIRA AK, COSTA RAP, et al. Prognostic biomarkers in oral squamous cell carcinoma: a systematic review[J]. Oral Oncol, 2017, 72: 38-47. DOI:10.1016/j.oraloncology.2017.07.003 |
| [3] |
QUINTERO-RONDEROS P, LAISSUE P. The multisystemic functions of FOXD1 in development and disease[J]. J Mol Med, 2018, 96(8): 725-739. DOI:10.1007/s00109-018-1665-2 |
| [4] |
JIN Y, LIANG Z, LOU H, et al. The emerging roles of fox family transcription factors in chromosome replication, organization, and genome stability[J]. Cells, 2020, 9(1): 258. DOI:10.3390/cells9010258 |
| [5] |
WANG J, LI W, ZHAO Y, et al. Members of FOX family could be drug targets of cancers[J]. Pharmacol Ther, 2018, 181: 183-196. DOI:10.1016/j.pharmthera.2017.08.003 |
| [6] |
LI D, FAN S, YU F, et al. FOXD1 promotes cell growth and metastasis by activation of vimentin in NSCLC[J]. Cell Physiol Biochem, 2018, 51(6): 2716-2731. DOI:10.1159/000495962 |
| [7] |
PAN F, LI M, CHEN W. FOXD1 predicts prognosis of colorectal cancer patients and promotes colorectal cancer progression via the ERK 1/2 pathway[J]. Am J Transl Res, 2018, 10(5): 1522-1530. |
| [8] |
WANG Y, QIU C, LU N, et al. FOXD1 is targeted by miR-30a-5p and miR-200a-5p and suppresses the proliferation of human ovarian carcinoma cells by promoting p21 expression in a p53-independent manner[J]. Int J Oncol, 2018, 52(6): 2130-2142. DOI:10.3892/ijo.2018.4359 |
| [9] |
ZHAO YF, ZHAO JY, YUE H, et al. FOXD1 promotes breast cancer proliferation and chemotherapeutic drug resistance by targeting p27[J]. Biochem Biophys Res Commun, 2015, 456(1): 232-237. DOI:10.1016/j.bbrc.2014.11.064 |
| [10] |
WU Q, MA J, WEI J, et al. FOXD1-AS1 regulates FOXD1 translation and promotes gastric cancer progression and chemoresistance by activating the PI3K/AKT/mTOR pathway[J]. Mol Oncol, 2021, 15(1): 299-316. DOI:10.1002/1878-0261.12728 |
| [11] |
LI X, JIAO M, HU J, et al. miR-30a inhibits androgen-independent growth of prostate cancer via targeting MYBL2, FOXD1, and SOX4[J]. Prostate, 2020, 80(9): 674-686. DOI:10.1002/pros.23979 |
| [12] |
CHEN SW, YANG MW, WANG CY, et al. Forkhead box D1 promotes EMT and chemoresistance by upregulating lncRNA CYTOR in oral squamous cell carcinoma[J]. Cancer Lett, 2021, 503: 43-53. DOI:10.1016/j.canlet.2020.11.046 |
| [13] |
LIN CH, LEE HH, CHANG WM, et al. FOXD1 repression potentiates radiation effectiveness by downregulating G3BP2 expression and promoting the activation of TXNIP-related pathways in oral cancer[J]. Cancers (Basel), 2020, 12(9): 2690. DOI:10.3390/cancers12092690 |
| [14] |
AUGUSTIN RC, DELGOFFE GM, NAJJAR YG, et al. Characteristics of the tumor microenvironment that influence immune cell functions: hypoxia, oxidative stress, metabolic alterations[J]. Cancers (Basel), 2020, 12(12): 3802. DOI:10.3390/cancers12123802 |
| [15] |
ALVES A, DIEL L, RAMOS G, et al. Tumor microenvironment and oral squamous cell carcinoma: a crosstalk between the inflammatory state and tumor cell migration[J]. Oral Oncol, 2021, 112: 105038. DOI:10.1016/j.oraloncology.2020.105038 |
 2021, Vol. 50
2021, Vol. 50




