文章信息
- 韩笑, 阎启昌, 王欣玲
- HAN Xiao, YAN Qichang, WANG Xinling
- 高糖微环境下小鼠晶状体上皮细胞中赖氨酸乙酰化的蛋白质组学分析
- Proteomic analysis of lysine acetylation in mouse lens epithelial cells under high-glucose microenvironment
- 中国医科大学学报, 2020, 49(8): 694-700
- Journal of China Medical University, 2020, 49(8): 694-700
-
文章历史
- 收稿日期:2019-10-12
- 网络出版时间:2020-07-28 16:42
研究[1-4]显示,翻译后修饰(post-translational modification,PTM)是探索疾病发病机制和防治靶点的重要研究对象。其中,乙酰化在生物体的炎症反应[5]、肿瘤发展[6]、糖尿病进程[7]中起着关键作用[8]。乙酰化可以影响蛋白质的各种生理过程(细胞周期、线粒体生物学过程、染色质重塑和核转运等)[9]。
研究[10-12]显示,白内障是导致失明的主要原因之一。糖尿病性白内障是1型糖尿病患者视力障碍的主要原因[13]。已有研究[14]显示2型糖尿病患者白内障发病率比正常人显著增加。可见高浓度血糖是促进白内障发生发展的重要诱因。但糖尿病性白内障的发病机制尚未明确。目前,糖尿病性白内障发病机制和防治靶点研究主要围绕长链非编码RNA(long non-coding RNA,lncRNA)[15]、GLUT葡萄糖转运蛋白[16]和白芦藜醇[17]等方面开展,但糖尿病性白内障的表观遗传调控和蛋白质组学改变尚不清楚。已有研究[18-20]证实PTM在糖尿病性白内障的发病过程中起重要作用。本研究利用稳定同位素标记氨基酸(stable isotope labeling by amino acids,SILAC)处理晶状体上皮细胞,采用泛乙酰化抗体富集,进行高分辨率液相色谱-串联质谱(liquid chromatography-mass spectro/mass spectro,LC-MS/MS)分析和生物信息学数据挖掘。
1 材料与方法 1.1 SILAC标记和高糖处理小鼠晶状体上皮细胞系α-TN4(中国医科大学附属第四医院晶状体学重点实验室提供)在37 ℃、95%空气和5%CO2环境下使用含有10%胎牛血清、100 U/mL青霉素、100 μg/mL链霉素和100 μg/mL潮霉素的DMEM培养基(葡萄糖5.5 mmol/L)培养。SILACTM蛋白定量试剂盒(美国Thermo Fisher Scientific公司)中重标同位素赖氨酸(L-13C-Lysine/L-13C615N4-Arginine,13C-Lysine)标记正常细胞;轻标同位素赖氨酸(L-Lysine/L-Arginine,12C-Lysine)标记高糖处理细胞。细胞在轻标和重标的培养基中培养至少6代(超过97%的标记效率)后,用45 mmol/L葡萄糖培养基处理轻标细胞24 h。处理后将2组细胞分别在轻标和重标SILAC培养基中培养48 h。扩增到所需数目后收集细胞,冷PBS洗涤2次,然后液氮快速冷冻收集。
1.2 蛋白质提取和乙酰化免疫沉淀(immunopreci-pitation,IP)富集将重标和轻标的晶状体上皮细胞等量混合,裂解缓冲液裂解后使用2-D Quant试剂盒确定蛋白质浓度。应用测序级的修饰胰蛋白酶(美国Promega公司)水解总蛋白。使用Agilent 300 Extend C18色谱柱,通过高pH反向高效液相色谱(high performance liquid chromatography,HPLC)分离肽。分离出80个组份后再融合成18个组份进行全蛋白测序;同样方法另外分离出80个组份融合成10个组份进行乙酰化IP富集。肽段溶解在IP缓冲液中,然后使用高质量的泛乙酰化抗体偶联树脂(抗体偶联的琼脂糖珠)进行乙酰化肽段富集,并在真空下冷冻干燥来进行LC-MS分析。
1.3 LC-MS/MS和数据库搜索通过超高性能液体分离系统EASY-nLC1000将肽段注射到纳升电喷雾源中进行串联质谱(mass spectro/mass spectro,MS/MS)分析。质谱仪Thermo ScientificTM Q ExactiveTM Plus(美国Thermo Fisher Scientific公司)连接超高效液相色谱(ultra performance liquid chromatography,UPLC),通过高分辨率分析仪Orbitrap检测并获得质谱数据。采用MaxQuan(tV.1.4.1.2)搜索二级质谱数据,并在Swissprot-Mouse数据库(https://www.uniprot.org/statistics/Swiss-Prot)进行数据匹配,同时使用反数据库排除随机匹配引起的误报。(1)消化酶设置为Trypin/P;(2)最小肽段长度设置为7个氨基酸残基;(3)最高耐受4个漏切位点、5种修饰和5个电荷;(4)进行质谱数据的质量控制。
1.4 蛋白质基因本体(gene ontology,GO)注释和亚细胞定位使用UniProt-GOA数据库(http://www.ebi.ac.uk/GOA/)对蛋白质进行GO注释。InterProScan软件用于功能性GO注释。根据GO注释将差异表达的蛋白质分为生物过程、细胞成分和分子功能3类进行分析。Wolfpsort软件用于蛋白质的亚细胞定位。
1.5 真核直系同源组(clusters of eukaryotic orthologous groups,KOG)分析利用KOG数据库(http://www.ncbi.nlm.nih.gov/KOG)将差异表达的蛋白分为信息存储和处理、细胞过程和信号传导、新陈代谢以及欠佳表征4类进行分析。
1.6 统计学分析采用GO富集分析、京都基因与基因组百科全书(Kyoto encyclopeida of genes and genomes,KEGG)富集分析(KEGG数据库,http://www.genome.jp/kegg)、蛋白质结构域富集分析(InterPro数据库,http://www.ebi.ac.uk/interpro/)和复合物富集分析(CORUM数据库,http://mips.gsf.de/genre/proj/corum/index.html),分别检测差异表达蛋白在功能、通路、结构域、复合物中的比例及变化情况,采用Fisher确切概率法(双侧)得到富集检验的P值,然后对数转换作为横坐标,富集分析的条形图越长,代表差异表达蛋白在该分类或者功能上的富集越显著。使用标准错误发现率控制方法对多种假设检验进行校正,校正后P < 0.05为差异有统计学意义。
2 结果 2.1 小鼠晶状体上皮细胞乙酰化频率和位点性质分析共鉴定出694个乙酰化蛋白,蛋白发生乙酰化的位点1~19个。共有224个(32%)蛋白具有多个赖氨酸乙酰化修饰位点,其中,Nucleolin(P09405)和Filamin-B(Q80X90)有9个乙酰化位点;Histone H2B type1-C/E/G(Q6ZWY9)有10个乙酰化位点;Myosin-9(Q8VDD5)有19个乙酰化位点。另外,得分 > 40的1 147个乙酰化肽段定量检测结果显示,肽段因长度不同表现出不同的丰度,多数肽段长度为8~14个氨基酸,与胰蛋白酶肽段的特性一致。质量误差设置为10/百万,质谱数据质控检验结果显示质谱仪处于性能稳定状态时,得到的肽段误差呈正态分布。
2.2 高糖环境下小鼠晶状体上皮细胞中乙酰化的GO分析和亚细胞定位结果显示,19%表达上调和23%表达下调的乙酰化蛋白在生物过程中参与了细胞过程;16%表达上调和21%表达下调的乙酰化蛋白参与了代谢过程。52%表达上调和48%表达下调的乙酰化蛋白具有结合功能;28%表达上调和37%表达下调的乙酰化蛋白具有催化活性。32%表达上调和32%表达下调的乙酰化蛋白发生在细胞中;27%表达上调和31%表达下调的乙酰化蛋白发生在细胞器。乙酰化蛋白的亚细胞定位结果显示,45%表达上调的乙酰化蛋白位于细胞核;44%表达下调的乙酰化蛋白位于线粒体。40%表达上调的乙酰化蛋白位于细胞质中,28%表达下调的乙酰化蛋白分布在细胞核中,见表 1。表明高糖微环境下晶状体上皮细胞内乙酰化蛋白发生了广泛的生物学变化。
| Item | Acetylation protein(%) | |
| up-regulation | down-regulation | |
| Biological process | ||
| Cellular process | 19 | 23 |
| Metabolic process | 16 | 21 |
| Single-organism process | 14 | 13 |
| Biological regulation | 9 | 11 |
| Response to stimulus | 9 | 7 |
| Cellular component organization or biogenesis | 8 | 5 |
| Multicellular organismal process | 7 | 6 |
| Developmental process | 7 | 5 |
| Localization | 3 | 3 |
| Reproduction | 2 | 0 |
| Signaling | 2 | 3 |
| Immune system process | 0 | 2 |
| Other | 4 | 1 |
| Molecular function | ||
| Binding | 52 | 48 |
| Catalytic activity | 28 | 37 |
| Structural molecule activity | 7 | 0 |
| Nucleic acid binding transcription factor activity | 3 | 2 |
| Protein binding transcription factor activity | 3 | 3 |
| Transporter activity | 0 | 3 |
| Enzyme regulator activity | 0 | 2 |
| Other | 7 | 5 |
| Cellular component | ||
| Cell | 32 | 32 |
| Organelle | 27 | 31 |
| Macromolecular complex | 21 | 9 |
| Membrane-enclosed lumen | 11 | 12 |
| Membrane | 5 | 11 |
| Extracellular region | 2 | 2 |
| Nucleoid | 0 | 2 |
| Other | 2 | 1 |
| Subcellular location | ||
| Nuclear | 45 | 28 |
| Cytoplasm | 40 | 18 |
| Extracellular | 9 | 5 |
| Cytoplasm and nuclear | 3 | 0 |
| Plasma membrane | 3 | 0 |
| Mitochondria | 0 | 44 |
| Endoplasmic reticulum | 0 | 3 |
| Peroxisome | 0 | 2 |
2.3 高糖环境下小鼠晶状体上皮细胞中乙酰化的KOG分析
将72个表达显著上调的蛋白分为19个类别(图 1A)。根据KOG注释,物质代谢类别(KOG类别G、F、I和P)和能量代谢类别(KOG类别C)蛋白发生了显著乙酰化。染色质结构和动力学,翻译、核糖体结构和生物发生,翻译后修饰、蛋白质更新、分子伴侣(分别为KOG分类B、J和O)有关蛋白也被乙酰化。将55个表达显著下调的蛋白分为16个类别(图 1B)。根据KOG注释,物质代谢类别(KOG类别G、E、F、H、I和P)和能量代谢类别(KOG类别C)蛋白是主要鉴定的乙酰化蛋白。翻译后修饰、蛋白质更新、分子伴侣,RNA加工和修饰和转录(分别为KOG类别O、A和K)相关蛋白也被乙酰化修饰。
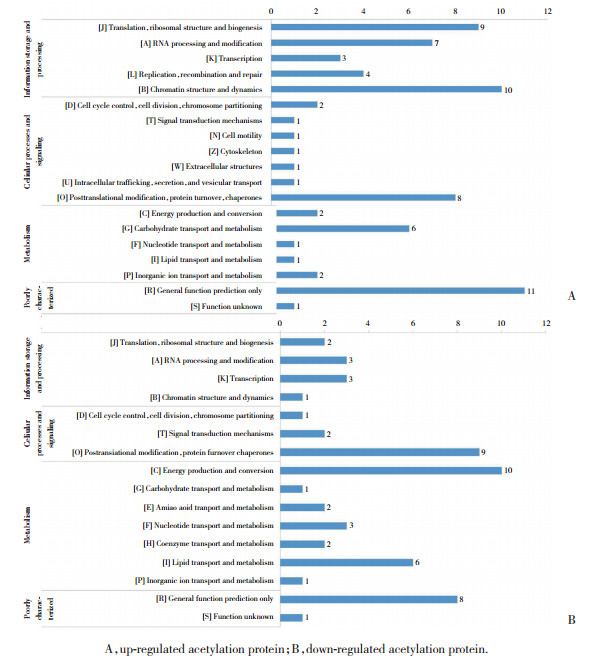
|
| A, up-regulated acetylation protein; B, down-regulated acetylation protein. 图 1 高糖环境下小鼠晶状体上皮细胞中乙酰化的KOG分析 Fig.1 KOG of acetylation in mouse lens epithelial cells under the high-glucose conditions |
2.4 高糖环境下小鼠晶状体上皮细胞中乙酰化的功能富集
生物过程、细胞成分和分子功能的富集程度结果显示:(1)生物过程,氧化还原过程、前体代谢产物和能量产生、免疫受体的体细胞多样化、体细胞DNA重组、二羧酸代谢过程和B细胞有关过程中均乙酰化蛋白富集水平显著。(2)细胞成分,乙酰化蛋白主要富集在线粒体、核小体、DNA弯曲复合体、线粒体部分、蛋白质-DNA复合体、细胞表面、剪接体复合体和线粒体基质中。(3)分子功能,蛋白质异二聚活性、DNA结合、氧化还原酶活性、脂肪酰基-CoA结合、分子内氧化还原酶活性不匹配的DNA结合相关的功能中存在显着富集。见图 2。
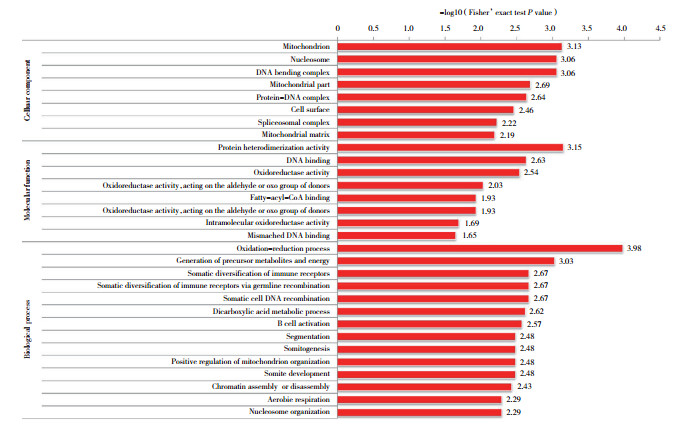
|
| 图 2 高糖环境下小鼠晶状体上皮细胞中乙酰化的GO功能富集 Fig.2 GO functional enrichment of acetylation in mouse lens epithelial cells under the high-glucose conditions |
KEGG通路富集分析结果显示,乙酰化蛋白主要富集全身性红斑狼疮,酒精中毒,甲状腺激素合成,碳代谢,剪接体,麻疹,RNA降解,过氧化物酶、果糖、甘露醇代谢等生理病理过程,见图 3。蛋白质结构域富集分析结果显示,蛋白质结构域富集在组蛋白折叠、组蛋白核心、烯醇酶、富含GC的序列DNA结合、共激活因子CBP、KIX域、锌指、SWAP、plectin/S10、核受体共激活因子和醛缩酶类型TIM,见图 4。乙酰化蛋白复合物显著富集在muts-α-组蛋白H4复合物、TRPV5-S100A10-annexin 2复合物和S100A10-annexin 2复合物,见图 5。
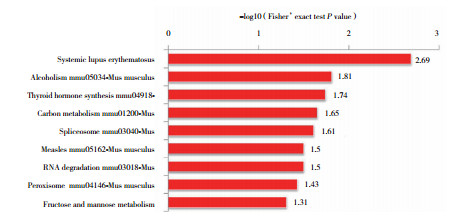
|
| 图 3 高糖环境下小鼠晶状体上皮细胞中乙酰化的KEGG功能富集 Fig.3 KEGG functional enrichment of acetylation in mouse lens epithelial cells under the high-glucose conditions |
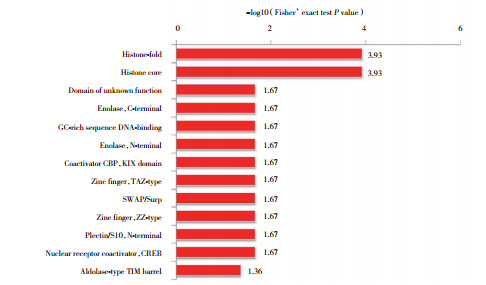
|
| 图 4 高糖环境下小鼠晶状体上皮细胞中乙酰化的蛋白质结构域功能富集 Fig.4 Domain functional enrichment of acetylation in mouse lens epithelial cells under the high-glucose conditions |

|
| 图 5 高糖环境下小鼠晶状体上皮细胞中乙酰化的蛋白质复合物功能富集 Fig.5 Complex functional enrichment of acetylation in mouse lens epithelial cells under the high-glucose conditions |
3 讨论
本研究在小鼠晶状体上皮细胞中共鉴定出694个乙酰化蛋白,蛋白发生乙酰化的位点1~19个。多数鉴定出的蛋白仅有1个或2个位点乙酰化,少数蛋白具有多个乙酰化位点。其中Myosin-9(Q8VDD5)具有19个乙酰化位点,该蛋白涉及多个重要功能(胞质分裂、细胞运动和细胞形态维持等)。已有研究[21]显示GroEL2是迄今为止报道的具有最多(24个)赖氨酸乙酰化位点的蛋白。一些蛋白质为何具有多个乙酰化位点,不同的乙酰化位点是同时还是逐步发生乙酰化问题仍有待深入研究。为了评估小鼠晶状体上皮细胞中乙酰化位点的分布特点,本研究计算了每个乙酰化蛋白的修饰位点数量。结果显示,共有224个(32%)蛋白具有多个赖氨酸乙酰化修饰位点。有研究[22]报道结核分枝杆菌中发现202个(26.2%)蛋白乙酰化,与枯草芽孢杆菌51个(18.8%)蛋白乙酰化比较,提示赖氨酸乙酰化在结核分枝杆菌中具有重要生理作用。由此推论,乙酰化在小鼠晶状体上皮细胞内也具有重要作用。
为了全面了解高糖对α-TN4细胞中蛋白赖氨酸乙酰化的影响,对所有已鉴定的乙酰化蛋白的GO功能分类、亚细胞定位和KOG功能分类进行分析。KOG可对真核细胞蛋白进行完整全面的分类,与GO和KEGG比较,KOG数据库提供的蛋白类别更多[23]。KOG分析结果显示,在高糖环境中晶状体上皮细胞内乙酰化主要参与了代谢过程,这与家蚕[23]、玫瑰孢菌[24]、大肠杆菌[25]和副溶血弧菌[26]等相关研究阐述乙酰化广泛存在于各物种的代谢过程一致。本研究结果表明晶状体上皮细胞内乙酰化在代谢过程中起主要作用,KOG分类也显示乙酰化广泛影响其他生物学功能。对乙酰化的生物学功能分析结果显示,乙酰化具有广泛的分子功能,并且参与了晶状体上皮细胞内的众多生物过程。这种广泛的功能对调节晶状体上皮细胞的内部环境和影响晶状体透明性等方面都起着重要作用,进而影响糖尿病性白内障的发生发展。
综上所述,本研究使用SILAC标记和泛乙酰化富集进行了高分辨率LC-MS/MS和生物信息学分析,成功揭示了高糖处理后晶状体上皮细胞中赖氨酸乙酰化的全面变化。本研究为首次在α-TN4细胞中进行高糖条件下的赖氨酸乙酰化和蛋白质组学分析。高糖诱导的赖氨酸乙酰化通过参与和改变多种生物功能、通路、结构域和蛋白质复合物,进而改变众多蛋白的表达及其功能。本研究为阐明小鼠晶状体上皮细胞中高糖微环境下致病机制,探索糖尿病性白内障治疗靶点提供了重要依据。
| [1] |
LIU J, QIAN C, CAO XT. Post-translational modification control of innate immunity[J]. Immunity, 2016, 45(1): 15-30. DOI:10.1016/j.immuni.2016.06.020 |
| [2] |
CHAKRABORTY J, BASSO V, ZIVIANI E. Post translational modification of Parkin[J]. Biol Direct, 2017, 12(1): 6. DOI:10.1186/s13062-017-0176-3 |
| [3] |
HEO KS. Regulation of post-translational modification in breast cancer treatment[J]. BMB Rep, 2019, 52(2): 113-118. DOI:10.5483/BMBRep.2019.52.2.017 |
| [4] |
LIN HN, CAROLL KS. Introduction:posttranslational protein modification[J]. Chem Rev, 2018, 118(3): 887-888. DOI:10.1021/acs.chemrev.7b00756 |
| [5] |
DASKALAKI MG, TSATSANIS C, KAMPRANIS SC. Histone methylation and acetylation in macrophages as a mechanism for regulation of inflammatory responses[J]. J Cell Physiol, 2018, 233(9): 6495-6507. DOI:10.1002/jcp.26497 |
| [6] |
AUDIA JE, CAMPBELL RM. Histone modifications and cancer[J]. Cold Spring Harb Perspect Biol, 2016, 8(4): a019521. DOI:10.1101/cshperspect.a019521 |
| [7] |
NAMMO T, UDAGAWA H, FUNAHASHI N, et al. Genome-wide profiling of histone H3K27 acetylation featured fatty acid signalling in pancreatic beta cells in diet-induced obesity in mice[J]. Diabetologia, 2018, 61(12): 2608-2620. DOI:10.1007/s00125-018-4735-7 |
| [8] |
LEE EJ, SEO JH, KIM KW. Special issue on protein acetylation:from molecular modification to human disease[J]. Exp Mol Med, 2018, 50(7): 1-2. DOI:10.1038/s12276-018-0103-4 |
| [9] |
DANCY BM, COLE PA. Protein lysine acetylation by p300/CBP[J]. Chem Rev, 2015, 115(6): 2419-2452. DOI:10.1021/cr500452k |
| [10] |
LI YJ, JIANG Q, CAO GF, et al. Repertoires of autophagy in the pathogenesis of ocular diseases[J]. Cell Physiol Biochem, 2015, 35(5): 1663-1676. DOI:10.1159/000373980 |
| [11] |
RICHTER GM, CHUNG J, AZEN SP, et al. Prevalence of visually significant cataract and factors associated with unmet need for cataract surgery:Los Angeles Latino Eye Study[J]. Ophthalmology, 2009, 116(12): 2327-2335. DOI:10.1016/j.ophtha.2009.05.040 |
| [12] |
CONGDON NG, FRIEDMAN DS, LIETMAN T. Important causes of visual impairment in the world today[J]. JAMA, 2003, 290(15): 2057-2060. DOI:10.1001/jama.290.15.2057 |
| [13] |
ŠIMUNOVIĆ M, PARADŽIK M, ŠKRABIĆ R, et al. Cataract as early ocular complication in children and adolescents with type 1 diabetes mellitus[J]. Int J Endocrinol, 2018, 2018: 6763586. DOI:10.1155/2018/6763586 |
| [14] |
BECKER C, SCHNEIDER C, ABALLÉA S, et al. diabetes is associated with an approximately two-fold increased detection rate of cataract[J]. Eye (Lond), 2018, 32(6): 1028-1035. DOI:10.1038/s41433-017-0003-1 |
| [15] |
GONG WF, ZHU GY, LI J, et al. lncRNA MALAT1 promotes the apoptosis and oxidative stress of human lens epithelial cells via p38MAPK pathway in diabetic cataract[J]. Diabetes Res Clin Pract, 2018, 144: 314-321. DOI:10.1016/j.diabres.2018.06.020 |
| [16] |
SWARUP A, BELL BA, DU JH, et al. Deletion of GLUT1 in mouse lens epithelium leads to cataract formation[J]. Exp Eye Res, 2018, 172: 45-53. DOI:10.1016/j.exer.2018.03.021 |
| [17] |
HIGASHI Y, HIGASHI K, MORI A, et al. Anti-cataract effect of resveratrol in high-glucose-treated streptozotocin-induced diabetic rats[J]. Biol Pharm Bull, 2018, 41(10): 1586-1592. DOI:10.1248/bpb.b18-00328 |
| [18] |
HAN X, DONG XX, SHI MY, et al. SUMOylation and deacetylation affect NF-κB p65 activity induced by high glucose in human lens epithelial cells[J]. Int J Ophthalmol, 2019, 12(9): 1371-1379. DOI:10.18240/ijo.2019.09.01 |
| [19] |
HAN X, WANG XL, LI Q, et al. HIF-1α SUMOylation affects the stability and transcriptional activity of HIF-1α in human lens epithelial cells[J]. Albrecht Von Graefes Arch Fur Klinische Und Exp Ophthalmol, 2015, 253(8): 1279-1290. DOI:10.1007/s00417-015-2999-x |
| [20] |
KIELMAS M, KIJEWSKA M, KLUCZYK A, et al. Comparison of modification sites in glycated crystallin in vitro and in vivo[J]. Anal Bioanal Chem, 2015, 407(9): 2557-2567. DOI:10.1007/s00216-015-8487-7 |
| [21] |
XIE LX, WANG XB, ZENG J, et al. Proteome-wide lysine acetylation profiling of the human pathogen Mycobacterium tuberculosis[J]. Int J Biochem Cell Biol, 2015, 59: 193-202. DOI:10.1016/j.biocel.2014.11.010 |
| [22] |
ZHANG YJ, IOERGER TR, HUTTENHOWER C, et al. Global assessment of genomic regions required for growth in Mycobacterium tuberculosis[J]. PLoS Pathog, 2012, 8(9): e1002946. DOI:10.1371/journal.ppat.1002946 |
| [23] |
NIE ZM, ZHU HL, ZHOU Y, et al. Comprehensive profiling of lysine acetylation suggests the widespread function is regulated by protein acetylation in the silkworm, Bombyx mori[J]. Proteomics, 2015, 15(18): 3253-3266. DOI:10.1002/pmic.201500001 |
| [24] |
LIAO GJ, XIE LX, LI X, et al. Unexpected extensive lysine acetylation in the trump-card antibiotic producer Streptomyces roseosporus revealed by proteome-wide profiling[J]. J Proteomics, 2014, 106: 260-269. DOI:10.1016/j.jprot.2014.04.017 |
| [25] |
ZHANG K, ZHENG SZ, YANG JS, et al. Comprehensive profiling of protein lysine acetylation in Escherichia coli[J]. J Proteome Res, 2013, 12(2): 844-851. DOI:10.1021/pr300912q |
| [26] |
PAN JY, YE ZC, CHENG ZY, et al. Systematic analysis of the lysine acetylome in Vibrio parahemolyticus[J]. J Proteome Res, 2014, 13(7): 3294-3302. DOI:10.1021/pr500133t |
 2020, Vol. 49
2020, Vol. 49




