文章信息
- 吴昆哲, 戴冬秋
- WU Kunzhe, DAI Dongqiu
- CRIM1基因对胃癌细胞增殖和凋亡的影响及其预后价值
- Effects of CRIM1 on the proliferation and apoptosis of gastric cancer cells and the prognostic value of CRIM1 expression
- 中国医科大学学报, 2020, 49(6): 565-569
- Journal of China Medical University, 2020, 49(6): 565-569
-
文章历史
- 收稿日期:2019-07-11
- 网络出版时间:2020-05-19 9:12
胃癌是最常见的消化道恶性肿瘤之一,在我国恶性肿瘤发病率中居第2位,死亡率中居第3位,且在新发病例中超过90%为中晚期胃癌[1]。尽管近年胃癌手术和药物治疗的效果有了很大的提升,但是胃癌极易发生转移,且晚期胃癌患者预后较差[2]。据报道,胃癌患者的5年生存率较低,2010年至2014年我国胃癌患者5年生存率仅为35.9%,较2000年至2004年的30.2%并未明显改善,这严重地危害了胃癌患者的生命健康[3]。胃癌的发生、发展是一个十分复杂的过程,受多种因素的调控,如促癌基因激活或抑癌基因失活均可导致胃癌细胞恶性增殖。因此,探索新的胃癌相关基因,对胃癌的治疗和预后具有重要意义。
富含半胱氨酸型运动神经元蛋白1 (cysteine-rich motor neuron 1,CRIM1)是一类糖基化的Ⅰ型跨膜蛋白,可以调控骨形态发生蛋白(bone morphogenetic protein,BMP)的表达。相关研究[4]表明,CRIM1在肿瘤细胞的增殖、凋亡、转移等过程中发挥着重要作用,CRIM1过表达可以促进肿瘤细胞增殖并延长肿瘤细胞周期。目前,CRIM1在胃癌中的作用仍未有报道。因此,本研究旨在通过沉默CRIM1基因探讨其对胃癌细胞增殖、凋亡的影响,为胃癌的临床治疗提供新的思路。
1 材料与方法 1.1 材料人胃癌细胞系AGS购自中科院上海细胞库;胎牛血清和DMEM培养基购自美国Gibco公司;脂质体Lipofectamine 3000购自美国Invitrogen公司;MTT购自美国Sigma公司;细胞凋亡检测试剂盒购自上海碧云天生物技术公司;兔抗人GAPDH抗体、CRIM1抗体、BCL-2抗体、BAX抗体均购自美国Cell Signaling公司;辣根过氧化物酶标记的山羊抗兔二抗购自博奥森生物公司。
1.2 方法 1.2.1 生物信息数据库分析基因表达谱交互式分析网站(gene expression profiling interactive analysis,GEPIA,http://gepia.cancer-pku.cn/index.html)是一个基于癌症基因组图谱(cancer genome atlas,TCGA)和基因型-组织表达项目(the genotype-tissue expression project,GTEx)中的数据,用于分析RNA测序的交互式Web服务器[5]。利用GEPIA网站中TCGA数据分析CRIM1基因在胃癌组织(408例)和癌旁正常组织(36例)中的表达水平。利用生物数据软件Kaplan-Meier plotter (http://kmplot.com/analysis/)的胃癌数据集,分析CRIM1表达与胃癌患者预后的关系。筛选条件:(1) mRNA RNA-seq:gastric cancer;(2) Probe set options:all probe sets per gene;(3) Gene symbol:CRIM1,其他选项按默认设置,共纳入876例患者数据。
1.2.2 细胞培养人胃癌细胞AGS购自中科院上海细胞库,使用含10%胎牛血清和1%青霉素-链霉素双抗混合液的DMEM培养基培养,并置于含5% CO2、恒温37 ℃的细胞孵育箱中。每1~2 d更换1次培养液,并按1:2的比例每2~3 d传代1次。
1.2.3 细胞转染转染前1 d将1×106个AGS细胞接种于6孔板中,置于37 ℃的细胞孵育箱中培养24 h后,使用脂质体Lipofectamine 3000转染小干扰RNA (small interfering RNA,siRNA),沉默CRIM1基因表达,所用siRNA片段由上海吉玛公司设计合成。转染分为3组:空白对照组、阴性对照组(si-NC组)和阳性转染组(si-CRIM1组)。采用Western blotting检测转染效果。每组设置5个复孔,转染后在细胞孵育箱中培养48 h,收集转染细胞用于后续研究。
1.2.4 MTT法检测细胞增殖能力取转染后各组AGS细胞,调整细胞浓度至3×103/mL并接种于96孔板中,置于恒温37 ℃的细胞孵育箱中培养48 h。每孔加入浓度为5 mg/mL的MTT溶液20 μL,继续培养4 h后弃去每孔中的上清液,随后向每孔加入150 μL DMSO,振荡混匀15 min后在酶联免疫检测仪上检测各孔在450 nm处读数,将光密度值(optical density,OD)视为细胞的密度,每组至少5个复孔,实验重复3次,将si-CRIM1组与si-NC组OD值的比值视为siRNA转染后AGS细胞的增殖率。
1.2.5 流式细胞术检测胃癌细胞的凋亡率取各组AGS细胞分别接种于6孔板中,并置于37 ℃、5% CO2条件下继续培养48 h。随后用PBS漂洗,用不含EDTA的胰酶消化,收集细胞,将细胞浓度调整为5×105/mL。向每孔中加入500 μL连接缓冲液使细胞重悬,随后每孔分别加入5 μL AnnexinⅤ-FITC和5 μL碘化丙啶染液,常温避光继续孵育10 min。将样品置于流式细胞仪上检测细胞凋亡率,每组检测1×105个细胞。实验重复3次。
1.2.6 Western blotting检测蛋白表达转染24 h后收集各组细胞,使用RIPA裂解液裂解细胞,并以12 000 r/min离心20 min,收集上清液,保存于-80 ℃冰箱中,BCA法测蛋白浓度。取30 μg蛋白进行SDS-PAGE电泳(80~120 V),100 V转膜1 h后,用5%脱脂奶粉室温封闭2 h。加入一抗,4 ℃孵育过夜,TBST缓冲液清洗3次,然后加入辣根过氧化物酶标记的二抗,37 ℃室温振荡1 h,再次TBST缓冲液清洗3次。滴加ECL显影剂,并曝光,应用Image J软件分析各组蛋白的灰度值。
1.3 统计学分析采用SPSS 22.0软件进行统计学分析。计量资料以x±s表示,组间比较采用成组设计t检验,P < 0.05为差异有统计学意义。
2 结果 2.1 生物信息数据库分析结果利用GEPIA网站获取CRIM1 mRNA在408例胃癌组织和36例癌旁正常组织中的表达情况。结果表明,胃癌组织中CRIM1 mRNA的表达水平明显高于癌旁正常组织,见图 1A。利用生物数据软件Kaplan-Meier plotter分析CRIM1基因表达与胃癌患者总生存期的关系。结果显示,CRIM1基因低表达患者的生存期显著高于CRIM1基因高表达患者,差异有统计学意义(P = 0.004 8),见图 1B。
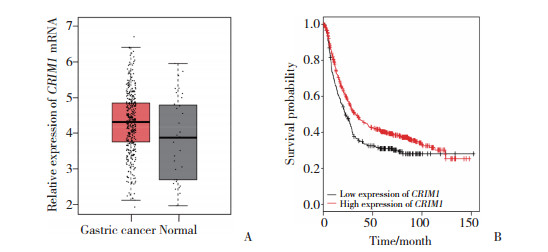
|
| A,expression of CRIM1 in gastric cancer tissue and normal gastric tissue based on analysis of the Gene Expression Profiling Interactive Analysis (GEPIA) database;B,survival curves constructed using the Kaplan-Meier plotter database. 图 1 CRIM1在胃癌中的表达情况以及CRIM1表达水平与胃癌患者总生存期的生存曲线 Fig.1 Expression levels of CRIM1 in gastric cancer and survival curves of patients with gastric cancer |
2.2 si-CRIM1沉默
si-NC组与空白对照组比较,AGS细胞中CRIM1蛋白表达水平无统计学差异(P > 0.05),而si-CRIM1组与空白对照组、si-NC组比较,AGS细胞中CRIM1蛋白相对表达量明显降低,差异均有统计学意义(分别为P = 0.037,P = 0.042),表明胃癌细胞转染siRNA后CRIM1基因被沉默。见图 2。
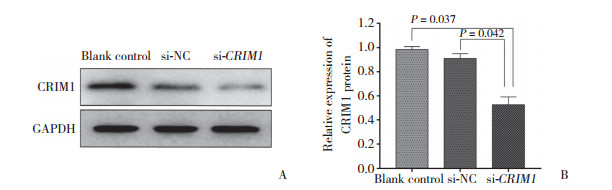
|
| A, the expression of CRIM1 protein was decreased by transfection with si-CRIM1; B, quantification of Western blotting results. 图 2 3组CRIM1蛋白的表达水平 Fig.2 Expression levels of CRIM1 protein in the three groups |
2.3 沉默CRIM1表达降低AGS细胞的增殖率
MTT结果表明,si-CRIM1组中AGS细胞增殖率明显低于空白对照组、si-NC组,且96 h时si-CRIM1组与空白对照组、si-NC组比较,AGS细胞增殖能力的差异均有统计学意义(分别为P = 0.035,P = 0.042)。见图 3。
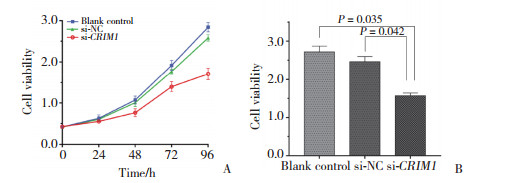
|
| A, proliferation rates of AGS cells in the three groups; B, quantification of the growth ability of AGS cells at 96 h. 图 3 沉默CRIM1后3组细胞的增殖情况 Fig.3 Proliferation of AGS cells in the three groups after silencing CRIM1 |
2.4 沉默CRIM1表达促进AGS细胞的凋亡率
流式细胞仪检测结果表明,空白对照组、si-NC组、si-CRIM1组中胃癌细胞凋亡率分别为(9.85±0.45) %、(10.74±0.73) %、(18.42±0.57) %,与空白对照组和si-NC组比较,si-CRIM1组中AGS细胞凋亡率明显升高,差异有统计学意义(P < 0.05)。见图 4。
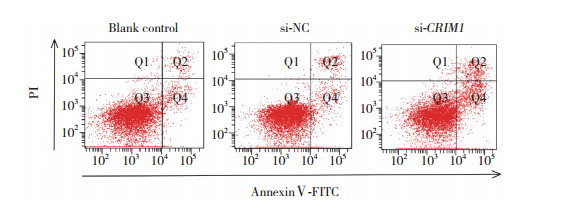
|
| 图 4 沉默CRIM1后3组细胞的凋亡率 Fig.4 Apoptosis rates of AGS cells in the three groups after silencing CRIM1 |
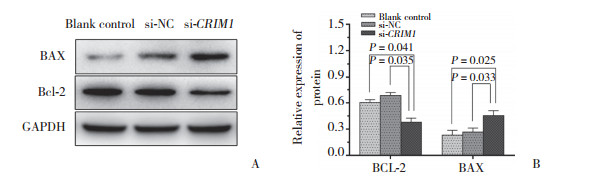
2.5 沉默CRIM1表达对AGS细胞中BCL-2、BAX蛋白表达的影响
Western blotting结果表明,si-CRIM1组AGS中凋亡相关蛋白BCL-2的相对表达量明显低于空白对照组和si-NC组(分别为P = 0.041,P = 0.035),而凋亡相关蛋白BAX的表达明显高于空白对照组和si-NC组,差异均有统计学意义(分别为P = 0.025,P = 0.033)。见图 5。
|
| A, expression of BCL-2 and BAX protein detected by Western blotting; B, quantification of Western blotting results. 图 5 Western blotting检测CRIM1沉默后3组AGS细胞的BCL-2和BAX蛋白水平 Fig.5 BCL-2 and BAX protein levels in AGS cells in the three groups detected by western blotting after silencing CRIM1 |
3 讨论
胃癌是世界上第三大常见的恶性肿瘤,其死亡率在世界范围内的肿瘤致死率中居第5位[6]。尽管近年来胃癌的治疗策略有了很大的改善,但胃癌患者的预后仍然很不乐观,其主要原因是胃癌的迅速进展与复发[7]。胃癌细胞获得较强的增殖能力和抗凋亡能力是导致胃癌迅速进展的主要原因。因此,找到与胃癌细胞增殖和凋亡相关的关键基因,可以为胃癌的靶向治疗提供新的分子靶点。目前,诸多基因已被报道参与胃癌的发生、发展,但仍未有文献报道CRIM1基因在胃癌中的作用。因此,本研究旨在通过沉默CRIM1基因探讨其在胃癌增殖和凋亡方面的作用。
CRIM1基因最早被报道与神经组织的发育有关,且在神经细胞构建中起着重要作用[8]。CRIM1基因位于人类第2号短臂染色体上(2p22.2),其翻译蛋白质量约为114×103。CRIM1是一种跨膜蛋白,含有胰岛素样生长因子结合蛋白功能和多个富半胱氨酸区域[10]。富半胱氨酸区域被认为可以与BMP特异性结合,CRIM1则可以通过富半胱氨酸区域结构抑制BMP前蛋白向成熟蛋白的转化[11]。近年来,BMP被报道在肿瘤细胞的增殖、凋亡和血管生成中起着重要作用[12]。CRIM1作为BMP拮抗因子,逐渐显现其在肿瘤中的作用。早期研究[13-15]证明,CRIM1在肾癌、肺癌、白血病等多种恶性肿瘤中表达升高,且与肿瘤的发生、增殖、凋亡和患者预后显著相关。此外,有研究[9]证实敲低CRIM1基因可以降低细胞与细胞以及细胞与基质的连接作用,从而抑制细胞的增殖。以上研究结果表明,CRIM1基因在多种恶性肿瘤中为促癌基因,但CRIM1在胃癌中的作用尚不明确。
本研究首先使用GEPIA平台中TCGA数据分析CRIM1基因,结果显示,CRIM1 mRNA表达水平在胃癌组织中显著高于正常胃黏膜上皮组织。此外,利用在线数据库Kaplan-Meier plotter分析CRIM1 mRNA表达与胃癌患者总生存期的关系,结果表明,CRIM1 mRNA高表达的胃癌患者总生存期较差。通过进一步实验验证,将沉默CRIM1的siRNA转染至胃癌AGS细胞中,探究下调CRIM1基因表达对胃癌细胞增殖和凋亡的影响。MTT和流式细胞术结果表明,沉默CRIM1基因表达后AGS细胞的增殖率显著降低,而凋亡率明显升高。Western blotting结果显示,沉默CRIM1基因表达可以抑制AGS细胞中BCL-2蛋白的表达,同时升高BAX蛋白表达,进一步说明CRIM1基因可能调控胃癌细胞凋亡过程。
综上所述,本研究结果提示,高表达的CRIM1基因与胃癌患者预后不良有关,并且该基因可能通过调控增殖和凋亡相关因子在胃癌的发生、发展中发挥作用。本研究为探究CRIM1在胃癌细胞增殖和凋亡中的作用提供了相关的实验依据。未来将进一步深入研究CRIM1基因在胃癌发生、发展中的作用机制,从而为胃癌的基因靶向治疗创造更多可能。
| [1] |
CHEN W, ZHENG R, BAADE PD, et al. Cancer statistics in China, 2015[J]. CA Cancer J Clin, 2016, 66(2): 115-132. DOI:10.3322/caac.21338 |
| [2] |
CHAU I, FUCHS CS, OHTSU A, et al. Association of quality of life with disease characteristics and treatment outcomes in patients with advanced gastric cancer:exploratory analysis of RAINBOW and REGARD phaseⅢ trials[J]. Eur J Cancer, 2019, 107: 115-123. DOI:10.1016/j.ejca.2018.11.013 |
| [3] |
ALLEMANI C, MATSUDA T, DI CARLO V, et al. Global surveillance of trends in cancer survival 2000-14(CONCORD-3):analysis of individual records for 37513025 patients diagnosed with one of 18 cancers from 322 population-based registries in 71 countries[J]. Lancet, 2018, 391(10125): 1023-1075. DOI:10.1016/S0140-6736(17)33326-3 |
| [4] |
ZENG H, TANG L. CRIM1, the antagonist of BMPs, is a potential risk factor of cancer[J]. Curr Cancer Drug Targets, 2014, 14(7): 652-658. DOI:10.2174/1568009614666140725094125 |
| [5] |
TANG Z, LI C, KANG B, et al. GEPIA:a web server for cancer and normal gene expression profiling and interactive analyses[J]. Nucleic Acids Res, 2017, 45(W1): W98-W102. DOI:10.1093/nar/gkx247 |
| [6] |
BRAY F, FERLAY J, SOERJOMATARAM I, et al. Global cancer statistics 2018:GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries[J]. CA Cancer J Clin, 2018, 68(6): 394-424. DOI:10.3322/caac.21492 |
| [7] |
WU DM, WANG S, WEN X, et al. Survival benefit of three different therapies in postoperative patients with advanced gastric cancer:a network meta-analysis[J]. Front Pharmacol, 2018, 9: 929. DOI:10.3389/fphar.2018.00929 |
| [8] |
ZHANG Y, FAN JQ, HO JW, et al. Crim1 regulates integrin signaling in murine lens development[J]. Development, 2016, 143(2): 356-366. DOI:10.1242/dev.125591 |
| [9] |
GLIENKE J, STURZ A, MENRAD A, et al. CRIM1 is involved in endothelial cell capillary formation in vitro and is expressed in blood vessels in vivo[J]. Mech Dev, 2002, 119(2): 165-175. DOI:10.1016/s0925-4773(02)00355-6 |
| [10] |
IYER S, PENNISI DJ, PIPER M. Crim1-, a regulator of developmental organogenesis[J]. Histol Histopathol, 2016, 31(10): 1049-1057. DOI:10.14670/HH-11-766 |
| [11] |
IYER S, CHHABRA Y, HARVEY TJ, et al. CRIM1 is necessary for coronary vascular endothelial cell development and homeostasis[J]. J Mol Histol, 2017, 48(1): 53-61. DOI:10.1007/s10735-016-9702-3 |
| [12] |
ANDREWS MG, DEL CASTILLO LM, OCHOA-BOLTON E, et al. BMPs direct sensory interneuron identity in the developing spinal cord using signal-specific not morphogenic activities[J]. elife, 2017, 6: e30647. DOI:10.7554/eLife.30647 |
| [13] |
OGASAWARA N, KUDO T, SATO M, et al. Reduction of membrane protein CRIM1 decreases E-cadherin and increases claudin-1 and MMPs, enhancing the migration and invasion of renal carcinoma cells[J]. Biol Pharm Bull, 2018, 41(4): 604-611. DOI:10.1248/bpb.b17-00990 |
| [14] |
ZENG H, ZHANG Y, YI Q, et al. CRIM1, a newfound cancer-related player, regulates the adhesion and migration of lung cancer cells[J]. Growth Factors, 2015, 33(5/6): 384-392. DOI:10.3109/08977194.2015.1119132 |
| [15] |
PRENKERT M, UGGLA B, TIDEFELT U, et al. CRIM1 is expressed at higher levels in drug-resistant than in drug-sensitive myeloid leukemia HL60 cells[J]. Anticancer Res, 2010, 30(10): 4157-4161. |
 2020, Vol. 49
2020, Vol. 49




