文章信息
- 王子璐, 刘立民, 孙晓
- WANG Zilu, LIU Limin, SUN Xiao
- 有氧运动对人骨骼肌基因表达的影响
- Effect of aerobic exercise on gene expression in human skeletal muscle
- 中国医科大学学报, 2020, 49(6): 556-560
- Journal of China Medical University, 2020, 49(6): 556-560
-
文章历史
- 收稿日期:2019-11-26
- 网络出版时间:2020-05-19 9:12
2. 中国医科大学 附属盛京医院药学部, 沈阳 110004
2. Department of Pharmacy, Shengjing Hospital, China Medical University, Shenyang 110004, China
体育锻炼的缺乏是诱发慢性病的重要原因之一[1-2]。体育活动已被证明可以改善和调节代谢稳态代谢效率[3]。年轻人和老年人久坐的生活方式和低体力活动是健康的不良因素。虽然有关运动对机体的有益作用已经达成共识,但很少有研究关注于评估有氧运动与机体基因表达之间的关系及其促进健康、预防疾病的分子机制。本研究拟通过生物信息学方法分别分析年轻人和老年人2个年龄段中运动组和非运动组股外侧肌活检样本基因表达谱,旨在探讨有氧运动对人骨骼肌全基因组表达的影响及其可能的作用机制。
1 材料与方法 1.1 数据来源本研究从美国国立生物技术信息中心公共基因芯片数据平台(gene expression omnibus,GEO)数据库(https://www.ncbi.nlm.nih.gov/geo/) [4]下载GSE9103,该数据库为mRNA基因芯片数据集,芯片平台是GPL570 [HG-U133_Plus_2] Affymetrix Human Genome U133 Plus 2.0 Array。用该芯片对股外侧肌活检样本进行骨骼肌转录谱分析,共40例样本,其中包含10例年轻久坐者、10例老年久坐者、10例年轻训练者和10例老年训练者。
1.2 差异表达基因(differentially expressed genes,DEGs)筛选应用R语言对芯片进行分析,读取下载矩阵文件,使用limma包分别对年轻久坐组和年轻训练组,以及老年久坐组和老年训练组进行分析,直接获取DEGs,并对结果进行去重[5]。筛选标准[4]为:log2基因表达差异倍数(fold change,FC)绝对值≥1,P < 0.05。用“ggplot2”R语言包绘制火山图,同时根据差异表达数据,用“VennDiagram”R语言包绘制韦恩图。
1.3 DEGs的基因本体论(gene ontology,GO)及京都基因和基因组百科全书(Kyoto Encyclopedia of Genes and Genomes,KEGG)分析本研究应用在线数据库DAVID (https://david.ncifcrf.gov/),分析提供所需的GO功能富集数据,分别按照生物途径(biological process,BP)、细胞定位(cellular component,CC)、分子功能(molecularfunction,MF)对基因进行注释和分类。除此之外,采用KEGG分析,用以发现可能涉及的生物学通路[6]。使用EASE Score或Fisher Exact统计方法,GO和KEGG各项筛选条件为P < 0.05[7]。应用“ggplot2”R语言包根据分析结果绘制气泡图。
1.4 DEGs的蛋白质相互作用(protein-protein interaction,PPI)网络构建和关键基因筛选STRING (https://string-db.org/)是一款在线分析工具(当前版本11.0),用来呈现与评估PPI网络[8]。将本研究筛选出的所有DEGs导入STRING,通过STRING分析工具可以进一步探寻这些DEGs之间潜在的联系[9]。筛选条件设置为:置信度≥0.15,互作最大值= 0。之后把STRING的计算结果导入Cytoscape (版本3.7.1) [10],挖掘PPI网络中连接最为紧密的集簇[5],应用的是分子复合物检测(molecular complex detection,MCODE)插件,筛选设置参数设置为默认参数。此外应用cytoHubba插件筛选出PPI网络中处于关键位置的前10个基因。
2 结果 2.1 DEGs筛选结果基因表达芯片GSE9103应用R语言分析后,在年轻组和老年组分别筛选出25个DEGs,在年轻组中,相较于久坐组,训练组8个mRNA表达上调,17个mRNA表达下调(FC≥2,P < 0.05)。在老年组中,相较于久坐组,训练组10个mRNA表达上调,15个mRNA表达下调(FC≥2,P < 0.05)。利用火山图(图 1)分析和观察FC值2倍以上变化的mRNA的分布及统计学差异,FC值2倍以下或者P > 0.05的基因标注为灰色,表达下调的基因标注为蓝色,表达上调的基因标注为红色。将年轻组和老年组筛选出的DEGs绘制韦恩图,如图 2所示,共有9个基因为2组群体共有的DEGs,包括ACTN3、AFAP1L1、CALML6、CKB、G0S2、IGFN1、LDHB、PDLIM5和SLC38A1。
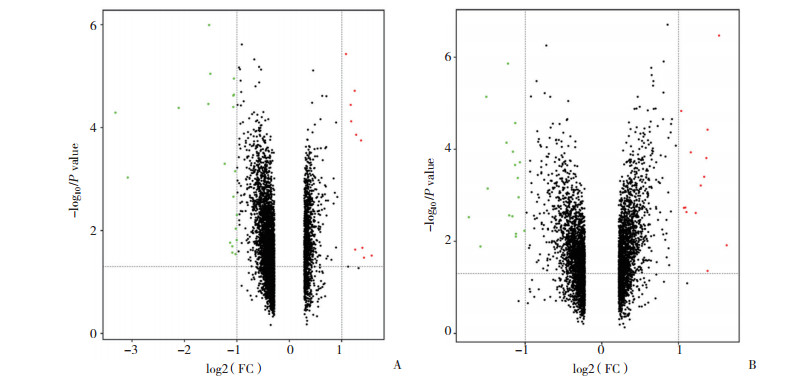
|
| A, young human subjects; B, old human subjects. 图 1 运动组和非运动组mRNA差异表达火山图分析 Fig.1 Differentially expressed genes between trained and sedentary subjects |
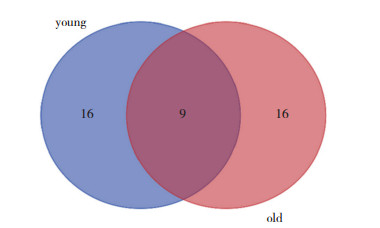
|
| 图 2 年轻组和老年组筛选出的差异表达基因韦恩图分析 Fig.2 The venn graph of DEGs in young and old human subjects |
2.2 DEGs的GO和KEGG分析结果
采用DAVID数据库对总的DEGs进行GO和KEGG信号通路富集分析后,结果如图 3所示,GO富集分析发现,DEGs主要定位在线粒体、细胞外和细胞质内,分子功能主要涉及细胞对饥饿的反应、乳酸代谢过程以及细胞生长调节等;KEGG分析显示,DEGs主要与胰高血糖素信号通路、细胞紧密连接和AMPK信号通路有关。
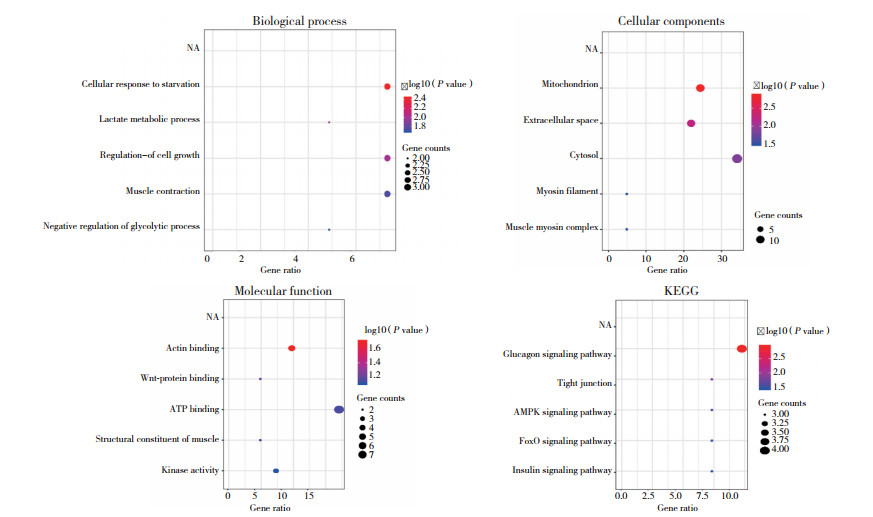
|
| 图 3 对差异表达的mRNA进行生物学过程、细胞组分分子功能的富集信息以及参与的细胞通路分析 Fig.3 Functional and signaling pathway analysis of DEGs, including biological process, cellular component, molecular function and KEGG pathway |
2.3 DEGs的PPI网络分析结果
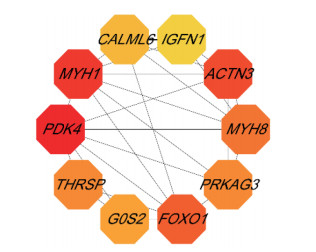
通过STRING得到的PPI网络由40个节点蛋白以及86条相互作用关系构成(enrichment P < 0.000 01),将PPI网络导入Cytoscape本地版,根据degree和log2FC大小2个因素美化PPI网络(图 4A)。通过MCODE插件分析发现1组集簇,此集簇包含9个节点(node)与16条连线(edge) (图 4B)。通过cytoHubba插件按照MCC为标准,最终筛选出处于关键位置的10个基因(图 5),分别为THRSP、MYH8、MYH1、PDK4、IGFN1、CALML6、PRKAG3、G0S2、ACTN3和FOXO1 10个关键基因。
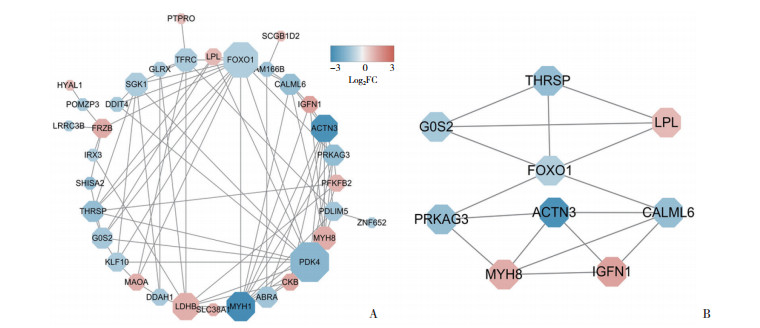
|
| A, PPI network construction:the size of node refers to the degree standard, and the color of node represents the values of log2FC; B, the clustering module is obtained by MCODE. 图 4 PPI网络分析结果和集簇模块构建 Fig.4 Protein-protein interaction network construction and module analysis |
|
| The color depth of nodes represents the importance in the network. 图 5 cytoHubba插件筛选出PPI网络中处于关键位置的前10个基因 Fig.5 The top 10 hub genes in the PPI network selected by cytoHubba |
3 讨论
研究[3]显示,身体长期或短期不活动都会对肌肉性能产生负面作用,并与胰岛素敏感性降低、有氧能力下降和基础代谢率降低有关。肌肉力量随着年龄的增长而下降,增加体育活动是目前已知的唯一能减轻这种下降趋势的方法[11]。有氧运动除了对机体健康有益外,还可以改善某些慢性疾病的症状,如体育活动对预防和(或)维持血糖控制及心血管危险因素有积极作用;在进行有氧运动之后,脑卒中和帕金森病患者除了在心肺健康方面有所改善外[12],在行走、功能和运动能力方面也有改善[13-14]。目前,有关有氧运动与机体基因表达之间的关系及其促进健康、预防疾病的分子机制的研究较少,本研究应用生物信息学方法分别对年轻人和老年人2个年龄段中运动组和非运动组股外侧肌活检样本基因表达谱进行了分析,筛选出的DEGs生物学功能主要涉及细胞对饥饿的反应、乳酸代谢过程、细胞生长调节,KEGG分析显示主要与胰高血糖素信号通路、细胞紧密连接和AMPK信号通路有关。
PPI网络筛选出THRSP、MYH8、MYH1、PDK4、IGFN1、CALML6、PRKAG3、G0S2、ACTN3和FOXO1这10个关键基因。其中,Thrsp被称为甲状腺激素应答点14蛋白,是脂质合成的重要调节基因,可被甲状腺激素和高糖饮食等刺激脂质合成的因子迅速上调,参与组织中的脂质合成调控[15];MYH8和MYH1编码肌球蛋白重链,参与肌肉的合成[16];PDK4增加胰岛素抵抗,升高血糖,减少非酯化脂肪酸水平,是线粒体能量代谢的调控因子[17];IGFN1参与成肌细胞融合和分化[18];CALML6的有关研究相对较少;G0S2为脂质代谢调控分子,PRKAG3是AMPK的调节亚基,在骨骼肌中有特异性表达,有研究[19-20]显示PRKAG3和G0S2均在AMPK影响葡萄糖或脂类相关代谢中发挥重要作用;ACTN3基因已经被证实为运动基因,编码一种α辅肌动蛋白,不仅与速度有关,而且也与耐力和力量有关,关于该基因的多态性的研究较多,其中某些基因型是成为优秀运动员的一种特殊标志[21];FoxO1作为Forkhead蛋白家族的成员,是调节细胞增殖、自噬、凋亡等作用的功能蛋白[22]。
以上的这些基因都不同程度地参与了运动对机体的调节作用,而本研究获得的结论尚需要更多的研究来进行验证,通过数据挖掘,探究有氧运动对机体作用的靶基因,可以为下一步的机制研究提供参考。
| [1] |
杨双燕, 周强. 久坐行为的流行病学研究与展望[J]. 中国运动医学杂志, 2016, 35(5): 500-505. DOI:10.16038/j.1000-6710.2016.05.017 |
| [2] |
周誉, 王正珍. 静坐少动与心血管风险因素[J]. 中国运动医学杂志, 2015, 34(8): 804-809. DOI:10.16038/j.1000-6710.2015.08.017 |
| [3] |
PALMNÄS MSA, KOPCIUK KA, SHAYKHUTDINOV RA, et al. Serum metabolomics of activity energy expenditure and its relation to metabolic syndrome and obesity[J]. Sci Rep, 2018, 8(1): 3308. DOI:10.1038/s41598-018-21585-6 |
| [4] |
柴毅, 谭峰, 樊巧玲. 用生物信息学分析预测绝经后骨质疏松症核心基因与互作miRNA的研究[J]. 中国骨质疏松杂志, 2018, 24(10): 1267-1272. DOI:10.3969/j.issn.1006-7108.2018.10.002 |
| [5] |
刘慧, 陆嘉骏, 樊璐, 等. 基于网络药理学和生物信息学的辛伐他汀分子生物学机制研究[J]. 中国临床药理学杂志, 2019, 35(14): 1510-1513. DOI:10.13699/j.cnki.1001-6821.2019.14.026 |
| [6] |
HUANG DW, SHERMAN BT, LEMPICKI RA. Systematic and integrative analysis of large gene lists using DAVID bioinformatics resources[J]. Nat Protoc, 2009, 4(1): 44-57. DOI:10.1038/nprot.2008.211 |
| [7] |
杨万霞, 潘云燕, 管沛文, 等. miR-129-5p调控的COL1A1作为胃癌潜在治疗靶点的生物信息学分析[J]. 南方医科大学学报, 2019, 39(5): 540-546. DOI:10.12122/j.issn.1673-4254.2019.05.07 |
| [8] |
SZKLARCZYK D, GABLE AL, LYON D, et al. STRING v11:protein-protein association networks with increased coverage, supporting functional discovery in genome-wide experimental datasets[J]. Nucleic Acids Res, 2019, 47(D1): D607-D613. DOI:10.1093/nar/gky1131 |
| [9] |
陈林波, 李先鹏, 姜昊, 等. 肝癌相关基因的生物信息学分析及功能预测[J]. 温州医科大学学报, 2018, 48(11): 828-832. DOI:10.3969/j.issn.2095-9400.2018.11.010 |
| [10] |
SHANNON P, MARKIEL A, OZIER O, et al. Cytoscape:a software environment for integrated models of biomolecular interaction networks[J]. Genome Res, 2003, 13(11): 2498-2504. DOI:10.1101/gr.1239303 |
| [11] |
UBAIDA-MOHIEN C, GONZALEZ-FREIRE M, LYASHKOV A, et al. Physical activity associated proteomics of skeletal muscle:being physically active in daily life may protect skeletal muscle from aging[J]. Front Physiol, 2019, 10: 312. DOI:10.3389/fphys.2019.00312 |
| [12] |
SAUNDERS DH, SANDERSON M, HAYES S, et al. Physical fitness training for stroke patients[J]. Cochrane Database Syst Rev, 2013, 10: CD003316. DOI:10.1002/14651858.cd003316.pub5 |
| [13] |
MACKAY CP, KUYS SS, BRAUER SG. The effect of aerobic exercise on brain-derived neurotrophic factor in people with neurological disorders:a systematic review and meta-analysis[J]. Neural Plast, 2017, 2017: 4716197. DOI:10.1155/2017/4716197 |
| [14] |
BANG DH, SON YL. Effect of intensive aerobic exercise on respiratory capacity and walking ability with chronic stroke patients:a randomized controlled pilot trial[J]. J Phys Ther Sci, 2016, 28(8): 2381-2384. DOI:10.1589/jpts.28.2381 |
| [15] |
吴静, 李晶, 门秀丽, 等. 甲状腺激素应答蛋白Thrsp在脂质合成中的调节作用[J]. 生理科学进展, 2012, 43(5): 393-397. DOI:10.3969/j.issn.0559-7765.2012.05.018 |
| [16] |
苏艳红, 周越, 王瑞元. 运动与肌球蛋白重链研究综述[J]. 天津体育学院学报, 2008, 23(4): 328-332. DOI:10.3969/j.issn.1005-0000.2008.04.013 |
| [17] |
杨蕊, 朱奕, 王颖, 等. 丙酮酸脱氢酶激酶PDK4在临床中的研究进展[J]. 中国医院药学杂志, 2019, 39(1): 101-105. DOI:10.13286/j.cnki.chinhosppharmacyj.2019.01.23 |
| [18] |
LI X, BAKER J, CRACKNELL T, et al. IGFN1_v1 is required for myoblast fusion and differentiation[J]. PLoS One, 2017, 12(6): e0180217. DOI:10.1371/journal.pone.0180217 |
| [19] |
RANDRIANARISOA E, LEHN-STEFAN A, KRIER J, et al. AMPK subunits harbor largely non-overlapping genetic determinants for body fat mass, glucose- and cholesterol metabolism[J]. J Clin Endocrinol Metab, 2020, 105(1): 14-25. DOI:10.1210/jcem/dgz020 |
| [20] |
马运芹, 张明亮, 陆俊茜, 等. 二甲双胍对非酒精性脂肪肝的缓解与AMPK-G0S2信号通路的关系[J]. 中国医药导刊, 2016, 18(4): 401-402. |
| [21] |
贾松竹, 李丽. ACTN3基因多态性与黑龙江省雪上项目运动员运动能力相关性分析[J]. 当代体育科技, 2018, 8(32): 219-220. DOI:10.16655/j.cnki.2095-2813.2018.32.219 |
| [22] |
ZHAO Y, YANG J, LIAO WJ, et al. Cytosolic FoxO1 is essential for the induction of autophagy and tumour suppressor activity[J]. Nat Cell Biol, 2010, 12(7): 665-675. DOI:10.1038/ncb2069 |
 2020, Vol. 49
2020, Vol. 49




