文章信息
- 贾茹, 孙媛媛, 富伟能
- JIA Ru, SUN Yuanyuan, FU Weineng
- 高迁移率族蛋白B-1通过细丝蛋白A影响喉癌细胞迁移
- Role of high mobility group box-1 in laryngeal cancer cell migration via filamin A
- 中国医科大学学报, 2020, 49(6): 490-494
- Journal of China Medical University, 2020, 49(6): 490-494
-
文章历史
- 收稿日期:2019-10-09
- 网络出版时间:2020-05-18 14:58
喉鳞状细胞癌(laryngeal squamous cell carcinoma,LSCC)是常见的喉癌[1],其细胞起源于喉上皮细胞。研究[1]显示LSCC的5年生存率仅为60%左右,主要死亡原因是转移和复发[2]。因此,研究喉癌细胞迁移、转移的分子机制具有重要的理论意义和潜在临床价值。高迁移率族蛋白B-1(high mobility group box-1,HMGB1)是核DNA结合蛋白,含有2个DNA结合区(boxA、boxB)和1个酸性C末端区[3]。研究[4]发现HMGB1促进细胞迁移和转移,说明其在肿瘤的发生发展过程中具有重要作用。有研究[5]证实乳腺癌中miR-200c可以通过调控HMGB1的表达来抑制乳腺癌细胞的迁移;在直肠癌中沉默HMGB1后肿瘤细胞迁移能力减弱[6],但其具体的分子机制尚不清楚。细丝蛋白A(filamin A,FLNA)是哺乳动物细胞内重要的细胞骨架蛋白[7]。有研究[8]证实FLNA在多种肿瘤细胞(乳腺癌等)中表达,但其在喉癌细胞系Hep2中的生物学功能尚不明确。研究[9]发现在FLNA启动子区存在2个HMGB1的潜在结合位点(5' -A/TAACAAT/A-3' )。因此推测HMGB1可能通过调控FLNA发挥癌基因作用。本研究应用分子生物学技术检测喉癌及癌旁组织中FLNA表达水平及其对喉癌Hep2细胞迁移能力的影响,初步探讨HMGB1基因在喉癌发生发展中的作用机制及生物学功能,为喉癌的分子诊治提供新线索。
1 材料与方法 1.1 材料 1.1.1 标本来源选取解放军第463医院耳鼻咽喉科30例喉癌患者术后组织标本,同时选取对应癌旁组织(距癌组织切缘 > 2 cm)作为对照组,选取的组织标本均经病理科确诊为LSCC,并具有完整临床样本信息,所有患者均知情同意。
1.1.2 细胞株及试剂人胚肾细胞系HEK293(中科院上海细胞库)、人喉癌Hep-2细胞(中科院上海细胞库)。胎牛血清(中国BI公司)、1640培养基(中国BI公司)、TRIzol(日本TaKaRa公司)、反转录试剂盒及PCR相关试剂(日本TaKaRa公司),RIPA裂解液(强)(碧云天生物技术公司)、BCA蛋白浓度测定试剂盒(碧云天生物技术公司)、Transwell小室(法国Coster公司)、结晶紫(碧云天生物技术公司)、Polyplus转染试剂(美国Afao公司)、FLNA蛋白抗体(武汉三鹰生物技术公司)、HMGB1-siRNA和FLNA-siRNA(上海吉凯公司)。
1.2 细胞转染提前24 h将处于对数生长期的Hep2细胞以2×105/mL密度铺于6孔板中。随机分为空白对照组(CON组)、阴性对照组(NC组)、HMGB1-siRNA组、FLNA-siRNA组以及HMGB1-siRNA+FLNA-siRNA组,然后将6孔板置于37 ℃、5% CO2的细胞培养箱中培养24 h,当细胞完全贴壁时,转染相关小RNA(NC、HMGB1、FLNA)和转染试剂。根据不同实验在不同时间点收集转染后的Hep2细胞。
1.3 cDNA合成和实时定量PCR使用PrimeSciptTM RT Master Mix Kit试剂盒将总RNA逆转录成cDNA,以cDNA为模板,采用TaKaRaSYBR Premix Ex TaqTMⅡ说明进行PCR扩增。引物序列:FLNA,上游引物5’-GACAGGTCGCTACACCATCC-3’,下游引物5’-CTAGCCCGTGACCTCCGATT-3’;GAPDH,上游引物5’-TAACATCAAATGGGGTGAGG-3’,下游引物5’-GGTTCACACCCATCACAAAC-3’。以GAPDH为内对照,反应结果用BIO-RAD CFX Connect Real-Time System软件分析,并计算每个反应管内的荧光信号到达设定的域值时所经历的循环数(cycle threshold,Ct)值。采用2-ΔΔCt计算喉癌及癌旁组织拷贝数的差异。
1.4 Western blotting检测收集转染48 h后NC组及HMGB1-siRNA组喉癌Hep2细胞,加RIPA裂解液冰浴30 min,提取喉癌细胞总蛋白并采用BCA法测定蛋白浓度,检测蛋白浓度后,将其调整至相同浓度,每孔加入35 μg蛋白上样。将蛋白SDS-PAGE电泳分离1.5 h、恒流300 mA电转2 h、5%脱脂牛奶封闭2 h、用TBST洗涤3次后,放入1:1 000稀释的FLNA一抗中摇床上4 ℃杂交过夜、次日取出后,用TBST洗涤3次后放入二抗中,37 ℃温箱中孵育1 h、用TBST洗涤3次后采用ECL化学发光法显影。BioRad图像分析系统测定条带吸光度,目标蛋白的相对表达量=目标蛋白吸光度值/GAPDH蛋白吸光度值。
1.5 细胞划痕实验收集各转染组48 h细胞,胰酶消化后计数,以1×104/mL密度接种于6孔板,接种24 h后,在6孔板中划痕,PBS冲洗3次,更换无血清培养基,分别于划痕后0、24、48 h观察细胞的迁移情况并拍照记录,实验重复3次。
1.6 Transwell实验转染细胞24 h后用胰蛋白酶消化细胞,以细胞密度3×104/孔接种于Transwell小室上层中(加入终体积200 μL双无1640培养基),小室下层加入600 μL10%胎牛血清的1640培养基。培养24 h后,依次进行PBS冲洗、4%多聚甲醛固定30 min、结晶紫染色30 min、PBS冲洗后用小刀将滤膜切下、中性树脂封片、显微镜计数上、下、左、右、中5个视野的穿膜细胞数并拍照。
1.7 统计学分析采用SPSS 23.0软件进行统计学分析,数据均以x±s表示,2组比较采用独立样本t检验,P < 0.05为差异有统计学意义。
2 结果 2.1 FLNA在喉癌组织及喉癌Hep2细胞中低表达应用实时PCR检测FLNA基因在30例喉癌组织及对应癌旁组织中的表达水平。结果显示,FLNA在11例(36.7%)癌组织中高表达。30例喉癌患者组织中FLNA mRNA表达量(0.632±0.057)显著低于癌旁组织(1.000±0.0,P < 0.01);应用实时PCR方法比较人胚肾细胞HEK293和喉癌Hep2细胞中FLNA表达水平,结果显示FLNA在HEK293细胞中表达水平(1.000±0.0)高于喉癌Hep2细胞(0.512±0.094,P < 0.05)。提示FLNA在喉癌中发挥抑癌基因作用。
2.2 HMGB1-siRNA抑制喉癌Hep2细胞HMGB1表达转染siHMGB1至喉癌Hep2细胞48 h后,应用实时PCR检测HMGB1表达。结果显示,转染HMGB1-siRNA后,Hep2细胞中HMGB1 mRNA表达(0.210 2±0.088 47)显著低于对照组(1.000 0±0.0,P < 0.001),说明HMGB1-siRNA能有效抑制喉癌Hep2细胞HMGB1的表达,证明转染有效。
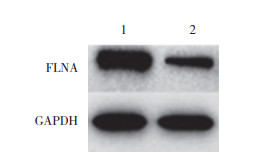
2.3 HMGB1基因沉默上调喉癌Hep2细胞中FLNA表达转染了HMGB1-siRNA后,应用Western blotting和实时PCR检测Hep2细胞中FLNA表达水平。结果显示,HMGB1-siRNA组中FLNA mRNA和蛋白水平均显著高于对照组(P < 0.05),见表 1、图 1。提示HMGB1基因沉默上调喉癌Hep2细胞中FLNA的表达,HMGB1负性调控FLNA。
| Group | FLNA mRNA | FLNA protein |
| NC | 1.000±0.0 | 0.407 0±0.105 50 |
| HMGB1-siRNA | 1.886±0.197 511) | 0.734 2±0.043 041) |
| 1)P < 0.05 vs NC group. NC,normal control. | ||
|
| 1, HMGB1-siRNA group; 2, NC group. 图 1 HMGB1基因沉默上调喉癌Hep2细胞中FLNA表达 Fig.1 HMGB1 silencing upregulated FLNA expression in laryngeal cancer Hep2 cells |
2.4 HMGB1通过FLNA促进喉癌Hep2细胞迁移(表 2、图 2)
| Group | Migration distance(μm) | Number of cell migrate per field |
| CON | 264.3±12.99 | 115.3±5.364 |
| NC | 269.0±17.39 | 105.7±5.548 |
| HMGB1-siRNA | 103.3±28.111) | 32.7±2.8482) |
| FLNA-siRNA | 377.3±11.571) | 211.3±11.0501) |
| HMGB1-siRNA+ FLNA-siRNA | 196.7±11.843) | 83.3±3.5283) |
| 1)P < 0.01,2)P < 0.001,3)P < 0.05 vs NC group. | ||

|
| A, wound-healing assay results (×50);B, transwell assay results (×200). 图 2 HMGB1 FLNA促进喉癌Hep2细胞迁移 Fig.2 HMGB1 promotes the migration of laryngeal cancer Hep2 cells by FLNA |
将FLNA-siRNA以及HMGB1-siRNA+FLNA-siRNA转染喉癌Hep2细胞48 h后,应用实时PCR检测FLNA mRNA表达水平。结果显示,与FLNA-siRNA组(0.188 8±0.066 31)比较,HMGB1-siRNA+FLNA-siRNA组(0.511 9±0.094 03)FLNA mRNA表达升高(P < 0.001),证明转染有效。同时,HMGB1-siRNA、FLNA-siRNA以及HMGB1-siRNA+FLNA-siRNA共转染喉癌Hep2细胞48 h后,采用划痕以及Transwell实验检测细胞迁移能力。结果显示,转染HMGB1-siRNA组迁移距离和数量明显低于NC组及对照组(P < 0.01),转染FLNA-siRNA组迁移距离和数量明显高于NC组及对照组(P < 0.01)。转染HMGB1-siRNA+FLNA-siRNA组与HMGB1-siRNA组相比,迁移能力明显升高(P < 0.05)。提示FLNA-siRNA可逆转HMGB1-siRNA对喉癌Hep2细胞迁移能力的促进作用,即FLNA介导HMGB1抑制喉癌Hep2细胞迁移。
2.5 喉癌组织中FLNA表达与临床病理特征的关系结果显示,FLNA表达与淋巴结转移、TNM分期和肿瘤分化程度密切相关(P < 0.05),而与患者年龄、性别等因素不相关(P > 0.05),见表 3。
| Clinicopathological characteristics | n | FLNA expression | t | P |
| Age(year) | 0.612 | 0.540 | ||
| ≥60 | 15 | 15.10±31.4 | ||
| <60 | 15 | 9.70±12.9 | ||
| Gender | 0.438 | 0.665 | ||
| Male | 25 | 12.30±24.8 | ||
| Female | 5 | 12.90±20.2 | ||
| T classification | 2.823 | 0.011 | ||
| T1+T2 | 20 | 18.20±27.5 | ||
| T3+T4 | 10 | 0.30±1.2 | ||
| Differentiation | 2.937 | 0.007 | ||
| Low and moderate | 23 | 1.07±1.4 | ||
| High | 7 | 9.57±14.2 | ||
| Lymphatic metastasis | 3.638 | 0.002 | ||
| Yes | 8 | 1.19±1.5 | ||
| No | 22 | 16.50±26.7 |
3 讨论
FLNA编码产物为一种细丝蛋白,是一种脚手架蛋白,可与肌动蛋白细丝交联,引导其与细胞膜上糖蛋白结合,引起细胞形状改变和迁移,在维持并稳定细胞骨架、细胞形态、细胞运动和信号转导等生物学行为中发挥重要作用[10]。
HMGB蛋白家族包含HMGB1、HMGB2和HMGB3 3种亚型蛋白,HMGB1是HMGB家族的重要成员[11]。本研究通过实时PCR检测喉癌组织及癌旁组织中FLNA基因的表达,发现喉癌中FLNA基因表达水平显著低于癌旁组织,且与喉癌患者淋巴结转移、临床分期以及肿瘤分化程度有关。提示FLNA在喉癌中可能发挥抑癌基因作用。研究发现HMGB1在FLNA启动子区存在潜在结合位点[9],本研究通过转染HMGB1特异性siRNA,应用实时定量PCR及Western blotting检测结果表明喉癌Hep2细胞中HMGB1沉默后FLNA表达上调,提示FLNA是HMGB1下游靶基因。
基于HMGB1在肿瘤中的重要作用,推测FLNA也可能对肿瘤细胞的生物学特性有影响。因此本研究采用划痕、Transwell实验分别检测了HMGB1、FLNA对喉癌Hep2细胞迁移和迁移的影响。结果显示,下调HMGB1表达后细胞迁移能力降低,且这种影响可被FLNA回复,提示HMGB1促进喉癌细胞迁移是通过调控FLNA实现的。FLNA作为一种新型肿瘤标志物,研究[12]已发现在鼻咽癌组织中的表达显著降低,且与TNM分期、淋巴结转移、临床分期、组织学分级显著相关。然而FLNA在调控喉癌细胞迁移的具体机制尚不清楚,需要进一步研究论证。
综上所述,HMGB1通过抑制FLNA的表达来促进喉癌细胞迁移。本研究以喉癌Hep2细胞为研究对象,首次报道了FLNA对喉癌迁移的影响及其初步机制,为喉癌分子靶向治疗的研究提供了新线索。
| [1] |
MARIONI G, MARCHESERAGONA R, CARTEI G, et al. Current opinion in diagnosis and treatment of laryngeal carcinoma[J]. Cancer Treat Rev, 2006, 32(7): 504-515. DOI:10.1016/j.ctrv.2006.07.002 |
| [2] |
BRITT CJ, GOURIN CG. Contemporary management of advanced laryngeal cancer[J]. Laryngoscope Investig Otolaryngol, 2017, 2(5): 307-309. DOI:10.1002/lio2.85 |
| [3] |
GOODWIN GH, SANDERS C, JOHNS EW. A new group of chromatin-associated proteins with a high content of acidic and basic amino acids[J]. Eur J Biochem, 1973, 38(1): 14-19. DOI:10.1111/j.1432-1033.1973.tb03026.x |
| [4] |
TRIPATHI A, SHRINET K, KUMAR A. HMGB1 protein as a novel target for cancer[J]. Toxicol Rep, 2019, 6: 253-261. DOI:10.1016/j.toxrep.2019.03.002 |
| [5] |
CHANG BP, WANG DS, XING JW, et al. MiR-200c inhibits metastasis of breast cancer cells by targeting HMGB1[J]. Huazhong Keji Daxue Xuebao Yixue Yingdewen Ban, 2014, 34(2): 201-206. DOI:10.1007/s11596-014-1259-3 |
| [6] |
李增军, 王海鹏, 宋宝, 等. RNA干扰沉默高迁移率族蛋白B1的表达对结直肠癌LoVo细胞增殖和侵袭的抑制作用[J]. 中华肿瘤杂志, 2015, 37(9): 664-670. DOI:10.3760/cma.j.issn.0253-3766.2015.09.008 |
| [7] |
NAKAMURA F, STOSSEL TP, HARTWIG JH. The filamins:organizers of cell structure and function[J]. Cell Adh Migr, 2011, 5(2): 160-169. DOI:10.4161/cam.5.2.14401 |
| [8] |
SAVOY RM, GHOSH PM. The dual role of filamin A in cancer:can't live with (too much of) it, can't live without it[J]. Endocr Relat Cancer, 2013, 20(6): R341-R356. DOI:10.1530/ERC-13-0364 |
| [9] |
HARLEY VR, LOVELL-BADGE R, GOODFELLOW PN. Definition of a consensus DNA binding site for SRY[J]. Nucleic Acids Res, 1994, 22(8): 1500-1501. DOI:10.1093/nar/22.8.1500 |
| [10] |
SHAO QQ, ZHANG TP, ZHAO WJ, et al. Filamin A:insights into its exact role in cancers[J]. Pathol Oncol Res, 2016, 22(2): 245-252. DOI:10.1007/s12253-015-9980-1 |
| [11] |
SOHUN M, SHEN HL. The implication and potential applications of high-mobility group box 1 protein in breast cancer[J]. Ann Transl Med, 2016, 4(11): 217. DOI:10.21037/atm.2016.05.36 |
| [12] |
SUN GG, LU YF, CHENG YJ, et al. Absent expression of FLNA is correlated with poor prognosis of nasopharyngeal cancer[J]. Tumour Biol, 2014, 35(4): 2967-2974. DOI:10.1007/s13277-013-1381-z |
 2020, Vol. 49
2020, Vol. 49




