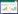文章信息
- 朱连成, 郭骞, 勾睿, 刘娟娟, 刘晴, 林蓓
- ZHU Liancheng, GUO Qian, GOU Rui, LIU Juanjuan, LIU Qing, LIN Bei
- 转染人附睾蛋白4基因对人卵巢癌细胞基因表达的影响
- Impact of transfection of the gene encoding human epididymis protein 4 on gene expression in ovarian cancer cells
- 中国医科大学学报, 2020, 49(5): 385-390, 395
- Journal of China Medical University, 2020, 49(5): 385-390, 395
-
文章历史
- 收稿日期:2019-09-10
- 网络出版时间:2020-05-06 16:55
卵巢癌作为女性生殖系统常见的恶性肿瘤,其死亡率居女性恶性肿瘤之首,大部分患者发现时已经是晚期[1],其原因包括早期转移、早期诊断困难及化疗耐药等[2]。人附睾蛋白4(human epididymis protein 4,HE4),又名乳清酸性蛋白4-二硫键核心结构域2(WAP four-disulfide core domain protein 2,WFDC2),是通过基因组及蛋白质组学筛选获得的卵巢癌肿瘤标志物,2008年被美国FDA指定为监测上皮性卵巢癌复发的血清标志物,其敏感性、特异性较高,近年来引起研究者广泛关注[3-5],然而大部分研究集中于早期诊断、判断预后等临床方面,关于其对卵巢癌恶性生物学行为影响的机制研究较少[6-9]。因此,本研究采用了基因芯片技术对转染了HE4基因的卵巢癌细胞系及其对应的空质粒转染组细胞进行基因表达序列分析,找到差异表达基因并对其进行基因和蛋白水平的验证,本研究采用基因本体(gene ontology,GO)及京都基因与基因组百科全书(Kyoto Encyclopedia of Genes and Genomes,KEGG)通路富集分析,初步探讨与HE4相关的分子机制,以期为卵巢癌中HE4相关的机制研究提供理论依据。
1 材料与方法 1.1 细胞培养、质粒构建及HE4基因转染人类上皮性卵巢癌细胞系ES-2购自美国ATCC,培养于含10%胎牛血清和1%盘尼西林/链霉素的RPMI-1640培养基,37℃恒温,5% CO2加湿。全长人类HE4 cDNA扩增pEGFP-N1或者pCMV6质粒,引物如下:P1,5’-TCCGCTCGAGATGCCTGCTTGTCGCCTAG-3’;P2,5’-ATGGGGTACCGTGAAATTGGGAGTGACACAGG-3’。转染采用脂质体转染试剂盒。细胞系分别标记为HE4高表达(HE4-H,O)和其对照空质粒转染细胞(HE4-H-Mock,OV),见表 1。
| Sample ID | label | OD260/280≥1.8 | OD260/230≥1.5 | Total amount | result |
| HE4-H | O | 2.02 | 2.21 | 16.10 | Pass |
| HE4-H-mock | OV | 2.02 | 2.35 | 12.10 | Pass |
1.2 实时PCR验证
TRIzol法分别提取各组细胞总RNA,SuperScript Ⅲ反转录成cDNA。使用Roche LightCycler480进行实时PCR反应。HE4引物及内参GAPDH引物见表 2。按照试剂说明和仪器操作规程将样品总RNA逆转录为cDNA,于Roche LightCycler 480进行实时PCR反应,利用仪器所匹配的软件计算每个基因的Ct值,常规采用ΔΔCt方法比较各自基因的表达量变化,并与基因芯片结果进行对比。反应至少进行3次。
| Gene | Primer sequence(5’-3’) | Product(bp) |
| HE4 | Sense: 5’-AGTGTCCTGGCCAGATGAAAT G | |
| Anti-sense: CAGGTGGGCTGGAACCAGAT | 147 | |
| FOXA2 | Sense: ATCCGCCACTCGCTCTCCTTC | |
| Anti-sense: GGTAGCAGCCGTTCTCGAACATG | 127 | |
| SERPIND1 | Sense: GGAGGAGGACGACGACTATCTGG | |
| Anti-sense: AGCGAACTTGGCGTTGAGGATG | 184 | |
| GAPDH | Sense: ACAACTTTGGTATCGTGGAAGG | |
| Anti-sense: GCCATCACGCCACAGTTTC | 101 |
1.3 总RNA提取及质控
每组细胞系采用2个样品进行准备,使用RNeasy Mini kit试剂盒提取总RNA,并以RNeasy MinElute Cleanup columns进一步纯化。使用NanoDrop DN-1000
对RNA质量及纯度进行评估,A260/A280≥1.8且A260/A230≥1.5作为标准。总RNA质量采用变性琼脂糖凝胶电泳检测。2组标本的RNA标记及检测数值见表 1。
1.4 基因芯片杂交、数据收集本研究中,每组标本采用3张芯片进行分析以充分降低误差。样品使用Phalanx杂交系统试剂盒进行扩增和标记,采用Human Whole Genome OneArray®(HOA6.1)芯片进行杂交。该芯片有30 275个DNA寡核苷酸探针,其中29 187个在RefSeq v38和Ensembl v56数据库中索引,含有1 088个质控探针。反应过程如下:50 ℃杂交16 h,3步法洗脱非特异性结合(步骤1,42 ℃洗脱5 min 1次;步骤2,42 ℃洗脱5 min 1次,25 ℃洗脱5 min;步骤3,冲洗20次),芯片离心后烘干并使用Axon4000B对芯片的信号强度数据进行扫描提取。
1.5 实时PCR验证芯片结果为了后续研究的持续性,在差异表达基因中挑选了上调的2个基因(FOXA2和SERPIND1),在2组细胞系中进行实时PCR分析,从而在RNA水平验证芯片结果。以GAPDH作为内参,引物由Invitrogen公司合成,方法同上。引物序列及产物见表 2。
1.6 免疫组化验证芯片结果选取经过实时PCR验证过的基因SERPIND1,同HE4一起,进行免疫组化验证其在卵巢癌组织中的表达。标本来源于2004年到2012年由中国医科大学附属盛京医院妇科收集的50例卵巢癌组织。按照常规SP方法对所有石蜡切片进行HE4及SERPIND1免疫组化染色,二者的一抗浓度为分别为1:100和1:400,肝癌组织作为阳性对照,结果判定标准为定位于胞质或胞膜中的棕黄色颗粒为阳性染色。在400倍光学显微镜下选择具有代表性的5个高倍镜视野,评分标准如下:染色程度0分,基本不着色;1分,淡黄色;2分,棕黄色;3分,棕褐色。着色细胞占计数细胞的百分率:0分,0%~5%;1分,> 5%~25%;2分,> 25%~50%;3分,> 50%~75%;4分,> 75%~100%。最后得分为两者的乘积:0~2分标记为-,3~4分标记为+,5~8分标记为++,9~12分标记为+++。-及+判定为低表达,++及+++判定为高表达。2位独立阅片者分别对标本进行评判。
1.7 统计学分析采用GenePix4.1软件对基因芯片采集的所有数据进行分析,筛选差异表达基因的条件是log2∣倍数变化∣≥1.5同时P < 0.05。对差异表达基因进行GO及KEGG信号通路等富集分析。应用SPSS 20.0进行独立样本t检验和χ2检验,相关性分析采用Spearman方法,应用GraphPad Prism进行制图。P < 0.05为差异有统计学意义。
2 结果 2.1 转染细胞验证及总RNA质量检验结果分别对3组细胞进行HE4的mRNA表达含量测定,发现HE4在HE4转染组细胞中的表达明显高于空质粒转染组细胞(2.403±0.473 vs 1.128±0.102,P < 0.001)及原始未处理组细胞(2.403±0.473 vs 1.112±0.099,P < 0.001),而后2组细胞的HE4表达水平差异无统计学意义(P=0.909)。将HE4转染组及空质粒转染组标本的RNA进行质量检测,发现2组的A260/A280比率为2.02,A260/A230比值为2.21~2.35(表 1),琼脂糖凝胶电泳检测可见RNA条带清晰,无向低分子量区域的成片拖带现象,说明2组样品总RNA完整,无降解(图 1),样品RNA的质量满足基因芯片杂交检验的质控要求。
|
| 图 1 转染细胞的总RNA凝胶电泳 Fig.1 Gel electrophoresis for total RNA of ovarian cancer cells |
2.2 芯片杂交结果
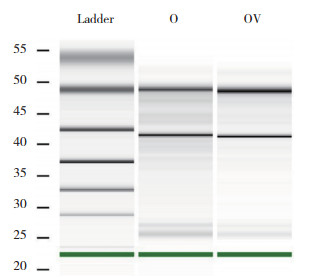
对所有采集的信号统一归一化log10计算,而后进行散点图分析以评估实验芯片的变异性,同时进行火山图分析以显示差异表达基因。结果显示,样品总体的数据变异性较小(图 2A),倍数变化存在显著的差异性,可挑选出差异表达基因(图 2B),其中356个基因表达下调,283个基因表达上调,数据可靠。按照倍数变化由高到低排序,列出2组10个代表性差异表达基因,见表 3。
|
| A, scatter plot; B, volcano plot. 图 2 数据归一化处理后的散点图及火山图 Fig.2 Scatter plot and volcano plot after normalization |
| Gene symbol | Description | Log2(fold change) | P |
| Up-genes | |||
| EPB41L3 | Erythrocyte membrane protein band 4.1-like 3 | 5.246 232 | < 0.001 |
| FOXA2 | Forkhead box A2 | 4.619 921 | < 0.001 |
| GDF15 | Growth differentiation factor 15 | 4.612 066 | < 0.001 |
| IL11 | Interleukin 11 | 3.887 501 | < 0.001 |
| EGR1 | Early growth response 1 | 3.217 277 | < 0.001 |
| DUSP5 | Dual specificity phosphatase 5 | 2.954 481 | < 0.001 |
| BCL2A1 | BCL2-related protein A1 | 2.911 151 | < 0.001 |
| HBEGF | Heparin-binding EGF-like growth factor | 2.719 461 | < 0.001 |
| IL12A | Interleukin 12A | 1.818 638 | < 0.001 |
| HIF1A | Hypoxia inducible factor 1,alpha subunit | 1.649 478 | < 0.001 |
| Down-genes | |||
| NNMT | Nicotinamide N-methyltransferase | -6.643 860 | < 0.001 |
| IGFBP7 | Insulin-like growth factor binding protein 7 | -4.130 080 | < 0.001 |
| HSPA1A | Heat shock 70kDa protein 1A | -4.109 380 | < 0.001 |
| ADGRE1 | Adhesion G protein-coupled receptor E1 | -4.046 958 | < 0.001 |
| SKP2 | S-phase kinase-associated protein 2 | -3.898 912 | < 0.001 |
| CD70 | CD70 molecule | -3.819 830 | < 0.001 |
| CFI | Complement factor Ⅰ | -3.766 450 | < 0.001 |
| IFITM1 | Interferon induced transmembrane protein 1 | -3.525 680 | < 0.001 |
| KIF20A | Kinesin family member 20A | -3.419 800 | < 0.001 |
| HSPA8 | Heat shock 70 kDa protein 8 | -1.947 670 | < 0.001 |
2.3 GO富集分析
GO富集分析结果显示,差异表达的基因在分子功能上,参与到了酶、转录因子及DNA结合;在细胞成分上,参与到了细胞核、胞质及胞外域作用;在生物过程上,参与到了生物高聚物代谢、程序性细胞死亡及凋亡等。见表 4。
| GO term | Genes in gene set | Genes in over-lap | P | FDR q-value |
| Molecular function | ||||
| Enzyme binding | 178 | 15 | < 0.001 | 2.62E-07 |
| Transcription factor binding | 307 | 19 | < 0.001 | 2.62E-07 |
| DNA binding | 602 | 26 | < 0.001 | 3.86E-07 |
| Transcription repressor activity | 152 | 12 | < 0.001 | 1.06E-05 |
| Transcription cofactor activity | 228 | 14 | < 0.001 | 1.71E-05 |
| Cellular component | ||||
| Nucleus | 1 430 | 54 | < 0.001 | 1.55E-12 |
| Cytoplasm | 2 131 | 62 | < 0.001 | 4.97E-10 |
| Extracellular region | 447 | 23 | < 0.001 | 7.04E-08 |
| Membrane | 1 994 | 53 | < 0.001 | 9.64E-08 |
| Extracellular region part | 338 | 19 | < 0.001 | 2.84E-07 |
| Biological process | ||||
| Biopolymer metabolic process | 1 684 | 63 | < 0.001 | 0.00E+00 |
| Programmed cell death | 432 | 31 | < 0.001 | 3.05E-14 |
| Apoptosis GO | 431 | 31 | < 0.001 | 3.05E-14 |
| Cell development | 577 | 34 | < 0.001 | 3.43E-13 |
| Nucleobase nucleoside nucleotide and nucleic acid metabolic process | 1 244 | 49 | < 0.001 | 1.26E-12 |
2.4 信号通路分析
对差异表达基因进行KEGG通路分析,发现MAPK、性激素生物合成、癌症、细胞周期、p53信号等通路显著富集,见表 5。
| Pathway | Genes in gene-set | Genes in over-lap | P | FDR q-value | Gene symbol |
| MAPK signaling pathway | 267 | 21 | < 0.001 | 7.46E-10 | TP53,FGF2,FGF1,FGF5,NFKB2,GADD45A,HSPA1A,RPS6KA1,IL1A,IL1R1,HSPA1B,HSPA2,HSPA8,DUSP1,MAP3K6,DDIT3,JUND,DUSP4,DUSP5,RELB,STMN1 |
| Steroid biosynthesis | 17 | 8 | < 0.001 | 7.46E-10 | CYP27B1,DHCR24,DHCR7,EBP,FDFT1,LSS,SQLE,TM7SF2 |
| Pathways in cancer | 328 | 21 | < 0.001 | 1.29E-08 | TP53,FGF2,FGF1,FGF5,NFKB2,CDKN1A,SKP2,CASP9,HIF1A,BMP2,BMP4,SMO,FZD4,FZD7,RAD51,PTGS2,LAMC2,RARB,DAPK2,MSH6,BCR |
| Cell cycle | 128 | 12 | < 0.001 | 1.58E-06 | TP53,GADD45A,CDKN1A,SKP2,PCNA,CDC25C,MCM2,MCM3,MCM5,CDKN2C,SMC1A,SMC3 |
| p53 signaling pathway | 69 | 8 | < 0.001 | 3.94E-05 | TP53,GADD45A,CDKN1A,CASP9,RRM2,GTSE1,PMAIP1,SESN2 |
2.5 实时PCR及免疫组织化学方法验证芯片结果
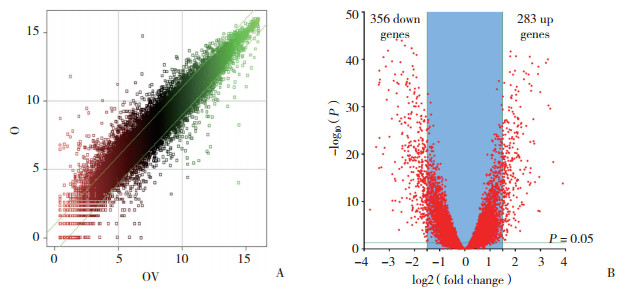
在差异表达基因中挑选2个表达上调的基因(FOXA2、SERPIND1)进行了实时PCR实验以验证芯片结果的可靠性,结果显示,基因FOXA2的mRNA相对表达在HE4转染组细胞中为4.712±1.504,明显高于空质粒转染组(1.177±0.264,P = 0.002)及原始未处理组(1.238±0.394,P = 0.002)的表达量,而后两组的表达量差异无统计学意义(P = 0.757);基因SERPIND1的mRNA相对表达在HE4转染组细胞中为1.703±0.487,明显高于空质粒转染组(0.956±0.246,P = 0.006)及原始未处理组(0.959±0.080,P = 0.006)的表达量,而后两组的表达量差异无统计学意义(P = 0.977)。实时PCR的结果与基因芯片显示的结果相一致。将经过实时PCR验证的SERPIND1通过免疫组化方法进行蛋白水平的验证。结果显示,类似于HE4,SERPIND1在细胞中的表达以胞质及胞膜中为主(图 3),HE4及SERPIND1在卵巢癌组织中的阳性表达率分别为78%及88%,高表达率分别为66%及62%。进一步分析发现,在50例标本中,HE4及SERPIND1同时阴性的有4例,同时阳性的有37例,Spearman相关性分析提示两者表达呈正相关(r = 0.402,P = 0.003)。以上通过基因及蛋白水平,验证了基因芯片结果的可靠性。见表 6。
|
| A, HE4;B, SERPIND1. Representative positive staining of HE4 and SERPIND1 in ovarian cancer samples by immunohistochemical staining.Scale bar= 50 μum. 图 3 免疫组化检查验证基因芯片结果 Fig.3 Immunohistochemical staining for validation of differentially expressed genes |
| HE4 | SERPIND1 | Total | |
| Negative | Positive | ||
| Negative | 4 | 7 | 11 |
| Positive | 2 | 37 | 39 |
| Total | 6 | 44 | 50 |
3 讨论
卵巢癌的发病率居于妇科恶性肿瘤第三位,每年可导致全球范围内15万患者死亡[1]。因此,找到能够早期诊断、监测预后的指标,对于提高卵巢癌患者的生存率有着重要作用。HE4在卵巢癌的早期诊断、预后评估等诸多方面引起大家的广泛关注[10-11],但关于它的基础研究甚少。本研究通过基因芯片分析了转染HE4基因后卵巢癌细胞基因表达的变化,这些差异基因的已知功能,可为卵巢癌中HE4的研究提供新思路。
SERPIND1基因也被称为肝素辅因子Ⅱ(heparin co-factorⅡ),属于丝氨酸蛋白酶抑制剂(Serpin)超家族成员[12],近年来发现许多Serpin家族的成员与肿瘤的发展有关,比如SERPINI2、SERPINB5、SERPINA9分别参与到了乳腺癌、前列腺癌和B细胞淋巴瘤等肿瘤的转移发展中[13-14]。SERPIND1的相互作用蛋白FGA、FGB、FGG与肺癌的上皮间质转化过程可能存在相关性,而其上游调控因子也可能参与卵巢癌的上皮间质转化[15]。在非小细胞肺癌中,SERPIND1可以通过PI3K途径促进肺癌细胞生成伪足,增强侵袭和迁移能力,它的过表达与更易复发及更短的生存时间相关[16]。本研究发现SERPIND1的基因及蛋白表达水平在卵巢癌中增高,且和HE4表达呈正相关,对该基因的进一步研究,有望为卵巢癌的发生发展机制、诊断标记物的研发等提供理论依据。
HE4可以促进卵巢癌细胞侵袭、转移、耐药[6, 17-18],但相关的作用机制仍存在争议。有研究[9]发现HE4可通过激活EGFR-MAPK信号通路促进卵巢癌细胞的黏附、迁徙和肿瘤生长,而其他研究者发现HE4通过抑制细胞增殖而在卵巢癌进展中起到保护性作用,推测该行为可能是通过激活MAPK和PI3K/Akt途径产生[19];HE4可与膜联蛋白A2形成复合体,通过MAPK和FOCAL信号通路促进肿瘤恶性进展[5, 7];HE4可以与EGFR,IGF1R和转录因子HIF1α相互作用,促进卵巢癌细胞的侵袭、耐药[17];HE4可激活AKT和Erk途径诱导卵巢癌细胞的耐药[4]。本研究找到了和HE4表达相关的通路,包括MAPK、性激素生物合成、细胞周期、p53等,首次在基因水平上为HE4相关的基础研究提供了切实可靠的参考。
综上,本研究通过基因芯片分析了转染HE4基因后卵巢癌细胞基因表达的变化,鉴别出了差异表达基因,其中,上调283个,下调356,并对结果进行基因及蛋白水平的验证,GO富集分析发现差异基因在生物过程中,参与到了生物高聚物代谢、程序性细胞死亡及凋亡等,KEGG通路分析发现差异基因参与到了MAPK、性激素生物合成、癌症、细胞周期、p53信号等。这些结果为卵巢癌中HE4的功能、机制和相互作用等基础研究提供了新的理念及参考。
| [1] |
WEBB PM, JORDAN SW. Epidemiology of epithelial ovarian cancer[J]. Best Pract Res Clin Obstet Gynaecol, 2017, 41: 3-14. DOI:10.1016/j.bpobgyn.2016.08.006 |
| [2] |
PLOTTI F, GUZZO F, SCHIRÒ T, et al. Role of human epididymis protein 4(HE4) in detecting recurrence in CA125 negative ovarian cancer patients[J]. Int J Gynecol Cancer, 2019, 211. DOI:10.1136/ijgc-2019-000211 |
| [3] |
ZHANG L, CHEN Y, WANG K. Comparison of CA125, HE4, and ROMA index for ovarian cancer diagnosis[J]. Curr Probl Cancer, 2019, 43(2): 135-144. DOI:10.1016/j.currproblcancer.2018.06.001 |
| [4] |
LEE S, CHOI S, LEE Y, et al. Role of human epididymis protein 4 in chemoresistance and prognosis of epithelial ovarian cancer[J]. J Obstet Gynaecol Res, 2017, 43(1): 220-227. DOI:10.1111/jog.13181 |
| [5] |
ZHU LC, ZHUANG HY, WANG HM, et al. Overexpression of HE4(human epididymis protein 4) enhances proliferation, invasion and metastasis of ovarian cancer[J]. Oncotarget, 2016, 7(1): 729-744. DOI:10.18632/oncotarget.6327 |
| [6] |
ZHU LC, GAO J, HU ZH, et al. Membranous expressions of Lewis y and CAM-DR-related markers are independent factors of chemotherapy resistance and poor prognosis in epithelial ovarian cancer[J]. Am J Cancer Res, 2015, 5(2): 830-843. |
| [7] |
ZHUANG HY, TAN MZ, LIU JJ, et al. Human epididymis protein 4 in association with AnnexinⅡpromotes invasion and metastasis of ovarian cancer cells[J]. Mol Cancer, 2014, 13: 243. DOI:10.1186/1476-4598-13-243 |
| [8] |
CHEN Y, MU X, WANG SH, et al. WAP four-disulfide core domain protein 2 mediates the proliferation of human ovarian cancer cells through the regulation of growth- and apoptosis-associated genes[J]. Oncol Rep, 2013, 29(1): 288-296. DOI:10.3892/or.2012.2114 |
| [9] |
LU RQ, SUN XH, XIAO R, et al. Human epididymis protein 4(HE4) plays a key role in ovarian cancer cell adhesion and motility[J]. Biochem Biophys Res Commun, 2012, 419(2): 274-280. DOI:10.1016/j.bbrc.2012.02.008 |
| [10] |
林贵玲. 卵巢癌化疗耐药标志物的研究进展[J]. 实用妇产科杂志, 2018, 34(12): 902-905. |
| [11] |
李芳, 吴素慧. HE4与妇科肿瘤相关性的研究进展[J]. 国际妇产科学杂志, 2017, 44(4): 369-373. DOI:10.3969/j.issn.1674-1870.2017.04.002 |
| [12] |
LIAO P, LI W, LIU RZ, et al. Genome-scale analysis identifies SERPINE1 and SPARC as diagnostic and prognostic biomarkers in gastric cancer[J]. Oncotargets Ther, 2018, 11: 6969-6980. DOI:10.2147/ott.s173934 |
| [13] |
HEIT C, JACKSON BC, MCANDREWS M, et al. Update of the human and mouse SERPIN gene superfamily[J]. Hum Genomics, 2013, 7: 22. DOI:10.1186/1479-7364-7-22 |
| [14] |
PATERSON MA, HOSKING PS, COUGHLIN PB. Expression of the serpin centerin defines a germinal center phenotype in B-cell lymphomas[J]. Am J Clin Pathol, 2008, 130(1): 117-126. DOI:10.1309/9QKE68QU7B825A3U |
| [15] |
WANG LL, XIU YL, CHEN X, et al. The transcription factor FOXA1 induces epithelial ovarian cancer tumorigenesis and progression[J]. Tumour Biol, 2017, 39(5): 1010428317706210. DOI:10.1177/1010428317706210 |
| [16] |
LIAO WY, HO CC, HOU HH, et al. Heparin co-factorⅡenhances cell motility and promotes metastasis in non-small cell lung cancer[J]. J Pathol, 2015, 235(1): 50-64. DOI:10.1002/path.4421 |
| [17] |
MOORE RG, HILL EK, HORAN T, et al. HE4(WFDC2) gene overexpression promotes ovarian tumor growth[J]. Sci Rep, 2014, 4: 3574. DOI:10.1038/srep03574 |
| [18] |
WANG H, ZHU LC, GAO J, et al. Promotive role of recombinant HE4 protein in proliferation and carboplatin resistance in ovarian cancer cells[J]. Oncol Rep, 2015, 33(1): 403-412. DOI:10.3892/or.2014.3549 |
| [19] |
KONG XL, CHANG XH, CHENG HY, et al. Human epididymis protein 4 inhibits proliferation of human ovarian cancer cells via the mitogen-activated protein kinase and phosphoinositide 3-kinase/AKT pathways[J]. Int J Gynecol Cancer, 2014, 24(3): 427-436. DOI:10.1097/IGC.0000000000000078 |
 2020, Vol. 49
2020, Vol. 49