文章信息
- 杨明丽, 黄哲, 王倩, 陈欢欢, 马赛男, 吴荣, 蔡炜嵩
- YANG Mingli, HUANG Zhe, WANG Qian, CHEN Huanhuan, MA Sainan, WU Rong, CAI Weisong
- 利用生物信息学分析筛选胰腺癌发生的潜在基因及机制
- Screening potential genes and mechanisms underlying pancreatic cancer using bioinformatics analysis
- 中国医科大学学报, 2020, 49(2): 134-138
- Journal of China Medical University, 2020, 49(2): 134-138
-
文章历史
- 收稿日期:2019-01-08
- 网络出版时间:2019-12-20 16:46
2. 中国医科大学附属盛京医院 普通外科, 沈阳 110015
2. Department of General Surgery, Shengjing Hospital, China Medical University, Shenyang 110015, China
胰腺癌是高度致命疾病,每5年就有43 090例患者死亡[1],5年生存率仅为6%[2]。造成胰腺癌生存率低的因素有多种,其中最重要的因素可能是超过一半患者是在胰腺癌晚期确诊的,而晚期胰腺癌的5年生存率仅为3%[3]。根据最新资料统计,至少有一半的胰腺癌患者无症状,直到疾病发展到晚期才出现症状[3]。因此许多患者错过了治疗的最佳时期。然而,胰腺癌的生物学特征不仅表现为早期复发和侵袭,还表现为化学抗性和放射抗性[4]。在过去的十余年中,大量研究已经发现了导致胰腺癌发生的较为敏感和有效的生物学标志物。MAZARICO等[5]研究发现CHKα可能是胰腺癌的治疗靶点,同时提出其可能是胰腺癌发生的新的关键基因。之前的研究[6]还发现SULF2表达是影响胰腺癌预后的独立危险因素,其可能是胰腺癌患者的治疗靶点。这些研究为胰腺癌发生和发展以及治疗的研究提供了良好的基础。但是之前的研究都是基于单个基因对于胰腺癌发生发展的影响,而癌症的发生是多基因多阶段的问题。因此关于胰腺癌发生的具体机制没有获得全面的阐释。
基因芯片可以从整个基因组水平的变化来观察胰腺癌发生机制,进而寻找其中的关键基因。本研究使用生物信息学方法分析GEO数据库[7]中胰腺癌的mRNA表达数据,以鉴定差异表达的基因,使用富集分析来观察差异表达基因的功能。还构建了蛋白质-蛋白质相互作用网络以鉴定与胰腺癌相关的核心基因,进而对核心基因进行预后分析,寻找影响胰腺癌发生和预后的关键基因。
1 材料与方法 1.1 芯片获取使用GEO数据库[7](https://www.ncbi.nlm.nih.gov/gds/)获得胰腺癌的表达谱芯片(GSE62452)。GSE62452是基于GPL6244构建的基因芯片,其中包括69个胰腺癌组织和61个癌旁非癌组织。
1.2 数据分析使用GEOquery(R语言中的模块)获得GSE52452的表达矩阵数据。使用R语言中的limma包对2组进行差异表达分析。在分析结果中,FDR < 0.05且|logFC| > 1作为差异表达基因。差异结果中不特异的探针以及重复的探针将被去掉。
1.3 差异基因富集分析使用Clusterprofiler(R语言的模块)对差异基因进行富集分析。GO分析[8]把差异表达基因富集到细胞成分、分析功能以及生物学过程3个方面。京都基因和基因组百科全书(Kyoto encyclopedia of genes and genomes,KEGG)是一个包含关于基因组、生物途径、疾病和化学物质信息的数据库集合。对差异表达基因进行分析后,选择P(矫正) < 0.05作为有差异的集合。
1.4 蛋白相互作用分析利用STRING数据库[9](https://string-db.org)寻找胰腺癌相关的差异表达基因的蛋白相互作用关系。综合得分 > 0.4作为相互作用存在的标准。通过拓扑网络可视化软件—cytoscope[10]对蛋白相互作用网络进行可视化,同时使用其中的插件—centiscape2.1对网络进行评分,筛选评分前十位的基因作为网络核心基因。
1.5 核心基因癌症基因组图谱(The Cancer Genome Atlas,TCGA)数据库预后分析TCGA数据库是美国国家人类基因组研究所(National Human Genome Research Institute,NHGRI)和美国国家癌症研究所(National Cancer Institute,NCI)之间的合作项目。通过测序,TCGA对33种肿瘤进行了基因组的分析。本研究利用TCGA数据库中的胰腺癌分组,对核心基因进行预后分析。其中P < 0.05表示预后存在意义。
2 结果 2.1 胰腺癌风险相关差异表达基因分析通过差异表达分析,最终得到了277个差异表达基因,其中98个基因在胰腺癌中低表达,179个基因在胰腺癌中高表达(图 1)。
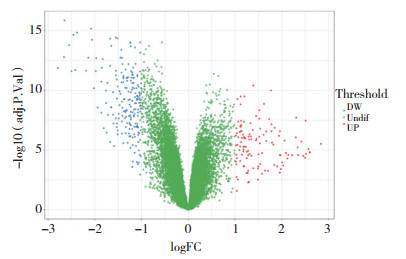
|
| 图 1 差异表达基因的火山图 Fig.1 Volcano plots of differentially expressed genes |
2.2 胰腺癌风险相关差异基因富集分析
结果显示,高表达基因与122个生物学过程、27个细胞组分以及25个分子功能有关。其中包括蛋白酶结合,生长因子结合以及消化系统过程等(图 2)。经过GO分析发现,低表达基因主要和26个集合有关,其中包括胆固醇代谢过程、酒精代谢过程以及消化在内9个生物学过程以及17个分子功能(图 2)。
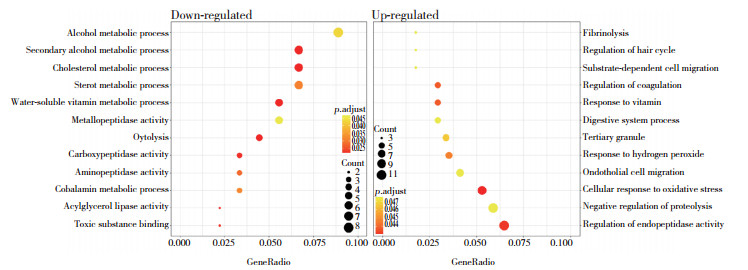
|
| 图 2 差异表达基因GO分析结果 Fig.2 GO analysis of differentially expressed genes |
经过通路分析发现,低表达基因主要和胰腺分泌物、脂肪消化吸收有关。而高表达基因则与PI3K-Akt信号通路及ECM-受体相互作用等有关。在通路分析中,高表达基因和低表达基因都与蛋白消化吸收有关(图 3)。
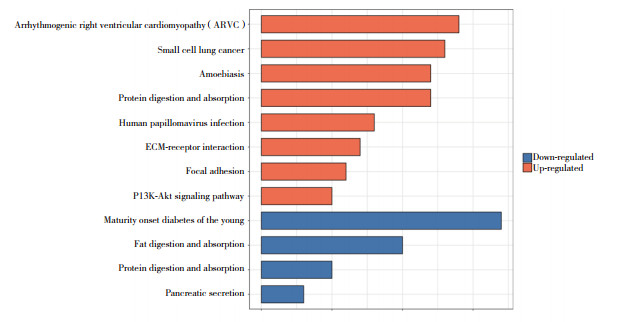
|
| 图 3 差异表达基因通路分析结果 Fig.3 Pathway analysis of differentially expressed genes |
2.3 风险相关基因蛋白相互作用分析
利用STRING数据库对差异表达基因进行蛋白相互作用分析,发现共有272个节点以及676条边。其中蛋白相互作用富集值< 1.0e-16。如图 4所示,在蛋白相互作用网络中寻找连接数前10位的作为核心基因。经过筛选发现,白蛋白(albumin,ALB)、表皮生长因子(epidermal growth factor,EGF)、纤连蛋白1(fibronectin 1,FN1)、胶原蛋白Ⅰ型Alpha 1链(collagen typeⅠalpha 1 chain,COL1A1)、胶原蛋白Ⅲ型Alpha 1链(collagen typeⅢalpha 1 chain,COL3A1)、整合素亚基Alpha 2(integrin subunit alpha 2,ITGA2)、胶原蛋白ⅩⅦ Alpha 1链(collagen typeⅩⅦ alpha 1 chain,COL17A1)、羧基酯脂肪酶(carboxyl ester lipase,CEL)、丝氨酸蛋白酶1(serine protease,PRSS1)以及DNA拓扑异构酶ⅡAlpha(DNA topoisomeraseⅡalpha,TOP2A)是胰腺癌风险相关蛋白相互作用网络中的核心基因。

|
| Blue circles represent low-expression genes, and orange circles represent high-expression genes. The more a gene is connected to other genes, the larger the circle. 图 4 胰腺癌风险相关的蛋白相互作用网络 Fig.4 Risk associated protein interaction network for pancreatic cancer |
2.4 核心基因预后分析
TCGA数据库包含了33种肿瘤的表达和临床数据。利用TCGA数据库进行预后分析探讨核心基因对于胰腺癌预后的影响发现,COL17A1、ITGA2、TOP2A基因影响胰腺癌的预后。见图 5。
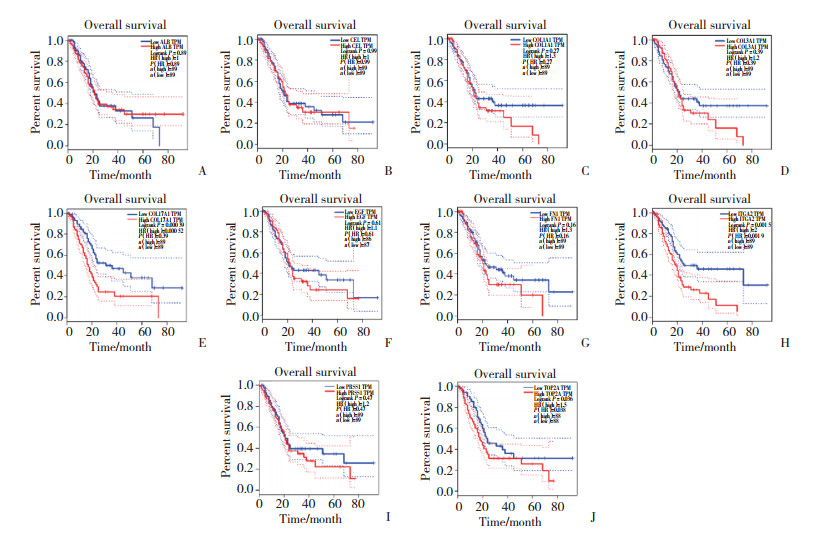
|
| A, ALB; B, CEL; C, COL1A1; D, COL3A1; E, COL17A1, F, EGF; G, FN1; H, ITGA2; I, PRSS1; J, TOP2A. 图 5 胰腺癌相关核心基因预后分析 Fig.5 Prognostic analysis for the core genes of pancreatic cancer |
3 讨论
在过去的研究中,胰腺癌的总体生存率和死亡率并没有显著的变化,这可能是由于缺乏可靠的生物学标志物来检测早期胰腺癌发生和预后有关。因此,在基因组学水平上全面评估胰腺癌发生的分子机制是相当必要的。随着芯片技术的发展,在基因组学水平宏观探讨胰腺癌发生的具体机制成为可能,进而可以寻找更多的基因靶标。本研究通过高通量方法对69个正常组织和61个癌组织进行了全基因组基因表达分析,以确定与胰腺癌发生相关的差异表达基因。通过差异表达分析得到了179个高表达基因,98个低表达基因。
GO分析结果发现,低表达基因主要与消化过程有关。胰腺的功能是参与消化,胰腺癌的发生会导致消化功能不全[11]。通路分析发现,低表达基因和胰腺分泌物有关,这与胰腺主要功能一致。另外,通过高表达基因研究发现,高表达基因与PI3K-Akt信号通路有关。张涓娟等[12]研究也发现PI3K-Akt信号通路可以影响胰腺癌的发生发展。另外高、低表达基因都与蛋白消化作用有关。胰腺的功能是分泌胰蛋白酶对物质进行消化,这与富集的结果一致。
本研究通过蛋白相互作用分析找到了10个核心基因:ALB、EGF、FN1、COL1A1、COL3A1、ITGA2、COL17A1、CEL、PRSS1、TOP2A。通过预后分析发现COL17A1、ITGA2、TOP2A基因既影响胰腺癌发生也影响胰腺癌的预后。TOP2A编码DNA拓扑异构酶,是一种在转录过程中控制和改变DNA拓扑状态的酶。这种核酶参与染色体浓缩、染色单体分离以及DNA转录和复制过程中发生的扭转应力缓解等过程。目前有研究[13]报道TOP2A通过β-catenin信号通路影响胰腺癌的发生。ITGA2是血小板和上皮细胞上重要的胶原蛋白受体,在正常上皮细胞中高表达。ITGA2的表达在正常细胞分化过程中受到调节,并在肿瘤发生过程中发生改变[14]。癌细胞中ITGA2的缺失与肝癌、结肠癌的转移行为有关[15-16]。然而胰腺癌癌细胞中ITGA2缺失的分子机制仍然未知。COL17A1编码ⅩⅦ型胶原的α链,与大多数胶原蛋白不同,胶原蛋白ⅩⅦ是跨膜蛋白,目前尚无此蛋白与胰腺癌相关的研究。
综上所述,本研究利用综合生物信息学方法分析了的参与胰腺癌发生的差异表达基因,找到了10个胰腺癌发病风险相关的核心基因和3个预后相关基因。本研究为探讨胰腺癌的分子机制提供依据,COL17A1、ITGA2、TOP2A等差异表达基因可作为胰腺癌治疗的潜在靶点。
| [1] |
SIEGEL RL, MILLER KD, JEMAL A. Cancer statistics, 2017[J]. CA:Cancer J Clin, 2017, 67(1): 7-30. DOI:10.3322/caac.21387 |
| [2] |
DESANTIS CE, LIN CC, MARIOTTO AB, et al. Cancer treatment and survivorship statistics, 2014[J]. CA Cancer J Clin, 2014, 64(4): 252-271. DOI:10.3322/caac.21235 |
| [3] |
ILIC M, ILIC I. Epidemiology of pancreatic cancer[J]. World J Gastroenterol, 2016, 22(44): 9694. DOI:10.3748/wjg.v22.i44.9694 |
| [4] |
ADAMSKA A, ELASKALANI O, EMMANOUILIDI A, et al. Molecular and cellular mechanisms of chemoresistance in pancreatic cancer[J]. Adv Biol Regul, 2018, 68: 77-87. DOI:10.1016/j.jbior.2017.11.007 |
| [5] |
MAZARICO JM, SÁNCHEZ-ARÉVALO LOBO VJ, FAVICCHIO R, et al. Choline kinase alpha(CHKα)as a therapeutic target in pancreatic ductal adenocarcinoma:expression, predictive value, and sensitivity to inhibitors[J]. Mol Cancer Ther, 2016, 15(2): 323-333. DOI:10.1158/1535-7163.MCT-15-0214 |
| [6] |
ALHASAN SF, HAUGK B, OGLE LF, et al. Sulfatase-2:a prognostic biomarker and candidate therapeutic target in patients with pancreatic ductal adenocarcinoma[J]. Br J Cancer, 2016, 115(7): 797-804. DOI:10.1038/bjc.2016.264 |
| [7] |
BARRETT T, WILHITE SE, LEDOUX P, et al. NCBI GEO:archive for functional genomics data sets:update[J]. Nucleic Acids Res, 2013, 41(Database issue): D991-D995. DOI:10.1093/nar/gks1193 |
| [8] |
GENE ONTOLOGY CONSORTIUM. The gene ontology(GO)project in 2006[J]. Nucleic Acids Res, 2006, 34(90001): D322-D326. DOI:10.1093/nar/gkj021 |
| [9] |
SZKLARCZYK D, FRANCESCHINI A, WYDER S, et al. STRING v10:protein-protein interaction networks, integrated over the tree of life[J]. Nucleic Acids Res, 2015, 43(Database issue): D447-D452. DOI:10.1093/nar/gku1003 |
| [10] |
SHANNON P, MARKIEL A, OZIER O, et al. Cytoscape:a software environment for integrated models of biomolecular interaction networks[J]. Genome Res, 2003, 13(11): 2498-2504. DOI:10.1101/gr.1239303 |
| [11] |
田明, 严俊, 胡进静, 等. 胰腺癌流行现状及危险因素综述[J]. 中国社会医学杂志, 2016, 33(2): 137-140. DOI:10.3969/j.issn.1673-5625.2016.02.012 |
| [12] |
张涓娟, 蒲宇, 李勇, 等. PI3K/AKT和MAPK/ERK1/2信号通路对胰腺癌PANC-1细胞VEGF表达的影响[J]. 川北医学院学报, 2014, 29(1): 44-48. DOI:10.3969/j.issn.1005-3697.2014.01.09 |
| [13] |
PEI YF, YIN XM, LIU XQ. TOP2A induces malignant character of pancreatic cancer through activating β-catenin signaling pathway[J]. Biochim Biophys Acta Mol Basis Dis, 2018, 1864(1): 197-207. DOI:10.1016/j.bbadis.2017.10.019 |
| [14] |
DING W, FAN XL, XU X, et al. Epigenetic silencing of ITGA2 by MiR-373 promotes cell migration in breast cancer[J]. PLoS One, 2015, 10(8): e0135128. DOI:10.1371/journal.pone.0135128 |
| [15] |
RAMIREZ NE, ZHANG ZH, MADAMANCHI A, et al. The α2β1 integrin is a metastasis suppressor in mouse models and human cancer[J]. J Clin Invest, 2011, 121(1): 226-237. DOI:10.1172/jci42328 |
| [16] |
SAWHNEY RS, COOKSON MM, OMAR Y, et al. Integrin alpha2-mediated ERK and calpain activation play a critical role in cell adhesion and motility via focal adhesion kinase signaling:identification of a novel signaling pathway[J]. J Biol Chem, 2006, 281(13): 8497-8510. DOI:10.1074/jbc.M600787200 |
 2020, Vol. 49
2020, Vol. 49




