文章信息
- 辛宁, 陈建康, 陈艳, 杨洁
- XIN Ning, CHEN Jiankang, CHEN Yan, YANG Jie
- 多芯片联合分析2型糖尿病发病相关基因及其与阿尔茨海默病的关系
- Multi-chip analysis of genes related to type 2 diabetes and their relationships with Alzheimer disease
- 中国医科大学学报, 2020, 49(12): 1106-1111, 1117
- Journal of China Medical University, 2020, 49(12): 1106-1111, 1117
-
文章历史
- 收稿日期:2020-04-30
- 网络出版时间:2020-12-03 12:19
根据国际糖尿病协会的报告,2017年全球有4.25亿糖尿病患者,其中2型糖尿病占90%以上。有研究[1]预测2045年糖尿病患者数量将增至6.29亿。糖尿病常导致全身血管及神经并发症,严重影响患者生活质量。2型糖尿病的发病机制尚不明确。因此,从基因水平阐明2型糖尿病的发病机制对于预防和治疗有重要意义。
随着高通量测序技术的发展,生物信息学分析成为深入了解2型糖尿病的一种新工具。基因表达汇编(gene expression omnibus,GEO)数据库存储许多表达谱芯片数据。有学者通过单个数据集对糖尿病患者和健康人群血液中的差异基因进行了研究[2-3],然而,国内尚未有使用R语言通过去除批次间差异的方式对单个数据集进行联合分析的报道。另外,糖尿病和阿尔茨海默病(Alzheimer disease,AD)关系密切[4],本研究将GEO数据库中2个人类2型糖尿病数据集进行联合分析,探索糖尿病发病的分子机制;同时利用基因集富集分析(gene-set enrichment analysis,GSEA)分析2型糖尿病关键基因与AD的关系。
1 材料与方法 1.1 数据来源GEO数据库(https://www.ncbi.nlm.nih.gov/geo/)下载芯片GSE85192(平台GPL16956)、GSE95849(平台GPL22448)、GSE97760(平台GPL16699)、GSE85426(平台GPL14500)。GSE95849中纳入12份样品,其中6份为2型糖尿病不伴糖尿病周围神经病变患者,6份为健康者。GSE85192中纳入18份样品,其中12份为治疗前的2型糖尿病患者,6份为健康者。GSE97760中纳入19份样品。其中10份非AD患者,9份为AD患者。GSE85426中纳入180份样品,其中90份为AD患者,90份为正常对照组。以上样本均来自外周血。下载芯片中的矩阵文件和平台文件。
1.2 方法 1.2.1 矩阵注释运用Perl软件(版本5.30.1)对GSE95849、GSE97760、GSE85426 3个数据集进行处理,利用平台文件中的基因名将原始矩阵文件ID重注释为基因名。对于GSE85192数据集,从Gencode数据库(ftp://ftp.ebi.ac.uk/pub/databases/gencode/Gencode_human/)下载人类转录本序列文件。使用BLAST软件将人类转录本序列与矩阵中探针核酸序列比对,得到含有基因名的重注释平台文件[5]。
1.2.2 多芯片矩阵合并及批次校正将矩阵探针表达量数据进行log2转化后,使用Perl软件将GSE95849、GSE85192 2个数据集的表达矩阵合并为1个矩阵。使用R软件(版本3.6.2)的sva包(sva包能使用对照探针消除批次间效应,还能直接构建新的替代变量用于分析,去除批间差[6])进行批次校正。
1.2.3 差异分析使用R软件的limma包分析矩阵中的差异基因,以基因表达量log2差异倍数(log2 fold change,logFC) > 0.5或者 < -0.5,并且校正后P值< 0.05为筛选参数,得到差异基因。用R软件绘制差异基因聚类热图。
1.2.4 加权基因共表达网络分析(weighted gene co-expression network analysis,WGCNA)利用R软件的WGCNA包,输入差异基因的表达矩阵,设置阈值为0.6,根据基因表达相似性,将基因划分成不同的模块,计算每个模块与2型糖尿病、年龄、性别3种临床性状的Pearson相关系数及P值,选取P < 0.01的模块内的基因用于后续分析。
1.2.5 基因功能注释和信号通路分析将与2型糖尿病相关模块中的基因输入DAVID6.8(https://david.ncifcrf.gov/)进行基因本体论(gene ontology,GO)功能分析,主要分析生物过程(biological process,BP)。再利用京都基因和基因组百科全书(Kyoto encyclopedia of genes and genomes,KEGG)工具对这些基因进行分析,以P < 0.05同时伴通路所含基因数 > 4个为筛选标准,按P值从小到大筛选前10条项目。
1.2.6 蛋白互作网络构建与模块分析使用STRING数据库(https://string-db.org/)分析差异基因编码蛋白的相互作用。再将得到的蛋白互作(protein-protein interaction,PPI)网络数据输入Cytoscape软件(版本3.7.2),使用cytoHubba插件进行分析。
1.2.7 GSEAToll样受体4(Toll-like receptor,TLR4)基因是关键基因,在GSE97760中AD患者和非AD患者间的表达量差异具有统计学意义(校正后P < 0.05),且在AD中高表达,因此对GSE97760矩阵中TLR4的表达量从小到大进行排序,以前10个为低表达组,后9个为高表达组,分析与TLR4高表达相关的信号通路。P < 0.05为差异有统计学意义。
1.2.8 TLR4表达量验证获得GSE85426中各样本中TLR4的表达量,对正常组和AD组的表达量进行t检验,用R语言绘制箱图使结果可视化。
2 结果 2.1 2型糖尿病患者差异表达的基因结果显示,与健康组比较,2型糖尿病组共获得626个差异表达基因,其中336个上调,290个下调。前100个(校正后P值从小到大排序)差异基因见图 1。

|
| Red represents high expression in the type 2 diabetes group, green represents low expression in the type 2 diabetes group, black represents the relative expression difference is not significant.DM, diabetes; NOR, healthy people. 图 1 差异表达基因分层聚类树形图和热图 Fig.1 Tree and heat map showing hierarchical clustering of differentially expressed genes |
2.2 WGCNA结果
将得到的差异基因输入WGCNA后,得到3个共表达基因模块,见图 2。结果显示,灰色和绿色模块内基因与2型糖尿病有关(P < 0.01);2个模块中共有567个基因。3个模块中基因与年龄、性别均无关(P > 0.01),见图 3。
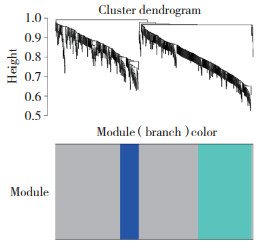
|
| 图 2 基因聚类树状图 Fig.2 Gene clustering dendrogram |
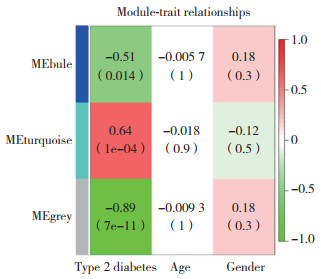
|
| The horizontal axis represents three clinical traits of type 2 diabetes, age and gender, and the vertical axis represents three gene modules:grey, blue and green.The value in parentheses is P value, and the number outside parentheses is Pearson correlation coefficient. 图 3 临床特征与模块特征相关性热图 Fig.3 Heat map showing the correlation between clinical features and module features |
2.3 GO分析结果
结果显示,差异基因主要参与的生物过程包括炎症反应,细胞对机械刺激的反应,Toll样受体(Toll-like receptor,TLR)信号通路,趋化因子、β干扰素产生的正向调节,组蛋白脱乙酰化等,见图 4。
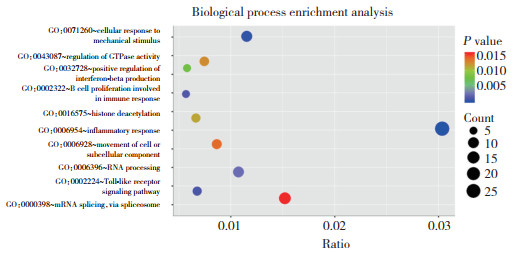
|
| Count,number of enriched genes;ratio,the ratio of enriched genes to all differential genes. 图 4 差异基因参与的生物学过程 Fig.4 Biological processes involved in differential genes |
2.4 KEGG分析结果
差异基因主要参与的KEGG信号通路有嘧啶代谢通路、TLR信号通路、疟疾、麻疹、RNA降解,见图 5。
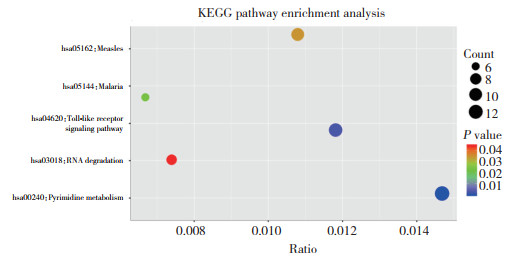
|
| Count, number of enriched genes; ratio, the ratio of enriched genes to all differential genes. 图 5 差异基因参与的KEGG通路 Fig.5 KEGG pathways involved in differential genes |
2.5 蛋白互作网络及模块分析结果
共有567个mRNA输入STRING数据库,以置信度得分 > 0.4为筛选参数,除外孤立的蛋白,共得到1 686对蛋白互作关系,470个蛋白。使用cytoHubba插件,按照度值和强度得分选取前10个基因,见表 1、2。之后取交集,最终得到的关键基因为ILF2、TLR4、POLR2G、MMP9。
| Order | Gene | Degree |
| 1 | SNRPE | 36 |
| 2 | MMP9 | 35 |
| 3 | HNRNPA1 | 35 |
| 4 | TLR4 | 33 |
| 5 | RPSA | 32 |
| 6 | POLR2G | 31 |
| 7 | RPS7 | 31 |
| 8 | ILF2 | 30 |
| 9 | HNRNPM | 30 |
| 10 | DDX17 | 29 |
| Order | Gene | Stress |
| 1 | MMP9 | 97 174 |
| 2 | ILF2 | 91 038 |
| 3 | POLR2G | 68 914 |
| 4 | CTPS1 | 64 604 |
| 5 | ITPKB | 63 520 |
| 6 | TLR4 | 61 560 |
| 7 | PSEN1 | 61 042 |
| 8 | ARG1 | 53 304 |
| 9 | CD40 | 50 318 |
| 10 | UBE2D3 | 48 378 |
2.6 GSEA分析结果
在GSE97760中,与TLR4高表达相关的KEGG通路富集于半胱氨酸和蛋氨酸代谢、基础转录因子、细胞周期、TLR信号通路、嘧啶代谢信号通路等(P < 0.05),见图 6A~6E。
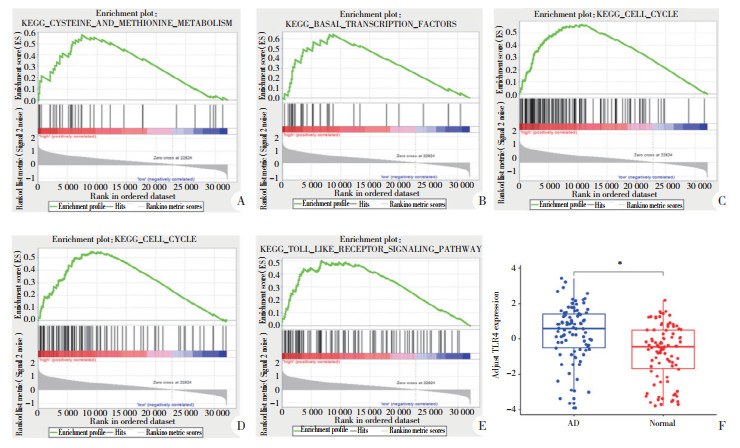
|
| A, cysteine and methionine metabolism; B, basal transcription factors; C, cell cycle; D, pyrimidine metabolism; E, toll-like receptor signaling pathway, F, adjust TLR4 expresson between AD group and normal group.*P < 0.01. 图 6 TLR4高表达有关的KEGG通路 Fig.6 Analysis of KEGG pathways related to high expression of TLR4 |
2.7 TLR4表达量验证
结果显示,在GSE85426中,正常人和AD患者外周血中的校正后TLR4水平差异有统计学意义(P < 0.01)。见图 6F。
3 讨论生物学过程显示,外周血中2型糖尿病患者与健康人差异基因的生物学功能富集在TLR信号通路、趋化因子产生的正向调节、β干扰素产生的正向调节、炎症反应,而且四者之间存在密切联系。TLR信号通路激活后,可以通过趋化因子和β干扰素产生2种途径诱导全身慢性炎症反应,进而引起胰岛素抵抗。研究[7]显示高脂饮食会导致肠道通透性增加,使脂多糖从肠道进入血液。脂多糖可以激活TLR4并与免疫细胞表面的CD14结合,触发多种细胞因子产生和趋化因子介导的炎症细胞募集,引起胰岛素抵抗,并导致低强度全身性炎症。炎性细胞因子还能使巨噬细胞在脂肪组织中募集,促进巨噬细胞释放促炎性细胞因子[8],从而干扰组织中的胰岛素信号,促进2型糖尿病发生[9]。还有学者[10-11]发现TLR3也能激活TLR信号通路,通过诱导β干扰素等激活Ⅰ型β干扰素,加速胰岛β细胞功能障碍和凋亡。动物实验[12]证实β干扰素突变小鼠就不会有TLR3介导的胰岛炎症反应。因此根据富集分析结果,可能存在“TLR-β干扰素/趋化因子-胰岛炎症反应轴”,这可能是导致2型糖尿病的重要原因之一。
KEGG通路显示,差异基因富集在嘧啶代谢、TLR信号通路、疟疾、麻疹、RNA降解上。有学者[13]通过动物实验对与2型糖尿病相关的诊断标志物进行分析发现,糖尿病大鼠体内核苷酸(包括嘧啶)水平较低,提示嘧啶代谢途径受到干扰。有学者[14]分析了糖尿病肾病大鼠和正常大鼠之间的代谢差异,发现差异富集在嘧啶代谢等代谢途径上,说明糖尿病肾病小鼠中存在嘧啶代谢紊乱。
本研究对关键基因的分析结果显示,TLR4、POLR2G、MMP9、ILF2可能是2型糖尿病发生的关键基因。糖尿病通过血管、葡萄糖代谢改变导致神经变性。AD通过下丘脑功能障碍、衰弱等影响全身葡萄糖代谢[15]。通过GSEA分析发现在AD患者中,TLR4升高也与嘧啶代谢和TLR信号通路有关,两者同时在2型糖尿病发生中起作用,可能是AD和2型糖尿病共同的发病机制。嘧啶是脑磷脂酰胆碱合成的原料。用尿嘧啶核苷源的饲料灌胃,可使沙土鼠脑磷脂酰胆碱增加,轴突神经突触膜的数量增加[16]。有学者[17]对AD模型小鼠海马进行分析,结果显示小鼠海马尿嘧啶明显增加,这可能是由于AD磷脂酰胆碱的合成减少而分解增多所致。AD可以表现为嘧啶代谢异常,但嘧啶代谢异常是否也是AD的病因尚待进一步验证。近年研究[18-20]发现,TLR通过识别病原体并启动炎症过程,在大脑中,尤其是在小胶质细胞中发现了TLR。肠道菌群与肠道和大脑(菌群-肠-脑轴)相互作用是引起AD的机制之一。肠道菌群失调可激活大脑中TLR信号通路,加重炎症反应,导致AD发生。因此,TLR4可能是2型糖尿病与AD之间的潜在联系。
综上所述,2型糖尿病发生可能与TLR-干扰素β/趋化因子-胰岛炎症反应轴密切相关。ILF2、TLR4、POLR2G、MMP9为2型糖尿病发病的关键基因,TLR4上调可通过影响嘧啶代谢及TLR信号通路影响2型糖尿病及AD的发生。本研究不足之处在于GSE95849数据集选取的对象为女性糖尿病患者,可能影响差异基因的分析结果,将来如果有更多的糖尿病外周血转录组数据,可以将组间性别、年龄等基线资料进行匹配后再进行分析,进而提高结果的准确性。
| [1] |
ZHONG M, WU YL, OU WJ, et al. Identification of key genes involved in type 2 diabetic islet dysfunction:a bioinformatics study[J]. Biosci Rep, 2019, 39(5): 2018-2072. DOI:10.1042/bsr20182172 |
| [2] |
JIAN LM, YANG GD. Identification of key genes involved in diabetic peripheral neuropathy progression and associated with pancreatic cancer[J]. Diabetes Metab Syndr Obes:Targets Ther, 2020, 13: 463-476. DOI:10.2147/dmso.s235011 |
| [3] |
DING LC, FAN L, XU XD, et al. Identification of core genes and pathways in type 2 diabetes mellitus by bioinformatics analysis[J]. Mol Med Report, 2019, 20(3): 2597-2608. DOI:10.3892/mmr.2019.10522 |
| [4] |
SHINOHARA M, SATO N. Bidirectional interactions between diabetes and Alzheimer's disease[J]. Neurochem Int, 2017, 108: 296-302. DOI:10.1016/j.neuint.2017.04.020 |
| [5] |
PEARSON WR. BLAST and FASTA similarity searching for multiple sequence alignment[M]//Methods in Molecular Biology. Totowa, NJ: Humana Press, 2013: 75-101. DOI: 10.1007/978-1-62703-646-7_5.
|
| [6] |
VARMA S. Blind estimation and correction of microarray batch effect[J]. PLoS One, 2020, 15(4): e0231446. DOI:10.1371/journal.pone.0231446 |
| [7] |
SINGER-ENGLAR T, BARLOW G, MATHUR R. Obesity, diabetes, and the gut microbiome:an updated review[J]. Expert Rev Gastroenterol Hepatol, 2019, 13(1): 3-15. DOI:10.1080/17474124.2019.1543023 |
| [8] |
AAMIR K, KHAN HU, SETHI G, et al. Wnt signaling mediates TLR pathway and promote unrestrained adipogenesis and metaflammation:therapeutic targets for obesity and type 2 diabetes[J]. Pharmacol Res, 2020, 152: 104602. DOI:10.1016/j.phrs.2019.104602 |
| [9] |
TAHA IM, ABDU ALLAH AM, ABD EL GAYED EM. Expression of toll-like receptor 4 and its connection with type 2 diabetes mellitus[J]. Cell Mol Biol (Noisy-Le-Grand), 2018, 64(13): 15-20. DOI:10.14715/cmb/2018.64.13.4 |
| [10] |
ZHOU ZX, ZENG CL, NIE LH, et al. The effects of TLR3, TRIF and TRAF3 SNPs and interactions with environmental factors on type 2 diabetes mellitus and vascular complications in a Han Chinese population[J]. Gene, 2017, 626: 41-47. DOI:10.1016/j.gene.2017.05.011 |
| [11] |
HUGHES CE, NIBBS RJB. A guide to chemokines and their receptors[J]. FEBS J, 2018, 285(16): 2944-2971. DOI:10.1111/febs.14466 |
| [12] |
CAESAR R, TREMAROLI V, KOVATCHEVA-DATCHARY P, et al. Crosstalk between gut microbiota and dietary lipids aggravates WAT inflammation through TLR signaling[J]. Cell Metab, 2015, 22(4): 658-668. DOI:10.1016/j.cmet.2015.07.026 |
| [13] |
WANG L, PI ZF, LIU S, et al. Targeted metabolome profiling by dual-probe microdialysis sampling and treatment using Gardenia jasminoides for rats with type 2 diabetes[J]. Sci Rep, 2017, 7(1): 1. DOI:10.1038/s41598-017-10172-w |
| [14] |
XIANG X, CAI HD, SU SL, et al. Salvia miltiorrhiza protects against diabetic nephropathy through metabolome regulation and wnt/β-catenin and TGF-β signaling inhibition[J]. Pharmacol Res, 2019, 139: 26-40. DOI:10.1016/j.phrs.2018.10.030 |
| [15] |
VIEIRA MNN, LIMA-FILHO RAS, DE FELICE FG. Connecting Alzheimer's disease to diabetes:underlying mechanisms and potential therapeutic targets[J]. Neuropharmacology, 2018, 136: 160-171. DOI:10.1016/j.neuropharm.2017.11.014 |
| [16] |
WURTMAN RJ, ULUS IH, CANSEV M, et al. Synaptic proteins and phospholipids are increased in gerbil brain by administering uridine plus docosahexaenoic acid orally[J]. Brain Res, 2006, 1088(1): 83-92. DOI:10.1016/j.brainres.2006.03.019 |
| [17] |
GONZÁLEZ-DOMÍNGUEZ R, GARCÍA-BARRERA T, VITORICA J, et al. Metabolomic screening of regional brain alterations in the APP/PS1 transgenic model of Alzheimer's disease by direct infusion mass spectrometry[J]. J Pharm Biomed Anal, 2015, 102: 425-435. DOI:10.1016/j.jpba.2014.10.009 |
| [18] |
LIN CX, ZHAO S, ZHU YL, et al. Microbiota-gut-brain axis and toll-like receptors in Alzheimer's disease[J]. Comput Struct Biotechnol J, 2019, 17: 1309-1317. DOI:10.1016/j.csbj.2019.09.008 |
| [19] |
HUANG NQ, JIN H, ZHOU SY, et al. TLR4 is a link between diabetes and Alzheimer's disease[J]. Behav Brain Res, 2017, 316: 234-244. DOI:10.1016/j.bbr.2016.08.047 |
| [20] |
MIRON J, PICARD C, LAFAILLE-MAGNAN MÉ, et al. Association of TLR4 with Alzheimer's disease risk and presymptomatic biomarkers of inflammation[J]. Alzheimers Dement, 2019, 15(7): 951-960. DOI:10.1016/j.jalz.2019.03.012 |
 2020, Vol. 49
2020, Vol. 49




