文章信息
- 张佳佳, 张斌, 袁莹, 高雪芹, 郭敏, 关一夫
- ZHANG Jiajia, ZHANG Bin, YUAN Ying, GAO Xueqin, GUO Min, GUAN Yifu
- 滚环扩增方法直接检测miR-93-5p的可行性研究
- Feasibility of rolling circle amplification for directly detecting miR-93-5p
- 中国医科大学学报, 2020, 49(1): 10-15
- Journal of China Medical University, 2020, 49(1): 10-15
-
文章历史
- 收稿日期:2018-12-13
- 网络出版时间:2019-12-12 9:44
2. 中国医科大学 附属口腔医院整形外科, 沈阳 110002
2. Department of Plastic Surgery, Hospital of Stomatology, China Medical University, Shenyang 110002, China
微小RNA(microRNA,miRNA)是一类内源性的非编码小分子RNA[1],介导mRNA降解或抑制其翻译,在转录后水平负调节基因的表达。miRNA与肿瘤的发生发展及肿瘤治疗耐药都有密切的关系[2]。研究[3-5]发现,miR-93-5p在多种肿瘤细胞中表达上调,其过度表达可导致肿瘤细胞对顺铂的敏感性降低,从而发生耐药。因此,推测可以通过检测miR-93-5p反映化疗过程中肿瘤细胞对顺铂的敏感性。如果检测到miR-93-5p过量表达,则提示肿瘤细胞可能已经对顺铂出现耐受,提醒医生及时调整治疗方案,对临床患者的治疗有积极意义。
滚环扩增(rolling circle amplification,RCA)技术作为恒温扩增技术中的一种,建立于90年代中期[6],该技术因其灵敏、特异、操作便捷的特点,已被应用于环状DNA扩增、单核苷酸多态性、病原体及miRNA的检测等多个方面[7-10]。本研究利用RCA技术建立了一种直接检测miR-93-5p的方法,并以RT-qPCR方法作为对照对该方法进行评估。
1 材料与方法 1.1 材料本研究所用miR-93-5p由宝生物工程(大连)有限公司合成。E.coli DNA Ligase、Exonuclease Ⅲ、dNTP、RNAiso for Small RNA试剂均购自宝生物工程(大连)有限公司,phi29 DNA Polymerase、SYBR GreenⅡ染料、TaqManTM MicroRNA逆转录试剂盒、TaqManTM Universal Master MixⅡ(no UNG)、TaqManTM MicroRNA Assay试剂盒(含miR-93-5p特异性逆转录茎环引物、PCR扩增引物、特异性TaqMan探针)均购自赛默飞世尔科技(中国)有限公司。本研究中所用寡核苷酸均由上海生工生物有限公司合成。
1.2 方法 1.2.1 引物设计miR-93-5p序列信息来自miRBase(http://www.mirbase.org/),Accession number为MIMAT0000093。利用DNAMAN和RNA structure软件优化寡核苷酸序列,使其自由能最低且无二级结构。本研究所用寡核苷酸序列见表 1。
| Oligonucleotide | Sequences(5'-3') |
| Padlock probe | p-$\mathsf{CAGCACTTTG}$TCTCTTCTACTTTGTCTCTATTCATATTCCACCCA$\mathsf{CTACCTGCACGAA}$ |
| Connection sequence | CAAAGTGCTGTTCGTGCAGGTAG |
| miR-93-5p | CAAAGUGCUGUUCGUGCAGGUAG |
| 3'A-miR-93-5p | CAAAGUGCUGUUCGUGCAGGUA$\underline {\mathsf{A}} $ |
| let-7a | UGAGGUAGUAGGUUGUAUAGUU |
| let-7b | UGAGGUAGUAGGUUGUGUGGUU |
| let-7c | UGAGGUAGUAGGUUGUAUGGUU |
| let-7d | AGAGGUAGUAGGUUGCAUAGUU |
| let-7e | UGAGGUAGGAGGUUGUAUAGUU |
| let-7f | UGAGGUAGUAGAUUGUAUAGUU |
| p-represents 5'-terminal phosphorylation;bold represents the sequence in which the padlock probe binds to the connection sequence/miR-93-5p;the bold is underlined to represent the mutation;the connection sequence is miR-93-5p DNA sequence,in which italic T is U in miR-93-5p. | |
1.2.2 反应原理
如图 1所示,首先,padlock probe的两端与connection sequence碱基互补配对结合形成一个开链的环状结构,其中padlock probe的3'-OH与5'-PO3紧密相邻,在DNA ligase的作用下形成磷酸二酯键,进而成为一个闭合的环状结构。用exonuclease将闭合环上的connection sequence以及未成环的线性padlock probe、未结合的connection sequence酶切去除,进而得到一个单链环状DNA,利用此单链环状DNA作为滚环扩增的环形模板,待测miR-93-5p作为引物,在phi29 DNA polymerase的作用下进行酶促延伸,产生大量与单链环状模板互补的重复串联拷贝序列,产物与SYBR Green Ⅱ荧光染料结合,在激发光的激发下发出强荧光,通过酶标仪收集荧光信号,从而达到检测miR-93-5p的目的。
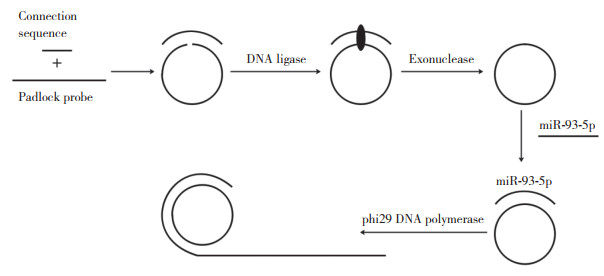
|
| 图 1 RCA检测miR-93-5p的原理 Fig.1 The principle of RCA detecting miR-93-5p |
1.2.3 单链环状模板的制备
连接反应体系包含padlock probe(600 nmol/L)2 µL,connection sequence(600 nmol/L)2 µL,10×E.coli DNA Ligase Buffer 2 µL,E.coli DNA Ligase 60 U,去离子水补齐至20 µL;反应条件为16 ℃连接16 h,65 ℃ 10 min终止反应。制备环形模板的酶切反应体系25 µL,连接产物20 µL,10×Exonuclease Ⅲ Buffer 2.5 µL,Exonuclease Ⅲ 125 U,去离子水补齐至25 µL;反应条件为37 ℃酶切16 h,95 ℃ 20 min终止反应。
1.2.4 RCA反应体系包含10×phi29 DNA Polymerase Buffer 10 µL,dNTP 5 µL,100×SYBR GreenⅡ1 µL,phi29 DNA Polymerase 0.25 U,环形模板(500 nmol/L)1 µL,miR-93-5p(浓度依不同实验而定)1 µL,去离子水补齐至100 µL;反应条件37 ℃ 30 min;酶标仪实时扫描荧光值。
1.2.5 逆转录反应制备引物池(RT Primer Pool)体系1 000 µL,包含5×RT primer10 µL,1×TE 990 µL;逆转录(参照逆转录试剂盒说明书)体系15 µL,RT Primer Pool 6 µL,dNTPs with dTTP 0.3 µL,Multiscribe Reverse Transcriptase 3 µL,10×RT Buffer 1.5 µL,RNase Inhibitor 0.19 µL,Nuclease-free water 1.01 µL,待测miRNA 3 µL;反应条件16 ℃ 30 min,42 ℃ 30 min,58 ℃ 5 min,4 ℃终止反应。产物于-20 ℃冰箱保存。
1.2.6 PCR反应反应体系20 µL,20×TaqMan MicroRNA Assays 1 µL,RT product 0.25 µL,TaqMan Universal Master MixⅡ(no UNG,2×)10 µL,去离子水8.75 µL。反应条件95 ℃ 10 min;95 ℃10 s;40 ℃ 60 s,50个循环;37 ℃终止反应。
1.2.7 实际样品检测取贴壁培养的A2780细胞,用RNAiso for Small RNA试剂提取RNA,-80 ℃保存。分别用RCA及PCR 2种方法检测miR-93-5p。
2 结果 2.1 padlock probe连接成环padlock probe与connection sequence经E.coli DNA Ligase连接后,一部分padlock probe形成了带有connection sequence的闭合环状结构,一部分padlock probe形成了与connection sequence首尾相连的串联长链结构,还有一部分padlock probe和connection sequence处于游离状态。为了得到单纯的单链环状DNA作为RCA的模板,用ExonucleaseⅢ处理连接产物,有效去除环状结构上的connection sequence、未成环的长链、游离的padlock probe和connection sequence,得到了单纯的单链环状DNA。理论上讲,此时样品中仅存闭合环状的padlock probe。将不同处理条件的样品加热变性后利用聚丙烯酰胺凝胶电泳(16%)对其进行了分析,电泳结果如图 2所示,经连接反应及酶切后,在60 bp处获得1个清晰的条带,这一条带为单链环状DNA,即single-stranded circular DNA。
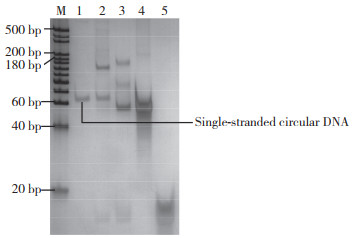
|
| M, 20 bp DNA Ladder; 1, padlock probe + connection sequence + E.coli DNA ligase + exonuclease Ⅲ; 2, padlock probe + connection sequence + E.coli DNA Ligase;3, padlock probe + connection sequence ;4, padlock probe; 5, connection sequence. 图 2 聚丙烯酰胺凝胶电泳(16%)分析padlock probe环化结果 Fig.2 Polyacrylamide gel electrophoresis (16%) analyze results of padlock probe cyclization |
2.2 灵敏度检测结果
将人工合成的miR-93-5p作为标准品,制备一系列不同浓度的样品,分别利用RCA方法和PCR方法进行扩增,用以评估RCA方法和PCR方法的检测灵敏度。RCA方法检测miR-93-5p的结果如图 3A和3B所示,检测范围为10~104 pmol/L,当miR-93-5p的浓度在250~1 000 pmol/L内显示了良好的线性关系,线性方程为y=862x-1974,R2=0.991 7。PCR方法检测miR-93-5p的结果如图 3C和3D所示,检测范围为10~104 pmol/L,线性方程为y =-5.68x+35.91,R2=0.999 8。结果显示,RCA方法检测miR-93-5p的灵敏度与PCR相近,但RCA方法线性检测范围较窄。
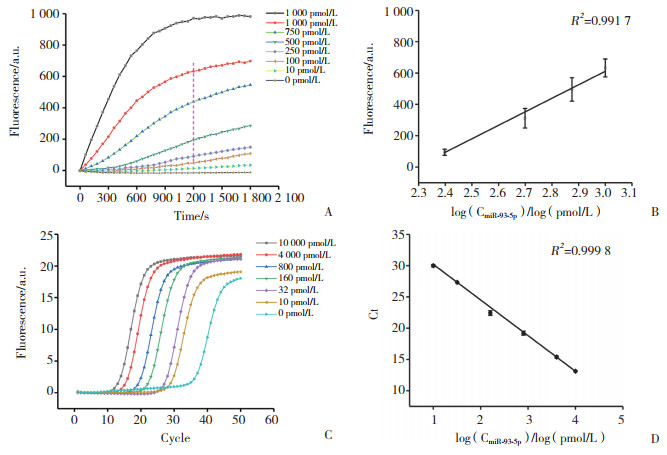
|
| A, rolling circle amplification (RCA) detects the sensitivity of miR-93-5p; B, RCA detects the standard curve of miR-93-5p constructed using the fluorescence value at 1 200 s of the amplification curve from graph A; C, PCR detects the sensitivity of miR-93-5p; D, PCR detects the standard curve of miR-93-5p constructed using the amplified Ct value of each concentration mentioned in graph C. 图 3 RCA及PCR方法检测miR-93-5p的灵敏度和标准曲线 Fig.3 Sensitivity of miR-93-5p and its standard curve as detected by RCA and PCR |
2.3 特异度检测结果
本研究将miR-93-5p作为引物结合在环形模板上进而引发扩增,扩增中可能会出现非检测目的序列结合在环形模板上导致非特异性扩增。为了确定这种检测方法的特异度,选取了与miR-93-5p非同源且不相关的miRNA let-7家族中的let-7a~let-7f及与miR-93-5p只有1个碱基差异的RNA序列3'A-miR-93-5p来进行特异度验证。同样是分别利用RCA方法和PCR方法进行扩增,用以评估RCA方法和PCR方法的检测特异度。结果如图 4所示,RCA和PCR方法检测miR-93-5p均显示了良好的特异性。
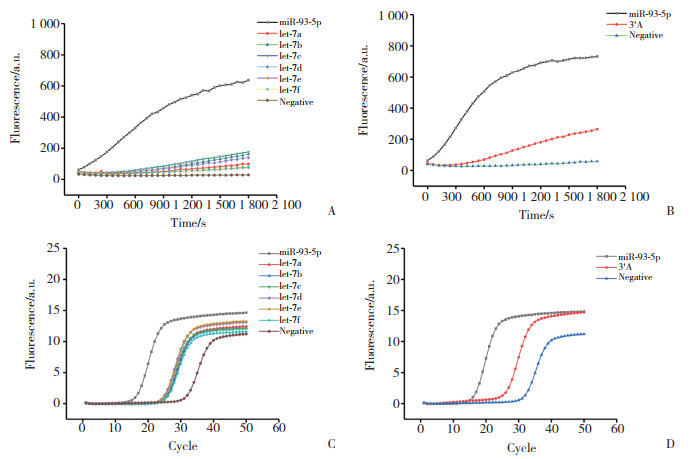
|
| A, RCA detects the specificity of miR-93-5p; B, RCA detects the specificity of miR-93-5p point mutation; C, PCR detects the specificity of miR-93-5p; D, PCR detects the specificity of miR-93-5p point mutation. 图 4 RCA及PCR检测miR-93-5p特异性和点突变特异性 Fig.4 Specificity of miR-93-5p and miR-93-5p point mutation as detected by RCA and PCR |
2.4 实际样品检测
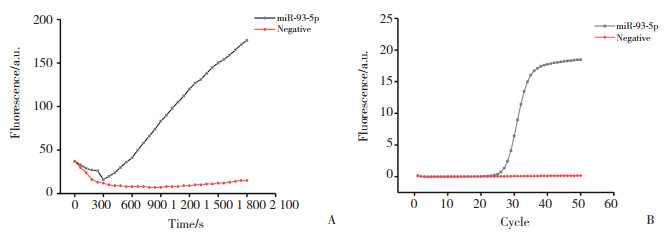
从贴壁培养的A2780细胞中提取RNA,然后对提取的RNA样品分别进行RCA和PCR检测,扩增结果如图 5所示,RCA和PCR均能检测出miR-93-5p,表明2种方法均可实现对样品的检测,适用于实际检测。
|
| A, RCA detects miR-93-5p in A2780 cells; B, PCR detects miR-93-5p in A2780 cells. 图 5 RCA及PCR检测A2780细胞中的miR-93-5p Fig.5 miR-93-5p in A2780 cells detected by RCA and PCR |
3 讨论
目前比较常用的miRNA检测方法有Northern印迹法、微阵列芯片技术、实时PCR,但是这些方法均存在一定的缺点,如Northern印迹法和微阵列芯片技术对样品需求量大、灵敏度低、特异性差、操作繁琐,应用时有诸多限制。在目前已经发表的检测miRNA的方法中,TaqMan探针stem-loop RT-qPCR方法是最经典且灵敏度、特异度均较高的方法,可视为检测miRNA方法中的金标准[11]。该方法虽然灵敏特异,但是价格昂贵,实验条件要求较高,应用范围受到局限。恒温扩增技术反应全程在同一温度下进行,对仪器要求大大简化,反应时间短,操作简单,相比于以上几种miRNA检测方法更能满足现代分子检测技术“快速简便”的要求,近年来逐渐应用于miRNA的检测。RCA是恒温扩增技术中的一种,模拟自然界中微生物环状DNA的自我复制过程,以环状单链DNA或RNA为模板,在具有链置换活性的DNA聚合酶作用下,引物沿着环形模板进行扩增,实现模板的信号放大。同样检测miRNA,PCR方法首先需要经过逆转录cDNA后,才能进行热循环PCR扩增检出目的RNA,而本研究所建立的RCA方法则无需经过逆转录和温度变换,恒温反应仅需30 min即可直接检出目的RNA,成本低廉,操作便捷,可实现样本的快速检测。
miR-93-5p作为一个与肿瘤调控相关的miRNA,与肿瘤耐药有着密切的联系[12]。在顺铂耐药的卵巢癌细胞中,miR-93-5p过表达,检测miR-93-5p可间接反映顺铂耐药的情况,有助于疾病治疗过程中及时发现肿瘤细胞耐药及调整治疗方案,对临床用药有指导意义。本研究所建立的RCA方法可以方便快捷地检测到不同浓度的miR-93-5p。但是,该方法在灵敏度方面还存在一定的局限,当待检测的miR-93-5p达到特定浓度时,曲线上升极快,线性度较差,这也是恒温扩增方法普遍存在的问题[13]。本研究的关键问题之一在于DNA与miRNA的杂交效率,不同浓度杂交效率的不同导致实际RCA反应时线性范围较窄。同时恒温扩增普遍还存在的一个问题就是没有理想的质控指标[14],这些都有待于进一步探究。
总之,本研究所建立的检测miR-93-5p的RCA方法所需设备简单,操作快捷,易于推广应用,具有较高的实际应用价值和广阔的应用前景。
| [1] |
LEWIS BP, BURGE CB, BARTEL DP. Conserved seed pairing, often flanked by adenosines, indicates that thousands of human genes are microRNA targets[J]. Cell, 2005, 120(1): 15-20. DOI:10.1016/j.cell.2004.12.035 |
| [2] |
MITCHELL PS, PARKIN RK, KROH EM, et al. Circulating microRNAs as stable blood-based markers for cancer detection[J]. Proc Natl Acad Sci USA, 2008, 105(30): 10513-10518. DOI:10.1073/pnas.0804549105 |
| [3] |
CHEN QY, QIN R, FANG Y, et al. Berberine sensitizes human ovarian cancer cells to cisplatin through miR-93/PTEN/akt signaling pathway[J]. Cell Physiol Biochem, 2015, 36(3): 956-965. DOI:10.1159/000430270 |
| [4] |
YANG W, BAI JQ, LIU DY, et al. MiR-93-5p up-regulation is involved in non-small cell lung cancer cells proliferation and migration and poor prognosis[J]. Gene, 2018, 647: 13-20. DOI:10.1016/j.gene.2018.01.024 |
| [5] |
MA DH, LI BS, LIU JJ, et al. MiR-93-5p/IFNAR1 axis promotes gastric cancer metastasis through activating the STAT3 signaling pathway[J]. Cancer Lett, 2017, 408: 23-32. DOI:10.1016/j.canlet.2017.08.017 |
| [6] |
FIRE A, XU SQ. Rolling replication of short DNA circles[J]. Proc Natl Acad Sci USA, 1995, 92(10): 4641-4645. DOI:10.1073/pnas.92.10.4641 |
| [7] |
BRASINO MD, CHA JN. Isothermal rolling circle amplification of virus genomes for rapid antigen detection and typing[J]. Analyst, 2015, 140(15): 5138-5144. DOI:10.1039/c5an00721f |
| [8] |
ZHU XL, SHEN YL, CAO JP, et al. Detection of microRNA SNPs with ultrahigh specificity by using reduced graphene oxide-assisted rolling circle amplification[J]. Chem Commun (Camb), 2015, 51(49): 10002-10005. DOI:10.1039/c5cc02039e |
| [9] |
CARINELLI S, KÜHNEMUND M, NILSSON M, et al. Yoctomole electrochemical genosensing of Ebola virus cDNA by rolling circle and circle to circle amplification[J]. Biosens Bioelectron, 2017, 93: 65-71. DOI:10.1016/j.bios.2016.09.099 |
| [10] |
DENG XJ, QIN SS, CHEN YQ, et al. B-RCA revealed circulating miR-33a/b associates with serum cholesterol in type 2 diabetes patients at high risk of ASCVD[J]. Diabetes Res Clin Pract, 2018, 140: 191-199. DOI:10.1016/j.diabres.2018.03.024 |
| [11] |
SMOCZYNSKA A, SEGA P, STEPIEN A, et al. MiRNA detection by stem-loop RT-qPCR in studying microRNA biogenesis and microRNA responsiveness to abiotic stresses[M]. New York: Human Press, 2019: 131-150. DOI:10.1007/978-1-4939-9042-9_10
|
| [12] |
CHEN X, CHEN S, XIU YL, et al. RhoC is a major target of microRNA-93-5P in epithelial ovarian carcinoma tumorigenesis and progression[J]. Mol Cancer, 2015, 14: 31. DOI:10.1186/s12943-015-0304-6 |
| [13] |
张宏萍, 周敏, 陆仁飞, 等. 恒温滚环扩增技术检测肠道病毒71型[J]. 实用医学杂志, 2015, 31(15): 2454-2457. DOI:10.3969/j.issn.1006-5725.2015.15.014 |
| [14] |
张俊, 曾照芳. 滚环扩增技术的原理及其应用的研究[J]. 生物信息学, 2012, 10(1): 12-14. DOI:10.3969/j.issn.1672-5565.2012.01.04 |
 2020, Vol. 49
2020, Vol. 49




