文章信息
- 崔大炜, 邢育柏, 金宏远, 彭雪强, 杨良, 范庆
- CUI Dawei, XING Yubai, JIN Hongyuan, PENG Xueqiang, YANG Liang, FAN Qing
- 肝癌高表达长链非编码RNA在肝癌中的表达及其对自噬水平的影响
- Expression of the Long Non-Coding RNA HULC and its Influence on Autophagy in Hepatocellular Carcinoma
- 中国医科大学学报, 2019, 48(11): 970-974
- Journal of China Medical University, 2019, 48(11): 970-974
-
文章历史
- 收稿日期:2018-12-29
- 网络出版时间:2019-11-20 11:20
2. 中国医科大学附属第四医院普通外科, 沈阳 110032
2. Department of General Surgery, The Fourth Affiliated Hospital, China Medical University, Shenyang 110032, China
肝细胞癌(hepatocellular carcinoma,HCC)是目前临床上最常见的恶性肿瘤之一,全世界每年约有43.7万人被确诊为HCC,其中约50%发生在中国[1-2]。尽管随着外科手术方法和介入、消融等技术的不断进步,HCC的治疗效果得到改善,但患者5年生存率仅约26%[3]。研究肝癌发生、发展的分子学机制,有助于探寻肝癌的早期诊断与靶向治疗的新靶点。自噬是细胞通过溶酶体或液泡分解自身组分,以达到维持细胞内正常生理活动和稳态的一种细胞代谢过程[4]。自噬在真核生物中保守存在,与人类疾病和健康息息相关,其中在肿瘤的发生发展过程中能够通过降解细胞器和蛋白质等为肿瘤细胞提供生长所需[5]。通过改变肿瘤细胞的自噬水平从而治疗肿瘤,已经成为攻克肿瘤的新思路。
长链非编码RNA(long non-coding RNA,lncRNA)是细胞中一类转录本长度大于200 nt的非编码RNA分子,其本身并不具备编码蛋白的功能[6]。起初认为其无任何编码功能,但近年来研究[7-8]显示lncRNA参与了多种生物学过程,尤其是肿瘤的发病机制。人们发现许多lncRNA在恶性肿瘤中都有差异性表达,并且lncRNA在这些恶性肿瘤中充当致癌基因或抑癌基因[9]。肝癌高表达(highly upregulated in liver cancer,HULC)的lncRNA是第一个在HCC中鉴定出的高表达的lncRNA,可通过调节邻近的蛋白质编码基因来促进肝癌细胞增殖,这意味着HULC在肿瘤细胞的发展过程中扮演了重要角色[10]。本研究通过检测lncRNA HULC在人肝癌组织和正常肝组织中的表达情况和体外实验,探讨其对肝癌细胞增殖及自噬水平的影响,为临床肝癌的靶向治疗提供新的靶点。
1 材料与方法 1.1 标本收集收集2016年3月至2018年6月间遵义医科大学第五附属(珠海)医院肝癌手术切除的60例HCC标本。60例HCC新鲜冰冻组织标本,置液氮冷却后送-80 ℃冰箱保存。所有组织经2名病理医师评估,确诊为肝癌组织且复查无误。本研究在医院伦理委员会批准和患者签署知情同意书后进行。
1.2 细胞系和培养条件肝癌细胞系(HepG2、Bel-7402、SMMC-7721及Huh7)和Chang Liver细胞系均购自中乔新舟生物科技有限公司。细胞培养选用高糖DMEM,添加10%胎牛血清,另加双抗(100 μg/mL青霉素和100 mg/mL链霉素)的培养基,置于37 ℃含5%CO2的培养箱中进行常规培养。每天更换新鲜培养基,当细胞融合度达到80%~90%时进行传代培养。
1.3 RNA提取与实时PCR采用TRIzol试剂(美国Invitrogen公司)裂解细胞,以用于细胞内总RNA的提取。使用Power SYBR Green(日本TaKaRa公司)做实时PCR分析。结果用GAPDH的表达量标准化。HULC上游引物:5’-TCATGATGGAATTG-GAGCCTT-3’,下游引物:5’-CTCTTCCTG-GCTTGCAGATTG-3’;GAPDH上游引物:5’-GTCAACGGATTTGGTCTGTATT-3’,下游引物5’-AGTCTTCTGGGTGGCAGTGAT-3’。实时PCR和数据收集基于ABI 7500平台。HULC相对于GAPDH的表达采用2-ΔΔCt法计算和标准化。
1.4 质粒的构建及细胞转染HULC过表达质粒为上海吉玛公司合成、鉴定后提供的商用产品(NR_002766.2),为HULC序列亚克隆至pcDNA3.1载体后构建成的质粒,pcDNA3.1空白载体作为阴性对照。将肝癌细胞株Bel-7402和HepG2按5×105/孔种于6孔板中,待细胞达到70%融合时,用pcDNA3.1-HULC以及相应的阴性对照(转染空载质粒)通过Lipofectamine 2000(美国Invitrogen公司)转染细胞,6 h后更换培养基,48 h后收获细胞用于实时PCR检测,72 h后用于Western blotting检测。
1.5 CCK-8测定细胞增殖CCK-8实验具体步骤按照制造商的标准方案进行。每隔24 h,采用CCK-8试剂(日本同仁化学研究所)检测1次细胞增殖。将对数生长期细胞接种在96孔板(1×104/孔),加入CCK-8溶液并孵育3 h。然后通过分光光度法测量每种溶液在490 nm下的吸光度值。设置空白对照组,同时每组分别设置4个复孔取平均值。
1.6 免疫荧光检测LC3将爬片放入12孔板中,胶原蛋白包被爬片,紫外线灯照30 min。分别将HepG2和Bel-7402细胞接种于12孔板中,1~2 d换液1次,待细胞贴壁且生长到65%左右,将pcDNA3.1-HULC质粒加入培养箱培养18 h,PBS清洗3次,每次5 min。随后用多聚甲醛固定20 min,再用PBS清洗3遍,每次5 min。Triton透膜,牛血清白蛋白封闭,孵育一抗和二抗,DAPI染核。分别以转染pcDNA3.1的HepG2和Bel-7402细胞为对照组,在荧光显微镜下检测各组肝癌细胞内LC3荧光斑点的数量与分布。
1.7 Western blotting对细胞用裂解液进行蛋白提取,然后使用SDS-PAGE等试剂,将蛋白转到NC膜(美国Sigma公司)上,用特定抗体孵育,进而检测特定蛋白(LC3抗体、Atg3和β-actin购自美国ProteinTech公司)的表达。通过放射密度测定法(Quantity One软件,美国Bio-Rad公司)对放射自显影图片进行量化。将β-actin作为对照。
1.8 统计学分析采用SPSS 17.0软件对结果进行统计与分析。定量资料用x±s表示,采用Student t检验进行比较。P < 0.05为差异有统计学意义。
2 结果 2.1 HULC在肝癌组织和正常肝组织中的表达水平采用实时PCR检测60例肝癌患者样本中HULC的表达情况。结果显示,肝癌组织中HULC的表达水平显著高于相邻正常肝组织(P < 0.05)(图 1A)。检测HULC在人肝癌细胞系HepG2、Bel-7402、SMMC-7721及Huh7中的表达水平,结果发现,上述4种肝癌细胞系中HULC的表达水平均高于正常肝细胞系Chang Liver(P < 0.05)(图 1B)。由于HULC在HepG2和Bel-7402中的表达相对较低,因此用于后续转染pcDNA3.1过表达细胞实验中。
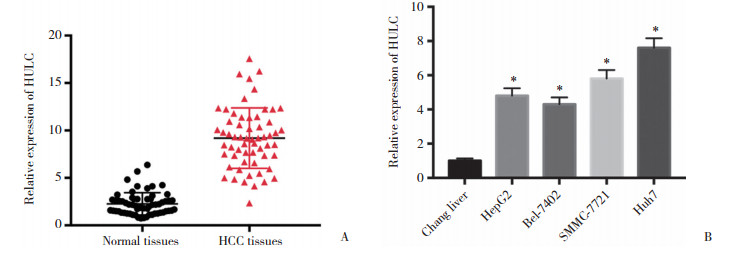
|
| A, expression level of HULC in HCC tissues was higher than that in normal tissues; B, expression levels of HULC in HepG2, Bel-7402, SMMC-7721, and Huh7 cells were higher than those in Chang liver cells. * P < 0.05 vs normal tissues in panel A; * P < 0.05 vs Chang liver cells in panel B. 图 1 HULC在肝癌组织和正常肝组织中的表达情况 Fig.1 Expression levels of HULC in HCC tissues and normal tissues |
2.2 过表达HULC促进肝癌细胞增殖
首先,在肝癌细胞HepG2和Bel-7402中成功转染pcDNA3.1-HULC质粒。通过实时PCR验证pcDNA3.1-HULC转染效率,结果显示,pcDNA3.1-HULC的转染效率分别为23倍与31倍(图 2A)。采用CCK-8试剂盒检测HULC过表达细胞系增殖能力的变化,结果显示,过表达HULC后肝癌细胞系增殖能力显著增加(图 2B)。
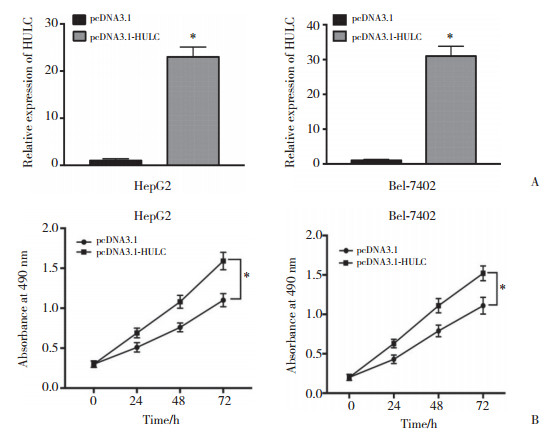
|
| A, pcDNA3.1-HULC increased the expression of HULC in HepG2 and Bel-7402 cells; B, overexpression of HULC promoted the proliferation of HepG2 and Bel-7402 cells. * P < 0.05 vs pcDNA3.1 group. 图 2 HULC对肝癌细胞HepG2和Bel-7402增殖的影响 Fig.2 Effect of pcDNA3.1-HULC on the proliferation of HepG2 and Bel-7402 cells |
2.3 过表达HULC表达能够增强肝癌细胞自噬水平
为了探讨HULC对肝癌细胞自噬水平的影响,在转染了pcDNA3.1-HULC质粒的肝癌细胞系HepG2、Bel-7402中,通过Western blotting检测过表达HULC的肝癌细胞中自噬标志蛋白LC3表达情况。结果显示,当HepG2和Bel-7402细胞中过表达HULC后,LC3-Ⅰ向LC3-Ⅱ的转化增多(图 3A)。这表明HULC能够促进肝癌细胞自噬。为了判断HULC对自噬体形成的影响,采用免疫荧光实验观察LC3荧光斑点的形成情况。结果显示,当肝癌细胞过表达HULC时,LC3荧光斑点形成增加(图 3B)。以上结果表明,HULC能够显著促进肝癌细胞自噬水平。
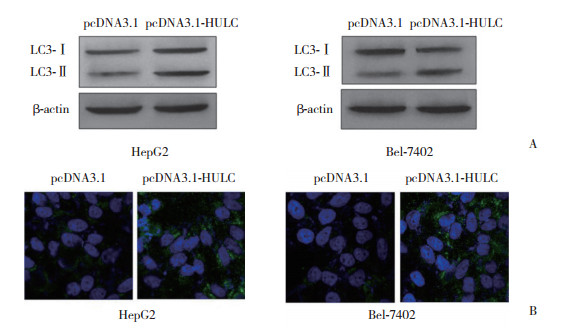
|
| A, overexpression of HULC increased the expression of LC3-Ⅱto LC3-Ⅰin HepG2 and Bel-7402 cells; B, overexpression of HULC increased the number of LC3 fluorescence points in HepG2 and Bel-7402 cells (×60). 图 3 HULC对肝癌细胞HepG2和Bel-7402自噬的影响 Fig.3 Effect of HULC on autophagy in HepG2 and Bel-7402 cells |
2.4 过表达HULC促进肝癌细胞中Atg3表达
为了明确HULC促进肝癌细胞自噬的机制,在转染pcDNA3.1-HULC质粒的肝癌细胞系HepG2、Bel-7402中,采用Western blotting检测细胞中自噬相关蛋白Atg3的表达。结果显示,在过表达HULC的肝癌细胞中Atg3表达显著上调(图 4A)。这表明HULC可以促进Atg3的表达。
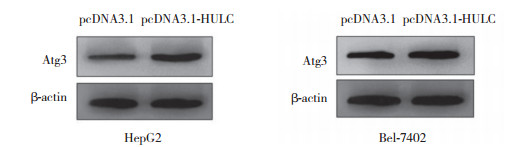
|
| 图 4 HULC上调促进肝癌细胞HepG2和Bel-7402中Atg3表达 Fig.4 Overexpression of HULC upregulated the expression of Atg3 protein in HepG2 and Bel-7402 cells |
3 讨论
lncRNA HULC是在肝癌中特异性高表达的lncRNA。由HULC基因转录而来,该基因定位在6p24.3,全长500 nt,含2个外显子(长度分别为303和182 bp)和1个内含子(长度为1 152 bp)。HULC基因转录后,产物经剪切、加工后形成长度为500 bp的RNA,与mRNA结构类似,具有且poly A尾结构。由于含有多个终止密码子,无编码蛋白功能,lncRNA HULC的保守性差导致其基因的多态性,与肿瘤易感性相关[11]。lncRNA HULC在肝癌组织中特异性高表达可能与肝癌微环境有关。近年来的研究[12-13]已经发现,一些异常表达的lncRNA对不同肿瘤的发生发展具有潜在作用,参与调控细胞免疫、凋亡、肿瘤侵袭与转移等恶性生物学行为。另外研究[14]发现,lncRNA能够通过表观遗传的调控方式影响肿瘤的生长。目前为止,人们对lncRNA的了解仍处于不断探索的阶段。
研究[15]表明,自噬与肿瘤的发生发展有着紧密的联系。自噬可通过调节肿瘤与肿瘤微环境的关系来促进肿瘤的存活与增殖。因此,通过促进或者抑制自噬从而治疗肿瘤,已经成为攻克肿瘤的新思路。随着人们对lncRNA的认识加深,其在自噬中的作用也逐渐受到人们的关注,一些lncRNA可以抑制自噬的发生,如Risa、H19等[16];与之相反,有些lncRNA能够促进自噬的发生,如BANCER、NEAT2、PTENT1、HOTAIR等[17]。
本研究发现,HULC在肝癌组织中的表达明显高于正常肝组织,并且在肝癌细胞系中的表达也高于正常肝细胞系。通过成功构建HULC过表达的HepG2和Bel-7402细胞系,本研究首先检测了肝癌细胞增殖能力的变化。结果发现,在过表达HULC的肝癌细胞系中细胞增殖能力显著增加。同时在自噬水平检测实验中,发现HULC能够促进肝癌细胞自噬的发生。而且本研究进一步发现,HULC能够显著诱导Atg3的上调。
综上所述,本研究结果提示,HULC在肝癌的发展过程中扮演着促癌基因的角色,能够促进自噬的发生,进而为肿瘤生存提供物质与能量基础。本研究结果可能为肝癌的靶向治疗提供新的方向,但具体分子机制有待进一步揭示。
| [1] |
TORRE LA, BRAY F, SIEGEL RL, et al. Global cancer statistics, 2012[J]. CA:Cancer J Clin, 2015, 65(2): 87-108. DOI:10.3322/caac.21262 |
| [2] |
VINEIS P, WILD CP. Global cancer patterns:causes and prevention[J]. Lancet, 2014, 383(9916): 549-557. DOI:10.1016/S0140-6736(13)62224-2 |
| [3] |
SIMARD EP, WARD EM, SIEGEL R, et al. Cancers with increasing incidence trends in the United States:1999 through 2008[J]. CA:Cancer J Clin, 2012, 62(2): 118-128. DOI:10.3322/caac.20141 |
| [4] |
GLICK D, BARTH S, MACLEOD KF. Autophagy:cellular and molecular mechanisms[J]. J Pathol, 2010, 221(1): 3-12. DOI:10.1002/path.2697 |
| [5] |
MOWERS EE, SHARIFI MN, MACLEOD KF. Functions of autophagy in the tumor microenvironment and cancer metastasis[J]. FEBS J, 2018, 285(10): 1751-1766. DOI:10.1111/febs.14388 |
| [6] |
BHAN A, SOLEIMANI M, MANDAL SS. Long noncoding RNA and cancer:a new paradigm[J]. Cancer Res, 2017, 77(15): 3965-3981. DOI:10.1158/0008-5472.CAN-16-2634 |
| [7] |
NECSULEA A, SOUMILLON M, WARNEFORS M, et al. The evolution of lncRNA repertoires and expression patterns in tetrapods[J]. Nature, 2014, 505(7485): 635-640. DOI:10.1038/nature12943 |
| [8] |
ZAVATTARI P, PERRA A, MENEGON S, et al. Nrf2, but not β-catenin, mutation represents an early event in rat hepatocarcinogenesis[J]. Hepatology, 2015, 62(3): 851-862. DOI:10.1002/hep.27790 |
| [9] |
MA Z, HUANG H, XU Y, et al. Current advances of long non-coding RNA highly upregulated in liver cancer in human tumors[J]. Onco Targets Ther, 2017, 10: 4711-4717. DOI:10.2147/OTT.S136915 |
| [10] |
DU Y, KONG G, YOU X, et al. Elevation of highly up-regulated in liver cancer (HULC) by hepatitis B virus X protein promotes hepatoma cell proliferation via down-regulating p18[J]. J Biol Chem, 2012, 287(31): 26302-26311. DOI:10.1074/jbc.M112.342113 |
| [11] |
KANG M, SANG Y, GU H, et al. Long noncoding RNAs POLR2E rs3787016 C/T and HULC rs7763881 A/C polymorphisms are associated with decreased risk of esophageal cancer[J]. Tumour Biol, 2015, 36(8): 6401-6408. DOI:10.1007/s13277-015-3328-z |
| [12] |
CHAN J, TAY Y. Noncoding RNA:RNA regulatory networks in cancer[J]. Int J Mol Sci, 2018, 19(5): 1310. DOI:10.3390/ijms19051310 |
| [13] |
YU WD, WANG H, HE QF, et al. Long noncoding RNAs in cancer-immunity cycle[J]. J Cell Physiol, 2018, 233(9): 6518-6523. DOI:10.1002/jcp.26568 |
| [14] |
HANLY DJ, ESTELLER M, BERDASCO M. Interplay between long non-coding RNAs and epigenetic machinery:emerging targets in cancer?[J]. Philos Trans R Soc Lond B Biol Sci, 2018, 373(1748): pii:20170074. DOI:10.1098/rstb.2017.0074 |
| [15] |
AMARAVADI R, KIMMELMAN AC, WHITE E. Recent insights into the function of autophagy in cancer[J]. Genes Dev, 2016, 30(17): 1913-1930. DOI:10.1101/gad.287524.116 |
| [16] |
MATHEW R, WHITE E. Autophagy in tumorigenesis and energy metabolism:friend by day, foe by night[J]. Curr Opin Genet Dev, 2011, 21(1): 113-119. DOI:10.1016/j.gde.2010.12.008 |
| [17] |
SUN T. Long noncoding RNAs act as regulators of autophagy in cancer[J]. Pharmacol Res, 2018, 129: 151-155. DOI:10.1016/j.phrs.2017.11.009 |
 2019, Vol. 48
2019, Vol. 48




