文章信息
- 郝鑫, 张媛媛, 陈菊芳, 刘明瑾, 田玉楼
- HAO Xin, ZHANG Yuanyuan, CHEN Jufang, LIU Mingjin, TIAN Yulou
- microRNA-30d/Runx2影响大鼠牙周辅助加速成骨正畸治疗模型成骨活性的研究
- Effect of the microRNA-30d/Runx2 Axis on Osteogenic Activity in PAOO-Facilitated Orthodontic Tooth Movement
- 中国医科大学学报, 2019, 48(11): 965-969
- Journal of China Medical University, 2019, 48(11): 965-969
-
文章历史
- 收稿日期:2019-06-05
- 网络出版时间:2019-11-20 13:10
正畸治疗的时间取决于错牙合畸形严重程度、患者的个体差异和配合性及正畸治疗手段等因素。较长的正畸治疗期可能增加牙齿脱钙、牙周炎伴牙槽骨吸收、牙髓炎、牙根吸收等并发症的风险[1-2]。因此,在纠正咬合的基础上,加速牙齿移动,缩短治疗周期,成为正畸临床医生需要解决的问题。牙周辅助加速成骨正畸治疗(periodontally accelerated osteogenic orthodontics,PAOO)基于骨皮质切开后产生的短暂脱矿—再矿化的局部加速现象[3],缩短正畸治疗时间的同时,又增加了正畸治疗的安全性与应用范围[4]。
本研究通过建立大鼠PAOO牙齿移动动物模型,观察正畸牙齿加速移动及传统移动过程中Runt相关转录因子2(Runt related transcription factor 2,Runx2)及microRNA-30d的表达情况。
1 材料与方法 1.1 主要试剂与仪器1%戊巴比妥钠、4%多聚甲醛、EDTA脱钙液、Trizol试剂、HiScript®Ⅱ Q RT SuperMix for qPCR试剂盒、QuantiFast®SYBR®Green PCR试剂盒(中国瑞沃德生命科技有限公司);定值拉簧(美国Ormco公司);体视显微镜(中国麦克奥迪实业集团有限公司);超微量分光光度仪、Ⅱ型荧光定量PCR仪(瑞士罗氏集团)。
1.2 动物实验 1.2.1 实验动物及分组选用清洁级、健康雄性SD大鼠21只,8周龄,体质量(300±20)g,由北京维通利华实验动物技术有限公司提供。分笼饲养,自由饮水,喂养SPF级全价颗粒维持鼠料。室温(20 ℃)、12 h/d照明下适应性饲养1周后进行实验操作。将21只SD大鼠随机分为传统牙齿移动组(TM组,n = 9),PAOO牙齿移动组(PAOO+TM组,n = 9),空白对照组(不做任何处理,n = 3)。TM组大鼠仅行正畸牵引(F=50 g),PAOO+TM组除行同等力量牵引外,行牙周辅助加速成骨,以上2组大鼠又按加力时间长短分为3、7、14 d 3个亚组。
1.2.2 大鼠牙齿移动模型建立TM组和PAOO+TM组大鼠行全身麻醉(1%戊巴比妥钠4 mL/kg腹腔注射)后,用牙科高速手机配裂钻车针在2个上颌切牙的牙根中部钻孔,用0.25 mm正畸不锈钢结扎丝穿过牙根上的孔洞,将上颌切牙结扎在一起增强支抗,并防止中切牙不断萌出。切开PAOO+TM组大鼠上颌第一磨牙颊、腭侧及近中牙槽黏膜,暴露牙槽骨面,用高速金刚砂车针在生理盐水冷却下距牙槽嵴顶约1 mm处打0.5 mm3的小孔,穿透骨皮质全层。将0.2 mm的结扎丝从TM组和PAOO+TM组大鼠的第一、二磨牙间穿过,上颌第一磨牙牙颈部平齐龈缘处的颊腭面及近中面磨出固位沟,将结扎丝安置于固位沟内,将拉簧固定在大鼠上颌第一磨牙与切牙之间,使拉簧拉伸,牵引第一磨牙近中移动。矫治力通过镍钛拉簧提供,测力计控制力值维持在50 g。术后3 d给予软质食物(如面包),之后恢复常规饮食。每天观察大鼠体征,检测拉簧固定状况,并清洁矫治装置,若拉簧损坏脱落,及时修复。分别于加力0 d(空白对照组)、3 d、7 d、14 d后处死大鼠。
1.3 牙齿移动距离测定在体视显微镜下,使用电子游标卡尺测量加力前TM组和PAOO+TM组大鼠上颌第一磨牙与上切牙之间的距离。处死大鼠后,去除矫治装置,取其上颌骨标本,再次测量大鼠上颌第一磨牙与上切牙之间的距离。取2次测量数值之差,为第一磨牙近中移动的距离。每只大鼠测量3次,取平均值。
1.4 HE标本的制备取大鼠上颌骨组织块,4%多聚甲醛固定48 h,4 ℃下10%EDTA脱钙3个月,乙醇脱水,透明,浸渍,石蜡包埋。制备第一磨牙近远中方向的纵向切片,厚度为5 μm,49 ℃展片,多赖氨酸包被的玻片捞片,60 ℃烤片2~3 h,行HE染色。
1.5 RT-PCR检测 1.5.1 提取总RNA分别于加力0、3、7、14 d处死大鼠,去除矫正装置,拔除上颌第一恒磨牙,取其周围牙槽骨,仔细去除牙槽骨表面软组织,放入冷冻管中,置于-80 ℃备用。用Trizol试剂提取总RNA,用NanoDrop2000超微量分光光度计测量RNA纯度及浓度,-80 ℃冻存备用。
1.5.2 合成cDNA用HiScript®Ⅱ Q RT SuperMix for qPCR(+gDNA wiper)试剂盒将加力0、3、7、14 d骨组织提取的mRNA逆转录成cDNA。用miScript Reverse Transcription Kit试剂盒将加力7 d获取的骨组织mRNA逆转录成cDNA。
1.5.3 定量RT-PCR将样品和引物按照说明书在QuantiFast®SYBR®Green PCR Master Mix中混合。以U6为内参基因进行实时定量PCR,对microRNA-30d进行相对定量,95 ℃ 5 min后,95 ℃ 10 s,60 ℃ 30 s,40个循环。引物为上海欧易生物医学科技有限公司设计,上海杰瑞生物工程有限公司合成。microRNA引物采用采用加PolyA尾合成,引物序列见表 1。分析扩增完毕后的熔解曲线和扩增曲线,采用2-△△Ct法进行mRNA相对定量分析。
| Gene | Sequence |
| Runx2 | Forward: TACTCTGCCGAGCTACGAA |
| Reverse: GTGAAACTCTTGCCTCGTC | |
| ALP | Forward:GGCGGATGATAAGGAGGAC |
| Reverse:GTCTTTCCCTCACTACACAG | |
| rno-miR-30d | Forward: TGTAAACATCCCCGACTGGAAG |
| Reverse: universal downstream primers in the kit | |
| U6 | Forward: CAAGGATGACACGCAAATTCG |
| Reverse: universal downstream primers in the kit |
1.6 统计学分析
采用SPSS 21.0软件进行统计学分析。每组实验重复3次,取x±s,对正态分布数据进行t检验,对非正态分布数据进行Wilcoxon秩和检验,双侧P < 0.05为差异有统计学意义。
2 结果 2.1 成功建立大鼠正畸牙齿移动模型所有大鼠均耐受实验过程,成功安装加力装置,未见正畸拉簧脱落现象。第一磨牙有松动现象,上颌第一及第二磨牙之间存在食物残渣。
2.2 大鼠正畸牙齿移动距离变化体视显微镜下测量上颌第一磨牙近中移动的距离,空白对照组上颌第一、第二磨牙紧密排列无间隙,加力3 d后有间隙。随着时间的增加,TM组、TM+PAOO组牙齿的移动距离不断加大,TM+PAOO组的移动速度大于TM组,在7 d时移动速率差值最大。见图 1。
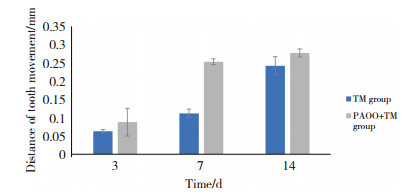
|
| 图 1 TM组、TM+PAOO组加力后牙齿移动距离 Fig.1 The tooth movement distance of TM and TM+PAOO groups |
2.3 HE染色结果
TM组牙周膜结构较致密,细胞质及细胞核结构清晰,纤维排列有序,牙周膜宽度均匀,成纤维细胞也均匀分布于牙周膜纤维之间。PAOO+TM组牙周膜宽度随加力时间延长而增大,在牙齿移动的第7天最为显著,牙周膜纤维被拉长,细胞成分增多,牙槽骨边缘可见新生血管,牙槽骨和牙骨质表面可见活跃的成骨细胞和新骨沉积,见图 2。

|
| TR, tooth root; PDL, parodontium; AB, alveolar bone. 图 2 不同加力时间大鼠第一磨牙近中移动牙槽骨HE染色 ×200 Fig.2 HE staining of alveolar bone around maxillary first molar of rats ×200 |
2.4 相关成骨因子的表达情况
qRT-PCR结果显示,加载正畸力后,与空白对照组相比,TM组、PAOO+TM组Runx2及碱性磷酸酶(alkaline phosphatase,ALP)表达量逐渐增加,第7天达峰值。第3、14天时,PAOO+TM组Runx2及ALP的表达水平高于TM组,而第7天时则低于TM组。见图 3。

|
| 图 3 qRT-PCR检测加力3、7、14 dTM组、PAOO+TM组牙槽骨组织Runx2、ALP的相对表达量 Fig.3 The relative expression levels of Runx2 and ALP mRNA in TM group and PAOO +TM group on the 3rd, 7th and 14th day |
为了检测microRNA-30d是否在加速正畸牙齿移动中产生作用,通过qRT-PCR检测了加力7 d后microRNA-30d在PAOO+TM组和TM组的表达情况,结果显示,PAOO+TM组microRNA-30d表达量是TM组的1.681 8倍,差异有统计学意义(P < 0.05)。
3 讨论正畸牙齿移动现象是由骨细胞、破骨细胞、成骨细胞与正畸力的耦合反应引起的[5]。牙齿移动速度主要取决于根部周围组织的重塑,而这又受到调节牙槽骨和牙周韧带中细胞行为的分子的调控[6]。Runx2作为骨特异性转录因子,能特异性结合许多成骨基因启动子上的顺式作用元件,调控成骨细胞特异性细胞外基质蛋白与基因表达,影响成骨细胞和破骨细胞分化,参与骨形成和吸收过程[7]。微小RNA是一类非编码的小RNA分子,由22~25个核苷酸组成,通过与靶mRNA的3’UTR互补或部分互补结合,使其降解或抑制翻译,进而调节其他蛋白质水平。近年来,越来越多的研究[8]报道微小RNA通过调控成骨细胞或破骨细胞的增殖、分化、凋亡等过程参与骨重构。本课题组之前的研究[9]表明,microRNA-30d参与成骨有关的生物功能和信号传导途径,在成骨细胞分化的过程中,microRNA-30d低表达,上调microRNA-30d能够抑制Runx2活性,降低ALP活性,抑制骨形成蛋白2(bone morphogenetic protein-2,BMP-2)诱导的成骨细胞分化。
SD大鼠为啮齿类动物,切牙较大,属不断生长的牙齿,磨牙为多根牙,结构和特性类似于人类牙齿,同时,磨牙的牙槽窝内骨更新率高,骨形成和骨吸收能够达到很好的平衡,受到外界刺激后牙槽骨的改建也非常明显且迅速[10]。大鼠牙齿移动模型装置脱落率较低,以拉簧作为加力装置,力值稳定,衰减较慢,各项力学性能都较好。据文献[11]报道,50 g的正畸力作用于大鼠第一磨牙,牙周组织改建最为明显。
目前普遍认为正畸牙齿移动是机械力作用于牙周韧带后产生牙周组织改建的生物力学现象,这种复杂的过程既与合成代谢和分解代谢时牙周韧带和牙槽骨细胞(破骨细胞、成骨细胞、骨细胞)活动水平相关[12],还与各种信号通路、炎症反应、细胞因子等变化有密切的关系,这些细胞和分子在转录、转录后和翻译水平的严格调控可影响牙齿移动的速度。破骨细胞激活是正畸牙齿移动中的第一步和限速步骤。然而,破骨细胞不能单独发挥作用,需要来自成骨细胞、骨细胞的信号,以促使其成熟、活化及靶向特异性骨吸收。成骨相关因子在加速正畸牙齿移动过程中也会起到一定的作用。研究发现微小RNA在成骨和破骨信号传递过程中起重要作用。microRNA-30d属于miR-30家族成员之一,位于人类染色体8q24.22。研究[13-14]发现miR-30家族的成员在心脏细胞中高度表达,与肺及乳腺肿瘤的发生、细胞的衰老等密切相关。miR-30家族绑定在Runx2 mRNA的3’末端翻译区,通过靶向Runx2负调节BMP-2诱导的成骨细胞分化[15]。PENG等[16]研究发现,microRNA-30家族通过靶向Smad1和Runx2负调节成骨分化。同时,转化生长因子-β可以通过BMP/Smad信号通路募集组蛋白去乙酰化酶下调micro-RNA-30d的表达[17]。
Runx2是骨组织生长发育过程中非常重要的转录因子,Runx2与成骨细胞异性顺式作用元件结合后,激活骨钙素、骨桥蛋白、骨涎蛋白和Ⅰ型胶原基因的转录与表达,还可以诱导骨保护素与Runx2结合,减少破骨细胞的产生[18]。Runx2表达受维生素D3、TGF-β/BMP-2、Wnt等多条信号传导通路的调控。同时,BMP/Smad信号通路对Runx2的表达也会产生影响,已经证实Runx2为Smad信号途径下游的效应分子,BMP的抑制和缺陷可以显著下调Runx2的表达。Runx2可以与Smad1和Smad5相互作用,增强成骨特异性基因的表达[19]。此外,微小RNA也可以控制Runx2的成骨活性并影响成骨细胞的成熟[20]。
本研究中,PAOO+TM组和TM组的Runx2和ALP的表达水平在加力3 d和7 d后逐渐增加,于7 d达到峰值,随后逐渐降低。7 d时,PAOO+TM组Runx2和ALP的表达量低于TM组,PAOO+TM组microRNA-30d的表达量明显高于TM组。因此,推测PAOO加速正畸牙齿移动可能是通过抑制成骨合成代谢过程,将移动牙区域的骨密度维持在相对较低的水平,进而加速正畸牙齿移动。
| [1] |
AMIT G, JPS K, PANKAJ B, et al. Periodontally accelerated osteogenic orthodontics (PAOO)-a review[J]. J Clin Exp Dent, 2012, 4(5): e292-e296. DOI:10.4317/jced.50822 |
| [2] |
ONG MMA, WANG HL. Periodontic and orthodontic treatment in adults[J]. Am J Orthod Dentofac Orthop, 2002, 122(4): 420-428. DOI:10.1067/mod.2002.126597 |
| [3] |
WILCKO WM, WILCKO T, BOUQUOT JE, et al. Rapid orthodontics with alveolar reshaping:two case reports of decrowding[J]. Int J Periodontics Restorative Dent, 2001, 21(1): 9-19. |
| [4] |
周昕, 陈行素, 耿忠楷, 等. PAOO技术辅助牙移动的临床应用进展[J]. 口腔医学, 2017, 37(5): 474-476. DOI:10.13591/j.cnki.kqyx.2017.05.021 |
| [5] |
ALIKHANI M, SANGSUWON C, ALANSARI S, et al. Biphasic theory:breakthrough understanding of tooth movement[J]. J World Fed Orthod, 2018, 7(3): 82-88. DOI:10.1016/j.ejwf.2018.08.001 |
| [6] |
HUANG HC, WILLIAMS RC, KYRKANIDES S. Accelerated orthodontic tooth movement:molecular mechanisms[J]. Am J Orthod Dentofacial Orthop, 2014, 146(5): 620-632. DOI:10.1016/j.ajodo.2014.07.007 |
| [7] |
任大鹏. ERK1/2-Runx2信号通路介导周期性张应力作用下牙周膜成纤维细胞成骨分化的机制研究[D].济南: 山东大学, 2016. http://cdmd.cnki.com.cn/Article/CDMD-10422-1017003673.htm
|
| [8] |
荆译萱, 赵红燕. 微小核糖核酸对成骨和破骨细胞分化及功能的调节作用[J]. 中华骨质疏松和骨矿盐疾病杂志, 2017, 10(6): 589-596. DOI:10.3969/j.issn.1674-2591.2017.06.015 |
| [9] |
TIAN YL, LIU J, BAI XF, et al. MicroRNA expression profile of surgical removed mandibular bone tissues from patients with mandibular prognathism[J]. J Surg Res, 2015, 198(1): 127-134. DOI:10.1016/j.jss.2015.04.071 |
| [10] |
韩旻轩.大鼠正畸牙齿移动过程中张力侧牙周组织RUNX2、OSX、OPN的表达[D].南京: 南京医科大学, 2013. http://cdmd.cnki.com.cn/Article/CDMD-10312-1013286446.htm
|
| [11] |
常悦, 张小平, 王明洁, 等. 不同正畸力对大鼠牙周组织改建的相关研究[J]. 牙体牙髓牙周病学杂志, 2015, 25(9): 529-533. DOI:10.15956/j.cnki.chin.j.conserv.dent.2015.09.004 |
| [12] |
FERGUSON DJ, VAID NR, WILCKO MT. Assessing accelerated tooth movement techniques on their own catabolic merits:a review[J]. J World Fed Orthod, 2018, 7(4): 122-127. DOI:10.1016/j.ejwf.2018.11.003 |
| [13] |
CHAN LW, WANG FF, MENG F, et al. MiR-30 family potentially targeting PI3K-SIAH2 predicted interaction network represents a novel putative theranostic panel in non-small cell lung cancer[J]. Front Genet, 2017, 8: 8. DOI:10.3389/fgene.2017.00008 |
| [14] |
OUZOUNOVA M, VUONG T, ANCEY PB, et al. MicroRNA miR-30 family regulates non-attachment growth of breast cancer cells[J]. BMC Genomics, 2013, 14: 139. DOI:10.1186/1471-2164-14-139 |
| [15] |
WU TT, ZHOU HB, HONG YF, et al. MiR-30 family members negatively regulate osteoblast differentiation[J]. J Biol Chem, 2012, 287(10): 7503-7511. DOI:10.1074/jbc.m111.292722 |
| [16] |
PENG SP, GAO D, GAO CD, et al. MicroRNAs regulate signaling pathways in osteogenic differentiation of mesenchymal stem cells (Review)[J]. Mol Med Rep, 2016, 14(1): 623-629. DOI:10.3892/mmr.2016.5335 |
| [17] |
LIU L, LIN WJ, ZHANG Q, et al. TGF-β induces miR-30d down-regulation and podocyte injury through Smad2/3 and HDAC3-associated transcriptional repression[J]. J Mol Med, 2016, 94(3): 291-300. DOI:10.1007/s00109-015-1340-9 |
| [18] |
李悦, 宫萍, 王燕, 等. Runx2基因调控致病的研究进展[J]. 医学综述, 2018, 24(11): 2081-2086. DOI:10.3969/j.issn.1006-2084.2018.11.001 |
| [19] |
李安娜.BMP——Smad信号通路在成骨与破骨细胞生成中的作用[D].济南: 山东大学, 2014.
|
| [20] |
ZHANG Y, XIE RL, CROCE CM, et al. A program of microRNAs controls osteogenic lineage progression by targeting transcription factor Runx2[J]. Proc Natl Acad Sci USA, 2011, 108(24): 9863-9868. DOI:10.1073/pnas.1018493108 |
 2019, Vol. 48
2019, Vol. 48




