文章信息
- 张艳芳, 孙瑞芬, 郭树春, 侯建华.
- ZHANG Yan-fang, SUN Rui-fen, GUO Shu-chun, HOU Jian-hua.
- 向日葵V-ATPase a3亚基基因的克隆及表达分析
- Cloning and Expression Analysis of V-type Proton ATPase Subunit a3 Gene in Sunflower (Helianthus annuus L.)
- 中国生物工程杂志, 2017, 37(5): 19-27
- China Biotechnology, 2017, 37(5): 19-27
- http://dx.doi.org/DOI:10.13523/j.cb.20170503
-
文章历史
- 收稿日期: 2016-12-08
- 修回日期: 2017-02-10
2. 内蒙古农牧业科学院 呼和浩特 010031
2. Inner Mongolia Academy of Agricultural & Animal Husbandry Sciences, Huhhot 010031, China
盐胁迫是自然界中主要的非生物胁迫之一,土壤中过多的可溶性盐会使植物吸收水分的能力受限,使植物在形态、生理、生化上发生改变,从而导致植物生长受阻。植物的抗盐性是指克服土壤高离子浓度造成的渗透胁迫和离子本身产生的毒害,在盐渍环境中生长和发育。植物对盐胁迫的反应形式多种多样,其中ATPase在胞内离子转运和植物抗盐过程中发挥着重要作用[1]。ATPase,又称为三磷酸腺苷酶,是一类能将三磷酸腺苷(ATP)催化水解为二磷酸腺苷(ADP)和磷酸根离子的酶。植物细胞膜上的ATP酶有3类:分别是位于细胞膜上的P型ATPase、位于液泡膜上的V型ATPase,以及位于线粒体内膜和叶绿体类囊体膜上的F型ATPase。V型ATPase是20世纪80年代Anraku等首次在酵母细胞的液泡膜上发现的一种运输质子的ATP酶,命名为液泡(vacuolar)ATP酶,简称为V-ATPase[2]。V-ATPase或称为V型泵(V-type pump),广泛分布在真核生物细胞原生质膜和细胞器(高尔基体、溶酶体、内膜体和分泌颗粒等)的内膜系统中,在植物细胞中主要存在于液泡膜上。V-ATPase利用水解细胞质中ATP产生的能量将细胞质中的H+泵入液泡,建立跨液泡膜的质子驱动力(proton motive force,PMF),从而驱动溶质在细胞质和液泡间跨液泡膜转运,维持细胞质溶质等相对恒定,保证生命活动的正常进行[3]。V-ATPase基因因其在生命活动中的重要性和实际意义,成为当前H+-ATPase家族中一个异常活跃的研究热点。柳树V-ATPase B亚基基因、甘蔗V-ATPase c亚基基因、二色补血草V-ATPase C亚基(Lb VHA-C)和大豆V-ATPase A亚基基因GmVHA-A的表达受盐胁迫强烈诱导,在盐胁迫的过程中发挥重要作用[4-7]。李平华[8]从盐地碱蓬中克隆了V-ATPase的B、H、c亚基基因,认为盐胁迫明显诱导了V-ATPase的B、H、c亚基基因表达,进一步证明这些基因表达存在协同作用。从碱蓬中克隆的V-ATPase B、C和H亚基基因转紫花苜蓿的过表达,提高了转基因苜蓿的耐盐碱性[9]。赵艳军[10]从小麦中克隆了V-ATPase B、F、G、H、c亚基基因,研究发现小麦耐盐突变体49 V-ATPase各亚基(A、B、C、D、F、G、H、a、c、d)在受到非生物胁迫时,表达量协调增高,并且V-ATPase B亚基基因的过量表达可以明显增强盐胁迫下转基因植株的耐盐性。刘娜等[11]克隆了棉花V-ATPase A亚基基因,认为棉花V-ATPase A亚基基因能够响应干旱胁迫应答。袁秀云等[12]克隆了蝴蝶兰V-ATPase E亚基基因,认为该基因受低温诱导,响应非生物胁迫。
向日葵(Helianthus annuus L.)作为盐渍土上的先锋作物,V-ATPase基因在其抵抗盐胁迫上起着至关重要的作用,但向日葵V-ATPase基因克隆与表达分析的相关研究报道甚少。本研究以油葵品种P50(内葵杂4号)为试验材料,克隆了向日葵V-ATPase a3亚基基因的cDNA全长,并对该基因的开放阅读框(open reading frame, ORF)进行了生物信息学分析;进行了不同非生物胁迫条件下V-ATPase a3亚基基因的表达模式分析。研究结果对探索向日葵V-ATPase基因的功能及利用具有重要意义,为进一步解析向日葵对盐胁迫的分子调节机制提供新线索。
1 材料与方法 1.1 试验材料处理油葵杂交种P50(内葵杂4号),由内蒙古农牧业科学院作物研究所向日葵课题组提供。试验在内蒙古农牧业科学院生物技术研究中心植物培养室中进行。材料处理参考之前的研究[13]。
1.2 试剂试剂RNAiso Plus、PrimeSript RT reagent Kit with gDNA Eraser(Perfect Real Time)、Gflex DNA Polymerase、SYBR Premix Ex Taq Ⅱ(Tli RNaseH Plus)、LA Taq购于TaKaRa公司;SMARTer RACE 5′/3′ Kit购于Clontech公司;High Pure PCR Product Purification Kit购于Roche公司;pGM-T Cloning Kit、E.coli TOP10购于天根生化试剂(北京)有限公司;其他常规试剂均为进口分装或国产分析纯。全部引物合成在TaKaRa公司完成,序列测定在南京金斯瑞生物工程技术服务有限公司完成,引物序列见表 1。
| Primer type | Primer name | Prime sequence(5′-3′) |
| 目的片段特异性引物 | ATP-F1 | TGGGCACTCAGCTTGGCACATT |
| Target gene primers | ATP-R1 | CTCACGTCTAAACACTAACC |
| 5′RACE引物 | ATP-R2 | TTCATCCTCGTCACCGGGTAAT |
| 5′RACE primers | ATP-R3 | TACGAGTAACACTCCAATCG |
| 3′RACE引物 3′RACE primers |
ATP-F4 | TTCCACCCTTTCTCCTTCGCATTA |
| cDNA的ORF特异性引物 | ATP-ORF-F1 | TTACGTGGTGAACTGATGAGAC |
| ORF primers of cDNA | ATP-ORF-R1 | GGGATAGAGCGACCTTGC |
| 实时荧光定量PCR引物 | ATP-qF1 | TACTACTATCCCTAATCGC |
| RT-PCR primers | ATP-qR1 | ATGACTTGCCTCCACTT |
| 内参基因18S rRNA引物 | QSP1 | AGAAACGGCTACCACATCCA |
| Inner control gene18S rRNA primers | QSP2 | TTGTTATTTATTGTCACTACCTCCC |
1.3 方法 1.3.1 总RNA的提取及cDNA合成
提取P50的总RNA,测其完整性和纯度。合成第一链cDNA。
1.3.2 向日葵V-ATPase a3亚基基因cDNA全长克隆(1) 向日葵V-ATPase a3亚基基因cDNA已知序列验证。根据高通量测序获得的盐胁迫向日葵转录组[14]结果得到的V-ATPase a3亚基基因(Unigene32233) 序列两端设计特异性引物ATP-F1和ATP-R1(表 1)。以120mmol/L NaCl胁迫处理3d的P50叶cDNA为模板,按Tks Gflex DNA Polymerase聚合酶说明书配制PCR反应体系,进行如下反应程序:94℃ 1min,98℃ 10s,55℃ 15s,68℃ 30s,30个循环,68℃ 10min。
(2) 向日葵V-ATPase a3亚基基因RACE全长克隆。以120mmol/L NaCl胁迫3d的P50幼苗叶片的Total RNA为模板,反转录合成5′ cDNA和3′ cDNA,按PrimeSript RT reagent Kit with gDNA Eraser(Perfect Real Time)说明书配制PCR反应体系。5′端采用巢式PCR扩增,Outer PCR反应程序:94℃ 1min,98℃ 10s,55℃ 15s,68℃ 60s,25个循环。Inner PCR反应程序:94℃ 1min,98℃ 10s,55℃ 15s,68℃ 60s,30个循环,68℃ 10min。3′端PCR反应程序:94℃ 5min,94℃ 30s,68℃ 30s,72℃ 3min,35个循环,72℃ 10min。将PCR产物回收纯化,加“A”尾,克隆并测序。将5′ RACE序列、中间序列和3′ RACE序列拼接。
1.3.3 向日葵V-ATPase a3亚基基因cDNA开放阅读框的克隆在其开放读码框两端设计特异引物,进行ORF的PCR扩增。反应程序:94℃ 1min;98℃ 10s,68℃ 30s,72℃ 3min,35个循环;72℃ 10min。将PCR产物回收纯化,克隆并测序。
1.3.4 向日葵V-ATPase a3亚基基因的生物信息学分析运用NCBI数据库(http://www.ncbi.nlm.nih.gov/gorf/gorf.html)中的查找和翻译V-ATPase a3亚基基因cDNA序列的CDS(coding sequence);运用在线分析软件ProtParam(http://www.expasy.org/tools/protparam.html)对该基因编码蛋白质进行理化性质分析;运用在线分析软件ProtScale(http://www.expasy.org/tools/protscale. html)的Kyte & Doolittle算法进行疏水性/亲水性预测;运用在线分析软件TMHMM Server 2.0(http://www.cbs.dtu.dk/services/TMHMM/)分析目的蛋白的跨膜结构域;运用PSORT(http//psort.hgc.jp/form.html)分析蛋白质的亚细胞定位;运用NCBI数据库中的Blast对氨基酸序列进行同源性搜索和比对分析;运用DNAMAN软件将目的序列与已报道其他物种的V-ATPase a3亚基基因同源序列进行多重比对,用MEGA5.0软件构建系统进化树。
1.3.5 向日葵V-ATPase a3亚基基因响应非生物胁迫的表达分析按SYBR Premix Ex Taq Ⅱ(Tli RNaseH Plus)试剂盒说明书配制反应体系,进行如下反应程序:95℃ 30s;95℃ 5s,60℃ 30s,40个循环。每个样品进行3次重复检测;利用2-ΔΔCT法计算目的基因相对表达量。
2 结果与分析 2.1 向日葵V-ATPase a3亚基基因全长序列的获得 2.1.1 向日葵V-ATPase a3亚基基因cDNA已知序列验证以盐胁迫向日葵转录组测序获得的已知序列设计特异性引物ATP-F1和ATP-R1,进行PCR扩增,得到1条约600bp(图 1)的片段,克隆测序结果与已知序列一致。
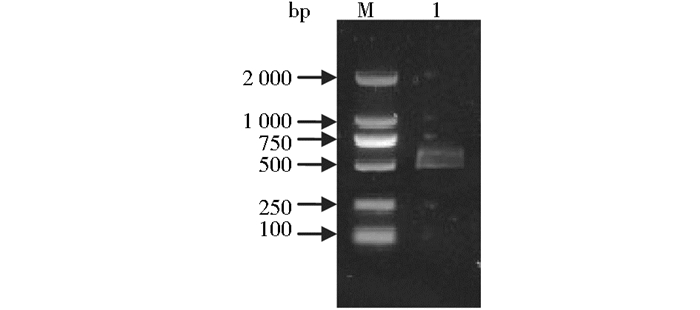
|
| 图 1 V-ATPase a3亚基基因序列验证PCR扩增结果 Figure 1 PCR amplification of sequence verification of V-type proton ATPase subunit a3 gene M:DL2 000 DNA marker |
用RNAiso plus提取NaCl处理的向日葵幼苗根、下胚轴和叶的总RNA,完整性好、纯度高,完全满足下一步RACE实验的要求。通过5′ RACE扩增,获得长2 301bp序列[图 2(a)]。通过3′ RACE扩增,获得长388bp序列[图 2(b)]。将两端序列与中间序列进行拼接,在其开放读码框两端设计1对特异引物,经RT-PCR扩增,获得2 601bp的序列[图 2(c)]。
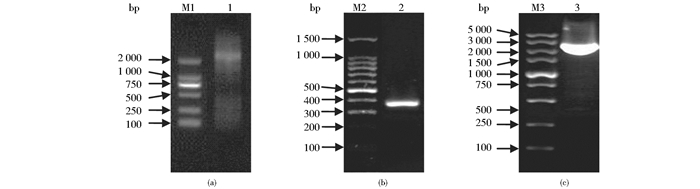
|
| 图 2 V-ATPase a3亚基基因RACE及cDNA全长PCR扩增结果 Figure 2 PCR amplification of V-type proton ATPase subunit a3 gene RACE and full-length cDNA (a)5′RACE M1 DL2 000 DNA Marker (b)3′RACE M2 100bp DNA marker (c)cDNA full Length M3 DL5 000 DNA marker |
向日葵V-ATPase a3亚基基因序列cDNA全长2 873bp,含有完整的CDS,自起始密码子ATG至终止密码子TAA长度为2 469bp,编码822个氨基酸(图 3),5′非编码区长109bp,3′非编码区长295bp,GenBank登录号为KU315054。该基因编码蛋白的理论分子质量为204.55kDa,等电点为6.29。对V-ATPase基因编码的氨基酸组成进行分析,其中含量最高的是亮氨酸(Leu),占11.6 %;其次是丙氨酸(Ala)和谷氨酸(Glu),分别占7.3 %;含量最少的为半胱氨酸(Cys),占1.1 %。分子式为C4226H6524N1054O1199S30,总原子数为13 033,亲水性平均系数为0.077。V-ATPase a3亚基基因编码的蛋白质为疏水性跨膜蛋白,有6个跨膜区, 其亚细胞定位:质膜=0.6、高尔基体=0.4、内质网(膜)=0.3,微体(过氧化物酶体)=0.3,最后将该蛋白质定位在质膜上的可能性大。通过Blast比对发现,向日葵V-ATPase a3亚基基因与已报道的朝鲜蓟(KVH88978.1)、芝麻(XP_011088459.1)、枣(XP_015892427.1)、川桑(XP_010096180.1)、辣椒(XP_016539814.1)、绒毛状烟草(XP_009602232.1)、烟草(XP_016478330.1)、林烟草(XP_009785999.1)、潘那利番茄(XP_015056376.1) 和番茄(XP_004230865.1) 的V-ATPase a3亚基基因氨基酸序列具有较高的一致性,分别为81%、80%、80%、80%、80%、80%、80%、80%、80%和80%。利用DNAMAN软件,将向日葵V-ATPase a3亚基基因推导的氨基酸序列与以上10个物种V-ATPase a3亚基基因推导的氨基酸序列进行多重序列比对分析,结果表明这些V-ATPase a3亚基基因在氨基酸序列上具有高度保守性(图 4)。对向日葵与朝鲜蓟等10种植物的V-ATPase a3亚基基因编码的氨基酸序列进行系统进化分析,表明相同科植物可以聚为一类,如辣椒、潘那利番茄和番茄都属于茄科,向日葵和朝鲜蓟都属于菊科,向日葵与朝鲜蓟的亲缘关系最近(图 5)。

|
| 图 3 V-ATPase a3亚基基因cDNA的CDS序列及其推导的氨基酸序列 Figure 3 CDS and its deduced amino acid sequence of V-type proton ATPase Subunit a3 gene cDNA |

|
| 图 4 V-ATPase a3亚基基因编码的氨基酸序列与其他物种同源氨基酸序列多重比对 Figure 4 Alignment of the deduced V-type proton ATPase subunit a3 gene amino acid sequence and its homologus amino acid sequences from other species |

|
| 图 5 V-ATPase a3亚基基因编码的氨基酸序列与其他物种同源序列的进化树分析 Figure 5 Phylogenetic analysis of the deduced V-type proton ATPase subunit a3 gene compare sunflower with other species |
实施荧光定量PCR分析表明,向日葵幼苗受到NaCl、ABA和PEG的胁迫后,V-ATPase a3亚基基因的表达量较对照均有不同程度增加。
2.3.1 V-ATPase a3亚基基因受盐胁迫的表达分析实施荧光定量PCR分析表明,随胁迫时间的延长,V-ATPase a3亚基基因的表达量呈现先升高后降低的趋势,其中胁迫24h时基因的表达量最大,120mmol/L、150mmol/L和180mmol/L胁迫处理的表达量分别是对照的57.95倍、82.33倍和134.67倍(图 6)。
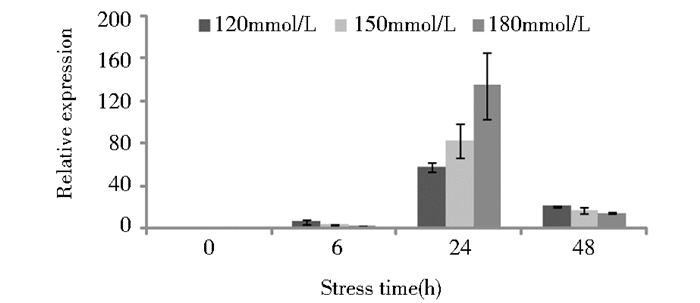
|
| 图 6 不同浓度NaCl胁迫下V-ATPase a3亚基基因在向日葵叶中的表达分析 Figure 6 Expression analysis of V-type proton ATPase subunit a3 gene in sunflower leaf under different NaCl concentration stress |
实施荧光定量PCR分析表明,向日葵幼苗受到不同浓度ABA诱导,随时间的延长,V-ATPase a3亚基基因的表达量呈现逐渐升高的趋势,其中诱导48h基因的表达量最大,5μmol/L、10μmol/L和50μmol/L诱导处理的表达量分别是对照的23.05倍、29.18倍和13.96倍(图 7)。
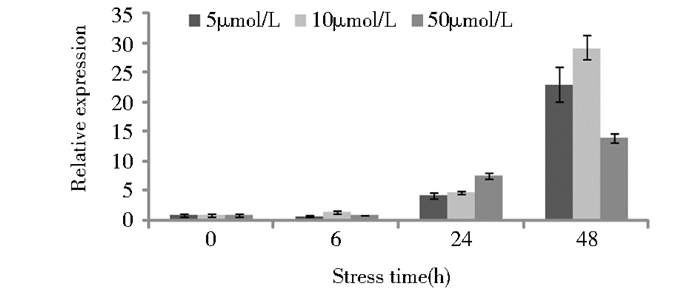
|
| 图 7 不同浓度ABA诱导下V-ATPase a3亚基基因在向日葵叶中的表达分析 Figure 7 Expression analysis of V-type proton ATPase subunit a3 gene in sunflower leaf under different ABA concentration stress |
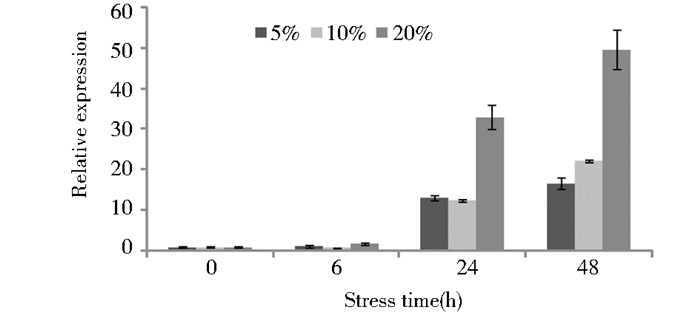
用不同浓度PEG处理模拟干旱胁迫,实施荧光定量PCR分析表明,向日葵幼苗受到不同浓度PEG胁迫,随时间的延长,V-ATPase a3亚基基因的表达量呈现逐渐升高的趋势,其中胁迫48h基因的表达量最大,5%、10%和20%胁迫处理的表达量分别是对照的16.76倍、22.27倍和49.52倍(图 8)。
|
| 图 8 不同浓度PEG胁迫下V-ATPase a3亚基基因在向日葵叶中的表达分析 Figure 8 Expression analysis of V-type proton ATPase subunit a3 gene in sunflower leaf under different PEG concentration stress |
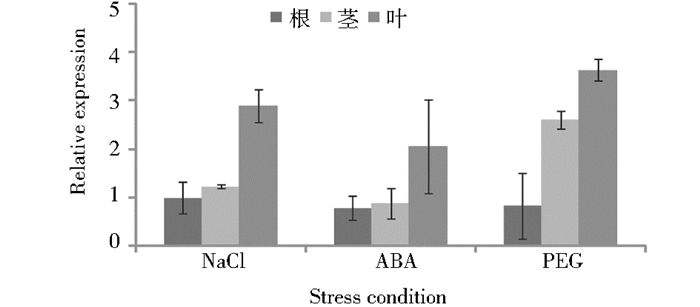
向日葵幼苗受到NaCl、ABA和PEG的胁迫后,V-ATPase a3亚基基因在根、下胚轴、叶中的相对表达量有差异,均为在叶中表达量最高,在茎中其次,在根中最少。其中,180mmol/L NaCl胁迫处理24h,叶中的表达量是根中的1.23倍;下胚轴中的表达量是根中的2.89倍。10μmol/L ABA胁迫处理48h,叶中的表达量是根中的1.13倍,下胚轴中的表达量是根中的2.62倍。20% PEG胁迫处理48h,叶中的表达量是根中的3.11倍,下胚轴中的表达量是根中的4.32倍(图 9)。
|
| 图 9 V-ATPase a3亚基基因在不同胁迫处理下的器官特异性表达 Figure 9 Expression analysis of V-type proton ATPase subunit a3 gene in various organs of sunflower under different kinds of stress |
新一代高通量测序技术的优点在于可以准确地分析基因表达差异、基因结构变异、筛选分子标记(SNP或SSR)等生命科学的重要问题。基因表达谱测序是直接对某一物种或特定细胞在某一功能状态下产生的mRNA进行高通量测序,可以用来研究基因的表达差异情况。本研究对高通量测序获得的盐胁迫向日葵数字化基因表达谱测序结果进行分析,发现V-ATPase a3亚基基因在120mmol/L NaCl胁迫处理P50幼苗叶片第3天的表达量最高,因此选择P50胁迫处理3天的叶片cDNA为模板,克隆了V-ATPase a3亚基基因cDNA全长。
V-ATPase与植物抗盐性密切相关。当植物处于盐胁迫条件下,V-ATPase显示出高度的灵敏性和可塑性,对保证细胞的正常生命活动具有重要的调节作用。在盐、干旱、寒冷、酸、缺氧和土壤中过量的重金属胁迫条件下,细胞依靠维持或调节V-ATPase活性得以存活[15]。V-ATPase由多亚基组成,其结构在进化中高度保守,其中很多亚基均具有多种亚型,由基因组上不同的基因编码[16]。邓林和陈少良[1]认为盐胁迫提高抗盐植物V-ATPase活性,主要是通过增加V-ATPase的主要功能亚基的基因表达以及蛋白质合成。研究发现盐胁迫后拟南芥、烟草悬浮细胞和甜菜V-ATPase a亚基基因的表达量增加[17-20]。V-ATPase a3亚基作为向日葵V-ATPase主要功能亚基,受NaCl、ABA和PEG胁迫调节,在转录水平参与了非生物胁迫应答,在不同胁迫条件下的表达模式不同,存在器官特异性表达差异。向日葵在盐碱地得以正常生长,V-ATPase a3亚基基因在将细胞质中的H+泵入液泡,维持细胞质的pH稳态平衡和建立跨膜的质子电化学梯度中发挥作用。然而,V-ATPase由多亚基组成,向日葵的V-ATPase基因耐盐的分子机制需要多亚基协同作用,在胁迫中的调控机制还是未知,有待于进一步研究。
综上所述,本研究完成了向日葵耐盐相关V-ATPase a3亚基基因的克隆与表达分析,为向日葵耐盐相关基因的利用和进一步揭示向日葵耐盐分子调控机制奠定坚实的基础。
| [1] |
邓林, 陈少良.
ATPase与植物抗盐性. 植物学报, 2005, 22(Supplemen) : 11–21.
Deng L, Chen S L. ATPase and salt resistance in plants. Chinese Bulletin of Botany, 2005, 22(Supplemen) : 11–21. |
| [2] | Ohsumi Y, Anraku Y. Active transport of basic amino acids driven by a proton motive force in vacuolar membrane vesicles of Saccharomyces cerevisiae. Journal of Biological Chemistry, 1981, 256(5) : 2079–2082. |
| [3] |
冯兰东. 盐地碱蓬液泡膜H+-ATPase的分离、纯化及亚基分析. 济南: 山东师范大学, 生命科学学院, 2006. Feng L D. Isolation, Purification and Subunit Analysis of Tonoplast H+-ATPase from Suaeda salsa L.. Jinan:Shandong Normal University, College of Life Sciences, 2006. |
| [4] |
李敏, 张健, 冯立国, 等.
柳树液泡膜ATP酶B亚基基因克隆及在盐胁迫下的表达分析. 江苏农业学报, 2013, 29(5) : 1149–1153.
Li M, Zhang J, et al. Cloning and expression analysis of vacuolar ATPase B subunit gene VHA-B in leaves of salt-stressed willow. Jiangsu Journal of Agricultural Sciences, 2013, 29(5) : 1149–1153. |
| [5] |
许志宇, 王俊刚, 杨本鹏, 等.
甘蔗液泡ATP酶C亚基基因克隆及序列分析. 生物技术通报, 2010(9) : 82–86.
Xu Z Y, Wang J G, Yang B P, et al. Cloning and sequence analysis of vacuolar ATPase subunit C gene from Saccharum officinarum L. Biotechnology Bulletin, 2010(9) : 82–86. |
| [6] |
蒋丽丽, 杨传平, 徐晨曦, 等.
二色补血草液泡膜H+-ATP酶C亚基(Lb VHA-C)基因克隆和表达分析. 植物生理学报, 2009, 45(12) : 1155–1159.
Jiang L L, Yang C P, Xu C X, et al. Cloning and expression analysis of Lb VHA-C Gene from Limonium bicolor(Bunge) Kuntze. Plant Physiology Communications, 2009, 45(12) : 1155–1159. |
| [7] |
孙艳香, 冯雪, 吴智艳, 等. 大豆液泡膜H+-ATPase A亚基cDNA的克隆与表达分析. 见: 中国植物学会, 中国植物学会七十五周年年会, 兰州, 2008, 兰州, 兰州大学出版社, 2008: 295-296. Sun Y X, Feng X, Wu Z Y, et al. Cloning and Expression Analysis of the cDNA of V-H+-ATPase Subunit A in Soybean. In:Chinese society of Botany, Documentary of abstracts of the seventy-five annual meeting of the Chinese society of Botany, Annual meeting of the seventy-five year anniversary of Botanical Society of China, Lan Zhou, 2008, Lanzhou, Lanzhou University Press, 2008:295-296. |
| [8] |
李平华. 盐胁迫下盐地碱蓬液泡膜质子泵表达分析及过量表达SsNHX1-AVP1对拟南芥耐盐性的影响. 济南: 山东师范大学, 生命科学学院, 2003. Li P H. Expression Analysis of Tonoplast Proton Pumps of Suaeda salsa and Effect of Co-overexpression of SsNHX1 and AVP1 on the Salt Tolerance of Arabidopsis thaliana Under Salt Stress. Jinan:Nanjing Normal University, College of Life Sciences, 2003. |
| [9] | Wang F W, Wang C, Sun Y, et al. Overexpression of vacuolar proton pump ATPase (V-H+-ATPase)subunitsB, C and H confers tolerance to salt and saline-alkali stresses in transgenic alfalfa (Medicagosativa L. Journal of Integrative Agriculture, 2016, 15(10) : 2279–2289. DOI:10.1016/S2095-3119(16)61399-0 |
| [10] |
赵艳军. 小麦V-H+-ATPase基因的克隆及耐盐性研究. 石家庄: 河北师范大学, 生命科学学院, 2008. Zhao Y J.Cloning and Salt-tolerance Research of V-H+-ATPase Subunit Genes in Wheat. Shijazhuang:Hebei Normal University, College of Life Sciences, 2008. |
| [11] |
刘娜, 倪志勇, 朱燕飞, 等.
棉花液泡膜H+-ATPase A亚基的克隆及表达载体的构建. 分子植物育种, 2015, 13(3) : 541–546.
Liu N, Ni Z Y, Zhu Y F, et al. Cloning of subunit H of V-H+-ATPase in cotton and constructing its plant. Expression Vector Molecular Plant Breeding, 2015, 13(3) : 541–546. |
| [12] |
袁秀云, 田云芳, 梁芳, 等.
蝴蝶兰液泡型ATP酶E亚基基因的克隆及序列分析. 河南农业科学, 2016, 45(6) : 104–110.
Yuan X Y, Tian Y F, Liang F, et al. Cloning and sequence analysis of vacuolar Proton-ATPase E subunit gene from Phalaenopsis amabilis. Journal of Henan Agricultural Sciences, 2016, 45(6) : 104–110. |
| [13] |
张艳芳, 孙瑞芬, 郭树春, 等.
向日葵E3泛素连接酶基因克隆及表达分析. 西北植物学报, 2016, 36(10) : 1933–1940.
Zhang Y F, Sun R F, Guo S C, et al. Cloning and expression analysis of E3 ubiquitin ligase gene HERC2 of sunflower (Helianthus annuus L.). Acta Botanica Boreali-occidentalia Sinica, 2016, 36(10) : 1933–1940. |
| [14] |
孙瑞芬, 张艳芳, 郭树春, 等.
基于RNA-Seq技术的盐胁迫向日葵转录组信息分析. 分子植物育种, 2015, 13(12) : 2736–2742.
Sun R F, Zhang Y F, Guo S C, et al. Analysis on transcriptome of sunflower under salt stress based on RNA-Seq technology. Molecular Plant Breeding, 2015, 13(12) : 2736–2742. |
| [15] | Dietz K J, Tavakoli N, Kluge C, et al. Significance of the V-type ATPase for the adaptation to stressful growth conditions and its regulation on the molecular and biochemical level. Journal of Experimental Botany, 2001, 52(363) : 1969–1980. DOI:10.1093/jexbot/52.363.1969 |
| [16] |
吴海平. 质子泵亚基Atp6vOd2在破骨细胞胞外酸化功能中的作用. 北京: 中国科学院研究生院, 2009. Wu H P. Role of Proton Pump Subunit Atp6vOd2 in Extracellular Acidification of Osteoclasts. Beijing:Graduate University of Chinese Academy of Sciences, 2009. |
| [17] | Hanitzsch M, Schnitzer D, Seidel T, et al. Transcript level regulation of the vacuolar H+-ATPase subunit isoforms VHA-a, VHA-E and VHA-G in Arabidopsis thaliana. Molecular Membrane Biology, 2007, 24(5-6) : 507–518. DOI:10.1080/09687680701447393 |
| [18] | Narasimhan M L, Binzel M L, Perezprat E, et al. NaCl regulation of tonoplast ATPase 70-kilodalton subunit mRNA in tobacco cells. Plant Physiology, 1991, 97(2) : 562–568. DOI:10.1104/pp.97.2.562 |
| [19] | Kirsch M, An Z, Viereck R, et al. Salt stress induces an increased expression of V-type H+-ATPase in mature sugar beet leaves. Plant Molecular Biology, 1996, 32(3) : 543–547. DOI:10.1007/BF00019107 |
| [20] | Lehr A, Kirsch M, Viereck R, et al. cDNA and genomic cloning of sugar beet V-type H+-ATPase subunit A and c isoforms:evidence for coordinate expression during plant development and coordinate induction in response to high salinity. Plant Molecular Biology, 1999, 39(3) : 463–475. DOI:10.1023/A:1006158310891 |
 2017, Vol. 37
2017, Vol. 37




