文章信息
- 吴林寰, 陆震鸣, 龚劲松, 史劲松, 许正宏.
- WU Lin-huan, LU Zhen-ming, GONG Jin-song, SHI Jin-song, XU Zheng-hong.
- 利用语义网技术实现的分布式异构食品微生物数据整合
- Integrating Distributed Heterogeneous Food Microorganism Data by Semantic Web Technology
- 中国生物工程杂志, 2017, 37(3): 124-132
- China Biotechnology, 2017, 37(3): 124-132
- http://dx.doi.org/DOI:10.13523/j.cb.20170317
-
文章历史
- 收稿日期: 2016-09-19
- 修回日期: 2016-12-13
随着基因组、转录组、代谢组等组学技术的大规模发展,生命科学的数据也呈现出了海量增长的趋势。在生命科学面临从实验驱动向大数据驱动转型的同时,对多维度的数据集成也提出了新的需求,包括从分子、细胞到生物系统的数据整合,跨物种的数据集成,多组学数据融合等。
长期以来,在异地、异构数据的数据整合方面已经形成了一些比较系统的理论和成熟的方法,包括建立数据仓库,或者通过建立视图 (view integration) 的方法进行整合。为了更好地进行数据整合,对于不同来源的数据,通常需要满足数据整合的一些基本原则[1],包括:①使用一个统一的名称和编码。无论使用什么样的数据整合框架或者方法,必须保证同样的一个描述对象用统一的名称来进行描述,并且具有唯一的编码。目前,尽管国际上已经提出了一些通用的规则和工具,如生命科学标识码 (life science identifier)、永久统一资源定位符 (persistent URLs) 等,但距离全面的应用还有比较大的差距[2]。尤其在生命科学领域,由于研究的目的、机构、技术手段各不相同,数据通常存储在不同机构维护的数据库中,采用不同的管理系统,因此,对于相同的描述内容,不同的数据库使用不同编码方式的现象还广泛存在。②尽可能采用国际通用的数据标准和语义学定义。生命科学领域的数据本身相对复杂,由于缺乏相应的标准,或者标准使用的局限性,对数据的定义和描述不一致,数据格式各异。因此,本体的使用就变得至关重要。目前,在生命科学领域广泛使用的本体如基因本体 (gene ontology)[3]对数据整合起到了极大的帮助。但是,尽管生物医学本体中心 (national center for biomedical ontologies) 项目已经发布了528个本体[4],其中真正为全球的研究团队接受并广泛使用的还是极少数。③在进行数据整合的同时,既要考虑各个数据库的独立性和数据的完整性,也需要保证数据的无缝链接,实现跨库检索和分析。但是由于数据处理过程的区别、数据质量控制的差异,以及数据之间存在着复杂的关联等原因,生命科学数据的无缝衔接往往难度极大,导致零散的数据难以成为系统的知识。
然而,异构数据的整合一直是一件极具挑战但却尤为必要的工作,尤其对于生命科学研究来说,通过将不同层面的数据进行整合,帮助发现隐藏的知识,才能真正实现数据驱动的科学研究的发展。
1 生命科学领域语义网数据库的现状语义网技术是一种数据描述和整合的方法,通过定义统一的资源标识符 (uniform resource identifier, URI) 和本体实现互联网上多源异构数据的关联。资源描述框架 (resource description framework, RDF) 是一个描述资源及其相互关系的简单模型,它是语义信息描述的有效手段,也是实现语义网的关键技术之一,通过定义包括主语、谓语和宾语的一套三元组的方式来描述资源,实现将互联网上的资源进行整合形成关联数据。
近年来,在生命科学领域,利用语义网技术进行数据整合已经有了较大的发展,一些重要的数据库已经开始利用网络本体语言 (web ontology language) 和RDF发布数据集。欧洲生物信息中心 (European Bioinformatics Institute, EBI) 目前利用RDF格式将其数据进行了关联,在平台上发布,提供利用SPARQL进行查询的接口[5],并且与UniProt数据库进行了合作。Uniprot[6]、GO等数据库也已经发布了基于RDF模型的数据集,并提供基于SPARQL语句进行查询的功能。BIO2RDF是一个利用语义网技术开发的开源项目,截至2014年7月,已经发布了包括KEGG、Pubmed、NCBI taxonomy等在内的35个RDF格式数据集,110亿个三元组。然而,由于该项目只是一个语义网技术上的实验性项目,所以在数据更新、数据关联和数据定义等方面,距离真正的应用还存在一定的差距[7]。另外,也有一些专题型数据库,依靠语义网技术进行了深度的数据整合。Ebola-KB是一个以埃博拉病毒为核心的知识库,利用语义网技术整合了包括基因组、蛋白质的结构和功能,以及来自于Pubmed的文献信息,同时提供了到Bio2RDF数据集的链接,利用这个平台,可以方便地将文献中关于埃博拉病毒的功能研究与生物信息学数据进行连接[8]。为了满足整合慢性肾脏疾病的实验数据和多层次生物学数据的需求,Simon等[9]利用语义学方法,整合了高通量肾脏实验数据,并利用肾脏和尿路系统疾病的本体,将多层次的生物学数据进行了系统的描述,再将其转换为RDF格式,提供基于SPARQL语句的查询,形成了一个肾脏疾病代谢途径的知识库。
2 FoodMicrobes数据库 2.1 主要数据源与数据内容食品发酵是微生物在工业中的一个重要应用,具有重大的产业价值。近年来,随着高通量测序技术的发展,产生了大量的基因组和元基因组数据,使得人们能够系统的分析食品微生物的代谢、微生物群落的相互作用及其对环境的反应机制。但是,数据分散的问题也同样存在,关于微生物的菌种、文献、基因和基因组、蛋白质、酶及代谢反应的信息存在不同的数据库中,对数据的检索和应用产生了极大的障碍。因此,我们利用关联数据的方法,整合了来自于不同数据源的食品微生物相关信息,形成了一个食品微生物的数据库,希望为食品微生物的研究人员提供一个方便的数据平台。
食品微生物主要是指用于食品生产的活的细菌、酵母或霉菌,由一种或多种微生物菌种或菌株组成。另外,微生物也会引起食物的腐烂、变质,引起人畜感染,这一类微生物通常称为食源性致病菌。在FoodMicrobes数据库中,我们总结了有文献记载用于食品微生物发酵过程的295种微生物,以及常见的食源性致病菌32种,系统整合了其物种、文献、基因和基因组序列、蛋白质、酶及代谢途径信息,同时整合了奶酪、泡菜等环境的元基因组信息, 形成了跨物种的,从宏观到微观、从分子到系统层面的数据整合。FoodMicrobes的主要数据源见表 1。
| 数据内容 | 数据来源 | 记录数 |
| Species | Global Catalogue of Microorganisms[10] | 295 |
| Strains | Global Catalogue of Microorganisms | 29 262 |
| Publications | Pubmed[11] | 126 718 |
| Patents | WIPOhttp:www.wipo/int/portal/en/index.html | 9 333 |
| Genomes | NCBI genome[12] | 552 |
| Genes | Genebank[13] | 49 630 |
| Proteins | Uniprot | 48 320 |
| GO | Gene ontology | 989 |
| Protein structure | PDB[14] | 1 025 |
| Enzyme | KEGG[15] | 1 342 |
| Pathway | KEGG | 118 |
| Metagenomes | MG-RAST[16] | 138 |
全球微生物菌种目录 (Global Catalogue of Microorganisms) 是由世界微生物数据中心 (world data center of microorganisms) 开发的微生物菌种资源数据库,系统搜集了来自全球43个国家103个微生物资源保藏中心所保藏,并可对外共享的微生物菌种资源信息。我们利用食品微生物发酵过程的295种微生物,从GCM中提取到了295条物种层面的信息和29 262条菌株层面的信息,原始数据以MySQL方式进行存储,所集成的物种信息包括微生物物种的分类地位、NCBI Taxonomy数据库的分类号、模式菌株、16S rRNA序列和参考文献。菌株层面的信息包括菌种的唯一菌株编号、名称、采集环境和采集人、历史信息、培养条件和生理生化性状等。文献与专利的原始数据以文本的方式进行存储,集成的内容包括文献的题目、关键词、摘要、作者、作者机构、发表期刊等。集成的专利信息包括专利的题目、摘要、专利号、专利申请人及专利权人等。基因组和基因信息来自NCBI数据库,包括基因的描述、起始位置和物种信息、基因组的描述、项目、状态及物种信息。蛋白质序列信息来自Uniprot数据库,主要包括蛋白质的定义,以及与基因组、基因功能、蛋白质结构和功能的关系信息。基因功能的信息来自GO数据库,主要包括特征、定义及分类信息。蛋白质结构数据库包括定义及其与蛋白质序列及物种的关系。酶及代谢途径信息来自KEGG数据库,包括酶的名字、历史及参与的反应以及代谢途径涉及的物种、反应、化合物和参考文献。微生物的元基因组数据来自MG-RAST数据库,包括元基因组测序项目的描述、采集环境、联系人信息和可公开的原始数据。可以看出,在FoodMicrobes数据库中进行整合的内容并不覆盖原始公开数据源中所有的数据项,这是因为FoodMicrobes数据库更侧重于建立数据的关联关系,并利用关联关系进行整合,所有这些数据记录所整合的内容也都包括数据源的链接,可以为用户提供查看详细信息的路径。
2.2 数据检索与使用FoodMicrobes数据库提供浏览和检索数据库等基本功能 (图 1)。数据检索分为对微生物的菌株和元基因组进行检索。针对微生物的菌株,可以对菌株的名称及用途进行检索;针对元基因组信息,可以检索元基因组的项目名称以及采集环境信息。在数据库的主页上,还提供了所集成数据的实时统计信息。
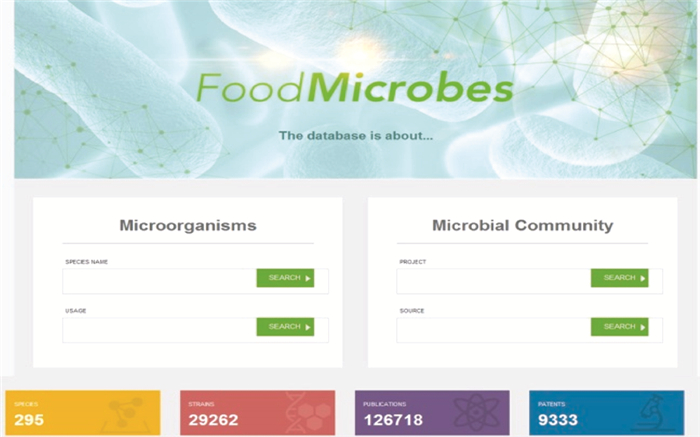
|
| 图 1 FoodMicrobes数据库检索界面 Figure 1 Search page of database |
检索数据库后,可以查看关于菌种和菌株的详细信息,其中菌株信息 (图 2) 来自于从全球主要保藏中心搜集的目录信息,此外,通过菌株的唯一编号所关联的菌株的文章、专利、基因组、核酸序列等信息也在此页面统一展示。菌种信息 (图 3) 是该物种所包括的所有菌株信息的集合,包括物种分类号、参考文献、模式菌株及关联的文献和专利信息。
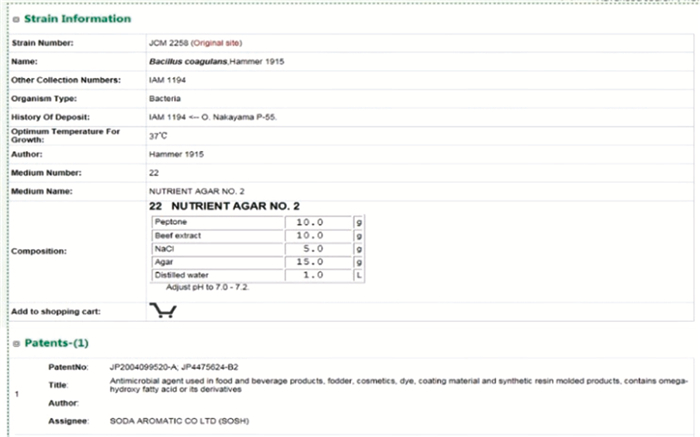
|
| 图 2 微生物的菌株目录信息 Figure 2 Strain catalogue of microorganisms |
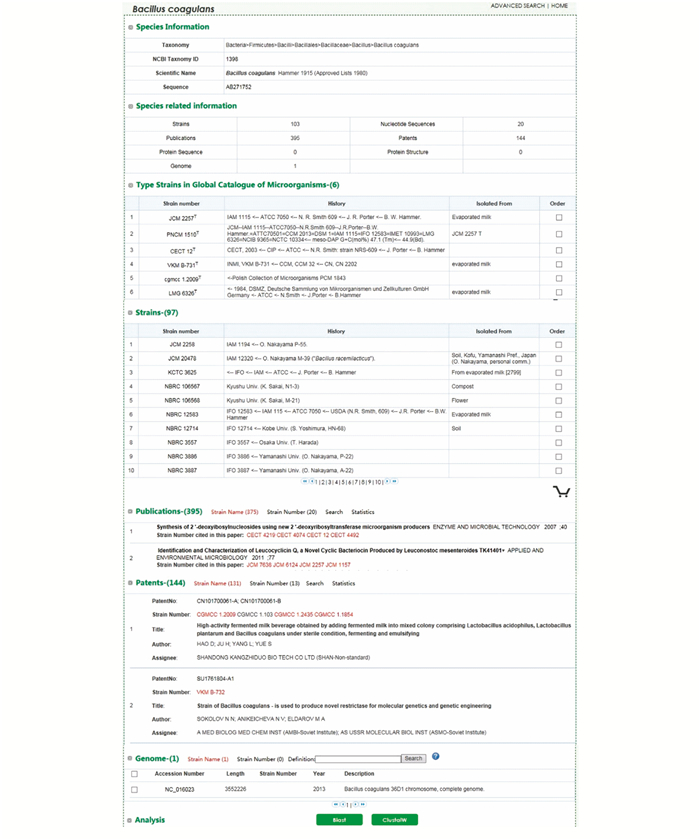
|
| 图 3 微生物的物种信息 Figure 3 Species information of microorganisms |
通过对元基因组的信息进行检索,可以获得目前在MG-RAST平台上已经公开的采集自食品发酵环境的元基因组测序项目的信息,包括项目的描述、采集地和联系信息,并且提供序列下载 (图 4)。

|
| 图 4 元基因组测序项目信息 Figure 4 Information of metagenomic sequencing project |
进一步对菌株信息进行查询,可以获得某一菌株所测序产生的基因组序列、基因序列,以及关联的蛋白质的序列、代谢途径和基因功能的列表 (图 5),并可查询其详细信息。从列表中可以看出,菌株与基因组、基因序列、菌株与基因功能、代谢途径与基因及酶之间都已经建立了关联。
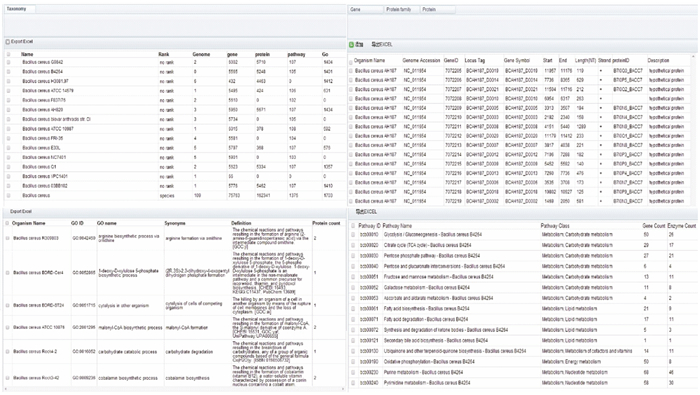
|
| 图 5 微生物物种的关联数据 Figure 5 Linked data of microorganisms |
RDF的基本数据模型包含三类对象:资源、属性和陈述。资源之间的关系通过属性和值来描述,资源是可拥有URI的任何事物,属性是拥有名称的资源,属性值是某个属性的值。
3.1 数据处理本体的目的是用来建立一套标准的描述和关系的定义,从而使不同数据之间能够准确的定义和相互理解,通常是由包括术语 (term) 以及术语间联系的一个列表组成的,同时还定义有属性、值的限制、不相交声明和对象间逻辑关系的说明。
在该数据库中,我们对来自不同数据源的数据进行了本体的定义 (图 6)。其中GO和Uniprot由于已经具有公开发表的RDF数据,为了促进数据的统一和共享,我们采用数据提供者发布的RDF格式数据并沿用了其所定义的本体。对未曾进行过定义的数据,如物种信息、基因和基因组信息、蛋白质序列与结构、代谢途径与酶等数据集,我们重新进行了本体的定义,并且在定义本体的时候,根据已经梳理的数据关联关系,加入了使数据进行关联的属性。
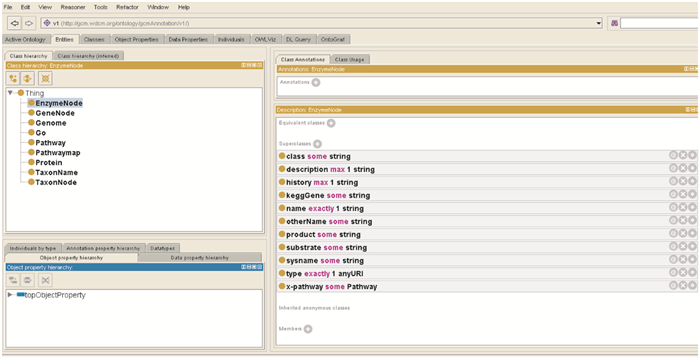
|
| 图 6 定义本体 Figure 6 Design of ontology |
由于在互联网上,内容相同或相似的资源可能使用不一致的名字,而内容不相同的资源,可能使用了相同的名称,这就会引起混淆和冲突。因此,需要利用命名空间来为所需要描述的资源进行定义。在该数据库中,除GO和Uniprot由于已经具有公开发表的RDF数据,我们沿用了其命名空间外,对其他数据集我们定义了唯一的命名空间,并赋予唯一的URI (图 7)。

|
| 图 7 定义基因组命名空间 Figure 7 Namespace of genome in Food Microbes databaes |
本数据库的主要数据内容分为组学相关的数据、物种相关数据和注释及功能相关数据 (图 8)。每一个数据集除了其本身的数据内容外,还需要在数据处理的过程中加入与其他数据集进行关联的内容。组学相关数据主要是以基因组为核心的数据,包括物种分类、基因组、基因序列和蛋白质序列。其中,物种分类信息通过NCBI的Taxonomy ID与基因组关联,基因序列通过NCBI的基因唯一标识与基因组和蛋白质序列关联。其中物种的NCBI的Taxonomy ID也与菌种信息进行关联,菌种信息再进一步通过菌种名称与菌株信息进行关联。每一个菌株都有一个唯一的菌株编号,该菌株编号在文献和专利发表时需要被引用。因此,菌株信息通过菌株的唯一编号同文献与专利进行关联。另外,蛋白质的序列信息通过Uniprot数据库的唯一编码,同GO数据库、PDB数据库的蛋白质结构进行关联。基因序列可以通过基因的ID与KEGG数据库中的酶进行关联,每一个酶也具有一个唯一的EC号,通过该编号可以与代谢途径信息进行关联。
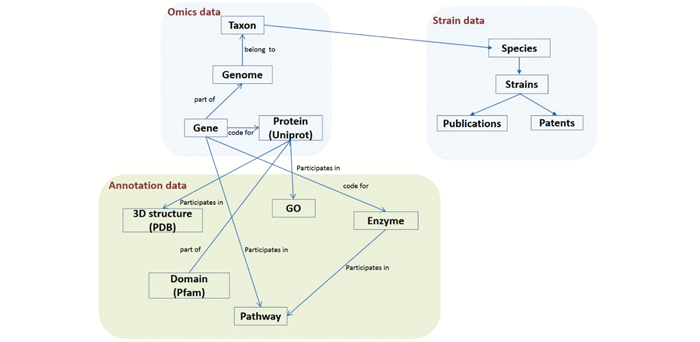
|
| 图 8 数据结构及关联关系 Figure 8 A system overview of database schema |
我们从公共数据库中下载了各数据集的原始数据,其中GO数据库和Uniprot数据库提供RDF格式的原始数据,在此基础上,我们将各种原始格式的数据在已经定义的本体和命名空间的基础上进行了RDF的转换,同时在处理数据的过程中加入了数据的关联关系,将关联关系也作为一个单独的属性加入到了数据集中。
3.3 平台及环境本数据库是采用Virtuoso数据库来实现存储的,支持RDF三元组的增、删、改、查等功能,通过构建SPARQL标准查询语句来查询Virtuoso数据库,前台的检索和展示页面采用Java web的方式构建。
4 结论与展望FoodMicrobes数据库通过RDF技术建立关联数据和建立数据仓库的方式,不仅实现了食品微生物相关的各层面数据的集成,同时系统的梳理了从菌株、菌种等宏观数据到基因组、蛋白质、酶和代谢途径等微观数据之间的关联关系,形成了一个紧密连接的系统。目前,在食品微生物领域,国内外还没有实现类似的工作。因此,该数据库不仅能为食品微生物研究提供一个重要的数据平台,同时,数据库所建立的关联关系、命名空间和本体,也可以作为其他领域微生物数据集成的一个重要借鉴和资源。
然而,一方面,除了数据的查询检索等功能外,数据的分析也是一个综合数据平台需要考虑的重要因素。RDF的数据格式与目前生物信息领域绝大多数的分析工具的兼容性上还存在问题。因此,如何实现数据格式的准确、高效转换,以及与数据分析工具之间的无缝对接,也是该数据库需要着重解决的问题。
另一方面,语义网和RDF还是一项相对较新的技术,在数据库的性能支持方面还有着一定的缺陷。目前,对超过百亿条三元组规模的大数据的性能支持方面还存在着较大的瓶颈。而大规模的生命科学数据已经是我们必须面对的现实,如何解决数据关联和运行效率方面的平衡还是一个重要的挑战,这也有赖于生命科学、生物信息学和信息技术多领域研究人员的交叉与合作。
| [1] | Carole G, Robert S. State of the nation in data integration for bioinformatics. Journal of Biomedical Informatics, 2008, 41(5) : 687–693. DOI:10.1016/j.jbi.2008.01.008 |
| [2] | Clark T, Martin S, Liefeld T. Globally distributed object identification for biological knowledge bases. Brief Bioinform, 2004, 5(1) : 59–70. DOI:10.1093/bib/5.1.59 |
| [3] | Ashburner M, Ball C A, Blake J A, et al. Gene ontology:tool for the unification of biology. Nat Genet, 2000, 25(1) : 25–29. DOI:10.1038/75556 |
| [4] | Mark A M, Natalya F N, Nigam H S, et al. The national center for biomedical ontology. J Am Med Inform Assoc, 2012, 19(2) : 190–195. DOI:10.1136/amiajnl-2011-000523 |
| [5] | Simon J, James M, Jerven B, et al. The EBI RDF platform:linked open data for the life sciences. Bioinformatics, 2014, 30(9) : 1338–1339. DOI:10.1093/bioinformatics/btt765 |
| [6] | SIB Swiss Institute of Bioinformatics Members. The SIB Swiss Institute of Bioinformatics' resources:focus on curated databases. Nucleic Acids Res, 2016, 44(D1) : D27–D37. DOI:10.1093/nar/gkv1310 |
| [7] | Alison C, Jose C, Peter A, et al. Bio2RDF release 2:improved coverage, interoperability and provenance of life science linked data. ESWC, 2013, 788(2) : 200–212. |
| [8] | Maulik R K, Michel D. An Ebola virus-centered knowledge base. Database, dio:10.1093/database/bav049.2015, 1-11. |
| [9] | Simon J, Julie K, Joost S. Developing a kidney and urinary pathway knowledge base. Journal of Biomedical Semantics, 2011, 2(Suppl 2) : S7. DOI:10.1186/2041-1480-2-S2-S7 |
| [10] | Linhuan W, Qinglan S, Hideaki S, et al. Global catalogue of microorganisms (gcm):a comprehensive database and information retrieval, analysis, and visualization system for microbial resources. BMC Genomics, 2013, 14 : 933. DOI:10.1186/1471-2164-14-933 |
| [11] | NCBI Resource Coordinators. Database resources of the National Center for Biotechnology Information. Nucleic Acids Res, 2016, 44(D1) : D7–D19. DOI:10.1093/nar/gkv1290 |
| [12] | Paul A K, Deanna M C, Francoise T, et al. Assembly:a resource for assembled genomes at NCBI. Nucleic Acids Res, 2016, 44(D1) : D73–D80. DOI:10.1093/nar/gkv1226 |
| [13] | Karen C, Ilene K M, David J. LGenBank. Nucleic Acids Res, 2016, 44(D1) : D67–D72. DOI:10.1093/nar/gkv1276 |
| [14] | Peter W R, Andreas P, Chunxiao B, et al. The RCSB Protein Data Bank:views of structural biology for basic and applied research and education. Nucleic Acids Res, 2016, 43(D1) : D345–D356. |
| [15] | Kanehisa M, Sato Y, Kawashima M, et al. KEGG as a reference resource for gene and protein annotation. Nucleic Acids Res, 2016, 44(D1) : D457–D462. DOI:10.1093/nar/gkv1070 |
| [16] | Keegan K, Glass E, Meyer F. MG-RAST, a Metagenomics Service for Analysis of Microbial Community Structure and Function. Methods Mol Biol, 2016, 1399 : 207–233. DOI:10.1007/978-1-4939-3369-3 |
 2017, Vol. 37
2017, Vol. 37




