文章信息
- 赵一瑾, 王腾飞, 汪俊卿, 王瑞明.
- ZHAO Yi-jin, WANG Teng-fei, WANG Jun-qing, WANG Rui-ming.
- 以CotC为分子载体在枯草芽孢杆菌表面展示海藻糖合酶
- Surface Display of Tres Using CotC as a Molecular Vector on Bacillus subtilis Spores
- 中国生物工程杂志, 2017, 37(1): 71-80
- China Biotechnology, 2017, 37(1): 71-80
- http://dx.doi.org/DOI:10.13523/j.cb.20170111
-
文章历史
- 收稿日期: 2016-06-27
- 修回日期: 2016-09-01
2. 山东省微生物工程重点实验室 济南 250353
2. Department of Biology Engineering, Jinan 250353, China
海藻糖(trehalose),又称为蘑菇糖(mushroom sugar)、覃糖(mycose),是由两个葡萄糖分子通过半缩醛羟基缩合而成的,由于不存在游离醛基,故其为一种非还原性双糖[1-2],在自然界中广泛存在于低等动物、细菌、酵母、真菌、昆虫和无脊椎动物中[3-4]。海藻糖是一种非特异性的保护剂,可以保护生物膜、蛋白质、脂质体及敏感细胞壁免受干旱、冷冻、渗透压的变化等造成的伤害[5-6],因此,海藻糖在工业上可用于不稳定食品、化妆品和医药的保存[7]。
研究发现,只有在恶劣环境下海藻糖才会合成,且在适宜环境下会迅速被降解[8],这给其生产带来了困难,因而找到可大量合成海藻糖的方式是当务之急。现阶段,海藻糖的制备工艺大体分为4种:微生物抽提法、发酵法、基因重组法和酶转化法[9]。微生物抽提法在制备海藻糖的过程中工艺较复杂,成本高,难以实现规模化生产;发酵法需要从微生物发酵体系中纯化得到海藻糖,但是发酵培养基中的成分比较复杂,分离结晶海藻糖困难,阻碍其大规模生产[10-11];基因重组法是对生物行基因改造,使不合成海藻糖的生物可以合成海藻糖,但其生产成本很高;酶转化法是目前认为最具工业化发展的制备方法,能够以葡萄糖、麦芽糖或直链淀粉为底物进行酶学反应生成海藻糖[12-13]。海藻糖合酶(trehalose synthase,Tres)属于糖苷水解酶第13家族,能够催化α,α-1,4-糖苷键连接的麦芽糖直接转化为α,α-1,1-糖苷键连接的海藻糖,该转化反应不需要磷酸盐的存在,不需要消耗高能物质,并且该酶的底物特异性较高,能够专一催化麦芽糖底物生成海藻糖,工艺简单,便于调控[14-15]。但目前发现的海藻糖合酶原始菌株中存在的内源性海藻糖合酶表达量低,发酵培养成本较高,分离提取困难,难以满足大规模工业生产要求的水平[16]。随着枯草芽孢杆菌基因组测序工作的完成,外源蛋白在枯草芽孢杆菌中表达成功的案例不断增加,枯草芽孢杆菌表达体系的安全、无内毒素特点,使其在食品与医药产业化生产领域备受认可[17]。构建重组枯草芽孢杆菌的芽孢表面展示体系时,利用芽孢衣壳蛋白融合展示海藻糖合酶,不仅能够避免因诱导剂的使用而造成的限制,而且融合酶不需要穿过细胞膜 [18-19],将海藻糖合酶直接应用于高端海藻糖的生产制备中,避免因细胞破碎,杂质分离造成的成本增加及热源物质对海藻糖的污染[20]。
芽孢表面展示技术对比噬菌体表面展示、细菌表面展示和酵母表面展示技术有明显的优势,即芽孢表面展示系统中形成的融合蛋白不需要穿过细胞膜就能有效的锚定在芽孢表面,这样可以有效地避免折叠不正确或定位不准确的问题[21]。本研究首先以增强型绿色荧光蛋白(enhanced green fluorescent protein,EGFP)作为报告蛋白,与芽孢衣壳蛋白CotC[22]融合,利用流式细胞仪检测荧光蛋白(EGFP)是否展示在枯草芽孢杆菌芽孢表面。并将展示成功的荧光蛋白基因利用分子生物学技术替换成海藻糖合酶基因,通过对其酶活的检测,结果表明海藻糖合酶被成功展示在枯草芽孢杆菌芽孢表面。
1 材料与方法 1.1 材料 1.1.1 菌株与质粒pEGFPN1质粒(含荧光蛋白基因egfp)为本实验室保存。感受态细胞E.coli DH5α为目的基因克隆宿主菌,零背景pTOPO-Blunt 平末端克隆筛选载体,购自艾德莱生物有限公司。Pseudomonas putide P06为海藻糖合酶基因treS的来源菌株,为本实验室保存。Bacillus subtilis 168 为芽孢衣壳蛋白基因cotC的来源菌株,同时也是重组质粒表达宿主菌。pHT01质粒中含有氯霉素(cmr)抗性标签,购自Invitrogen公司。
1.1.2 实验试剂即用PCR扩增试剂盒(pfu)、2×Phanta Max Master Mix、Alkaline Phosphatase(Calf intestine)、限制性内切核酸酶和T4 DNA连接酶均购自宝生物(大连)技术有限公司;质粒提取试剂盒、DNA Maker以及T载体购自北京全式金公司;细菌基因组快速提取试剂盒、DNA片段回收试剂盒和氯霉素购自生工生物工程(上海)股份有限公司;ClonExpress MultiS One Step Cloning Kit重组克隆试剂盒购自南京诺唯赞生物科技有限公司;其他试剂购自国药集团试剂有限公司。
1.1.3 培养基LB(Luria-bertani medium)液体培养基:蛋白胨10g/L、酵母浸粉5g/L、氯化钠10g/L,用于培养大肠杆菌。
LB固体培养基:在LB液体培养基中加入1.5%~2%(m/V)琼脂粉。
LB淀粉培养基:在LB培养基中添加1%(m/V)淀粉,用于筛选枯草芽孢杆菌转化子。
DSM(difico sporulation medium) [23]:营养肉汤8g/L、KCl 1g/L、MgSO4·7H2O、0.25g/L、一水氯化锰0.002g/L、Ca(NO3)2 1.0mmol/L,MnCl2 10μmol/L,FeSO4 1.0μmol/L。
1.1.4 引物细菌基因组DNA提取试剂盒提取Bacillus subtilis 168菌株基因组DNA,并以此为模板克隆芽孢衣壳蛋白基因cotC,以及上游同源臂基因amyEF、下游同源臂基因amyER。质粒提取试剂盒提取pEGFP-N1质粒和pHT01质粒,并分别为模板克隆目的基因egfp和氯霉素抗性基因cmr,其中插入片段cmr和amyER使用ClonExpress无缝克隆技术与克隆载体发生重组反应,其引物由CE Design软件设计,表面展示荧光蛋白的引物序列如表 1所示。
| Primer name | Primer sequence (5′-3′) | Restriction site |
| amyEF(F1) | AAGCTTCTGCAGCGATTCAAAACCTCTTTACTGCC | HindIII |
| amyEF(R1) | ATTTATCGAGCTCGCATCAAATCGAAAACCGTCT | SacI |
| cotC(F2) | CGATTTGATGCGAGCTCGATAAATCGTTTGGGCCG | SacI |
| cotC(R2) | TCCACCAGACCCTCCACCGGTACCGTAGTGTTTTTTATGCTTTTTATAC | KpnI |
| egfp(F3) | CGGTGGAGGGTCTGGTGGAGGATCAATGGTGAGCAAGGGCGAG | |
| egfp(R3) | GGATCCGTTCCAGGTAAGGTATAAACT | BamHI |
| cmr(F4) | tcgcgtgtcgcccttggatccTTTTGCTGGCCTTTTGCTCA | |
| cmr(R4) | tccattcgggtTAGTGACTGGCGATGCTGTCGTTTGCTCA | |
| amyER(F5) | cagtcactaACCCGAATGGAAACAACCAGA | |
| amyER(R5) | taccttacctggaacggatccCGTGTAAATTCCGTCTGCATTTT | |
| Note:The underlined sequences correspond to the restriction site HindIII in F1 and the site and the underlined sequences correspond to the SacI in R1 and F2. The underlined sequences correspond to the restriction site KpnI in R2 and the site BamHI in R3 | ||
1.2 方法 1.2.1 cotC基因、amyEF基因和egfp基因的获得
提取Bacillus subtilis 168菌株基因组DNA,以基因组DNA作为模板,利用引物cotC(F2) 、cotC(R2) 通过PCR扩增cotC片段;以amyEF(F1) 、amyEF(R1) 为引物,PCR扩增amyEF片段;以amyER(F5) 、amyER(R5) 为引物,PCR扩增amyER片段。上述反应条件为:预变性温度:95℃ 5min;变性温度:95℃ 30s;退火温度分别为51℃(cotC)/55℃(amyEF)/57℃(amyER)30s;延伸温度:72℃ 3.5min,30个循环;最后72℃延伸10min。反应结束后以1%的琼脂糖凝胶电泳检测产物并进行柱回收。
提取pEGFP-N1质粒,以质粒作为模板,利用引物egfp(F3) 、egfp(R3) PCR扩增egfp片段;提取pHT01质粒,以质粒作为模板,利用引物cmr(F4) 、cmr(R4) PCR扩增cmr片段。反应条件均为预变性温度:95℃ 5min;变性温度:95℃ 30s;退火温度:55℃ 30s;延伸温度:72℃ 2min,30个循环;最后72℃延伸10min。反应结束后分别以1%的琼脂糖凝胶电泳检测产物并进行柱回收。
1.2.2 线性化克隆载体pTOPO-amyEF-CotC-egfp的制备经上述PCR扩增得到的芽孢衣壳蛋白基因cotC和荧光蛋白基因egfp通过重叠PCR技术,得到融合基因片段cotC-egfp。同样将融合基因片段cotC-egfp与上游同源臂基因amyEF进行重叠PCR,得到融合基因片段amyEF-cotC-egfp。将融合基因片段与零背景pTOPO-Blunt平末端载体连接,得到克隆载体pTOPO-amyEF-cotC-egfp。选择BamHI酶切位点,对克隆载体进行单酶切线性化。
1.2.3 重组质粒pTOPO-egfp的构建将线性化克隆载体与插入片段于冰水浴中配制实验组和对照组的反应体系:线性化克隆载体、插入片段扩增产物cmr和amyER分别添加1μl,在实验组加入4μl 5×CE MultiS Buffer和2μl Exnase MultiS,补加ddH2O至20μl;对照组直接补加补加ddH2O至20μl。使用移液枪上下轻轻吹打混匀,于37℃反应30min。待反应完成后,立即将反应管置于冰水浴中冷却5min。取10μl冷却反应液加入到100μl DH5α感受态细胞中,并将转化产物涂布于LB平板,于37℃恒温培养箱培养过夜,筛选克隆子。进一步对所构建的重组质粒pTOPO-egfp进行三酶切SacI/KpnI/BamHI验证,再次确保重组质粒构建成功验证携带目的基因的正确性。整个构建过程如图 1所示。
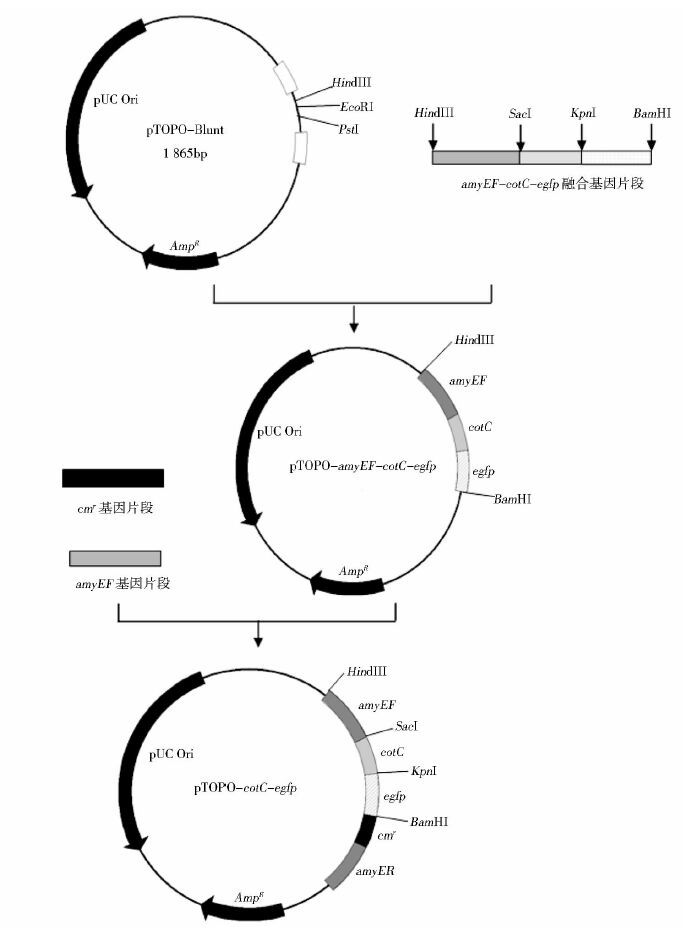
|
| 图 1 重组质粒pTOPO-egfp的构建流程图 Figure 1 Flow chart of recombinant plasmid construction |
提取pTOPO-egfp质粒用Pst I限制酶线性化后,Spizizen法[24]转至Bacillus subtilis 168感受态细胞,涂布于含氯霉素的LB平板(氯霉素浓度为10μg/ml),37℃条件下倒置培养12~16h,筛选阳性转化子。挑取含氯霉素的LB抗性平板上长出的单菌落进行菌落PCR检验。
1.2.5 重组芽孢的制备利用饥饿法在DSM培养基上诱导重组菌产生芽孢。将上述重组菌点接含5ml LB的试管中37℃过夜培养,然后按照1%(V/V)接种量转接至含50ml DSM的250ml的三角瓶中,37℃、200r/min继续培养48h。再将菌体以8 000r/min离心20min,弃去上清液,用无菌去离子水洗涤沉淀2次,并用去离子水悬浮。加入溶菌酶使其终浓度为2mg/ml,37℃水浴2h。再用1mol/L无菌氯化钠和1mol/L无菌氯化钾各洗涤一次,最后用无菌去离子水悬浮,制备芽孢悬浮液。
1.2.6 流式细胞仪检测荧光强度按照1.2.5 方法诱导重组菌株产生芽孢,得到芽孢悬浮液。取10μl芽孢悬浮液涂于载玻片上,室温2h自然晾干盖上盖玻片,用PBS漂洗3次,加入含2%(m/V)BSA的PBS室温封闭15min,再用PBS漂洗3次,将处理好的样品置于流式细胞仪上检测。该流式细胞仪配置了488nm的氩激发器和525nm的发射过滤器,样品流速设为150~300个芽孢/s,通道内需检测至少10 000个芽孢的荧光信号。
1.2.7 整合型重组质粒pTOPO-tres的构建以Pseudomonas putide P06菌株基因组DNA为模板,tres(F6) 、tres(R6) 为引物(表 2) ,PCR扩增tres片段;反应条件:预变性温度,95℃ 5min;变性温度,95℃ 30s;退火温度,51℃ 30s;延伸温度,72℃ 3.5min,30个循环;最后72℃延伸10min。
| Primer name | Primer sequence (5′-3′) | Restriction site |
| tres(F6) | GGTACCTAAGTATTTTATGACCCAGCCCGACCC | KpnI |
| tres(R6) | CTCACCATGGATCCAACATGCCCGCTGCTGT | BamHI |
| Note:The underlined sequences correspond to the restriction site KpnI in F6 and the site BamHI in R6 | ||
对上述PCR扩增得到的海藻糖合酶基因tres和重组质粒pTOPO-egfp分别用KpnI/BamHI双酶切,柱纯化酶切的pTOPO-egfp重组质粒,胶回收tres基因片段,连接得到pTOPO-Tres整合型重组质粒,然后转化至大肠杆菌DH5α感受态细胞,并将转化产物涂布于LB平板,于37℃恒温培养箱培养过夜,筛选克隆子,用灭菌的牙签挑取单菌落,以tres(F6) 、tres(R6) 作上、下游引物,以菌落PCR初步筛选。将初步筛选出的阳性克隆子按对应序号在含氨苄抗性的LB培养基中扩培,送至上海生工测序验证。整个构建过程如图 2所示。
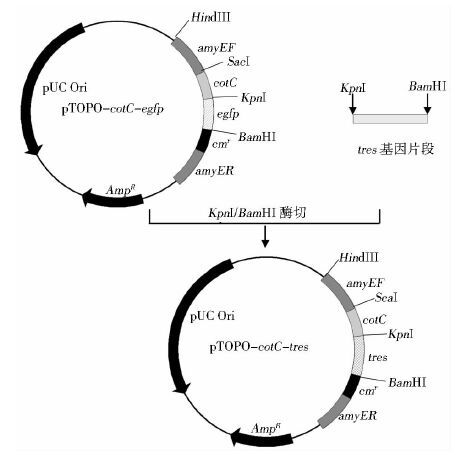
|
| 图 2 重组质粒pTOPO-Tres的构建 Figure 2 Flow chart of recombinant plasmid construction |
提取测序正确的pTOPO-Tres质粒,用Pst I限制酶线性化后,按照1.2.4 的方法筛选阳性克隆子,挑取含氯霉素的LB抗性平板上长出的单菌落进行菌落PCR检验。
1.2.9 整合型表达重组菌的芽孢表面展示Tres的酶活测定以重组菌株Bacillus subtilis-Tres为菌种,经活化后接种到发酵培养基中培养。将活化后的枯草芽孢杆菌接种发酵培养基中培养,发酵培养基为:葡萄糖800g、淀粉110g、豆粕1 500g、硫酸锰50g、磷酸氢二钾150g、磷酸二氢钾75g、硫酸镁25g、酵母膏11g、氯化铁5g、碳酸钙5g,定容至5L,发酵48h[25]。每隔5h取一次100ml发酵液,将发酵液8 000r/min离心20min,弃上清液,用去离子水洗涤沉淀2次,并用去离子水悬浮,加入溶菌酶使其终浓度为2mg/ml,37℃处理30min破坏营养细胞,8 000r/min离心10min,烘干,称量干菌体重量,最后用去离子水悬浮得到芽孢悬浮液。取5ml发酵48h后的重组菌芽孢悬浮液与10ml pH 7.5的磷酸盐缓冲液配制的终浓度30%的麦芽糖浆混匀后制备成海藻糖合酶的反应体系。将此反应体系置于50℃恒温水浴震荡摇床中反应2h,反应结束后,80℃水浴20min终止酶的转化反应。室温,1 000r/min离心10min,取上清液进行HPLC检测转化体系中海藻糖含量。转化有空载体的枯草芽孢杆菌经相同方法处理之后做空白对照,测定单位体积酶活。HPLC测定方法:色谱柱(Hypersil NH2,300×4.6mm,5μmol/L);流动相(乙腈:水=3:1) ;流速(l.0ml/min);检测器(示差折光检测器);进样量10μl;40℃测定。海藻糖计算公式:
| $T = {{{S_T}\cdot{M_S}} \over {{S_S}\cdot{M_T}}} \times 100\% $ |
式中,T为海藻糖含量(%);SS为标准品峰面积;ST为样品峰面积;MT为样品质量(g);MS为标准品量(g)。
1.2.10 酶活单位的定义酶活力单位定义:在pH 7.5、50℃条件下,每小时催化转化30%麦芽糖溶液产生1μmol海藻糖的酶量为1单位(U)。
2 结果与分析 2.1 目的基因扩增以Bacillus subtilis 168菌株基因组DNA为模板,以不同的引物分别PCR扩增得到大小分别约为372bp、644bp、641bp的目的片段为芽孢衣壳蛋白基因cotC,以及上游同源臂基因amyEF、下游同源臂基因amyER;以pEGFPN1质粒为模板,PCR扩增得到大小约为850bp的目的片段egfp;以pHT01质粒为模板,PCR扩增大小约为1 245bp的cmr。以Pseudomonas putide P06菌株基因组DNA为模板,PCR扩增得到大小约为2 064bp的海藻糖合酶基因tres片段。上述PCR产物电泳结果如图 3所示,均显示扩增出与目的基因大小相符的DNA片段。
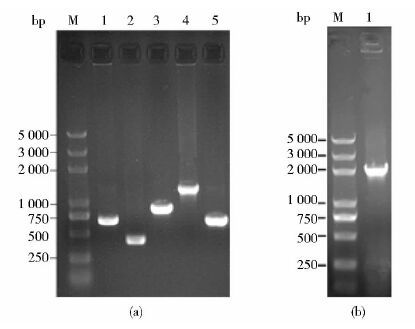
|
| 图 3 目的基因PCR电泳图 Figure 3 PCR results of target gene (a)Lane 1: PCR results for the purpose of amyEF gene; Lane 2: PCR results for the purpose of cotC gene; Lane 3: PCR results for the purpose of egfp gene; Lane 4: PCR results for the purpose of cmr gene; Lane 5: PCR results for the purpose of amyER gene; Lane M: Trans 5K DNA Marker (b) Lane 1: PCR results for the purpose of tres gene; Lane M: Trans5K plus DNA marker |
经融合PCR技术将上游同源臂amyEF扩增片段、芽孢衣壳蛋白基因cotC扩增片段与荧光蛋白基因egfp扩增片段连接成融合片段,再与pTOPO-TA载体连接,构建成重组T载体。经菌落PCR初步筛选,PCR重组T载体酶切验证及融合片段测序后,将测序结果经BLAST比对分析,结果显示融合片段基因序列与原始模板中的基因序列相似度达到100%,目标基因amyEF-cotC-egfp融合成功。将融合基因片段与零背景pTOPO-Blunt平末端载体连接,转化至E.coli DH5α感受态细胞,经菌落PCR筛选后使用HindIII单酶切,经琼脂糖凝胶电泳图验证得到大约在1 865bp的条带(图 4) ,再次确定融合片段与平末端载体连接成功,即得到重组菌Bacillus subtilis-Tres。选择BamHI酶切位点,对克隆载体进行单酶切线性化。
|
| 图 4 BamHI单酶切线性化克隆载体电泳图 Figure 4 Results of the linearized cloning vector use BamHI single restriction result analysis |
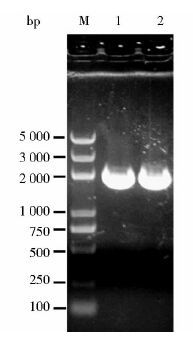
将线性化克隆载体与插入片段cmr和amyER按照1.2.3 的反应体系混匀各组分,置于37℃反应30min,立即置于冰水浴中冷却5min。按照1.2.4的方法筛选克隆子进行菌落PCR鉴定。成功获得重组质粒pTOPO-egfp,如图 5所示。
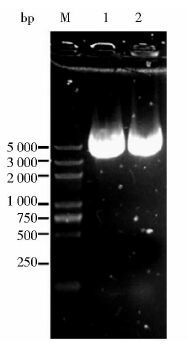
|
| 图 5 重组质粒pTOPO-egfp电泳图 Figure 5 Results of the recombinant plasmid pTOPO-egfp result analysis |
将构建完成且测序准确的重组质粒利用Spizizen法转至Bacillus subtilis 168感受态细胞中,挑取含氯霉素的LB抗性平板上长出的单菌落进行菌落PCR检验。经LB培养基摇瓶培养24h提取枯草芽孢杆菌基因组,以阳性转化子基因组为模板,以egfp(F3) 、egfp(R3) 为引物进行PCR鉴定,成功克隆出egfp基因,说明重组菌构建成功,命名为Bacillus subtilis-egfp。将重组质粒pTOPO-egfp经SacI/Kpn I/BamHI 3种酶酶切鉴定分析正确。三酶切之后的电泳结果如图 6所示。
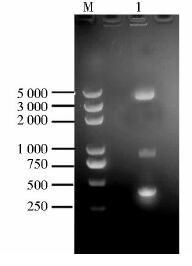
|
| 图 6 重组质粒pTOPO-egfp片段的三酶切验证 Figure 6 Results of the pTOPO-egfp recombinant plasmid restriction enzyme analysis |
为了检测重组菌Bacillus subtilis-egfp是否将荧光蛋白基因egfp展示在芽孢表面,选择流式细胞仪检测。结果见图 7。由图 7可知,原始菌株的芽孢表面发出的荧光强度要小于重组菌株Bacillus subtilis-egfp。结果表明,使用该种方法,芽孢衣壳蛋白cotC与egfp融合后被成功展示在芽孢的表面,但表达量偏低。
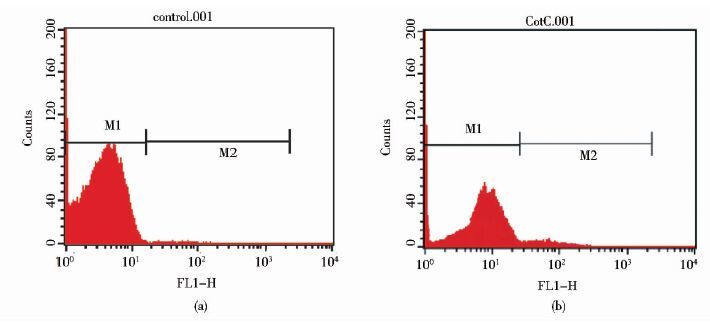
|
| 图 7 流式细胞仪分析菌株Bacillus subtilis-egfp的重组芽孢表面展示的EGFP Figure 7 Flow cytometry analysis of the TreSon the spore surface (a) Original bacterial spores (b) Recombinant Bacillus subtilis-egfp spores |
对上述重组质粒pTOPO-EGFP和PCR扩增得到的海藻糖合酶基因tres分别用KpnI/BamHI双酶切,经连接后,成功获得重组质粒pTOPO-Tres。采用Spizizen法对重组质粒进行化学法转化至Bacillus subtilis 168感受态细胞中,涂布于含氯霉素的LB平板(氯霉素浓度为10μg/ml),37℃条件下倒置培养12~16h,筛选阳性转化子。挑取含氯霉素的LB抗性平板上长出的单菌落进行菌落PCR检验。结果如图 8所示。
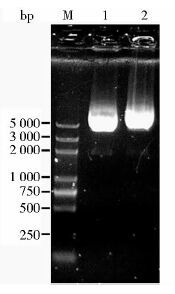
|
| 图 8 重组质粒pTOPO-Tres菌落PCR电泳图 Figure 8 The recombinant plasmid pTOPO-Tres colony PCR result analysis |
为了证实芽孢表面展示Tres是否有活性,根据1.2.9 中检测方法,利用HPLC对重组菌芽孢悬浮液进行液相检测。由HPLC测得液相图谱如图 9所示,图中A样品为重组菌芽孢悬浮液转化结果,表明有麦芽糖转化为海藻糖;而对照组B样品的芽孢悬浮液转化结果未见海藻糖峰,这表明海藻糖合酶基因通过与芽孢衣壳蛋白基因cotC融合成功,在枯草芽孢杆菌芽孢表面成功展示。
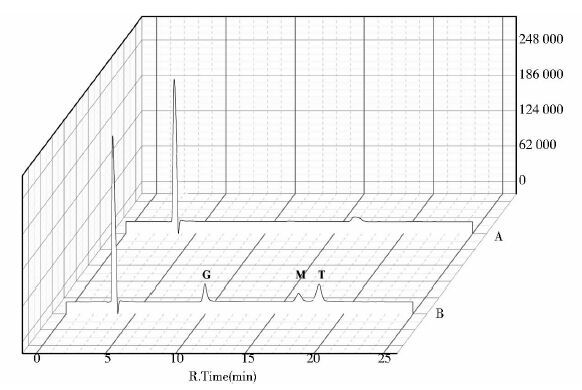
|
| 图 9 重组菌、野生菌芽孢悬浮液转化生成海藻糖高效液相图谱 Figure 9 HPLC results of intracellular and extracellular Recombinants G,M,T represents a glucose peak,a maltose peak,and a trehalose peak. A: Recombinant spore suspension; B: Primordial spore suspension |
根据计算结果,绘出重组菌的干菌体生长曲线和酶活曲线,如图 10所示,结果显示重组菌的芽孢悬浮液从26h开始有酶活,酶活较低,到33h酶活达到最大,酶活为252U/ml。这说明芽孢衣壳蛋白基因cotC是展示海藻糖合酶的合适分子载体,海藻糖合酶基因通过与芽孢衣壳蛋白基因cotC融合,成功的在枯草芽孢杆菌芽孢表面展示出来。
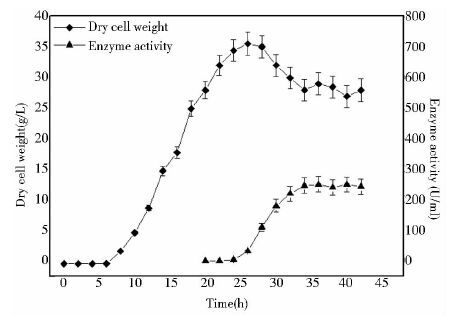
|
| 图 10 重组菌的干菌体生长曲线和酶活曲线 Figure 10 Results of the dry cell weight of recombinant bacteria growth curve and activity curve result analysis |
实验成功构建了来源于pEGFPN1质粒的荧光蛋白基因和来源于Pseudomonas putide P06菌株的海藻糖合酶基因的枯草芽孢杆菌芽孢表面展示重组菌。
本研究首先将芽孢衣壳蛋白基因cotC与荧光蛋白基因egfp融合,构建融合蛋白表达载体pTOPO-egfp,获得能够表面展示荧光蛋白的枯草芽孢杆菌重组菌,并通过流式细胞仪进行验证,表明所构建载体pTOPO-EGFP能够成功用于B. subtilis 168中外源蛋白的芽孢表面展示。同时,为实现海藻糖合酶的芽孢表面展示,通过酶切连接的方法将pTOPO-EGFP来源于Pseudomonas putide P06菌株的海藻糖合酶基因tres替换荧光蛋白基因egfp,最终获得芽孢表面展示海藻糖合酶的重组菌。将所获重组菌芽孢收集后作为全细胞催化剂与麦芽糖反应,避免了破碎细胞提纯海藻糖合酶的繁琐步骤,节约生产成本,利于工业化生产。反应产物使用HPLC法分析发现有海藻糖生成,经计算发现发酵33h后酶活最高可达252U/ml,表明海藻糖合酶Tres已成功展示在B. subtilis 168的芽孢表面。
虽然本研究首次利用芽孢衣壳蛋白基因cotC进行tres的芽孢表面展示,但与在大肠杆菌中表达[26]相比tres的表达量依然偏低,这可能是由于CotC在B. subtilis 168表面展示位点有限有关[27],同时利用CotC进行表面展示的Tres处于crust层和衣壳外层的包裹中[28],因而催化能力受限,也可能会间接降低Tres的表观酶活力。但本研究所利用的生产菌种为枯草芽孢杆菌,是国际认同可以运用到人类与动物的相关领域的安全菌种,因此产品的安全性更高,并能够实现食品级和医药级海藻糖的生产,同时芽孢表面展示也避免了细胞破碎引起的污染及成本问题,也有利于酶的重复利用,因此具有一定的应用价值。在后续实验中本研究将继续进行包括crust层蛋白在内的更多芽孢表面展示位点的展示,探索最优化的组合展示方法,同时对遗传基因的稳定性等方面进行探究,以期进一步提高Tres在B. subtilis 168表面的展示水平,继续降低生产成本,并完善相关生产工艺,最终实现高品质海藻糖合酶工业化生产。
| [1] | Leslie S B, Israeli E, Lighthart B, et al. Trehalose and sucrose protect both membranes and proteins in intact bacteria during drying. Applied & Environmental Microbiology, 1995, 61(10) : 3592–3597. |
| [2] | 齐向辉, 陈华友, 蒙健宗, 等. 海藻糖在生物组织器官保护方面的新应用. 安徽农业科学, 2009, 37(33) : 16729–16732. Qi X H, Chen H Y, Meng J Z, et al. Novel application of trehalose in the bioprotection of biological tissue and organ. Journal of Anhui Agricultural Sciences, 2009, 37(33) : 16729–16732. |
| [3] | Elbein A D. The metabolism of alpha, alpha-trehalose.. Advances in Carbohydrate Chemistry & Biochemistry, 1974, 30:227-256. |
| [4] | Avonce N, Mendoza-Vargas A, Morett E, et al. Insights on the evolution of trehalose biosynthesis. Bmc Evolutionary Biology, 2006, 6(1) : 1–15. DOI:10.1186/1471-2148-6-1 |
| [5] | Elbein A D, Pan Y T, Pastuszak I, et al. New insights on trehalose:a multifunctional molecule.. Glycobiology, 2003, 13(4):17R-27R. |
| [6] | Benaroudj N, Lee D H, Goldberg A L. Trehalose accumulation during cellular stress protects cells and cellular proteins from damage by oxygen radicals. J Biol Chem, 2001, 276(26) : 24261–24267. DOI:10.1074/jbc.M101487200 |
| [7] | 靳文斌, 李克文, 胥九兵, 等. 海藻糖的特性、功能及应用. 精细与专用化学品, 2015, 23(1) : 30–33. Jin W B, Li K W, Xu J B, et al. The character and function of trehalose and its application. Fine & Specialty Chemicals, 2015, 23(1) : 30–33. |
| [8] | Jules M, Beltran G, François J, et al. New insights into trehalose metabolism by Saccharomyces cerevisiae:NTH2 encodes a functional cytosolic trehalase, and deletion of TPS1 reveals Ath1p-dependent trehalose mobilization.. Applied & Environmental Microbiology, 2008, 74(3):605-614. |
| [9] | 薛鸿毅, 张莉, 徐汝意, 等. 酶法生产海藻糖研究进展. 中国食品添加剂, 2011(4) : 159–162. Xue H Y, Zhang L, Xu R Y, et al. Study on the production of trehalose through enzyme method. China Food Additives, 2011(4) : 159–162. |
| [10] | Ryu S I, Park C S, Cha J, et al. A novel trehalose-synthesizing glycosyltransferase from Pyrococcus horikoshii:Molecular cloning and characterization. Biochemical & Biophysical Research Communications, 2005, 329(2) : 429–436. |
| [11] | Ryu S I, Kim J E, Kim E J, et al. Catalytic reversibility of Pyrococcus horikoshii trehalose synthase:Efficient synthesis of several nucleoside diphosphate glucoses with enzyme recycling. Process Biochemistry, 2011, 46(1) : 128–134. DOI:10.1016/j.procbio.2010.07.030 |
| [12] | Kim H M, Chang Y K, Ryu S I, et al. Enzymatic synthesis of a galactose-containing trehalose analogue disaccharide by Pyrococcus horikoshii, trehalose-synthesizing glycosyltransferase:Inhibitory effects on several disaccharidase activities. Journal of Molecular Catalysis B Enzymatic, 2007, 49(1-4) : 98–103. DOI:10.1016/j.molcatb.2007.08.012 |
| [13] | Avonce N, Mendoza-Vargas A, Morett E, et al. Insights on the evolution of trehalose biosynthesis. Bmc Evolutionary Biology, 2006, 6(1) : 1–15. DOI:10.1186/1471-2148-6-1 |
| [14] | Yoshida M, Nakamura N, Horikoshi K. Production of trehalose from starch by maltose phosphorylase and trehalose phosphorylase from a strain of plesiomonas. Starch-Starke, 1997, 49(49) : 21–26. |
| [15] | Woo E J, Ryu S I, Song H N, et al. Structural insights on the new mechanism of trehalose synthesis by trehalose synthase TreT from Pyrococcus horikoshii. Journal of Molecular Biology, 2010, 404(2) : 247–259. DOI:10.1016/j.jmb.2010.09.056 |
| [16] | Wernerus H, Stahl S. Biotechnogical applications for surface-engineered bacteria. Biotechnology and Applied Biochemistry, 2004, 40(3) : 209–228. DOI:10.1042/BA20040014 |
| [17] | 余小霞, 田健, 伍宁丰. 枯草芽胞杆菌芽胞表面展示外源蛋白的研究进展. 中国农业科技导报, 2013, 15(5) : 31–38. Yu X X, Tian J, Wu N F. Research progress on Bacillus subtilis spore display of recombinant proteins. Journal of Agricultural Science & Technology, 2013, 15(5) : 31–38. |
| [18] | 李倩, 宁德刚, 吴春笃. 以CotX为分子载体在枯草芽胞杆菌芽胞表面展示绿色荧光蛋白. 生物工程学报, 2010, 26(2) : 264–269. Li Q, Ning D G, Wu C D. Surface display of GFP using CotX as a molecular vector on Bacillus subtilis spores. Chinese Journal of Biotechnology, 2010, 26(2) : 264–269. |
| [19] | Ryu S I, Kim J E, Huong N T, et al. Molecular cloning and characterization of trehalose synthase from Thermotoga maritima, DSM3109:Syntheses of trehalose disaccharide analogues and NDP-glucoses. Enzyme & Microbial Technology, 2010, 47(6) : 249–256. |
| [20] | 王贺, 杨瑞金, 华霄, 等. 利用枯草芽孢衣壳蛋白表面展示β-半乳糖苷酶. 食品与发酵工业,, 2012, 38(7) : 1–5. Wang H, Yang R J, Hua X, et al. Functional display of β-galactosidase on the spore surface of Bacillus subtilis using spore coat protein as anchor motif. Food & Fermentation Industries, 2012, 38(7) : 1–5. |
| [21] | Tavassoli S, Hinc K, Iwanicki A, et al. Investigation of spore coat display of Bacillus subtilis β-galactosidase for developing of whole cell biocatalyst. Arch Microbiol, 2013, 195(3) : 197–202. DOI:10.1007/s00203-013-0867-9 |
| [22] | Nicholson W L, Setlow P. In Molecular Biological Methods for Bacillus. Chichester: John Wiley and Sona Inc, 1990: 391-450. |
| [23] | Santos C A F, Rodrigues M A, Zucchi M I. Proceedings of the National Academy of Science of the United States of America. Pesquisa Agropecuária Brasileira, 2013. |
| [24] | 郭夏丽, 狄源宁, 王岩. 枯草芽孢杆菌产芽孢条件的优化. 中国土壤与肥料, 2012(3) : 99–103. Guo X L, Di Y N, Wang Y. Optimization of sporulation conditions of Bacillus subtilis. Soil & Fertilizer Sciences in China, 2012(3) : 99–103. |
| [25] | 邵引刚, 李峰, 孙辉, 等. 海藻糖的应用及基因工程研究. 安徽农业科学, 2008, 36(3) : 857–859. Shao Y G, Li F, Sun H, et al. Study on the genetic engineering of trehalose and its application. Journal of Anhui Agricultural Sciences, 2008, 36(3) : 857–859. |
| [26] | 薛鸿毅. 恶臭假单胞菌海藻糖合酶基因在大肠杆菌中的表达. 济南:山东轻工业学院, 食品与生物工程学院,2012. Xue H Y. Study on Expression of Trehalose Synthase Genes from Pseudomonas putida P06 in E.coli.Jinan:Shandong Polytechnic University, School of Food and Bioengineering,2012. |
| [27] | Isticato R, Esposito G, Zilh o R, et al. Assembly of multiple CotC forms into the Bacillus subtilis spore coat. Journal of Bacteriology, 2004, 186(4) : 1129–1135. DOI:10.1128/JB.186.4.1129-1135.2004 |
| [28] | Isticato R, Cangiano G, Tran H T, et al. Surface display of recombinant proteins on Bacillus subtilis spores. Journal of Bacteriology, 2001, 183(21) : 6294–6301. DOI:10.1128/JB.183.21.6294-6301.2001 |
 2017, Vol. 37
2017, Vol. 37




