文章信息
- 饶菁菁, 景一娴, 邹明月, 胡小蕾, 廖飞, 杨晓兰.
- RAO Jing-jing, JING Yi-xian, ZOU Ming-yue, HU Xiao-lei, LIAO Fei, YANG Xiao-lan.
- 季也蒙毕赤酵母菌尿酸酶基因的克隆、重组表达及表征
- Clone, Expression and Characterization of the Uricase from Meyerozyma guilliermondii
- 中国生物工程杂志, 2017, 37(11): 74-82
- China Biotechnology, 2017, 37(11): 74-82
- http://dx.doi.org/DOI:10.13523/j.cb.20171110
-
文章历史
- 收稿日期: 2017-07-31
- 修回日期: 2017-10-15
血清尿酸增高造成高尿酸血症,从而导致痛风、心血管病变等系列疾病[1-2]。尿酸酶(uricase, EC1.7.3.3)能够专一催化尿酸氧化成易溶的尿囊素再主要经肾脏排泄。尿酸酶是酶法测定血清尿酸诊断高尿酸血症相关疾病的关键工具酶[3-4],也是治疗高尿酸血症的候选蛋白质药物,更是当前治疗难治性痛风的唯一有效药物[5-6]。尿酸酶的应用都要求其生理pH下比活性高、稳定性好。但现有天然尿酸酶即使在最适条件下比活性都很低,稳定性也有限,尤其现有尿酸酶最适pH都偏碱性,在生理pH下保留活性都没有超过60%[1]。所以,热稳定性好、在生理pH下比活性更高的尿酸酶有更好应用潜力。
为便于重组表达,微生物是尿酸酶的最重要来源。很早发现,中国微生物菌种保藏中心的酵母细胞株C.G.M.C.C 2.1008在尿酸诱导下表达尿酸酶,其热稳定性优良且比活性较高,有显著应用潜力[1];最有吸引力的是此尿酸酶最适pH接近中性,是分子改造提高催化效力的理想起始尿酸酶(数据未发表)。用DEAE-纤维素反复负吸附纯化后确认纯度满足要求的天然尿酸酶在上海进行Edman降解,发现其酶蛋白N端被封闭,而进一步串联质谱分析发现其氨基酸序列与已知尿酸酶都显著不同[7]。此前据苛求芽孢杆菌尿酸酶N端Edman降解所得氨基酸序列,以基因组DNA为模板,经简并引物PCR成功克隆出其绝大部分编码序列[8]。但糟糕的是,据上海进行串联质谱分析所得肽段的氨基酸序列,以该真菌基因组DNA为模板,反复尝试简并引物PCR,既没有克隆出任何与尿酸酶相似基因序列,也没有克隆出串联质谱分析所发现序列相似蛋白质的肽段。为快速克隆获得此真菌尿酸酶编码序列,本文先测定rRNA鉴定此菌种确为季也蒙毕赤酵母(Meyerozyma guilliermondii),用制备型SDS-PAGE纯化尿酸诱导的天然酶重新进行串联质谱分析发现其氨基酸序列与据季也蒙毕赤酵母基因组序列推定的假定尿酸酶(Uniprot id: A5DFP1)氨基酸序列高度相似,转录组测序发现此序列高效诱导表达;进一步据A5DFP1编码序列设计精确引物从cDNA经PCR克隆出目标基因,并重组表达及与天然酶性质进行比较,确认获得来自此季也蒙毕赤酵母细胞株尿酸酶的编码序列。
1 材料与方法 1.1 材料 1.1.1 菌株与质粒载体菌株购自于中国微生物菌种保藏中心(C.G.M.C.C 2.1008)。pDE1及pDE2质粒载体来源于定向克隆试剂盒。感受态大肠杆菌DH5α及BL21(DE3)细胞购自于北京鼎国昌盛生物技术公司。
1.1.2 试剂DNA ladder(DL2000)和rTaq酶购自于TaKaRa公司,蛋白质Marker购自于赛默飞世尔科技有限公司;T载体试剂盒及定向克隆试剂盒购自于北京擎科新业生物技术有限公司,质粒提取试剂盒购自于美国Omega公司,真菌RNA提取试剂盒及胶回收试剂盒购自于Mage公司,逆转录试剂盒购自于罗氏公司;DEAE-cellulose 52层析柱和Ni柱购自于索莱堡公司。其他试剂均为国产分析纯。
1.2 方法 1.2.1 菌种鉴定适量菌液涂布于麦芽糖浸膏平板上,37℃孵箱培养24h。平板于干冰运输至北京中美泰和生物技术公司和重庆盘点生物科技有限公司分别进行菌种鉴定。
1.2.2 天然尿酸酶的诱导表达及纯化此酵母经过扩大培养及尿酸诱导后获取天然尿酸酶(MGU),经DEAE-cellulose 52纤维素负吸附纯化3次[1],浓缩,20mmol/L Tris-HCl (pH8.0)透析过夜,取1.5ml酶液经超滤管超滤至300μl。取25 l进行SDS-PAGE。
1.2.3 串联质谱鉴定SDS-PAGE后进行考马斯亮蓝染色,脱色充分后将胶上可见的条带依次切下并编号为1、2、3、4,保存于灭菌的EP中。将条带用灭菌后的双蒸水清洗3次,送第三军医大学生物医学分析测试中心进行串联质谱鉴定。
1.2.4 转录组测序分析取尿酸诱导培养后菌液,于干冰运输至上海美吉生物有限公司转录组测序。
1.2.5 尿酸酶基因的扩增取尿酸诱导后的真菌培养液,用真菌RNA提取试剂盒提取RNA后进行逆转录得到cDNA模板,分装后于-20℃保存。以Uniprot id A5DFP1编码序列为模板,针对此蛋白质N端前8个氨基酸残基的编码序列及C端含终止密码及以外21个碱基的基因组序列,设计第一对精确PCR引物(委托北京擎科新业生物技术有限公司合成);其中正向引物PF1序列为CACCATGTCTTTGGTCGCATCTTCATACG,反向引物PR1序列为TTATAACTTGGCGTTGGCGTCTCTG。再设计第二对精确引物以便PCR产物与表达载体连接;正向引物PF2为CACCATGTCTTTGGTCGCATCTTCATACG,反向引物PR2为TAACTTGGCGTTGGCGTCTCTG。PCR反应体系为100μl:rTaq酶1μl,10×buffer 10μl,MgCl2 6μl,dNTPmix 8μl,PF1/ PF2 4μl,PR1/ PR2 4μl,再用无菌水补足至100μl。PCR参数设置:95℃预变性3min,95℃变性30s,60℃退火30s,72℃延伸1min,35个循环后72℃延伸5min。PCR产物用1.7%的琼脂糖凝胶电泳后,胶回收备用。
1.2.6 T载体连接测序将胶回收后的产物根据试剂盒说明连接至T载体后转化入DH5感受态中,加入不含抗生素的LB培养基复苏后取适量菌液涂布于含100mg/L氨苄青霉素抗性的LB选择平板上筛选转化子,37℃倒置培养过夜。次日挑取单克隆于含100mg/L氨苄青霉素抗性的培养基中扩大培养后,送菌液测序。
1.2.7 重组尿酸酶表达质粒的构建引物PF1和PR1、PF2和PR2能使PCR产物带上CACC接头,便于与定向克隆表达载体pDE1及pDE2直接连接。构建的质粒pDE1-MGU重组表达后的尿酸酶蛋白N端有6His标签,pDE2-MGU重组表达后的尿酸酶蛋白C端有6His标签,pDE1-MGU送至北京泰和基因有限公司进行突变去掉N端6His标签及连接肽序列构建不带6His标签的重组尿酸酶质粒R-MGU。将上述质粒采用42℃热激法转化入大肠杆菌感受态DH5α中,在含卡那霉素抗性的LB平板上筛选转化子,37℃倒置培养过夜。次日挑阳性克隆进行菌落PCR鉴定,并扩大培养后用质粒提取试剂盒提取质粒,于-20℃中保存待用。
1.2.8 重组尿酸酶的诱导表达将重组尿酸酶质粒经42℃热激法转化大肠杆菌感受态BL21(DE3),于含有100mg/L卡那霉素抗性的LB选择平板上筛选转化子,37℃倒置培养过夜。次日分别挑取单克隆接种到含卡那霉素的3ml培养基中,37℃ 180r/min摇床培养过夜后按1:1 000比例接种至新的250ml培养基中。37℃ 180r/min摇床培养至菌液OD600达到0.6~1.0时,加入终浓度为0.2mmol/L的异丙基-β-D-硫代半乳糖苷(IPTG),16℃ 110r/min摇床培养20~22h。4℃ 8 000r/min离心15min收集菌体。用20mmol/L Tris-HCl(pH8.0)缓冲液重悬菌体,冰浴下超声波碎菌(超声振幅33%,工作时间5s,间隔时间5s,超声30min)。4℃ 12 000r/min离心20min离心破碎菌液,分别收集上清液即为粗酶液。
1.2.9 重组尿酸酶的纯化N端和C端带有6His标签的重组尿酸酶蛋白用镍柱纯化。收集300mmol/L咪唑洗脱的洗脱液即为纯化一次的酶液。重复镍柱纯化一次,收集目的蛋白洗脱液保存于4℃。重组表达后无6His标签尿酸酶蛋白等电点约为8.97,故根据负吸附原理采用DEAE-cellulose 52层析柱纯化。将粗酶液上样于经20mmol/L Tris-HCl(pH8.0)缓冲液平衡的DEAE-cellulose 52层析柱,控制流速为5~7s/滴,吸附完成后,用平衡缓冲液洗涤/洗脱层析柱。同时每管1~2ml收集洗涤洗脱液。测定洗脱液中尿酸酶活力,合并活力最高部分。重复DEAE-cellulose 52层析柱纯化一次,收集目的蛋白于4℃保存。
1.2.10 重组尿酸酶的表征取适量酶液进行10%的SDS-PAGE。尿酸酶为底物测定尿酸酶活力[9-10],考马斯亮蓝法测其蛋白质浓度以计算比活性。双倒数分析法测定米氏常数(Km)[11]。测定酶活性抑制率对抑制剂浓度对数的响应,用MS Excel 6.0内嵌函数拟合线性部分确定半抑制浓度(IC50),根据不同重组尿酸酶Km换算竞争性抑制剂氧嗪酸钾和黄嘌呤的抑制常数。取纯化后的尿酸酶滤菌后,分别加至滤菌的pH7.4磷酸盐缓冲液及pH9.2硼砂缓冲液中测定热稳定性(热稳定性测定体系含终浓度为0.1g/ml的氨苄霉素和卡那霉素、0.1mmol/L EDTA、2.0mmol/L对氨基苯甲脒二磷酸盐)。同时分别考察终浓度为30 μmol/L的抑制剂氧嗪酸钾对其热稳定性的影响。将稳定性测定体系置于37℃恒温细胞培养箱中,20min后超净台取样测定初始活性,然后间隔不同时间测定活性记录活性衰减过程。将离子交换负吸附纯化后的天然尿酸酶MGU和重组尿酸酶R-MGU再进行酸性非变性电泳后胶回收[7],样品用超滤管浓缩脱盐后送至第三军医大学生物分析测试中心进行MODLI-TOF-MS检测其分子质量进行比较。
2 结果 2.1 MGU的编码序列片段信息酵母菌C.G.M.C.C 2.1008尿酸酶(MGU,以下指来自此细胞株的尿酸酶)需尿酸诱导表达[1, 7],其最大优势是最适pH接近中性(数据未发表)。获得酶蛋白部分肽段或编码基因序列就能用简并引物PCR或筛选cDNA快速克隆其完整编码序列[8]。将MGU纯化到HPLC显示为单一肽段后,Edman降解测序发现其N端被封闭,用胰蛋白酶解后串联质谱分析肽段序列发现其序列与已知真菌尿酸酶相差都很大,但与一种特殊脱氢酶氨基酸序列相似(完成于上海)[7]。据此相似蛋白质肽段序列设计简并引物用于以基因组DNA为模板经PCR扩增其编码序列,但没有获得任何有意义的序列信息。所以,迫切需其他路线快速确定MGU的部分氨基酸序列甚至部分编码序列,以便快速克隆其基因。
Uniprot数据库中有季也蒙毕赤酵母ATCC6260基因组序列推断的假定尿酸酶A5DFP1,但没有进行重组表达确认。为尽快确认MGU与尿酸酶A5DFP1氨基酸序列相似性,先测定rRNA序列鉴定菌种,结果发现该细胞属于季也蒙毕赤酵母,但无法确定到细胞株(其28S rRNA序列D1/D2区和ITS区序列分别见Genbank登录号KY688098和KY688099)。首次串联质谱发现MGU氨基酸序列与A5DFP1无明显相似性,有可能源自未知实验环节出错。该酵母菌细胞用尿酸诱导表达天然MGU后,用DEAE-cellulose 52柱层析纯化3次,再用SDS-PAGE分辨出4个蛋白成分进行串联质谱分析(图 1),发现分子质量由大到小4种多肽对应的最相似蛋白质分别为A5DJ39、A5DR94、A5DFP1、A5DNM4。其中,A5DJ39为甲酸脱氢酶;A5DR94为一种未表征过的蛋白质,结构域预测其有水解酶活性;A5DNM4为肽基脯氨酰顺式反式异构酶。意外的是,最高丰度成分3胰蛋白酶水解后,串联质谱解析成功的所有高丰度肽段都在季也蒙毕赤酵母尿酸酶A5DFP1氨基酸序列中找到全同肽段,这些高丰度肽段合并已覆盖A5DFP1氨基酸序列中近90%区域(图 2),即使部分低丰度肽段也能与A5DFP1的肽段序列匹配。用MALDI-TOF-MS发现MGU肽段分子质量约为17.4kDa,而SDS-PAGE分析肽段分子质量约为35kDa[1, 7],但按A5DFP1氨基酸序列计算的肽段分子质量约为33.8kDa[1, 7]。所以,MGU与A5DFP1序列高度相似,但MGU肽段可能更长或者存在化学修饰。
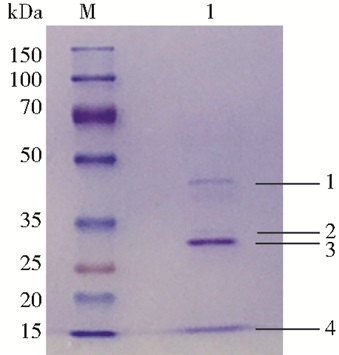
|
| 图 1 天然季也蒙毕赤酵母尿酸酶SDS-PAGE Figure 1 SDS-PAGE for MGU M: Protein maker; 1: Purified MGU |

|
| 图 2 串联质谱解析MGU肽段与A5DFP1氨基酸序列的比对 Figure 2 Alignment for amino acid sequence of A5DFP1 and MGU peptides by MS/MS 1: Amino acid sequence of A5DFP1; 2: Peptides by MS/MS |
为确认A5DFP1氨基酸序列与MGU序列的相似度,进一步进行转录组测序。MGU依赖于尿酸诱导表达[1, 7]。转录组测序得到两个编码片段的read与基因PGUG_02092的DNA序列对应完全一致,其转录本EDK37994与A5DFP1完全一致,且表达量很高(fpkm位列第11) (图 3)。可见,酵母菌C.G.M.C.C 2.1008诱导型尿酸酶MGU氨基酸序列与A5DFP1高度相似,应该可用PCR从其cDNA中快速克隆其绝大部分编码序列。

|
| 图 3 转录组测序验证目的基因 Figure 3 Confirmation of the target gene by RNA-seq (a) Fpkm of the transcript EDK37994 (b) Alignment of PGUG_02092 and read1 (c) Alignment of PGUG_02092 and read2 |
以cDNA为模板,据A5DFP1蛋白N端前8个残基编码序列和C端终止密码及其后基因组序列设计精确引物,PCR扩增直接获得唯一900bp左右产物(图 4)。
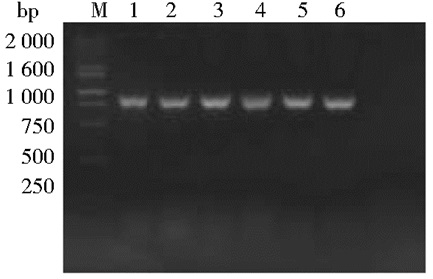
|
| 图 4 PCR产物及阳性克隆的琼脂糖凝胶电泳 Figure 4 Agarose gel electrophoresis for the PCR product and positive monoclone M: Maker DL2000; 1:PCR product of the primer PF1/PR1; 2:PCR product of the primer PF2/PR2; 3-4:Positive clones bearing N-6His; 5-6:Positive clones bearing C-6His |
将胶回收纯化PCR产物连接至T载体用通用引物测序,发现其与A5DFP1编码序列仅有第435位碱基不同;A5DFP1中此位置为G,而在此C.G.M.C.C 2.1008酵母菌中为C,但对应氨基酸序列仍相同(详见Genbank登录号KY706244.1)。重复用cDNA为模板再次PCR扩增后测序确认该位点仍为C。用基因组DNA和相同引物扩增,所得PCR产物序列也一致。将PCR复性温度升高3℃,仍然获得相同的唯一PCR产物(数据未给)。MALDI-TOF-MS测定天然酶分子质量与按A5DFP1氨基酸序列推算相差近1.0kDa,但天然MGU的N端被封闭而无法Edman降解测序。因此,此MGU氨基酸序列可能除了N端外应与A5DFP1氨基酸序列相同,或天然酶有较大体积的修饰基团。
2.3 MGU的重组表达和表征初步用5′-RACE扩增MGU的N端未知编码序列未获得有意义新序列。真菌尿酸酶的重组表达较为容易,故先重组表达考察产物与天然MGU是否有相同性质。真核尿酸酶适合用6His标签融合表达以方便纯化[12]。同时尿酸酶多肽链两端参与构成了活性中心[13];在此真菌尿酸酶任一端添加6His标签可能显著降低其活性,但无法确认是因为添加标签肽造成活性下降,还是克隆出错或N端编码序列不全使所得尿酸酶活性下降。所以,分别设计了N端、C端带6His标签,以及无标签的表达载体进行表达并比较。
三种形式的MGU诱导表达后都有显著的尿酸酶活性(大肠杆菌宿主细胞无内源尿酸酶活性)。将重组尿酸酶pDE1-MGU和pDE2-MGU经镍柱纯化;无标签R-MGU经两次DEAE纤维素柱层析纯化后,再用SDS-PAGE比较。结果发现三种表达载体所得肽段分子质量相差很小(图 5)。R-MGU经DEAE纤维素柱层析纯化效果太差。但参照此前酸性电泳分离MGU的条件[7],用非变性酸性电泳获得高纯度肽段却都没有尿酸酶活性。
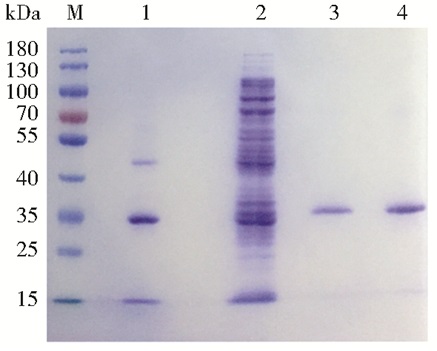
|
| 图 5 天然及重组尿酸酶SDS-PAGE Figure 5 SDS-PAGE for MGU and recombined uricases M: Protein maker; 1: MGU; 2: R-MGU; 3: pDE1-MGU; 4: pDE2-MGU |
测定三种形式重组MGU的酶学性质,发现三者比活性差异显著(表 1),但显然这主要来自纯化效果差异。三者Km无显著差异(表 2)。氧嗪酸钾和黄嘌呤是MGU的代表性强竞争性抑制剂[1, 7];三种重组尿酸酶对这两种竞争性抑制剂的敏感性也无显著差异(表 2)。令人惊喜的是,尿酸酶三种重组表达形式在pH 7.4时活性都比在pH 9.2时更高,与天然MGU一样表现为最适pH接近中性。
| 尿酸酶 | pH 7.4 | pH 9.2 | |||||
| Km | 氧嗪酸钾Ki | 黄嘌呤Ki | Km | 氧嗪酸钾Ki | 黄嘌呤Ki | ||
| MGU | 42±4 | (3.0±0.8)×10-2 | 4±1 | 31±4 | 1.4±0.2 | 7.5±0.3 | |
| pDE1-MGU | 31±4 | (2.0±0.6)×10-2 | 5.7±0.2 | 33±2 | 1.8±0.3 | 2.7±0.3 | |
| pDE2-MGU | 35±2 | (1.0±0.1)×10-2 | 3.3±0.3 | 38±4 | 2.6±0.3 | 4.3±0.4 | |
| R-MGU | 30±3 | (2.2±0.1)×10-2 | 3.9±0.4 | 28±4 | 1.4±0.2 | 3.5±0.2 | |
分别用制备型酸性电泳纯化R-MGU和天然MGU至单一蛋白质带[7],再比较其分子大小。在相同MALDI-TOF-MS设备用相同校准品测定天然MGU和R-MGU肽段分子质量;天然MGU分子质量为17.434kDa,而R-MGU分子质量为17.429kDa (图 6),二者的差别在MALDI-TOF-MS测量误差范围内。特殊的是,将MGU和R-MGU基本等量混合后再用MALDI-TOF-MS测定分子质量时,仍然得到单峰且肽段分子质量为17.428kDa。可见,重组表达的无标签R-MGU与天然MGU氨基酸序列和化学修饰可能都相同。
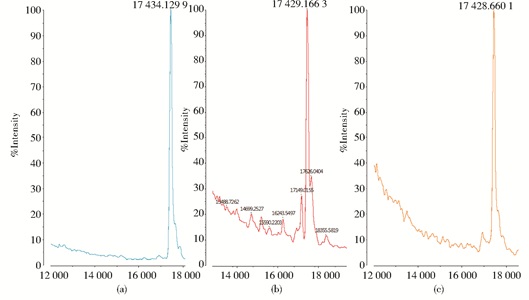
|
| 图 6 MALDI-TOF-MS比较天然MGU及重组R-MGU的分子质量 Figure 6 Comparison of molecular weights of MGU, R-MGU and their mixture (a): MGU (b): R-MGU (c): mixture of MGU and R-MGU |
天然MGU在pH 7.4加氧嗪酸钾及在pH 9.2时热失活初期都出现活性基本不降的平台期[7],三种重组表达形式在热失活初期都无这种平台期(图 7)。在pH 7.4,无配体时重组R-MGU的热失活半衰期比天然MGU短,但有配体时半衰期基本无差异;在pH 9.2,无配体时C端带6His MGU热失活半衰期显著短于天然MGU,但有配体时差距减小。
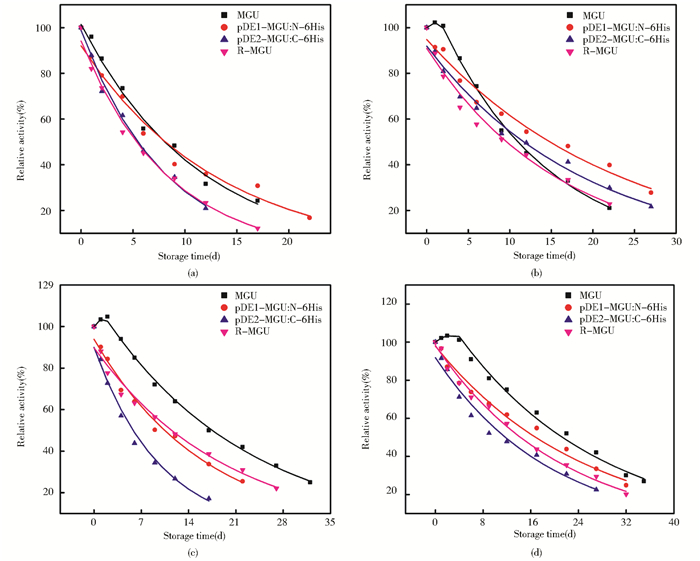
|
| 图 7 天然及重组尿酸酶的热失活过程 Figure 7 Thermoinactivation processes for MGU andthe recombined uricases (a) pH 7.4 (b) pH 7.4 plus oxonate (c) pH 9.2 (d) pH 9.2 plus oxonate |
图 5及表 1结果表明无标签R-MGU在大肠杆菌中重组表达后比在天然宿主细胞中更难纯化[1, 7],可能大肠杆菌宿主细胞中有较多等电点相近的蛋白质。文献报道用6His标签成功表达真核尿酸酶[12]。但尿酸酶N端和C端序列显著影响其活性和热稳定性[13]。比较在N端和C端添加6His标签的重组表达MGU表明,6His标签添加到N端或C端对其最大活性和对底物亲和力等性质影响较小(表 1和表 2),特别是其活性的pH效应未显著改变(数据未给)。可见,MGU适合用6His标签融合表达以便于快速纯化。
SDS-PAGE分析天然及重组R-MGU蛋白分子质量均在35kDa左右,但可能由于多肽在MALDI-TOF-MS时带有两个正电荷故其肽链分子质量接近17.4kDa。总体而言,本文结果支持天然MGU氨基酸序列与在大肠杆菌中按A5DFP1氨基酸序列重组表达的R-MGU一致。Edman降解测序时发现其肽链的N端封闭[7],表明其存在N端化学修饰。多次用MALDI-TOF-MS测定所得MGU肽段的分子质量都比按A5DFP1氨基酸序列计算分子质量大,接近1.0kDa;如此大差距不可能来自实验测定误差。球形节杆菌尿酸酶的N端有超出其链间β折叠的短肽段,该酶的热稳定性非常优良[13-16]。苛求芽孢杆菌尿酸酶的N端和C端序列缺失都会造成其热稳定性显著下降[13, 17-18]。所以,可能天然MGU有特殊修饰并且此修饰与其热稳定性相关;R-MGU能再现天然MGU的N端修饰,为鉴定其化学修饰及此修饰在MGU酶学性质中的意义奠定了基础。
医药用酶的价值依赖于其比活性及热稳定性。三种重组MGU热稳定性在pH 7.4与天然MGU差距很小,但在pH 9.2差距增大(图 7)。可能N端或C端6His标签影响重组MGU热稳定性。R-MGU纯度低,污染蛋白酶对蛋白质降解肯定会降低R-MGU热稳定性。现有尿酸酶最适pH都偏碱,降低了其作为诊断工具酶及药用酶的价值。MGU是至今发现的第一个最适pH接近中性的尿酸酶,解析其最适pH的决定因素,对研制在生理条件下活性更高的药用尿酸酶、适合双试剂法测定血清尿酸的诊断工具酶[16],无疑都有重要价值。MGU存在还未鉴定的化学修饰,且该修饰可能与其最适pH及热稳定性有关;解析其化学修饰本质及在此酶最适pH和热稳定性中的意义,并解析其最适pH的决定因素,是下一步的研究重点。首次在上海委托串联质谱分析所得MGU肽段信息,误导了MGU编码序列克隆的方向;多角度交叉验证是获得可靠实验结果的必要途径。正是基于多角度交叉验证MGU与A5DFP1的序列相似性,本文才能快速确认来自细胞株C.G.M.C.C 2.1008最适pH为中性尿酸酶的编码序列,为后续研究奠定基础。
| [1] |
武文明, 曾昭淳, 李小彦, 等. 产朊假丝酵母尿酸酶的纯化和特性研究. 西南大学学报(自然科学版), 2008, 30(3): 84-89. Wu W M, Zeng Z C, Li X Y, et al. Purification and characterization of the uricase from Candida utilis. Journal of Southwest University (Natural Science Edition), 2008, 30(3): 84-89. |
| [2] |
Ishizaka N, Ishizaka Y, Toda E, et al. Association between serum uric acid, metabolic syndrome, and carotid athero-sclerosis in Japanese individuals. Arterioscler Thromb Vasc Biol, 2005, 25(5): 1038-1044. DOI:10.1161/01.ATV.0000161274.87407.26 |
| [3] |
Duncan P, Gochman N, Bayse D, et al. A candidate reference method for uric acid in serum. Ⅱ. Interlaboratory testing. Clinical Chemistry, 1982, 28(2): 291-293. |
| [4] |
Koyama Y, Ichikawa T, Nakano E. Cloning, sequence analysis, and expression in Escherichia coli of the gene encoding the Candida utilis Urate Oxidase (Uricase). Journal of Biochemistry, 1996, 120(5): 969-973. DOI:10.1093/oxfordjournals.jbchem.a021514 |
| [5] |
Pui C H, Relling M V, Lascombes F, et al. Urate oxidase in prevention and treatment of hyperuricemia associated with lymphoid malignancies. Leukemia, 1997, 11(11): 1813-1816. DOI:10.1038/sj.leu.2400850 |
| [6] |
朱献军, 刘建国, 黎高翔. 尿酸氧化酶基因的克隆、表达及其产物的应用. 生物工程学报, 2001, 17(1): 68-72. Zhu X J, Liu J G, Li G X. Cloning and expression of urate oxidase and its application in serum uric acid analysis. Chinese Journal of Biotechnology, 2001, 17(1): 68-72. |
| [7] |
Wu J, Yang X, Wang D, et al. A numerical approach for kinetic analysis of the nonexponential thermoinactivation process of uricase. Protein Journal, 2016, 35(4): 1-12. |
| [8] |
赵运胜, 卜友泉, 廖飞. 用简并引物快速克隆苛求芽孢杆菌尿酸酶的编码序列. 重庆医科大学学报, 2017, 42(2): 219-224. Zhao Y S, Bu Y Q, Liao F. Rapid cloning gene sequence of the uricase from Bacillus fastidious by degenerated primer. J Chongqing Med Univ, 2017, 42(2): 219-224. |
| [9] |
赵运胜, 赵利娜, 杨根庆, 等. 苛求芽孢杆菌胞外尿酸酶的表征及其在血清尿酸测定中的应用. 华中科技大学学报(医学版), 2007, 36(2): 239-242. Zhao Y S, Zhao L N, Yang G Q, et al. Characterization of an extracellular uricase from Bacillus fastidious and its application in direct kinetic assay of serum uric acid. Acta Med Univ Sci Technol Huazhong, 2007, 36(2): 239-242. |
| [10] |
Liao F, Zhu X Y, Wang Y M, et al. The comparison of the estimation of enzyme kinetic parameters by fitting reaction curve to the integrated Michaelis-Menten rate equations of different predictor variables. Journal of Biochemical & Biophysical Methods, 2005, 62(1): 13-24. |
| [11] |
Liu X, Wen M, Li J, et al. High-yield expression, purification, characterization, and structure determination of tag-free Candida utilis uricase. Applied Microbiology & Biotechnology, 2011, 92(3): 529-537. |
| [12] |
吴双林, 陈斌, 刘成倩, 等. 猪尿酸氧化酶在大肠杆菌中的表达、纯化与部分酶学性质分析. 生物工程学报, 2009, 25(11): 1664-1670. Wu S L, Chen B, Liu C Q, et al. Expression in Escherichia coli, purification and enzymatic properties of porcine urate oxidase. Chin J Biotech, 2009, 25(11): 1664-1670. DOI:10.3321/j.issn:1000-3061.2009.11.011 |
| [13] |
Feng J, Wang L, Liu H, et al. Crystal structure of Bacillus fastidious uricase reveals an unexpected folding of the C-terminus residues crucial for thermostability under physiological conditions. Applied Microbiology & Biotechnology, 2015, 99(19): 7973-7986. |
| [14] |
陈丹, 韩晶, 史洪娜, 等. 球形节杆菌尿酸酶的表达、纯化及其活性. 中国生物制品学杂志, 2014, 27(3): 351-355. Chen D, Han J, Shi H N, et al. Expression, purification and activity of Arthrobacter globiformis uricase. Chinese Journal of Biologicals, 2014, 27(3): 351-355. |
| [15] |
Suzuki K, Sakasegawa S I, Misaki H, et al. Molecular cloning and expression of uricase gene from Arthrobacter globiformis in Escherichia coli and characterization of the gene product. Journal of Bioscience & Bioengineering, 2004, 98(3): 153-158. |
| [16] |
Huang Y, Chen Y, Yang X, et al. Optimization of pH values to formulate the bireagent kit for serum uric acid assay. Biotechnology & Applied Biochemistry, 2015, 62(1): 137. |
| [17] |
Zhang C, Yang X, Feng J, et al. Effects of modification of amino groups with poly(ethylene glycol) on a recombinant uricase from Bacillus fastidiosus. Biosci Biotechnol Biochem, 2010, 74(6): 1298-1301. DOI:10.1271/bbb.100080 |
| [18] |
Zhang C, Yang X, Gao A, et al. Comparison of modification of a bacterial uricase with N-hydroxysuccinimide esters of succinate and carbonate of monomethoxyl poly(ethylene glycol). Biotechnol Appl Biochem, 2014, 61(6): 683-690. DOI:10.1002/bab.2014.61.issue-6 |
 2017, Vol. 37
2017, Vol. 37




