文章信息
- 许妍, 赵雪, 堵晶晶, 杨琼, 杨大洪, 蒲红州, 张顺华, 朱砺.
- XU Yan, ZHAO Xue, DU Jing-jing, YANG Qiong, YANG Da-hong, PU Hong-zhou, ZHANG Shun-hua, ZHU Li.
- 环状RNA研究进展
- Research Progress of CircularRNAs
- 中国生物工程杂志, 2017, 37(10): 93-102
- China Biotechnology, 2017, 37(10): 93-102
- http://dx.doi.org/DOI:10.13523/j.cb.20171013
-
文章历史
- 收稿日期: 2017-06-06
- 修回日期: 2017-07-13
2 成都农业科技职业学院 成都 611130
3 四川省雅安市名山区农业局 雅安 625100
4 四川省南江县农业局 巴中 635600
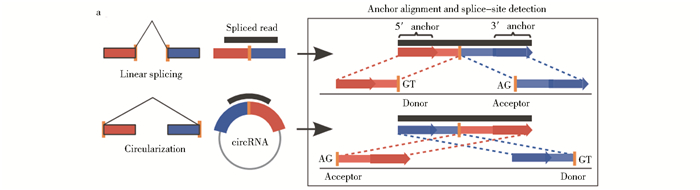
circRNA是一类内源性非编码RNA(ncRNA),最早发现于1971年。当时,研究者在研究马铃薯纺锤块茎病时发现类病毒(viroids)能侵染植株并导致其死亡。但与病毒不同的是,类病毒没有蛋白质外壳包被,且其基因组是单链闭合的RNA分子[1]。1979年,Hsu等[2]通过电子显微镜观察到真核细胞细胞质中存在圆环形RNA。1993年,研究者发现小鼠精子决定基因Sry在转录过程中存在环状转录[3]。但这些发现在当时并没有引起研究者太大注意。直到2012年,借助于高通量测序技术,Salzman等[4]研究发现无论是白血病细胞、Hela细胞系还是正常人原代血细胞均存在通过外显子重排形成非线性的环状转录物现象,约80个环状RNA被首次报道。自此,大量的circRNA分子相继得到鉴定。然而,circRNA没有系统的命名规则,研究者一般根据circRNA的亲本基因或者与其结合的miRNA来命名,但这种命名法存在诸多弊端,如会出现同一个circRNA有多个命名。如何建立一套完善的、公认的circRNA命名系统也是当前研究circRNA亟需解决的重要问题。
在很长一段时间里,研究者认为无5′端帽子和3′端多聚腺苷酸尾巴的环状闭合结构的circRNA是错误剪接或者由低丰度mRNA转录产生的副产物[5-6]。最近发现circRNA可在哺乳动物的细胞质中大量地稳定存在[4, 7]。尽管如此,对circRNA的形成机制依然不清楚。目前,学界存在两种主流的circRNA形成机制,即套索驱动模型和内含子配对驱动模型。如图 1所示,套索驱动模型认为pre-RNA在转录过程中由于RNA发生了部分折叠,拉近相邻的外显子,从而致使外显子跳跃(exon skipping),之后被跨越的区域由剪接供体(splice donor;SD)与剪接受体(splice acceptor;SA)结合形成了circRNA中间体,并进一步通过套索剪接形成由外显子构成的circRNA分子。该模型认为,任何的外显子跳跃事件都有可能产生一个外显子circRNA[4, 8]。而内含子配对驱动模型认为,位于内含子区域的反向互补序列导致了内含子区域配对,使与内含子接壤的两个外显子相互靠近,进一步反向剪接形成circRNA分子。众所周知,内含子上的ALU重复序列可以促使外显子环化[9],也有研究表明,在线虫和人的成环外显子的侧翼内含子上还存在反向互补序列,通过双链RNA编辑酶ADAR1的参与,使内含子反向互补序列在生成circRNA时起到关键作用,当敲除ADAR1时circRNA表达显著上调。这使内含子间的互补配对序列通过竞争性结合的方式介导外显子环化模型得到支持[10]。两种模型的不同之处在于剪接事件发生的顺序不同,剪接受体需要的环化外显子的3′位点不同,以及最终剪接所形成的包含了被跳跃的外显子的线性转录本不同。
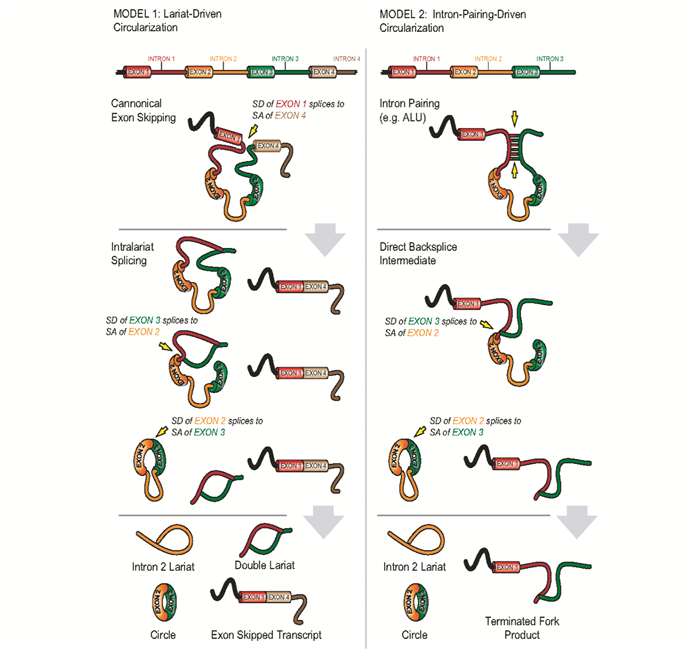
|
| 图 1 索尾插接(Backsplicing)形成模型和circRNA形成机制 Figure 1 Backsplicing and circRNA formation mechanisms In Model 1, exon-skipping leads to a lariat whose restricted structure promotes circularization. In Model 2, exon-skipping is not required, with ALU complementarity or other RNA secondary structures bringing nonsequential donor-acceptor pairs into apposition, allowing for circularization[7] |
细胞中除了大量的外显子circRNA外,还存在少量ciRNA(circular intronic RNA;ciRNA)。大部分内含子在剪切作用后会被降解,但有研究发现,一些存在特殊核酸序列(在5′连接位点上包含了7ntGU序列和在3′分支位点上包含11ntC序列的共有基序)的内含子在剪切后不会被脱分支酶降解,从而形成了ciRNA[11-12]。
2 circRNA的特征大量的研究表明,circRNA在哺乳动物细胞中具有内生、丰富、保守、稳定等特点[7, 13]。通过测序和计算分析人类、老鼠和线虫RNA,发现大量circRNA可稳定表达,并经常表现出组织特异性或时空特异性[14]。circRNA的特征与其作用紧密相关,因此分析circRNA特征对于研究其生物学功能具有重要意义。
2.1 稳定性与线性RNA不同,circRNA可形成不具有5′端帽子和3′端多聚腺苷酸尾巴的共价闭合环状结构(图 2)。这使得circRNA可免受核糖核酸酶和核酸外切酶的降解,具有比线性RNA更长的半衰期、更强的稳定性[15]。如Bahn等[16]通过RNA-Seq对人类无细胞唾液进行circRNA全基因组分析,发现在唾液中存在超过400种circRNA,由于体液中含有大量核酸酶,circRNA在体液中大量表达证明其具有较高的稳定性。目前,大量研究者运用核酸酶处理RNA样品以鉴定、辨别circRNA [11]。此外,不含开放阅读框的闭合环状结构也被认为是circRNA不具备编码蛋白能力的原因之一。
生物分子的进化使得物种从低级到高级不断提高,蛋白质和核酸等生物大分子必须具有保守性才能在进化中得以保存。circRNA在不同物种间具有高度的进化保守性。Salzman等[13]发现人类与小鼠的circRNA有4%来自直系同源基因。还有研究发现,大脑和睾丸中有44%的circRNA是相同的[7]。此外,Werfel等[17]分析了人类、大鼠和小鼠心肌的circRNA,发现每个物种中都能筛选出超过9 000个circRNA,其中大鼠和小鼠间有30%的circRNA具有保守性,在三个物种中都具有保守性的circRNA达到10%。
2.3 亚细胞定位circRNA在细胞质和细胞核内均有分布。大部分circRNA在细胞浆中富集,其丰度有时甚至比相应的线性mRNA高10余倍,这可能是由于circRNA比线性RNA更稳定造成的。核酸酶往往通过识别线性RNA分子末端发挥作用,circRNA是一个闭合结构,因此对核酸酶具有高耐受性[7]。细胞质中的外显子circRNA上存在miRNA反应元件(miRNA response elements;MREs),作为miRNA的海绵分子(Sponge),在转录后水平发挥调控作用[13, 18]。也有研究表明,内含子形成的circRNA会大量存在于细胞核。例如,ci-ankrd52的形成过程取决于在5′连接位点上包含了7ntGU序列和在3′分支位点上包含11ntC序列的共有基序,这些ciRNA上的miRNA结合位点很少,并富集在miRNA转录位点上,可作为RNA聚合酶Ⅱ翻译的正向调控者,敲除这些circRNA会导致它们的亲本基因表达量减少。故ciRNA主要作用是调控基因转录[19]。
2.4 组织特异性circRNA具有组织特异性。例如,hsa_circRNA_2149在CD19+粒细胞中被发现,而CD34+粒细胞,嗜中性粒细胞或HEK293细胞中则没有。线虫的一些circRNA只在卵母细胞中发现,而在1、2期胚胎细胞中没有发现这些circRNA[18]。我们可以通过circRNA在不同组织中的表达特异性初步判断该circRNA对哪些组织的生长发育过程中起调控作用。在疾病研究中,circRNA的组织特异性可以使它成为特定组织疾病的诊断因子。
2.5 较高的丰度和丰富的种类circRNA丰度高。Zheng等[20]通过对circRNA的宿主基因分析,发现一个基因可产生多个circRNA。例如致癌基因PTK2,可生成47个不同的circRNA。并且,一个基因位点产生的circRNA往往有一个主导的circRNA亚型,剪接的外显子或内含子位置不同还会产生其他丰度较少的亚型,所以circRNA的丰度比其线性的mRNA要高。
circRNA可来自基因组上多种基因结构,其类型远比想象的要复杂,目前,研究者将circRNA主要分为以下几类:(1)外显子来源的circRNA(exonic circRNA):该类circRNA可以由一个外显子形成,也可由多个外显子通过外显子跳跃或内含子区域反向互补序列驱动外显子环化形成;(2)内含子来源的circRNA(circular intronic RNA):该类circRNA位于已知基因的某个内含子内,由内含子自身环化形成,依赖于5′剪切位点附近的7ntGU序列和3′分支位点的11nt C序列共有基序;(3)外显子-内含子来源的circRNA(Exon-intron circular RNA):该类circRNA是由外显子与保留在外显子之间的内含子通过套索驱动模型环化形成。
3 circRNA的作用机制circRNA发挥作用的机制主要包括:作为竞争性內源RNA[21-23];调节选择性剪接或翻译[24];调控亲本基因的表达[19, 25];与蛋白互作[18, 26]等方式发挥生物学功能。
3.1 作为竞争性內源RNAMicroRNA是一类重要的基因表达转录后调控因子,长度约为22nt。可与靶基因mRNA的3′UTR区域结合,降解或抑制mRNA翻译[27]。生物体内的竞争性內源RNA(competitive endogenous RNA,ceRNA),如mRNA,假基因和长链非编码RNA都可竞争性地与miRNA结合[27-28],从而影响miRNA对基因表达的调控。
如图 3所示,ceRNA假说是指miRNA可通过结合mRNA导致基因沉默,而ceRNA可通过应答元件(microRNA response elements,MREs)竞争性地结合microRNA来调节靶基因表达[29]。ceRNA并不是一种新的RNA分子,而是一种新发现的基因表达调节机制。ceRNA的种类有很多,如lncRNA,假基因等。最近,circRNA也被认为是一种ceRNA,它可通过碱基互补配对作用像海绵一样吸附miRNA,从而调节miRNA靶基因的表达,以发挥作用[30] (表 1)。如在胃癌组织中随病程逐渐上调的circPVT1可与miRNA-125家族(miRNA-125a, miRNA-125b, let-7b)结合,促进其靶基因E2F2的表达,进而促进癌细胞的增殖[31]。在心肌肥大和心力衰竭模型中表达量显著降低的circRNA-HRCR可作为内源性miRNA-223的海绵,上调细胞凋亡抑制因子ARC,从而抑制心肌肥大和心力衰竭[32]。circRNA的发现极大地丰富了人们对真核生物和ceRNA调控作用网络的认识。
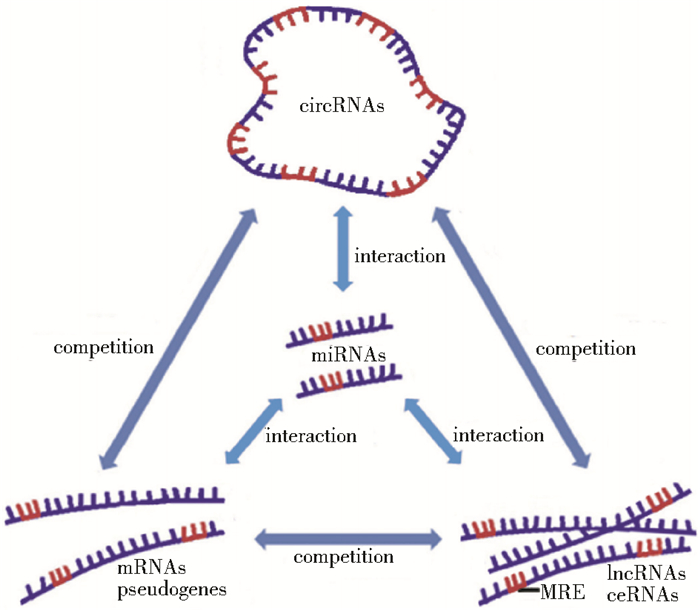
|
| 图 3 ceRNA网络 Figure 3 Network of ceRNAs circRNAs, mRNAs, pseudogenes and lncRNAs act as ceRNAs to regulate their respective roles, which contain shared MREs to compete for microRNA binding. They maintain dynamic balance to regulate cellular homeostasis. If homeostasis of ceRNAs is dysregulated, the diseases may occur[33] |
| circRNA | 物种 | 细胞/组织 | Sponge miRNA | 靶基因 | 功能 | 文献 |
| CDR1as | 斑马鱼 | 神经组织 | miRNA-7 | —— | 损伤中脑发育 | [30] |
| CDR1as | 小鼠 | 胰岛β细胞 | miRNA-7 | Pax6 Myrip | 促进胰岛素分泌 | [34] |
| Circ100284 | 人 | 角质细胞 | miRNA-217 | EZH | 促进癌细胞增殖 | [35] |
| CircPVT1 | 人 | 胃组织 | miRNA-125 cluster | ERF2 | 促进胃癌细胞增殖 | [31] |
| HRCR | 小鼠 | 心脏组织 | miRNA-223 | ARC | 抑制心肌肥大及心力衰竭 | [32] |
| CircHIPK3 | 人 | 癌组织 | miRNA-124 | IL6R DLX2 | 抑制癌细胞增殖 | [20] |
| Cir-ITCH | 人 | 食管鳞状细胞 | miRNA-7 miRNA-17 miRNA-214 |
-- | 减慢癌细胞周期, 抑制食管癌 | [23] |
| Cir-ZNF609 | 人 | 结肠组织 | miRNA-150-5p | AKT3 | 可用于诊断先天性巨结肠症 | [36] |
3.2 调节选择性剪接或翻译
选择性剪接(也叫可变剪接)是指从一个mRNA前体中通过不同的剪接方式(选择不同的剪接位点组合)产生不同的mRNA剪接异构体的过程,最终的蛋白产物会表现出不同或者是相互拮抗的功能和结构特性,或者在相同细胞中由于表达水平的不同而导致不同表型[37]。最近,Chen[38]发现circRNA可参与剪接和转录的调控。Chao等[39]发现小鼠的Fmn基因可以通过“索尾拼接(backsplicing)”来产生circRNA,这个circRNA包含了作为“mRNA trap”的转录起始位点,且遗留了一个非编码线性转录从而减少黄素蛋白的表达水平。
翻译是根据遗传密码的中心法则,将成熟的信使RNA分子(由DNA通过转录而生成)中“碱基的排列顺序”(核苷酸序列)解码,并生成对应的特定氨基酸序列的过程。大多数研究者将circRNA归类为非编码RNA就是因为其在通常情况下不能被翻译。但近期研究人员发现许多单外显子circRNA包含了人类成纤维细胞翻译起始位点[26]。circRNAs可通过螯合翻译的起始位点来作为mRNA的Trap从而调节蛋白的表达水平。此外,Abe等[40]发现,包含多个开放阅读框(open reading frame, ORF)且没有终止密码子的circRNA在大肠杆菌中可以有效地翻译为蛋白质,类似于聚合酶的滚环扩增(rolling circle amplification, RCA)。这些研究说明在某些特定条件下circRNA可以翻译为有功能的蛋白质。
3.3 调节亲本基因的表达调控其亲本基因的表达是非编码RNA主要作用之一,然而不同类型非编码RNA对亲本基因的调控机制不同。如长链非编码RNA可通过染色质状态的表观遗传修饰来调控亲本基因的表达[41],roX1, roX2等非编码RNA可反式调节基因转录[42]。最近,研究者首次在人类细胞中找到了ciRNA并证明它可以富集在编码自身基因的转录位点。Zhang等[43]发现ciRNA的形成依赖于关键侧翼RNA分子,这些ciRNA很少富集在microRNA的靶位点上,暗示它们在功能上可能与外显子circRNA存在差异。外显子之间保留有内含子的一类circRNA称为exon-intron circRNA或EIciRNA,研究者发现它们主要定位于细胞核,可通过与特异性RNA相互作用顺式调节其亲本基因聚合酶Ⅱ的转录,从而促进其亲本基因转录[12, 44]。如通过紫外交联-免疫共沉淀RNA聚合酶Ⅱ,Zhang等[12]发现Hela细胞中存在111个EIciRNA。进一步研究表明,表达量较高的circEIF3J和circPAIP2可以和U1互补配对,从而与U1小核糖核蛋白相互作用顺式促进EIF3J和PAIP2基因的转录。此外,circRNA(cir-ITCH)不仅能与其亲本基因ITCH的3′UTR共享一个miRNA结合位点,且可与miRNA-7,miRNA-17,miRNA-214相互作用,提高ITCH的表达水平[23]。
3.4 与蛋白互作研究表明,在RBP装配或者变构调节中起作用的circRNA可绑定RNA结合蛋白(RNA binding proteins, RBP),进而发挥生物学功能,如circMbl与MBL蛋白有强烈的直接相互作用,MBL蛋白的多种亚型可以通过自身外显子促进circMBI产生。特别是当MBI蛋白过量时,它通过促进circMBI的产生来降低自身mRNA产量。此外,circMBI也可通过绑定MBI蛋白从而清除多余的MBI蛋白[24]。外显子来源的circRNA能够和AGO蛋白以及RNA聚合酶Ⅱ相互作用,从而作为microRNA的海绵分子来参与转录调控[26, 45]。因此,circRNA可以与不同的蛋白分子相结合,以提高DNA, RNA, RBP之间的相互作用进而发挥其生物学功能。
4 circRNA的研究方法获取海量circRNA数据(表 2)是进一步分析、研究circRNA特征、功能、作用机制等的基本前提。目前,对特定组织或细胞进行circRNA高通量测序的方法主要有两种:一种是根据circRNA闭合环状特性,通过核酸酶处理去除线性RNA分子,特异地富集circRNA分子后测序,另一种是不经核酸酶处理直接测序,之后再通过生物信息学对潜在的circRNA进行筛选。前者测出的circRNA纯度较高,更有针对性地分析受检样本中特定的circRNA谱、circRNA剪接模式、组间差异和功能等,但由于部分circRNA可以被消化,就使低丰度的circRNA被去除,导致circRNA信息不全。后者虽能检测出全部的circRNA,但由于生物信息分析手段的局限性,可能会将线性RNA误判为circRNA,导致假阳性率较高。因此,经核酸酶处理后进行高通量测序是目前发现circRNA的主要手段。尽管如此,高通量测序发现的circRNA仍然需要进一步用实验进行最终鉴定。由于circRNA具有稳定的环状结构,而线性RNA不具备此特点,所以用核酸外切酶RNase R,5′端核酸外切酶等处理可以使大部分线性RNA降解,而circRNA不被降解[20]。取1μg总RNA用1U(1个单位)的RNase R,在37℃下消化15min左右(不同品牌的酶有不同消化时间,根据说明书使用)。用放线菌素D处理细胞也可发现circRNA的半衰期明显要比线性RNA高得多,这样可以初步鉴定出circRNA[46];同时可以设计circRNA的发散性引物(divergent primers)和收敛性引物(Convergent primers)对cDNA和全基因组DNA(gDNA)进行circRNA的扩增,通过跑胶可以发现circRNA在cDNA中用发散性引物可以扩增出来,但在gDNA中扩增不出来。这是由于发散性引物促使circRNA成环,只有在单链的cDNA中可以发挥作用,而双链的gDNA不可成环[32]。对于circRNA功能的验证方法较多,例如RNA结合蛋白免疫沉淀技术(RNA Binding Protein Immunoprecipitation, RIP),可以用来研究细胞内circRNA与蛋白结合情况,可以通过AGO2蛋白的免疫共沉淀的富集结果来验证circRNA与其相结合的miRNA是靶向结合的[47]。荧光原位杂交技术(Fluorescence in situ hybridization, FISH),可以用来对circRNA进行细胞亚定位,利用siRNA和反义寡核苷酸可以干扰circRNA的表达从而进行circRNA的功能验证[20]。
| 名称 | 描述 | 网址 |
| circBase | 汇集大量已知或未知功能的circRNA数据 | http://www.circb ase.org/ |
| circRNABase | miRNA-circRNA相互作用网络预测与分析 | http://starbase.sy su.edu.cn/mircircRNA.php |
| Circ2Traits | 汇集circRNA相关的疾病及表征形状数据 | http://gyanxet-b eta.com/circdb/ |
| deepBase | 包含约15万circRNA基因(人、鼠、果蝇、线虫等),构建了最全面的circRNA的表达图谱。有效且灵活地整合在线circRNA相关数据库数据,可为实验提供思路和指导 | http://deepbase.sysu.edu.cn/ |
| CircNet | 数据库数据基于转录测序结果,并进一步联合microRNA等信息进行了整合分析,主要包含:circRNA分子序列;circRNA与miRNA及靶基因的调控网络;circRNA分子的表达谱;circRNA分子的基因组注释 | http://circnet.mbc.nctu.edu.tw/ |
5 circRNA与疾病的关系
人类基因组可广泛转录生成大量非编码RNA,如miRNA、lncRNA、piRNA等,它们很多都与疾病的发生、发展密切相关[28]。最近研究表明,circRNA在多种疾病中扮演着重要的角色[48-49]。
5.1 心血管疾病心力衰竭是心脏疾病发展的终末阶段,属世界性的高发性致死疾病。研究表明,心脏肥大与心衰风险增加显著相关[50]。一些因子如G蛋白偶联受体,肾上腺素,血管紧缩素,内皮缩血管肽(ET-1)感受器,都是心脏功能和心肌肥大生长的关键调节因子[45, 51-52]。最近,越来的越多的研究表明circRNA在调控心肌细胞发展、分化、增殖和凋亡中发挥重要作用[53]。在小鼠中过表达在病理性缺陷心脏组织中高表达的miRNA-223后发现随着年龄增长,实验鼠表现出心肌肥大,间质纤维化增加,心脏应激基因表达增加等特征。在ISO诱导的心肌肥大小鼠中强制表达circRNA HRCR可显著缓解心肌肥大。进一步的研究表明,circRNA HRCR可竞争性结合内源性miRNA-223,释放靶基因ARC,进而保护心脏不受病理性肥大和心力衰竭影响。该结果提示circRNA可作为治疗心肌肥大和心力衰竭的潜在靶标[32]。此外,circRNA在血管及其相关疾病中的作用也有所研究。例如吸烟,肥胖和高血脂等因素诱发的动脉粥样硬化。cANRIL(circular antisense non-coding RNA in the INK4 locus)是由INK4/ARF基因反转录合成的。cANRIL亚型的表达与INK4/ARF转录和动脉粥样硬化风险有关。通过使用下一代DNA测序和剪接预测算法,确定染色体9p21基因多态性在有动脉粥样硬化风险患者中可调节ANRIL剪接和cANRIL的产生[54],故circRNA在动脉粥样硬化中可能也扮演着重要角色。
5.2 糖尿病在Ⅱ型糖尿病晚期,患者常出现各种并发症。然而,目前的Ⅱ型糖尿病的早期诊断存在很多不足,需要一个新的高灵敏度的生物标记物来作为Ⅱ型糖尿病的预后指标。Zhao等[55]通过基因芯片技术分析比较了Ⅱ型糖尿病患者和对照组的外周血中circRNA表达谱,结果表明hsa_circ_0054633对于诊断糖尿病前期和糖尿病,是一个有较高灵敏度和特异性的生物标志物。已经证实了circRNA Cdr1as是miRNA-7的海绵分子,由于miRNA-7在胰岛细胞中大量表达,并且可导致糖尿病。研究表明,过表达Cdr1as可抑制miRNA-7的表达,从而上调miRNA-7的靶基因Myrip和Pax6的表达,导致胰岛素分泌增加,也发现过表达Cdr1as,使胰岛素1和胰岛素2基因的mRNA表达量增加,这就证明circRNA Cdr1as会影响胰岛素分泌和胰岛B细胞的更新[34]。
5.3 癌症研究表明miRNA、lncRNA可作为胃癌、结直肠癌等癌症疾病的标志物用于临床诊断。circRNA在哺乳动物中因具有较高的保守性和稳定性,更有潜力成为诊断癌症的理想的生物标志物[56-57]。Li等[58]发现在胃癌组织中显著下调的Hsa_circ_002059,其表达水平与胃癌细胞的转移,TNM分期,性别和年龄都相关。如对于女性或其他年龄段,Hsa_circ_002059在60岁以上的男性胃癌组织中表达量更低。相比于手术前,胃癌治疗手术后的Hsa_circ_002059在血浆中的表达水平显著升高。由于基因组位点的扩增而表达上调的circPVT1可通过“海绵作用”吸附miR-125家族而抑制胃癌细胞增殖。经单变量和多变量的Cox比例风险分析表明,circPVT1可作为胃癌患者生存的独立预后指标,它与TNM分期结合能更好地预测胃癌患者的存活率[31]。最近的研究表明,circPVT1的宿主基因PVTI主要通过调节原癌基因c-myc的蛋白稳定性来参与癌症的发生[59-60]。然而,与PVTI不同的是,circpvt1不与原癌基因c-myc直接作用,而是作为let-7b的海绵分子促进c-myc的表达。因此,circpvt1和PVT1可以不同的方式协同调节c-myc的表达来进一步调控癌症的发生。结直肠癌相关研究表明,hsa_circ_001988在癌组织中减少,与肿瘤细胞分化的程度及预后相关[61]。这些结果提示circRNA可作为疾病诊断的生物学新标志物,在药物开发和疾病诊治中具有良好的应用前景。
6 展望目前的研究表明,circRNA并非转录噪音,而是一类具有重要生物学功能的非编码RNA,可通过多种作用机制调控机体正常的生长发育以及疾病的发生发展。circRNA作为miRNA的海绵分子发挥生物学功能,可调控基因的表达以及作为疾病诊断和预后的生物标志物,提供新的潜在治疗靶点。circRNA编码蛋白并与蛋白质互作能力已成为现阶段的研究热点。人、小鼠、线虫等物种的circRNA信息已收录在circbase数据库中(表 2),并且在Circ2Traits中已建立了与疾病相关的circRNA的数据库,这些数据库丰富了人们对circRNA的分子结构和作用机制等方面的认知。目前,circRNA的发生机制和作用机制尚未完全明确,其在生命活动中发挥的作用以及分子机制都还需要进一步的探究。circRNA作为非编码RNA的研究热点之一,与编码RNA、miRNA、lncRNA比较,相关研究较少,对其进一步的深入研究可进一步完善人们对生物机体遗传调控网络的认知。
| [1] |
Diener T O. Potato spindle tuber "virus":Ⅳ. A replicating, low molecular weight RNA. Virology, 1971, 45(2): 411-428. DOI:10.1016/0042-6822(71)90342-4 |
| [2] |
Hsu M, Cocaprados M. Electron microscopic evidence for the circular form of RNA in the cytoplasm of eukaryotic cells. Nature, 1979, 280(5720): 339-340. DOI:10.1038/280339a0 |
| [3] |
Qiu P C, Gaudette M F, Robinson D H, et al. Expression of the mouse testis-determining gene Sry in male preimplantation embryos. Molecular Reproduction & Development, 1995, 40(2): 196-204. |
| [4] |
Salzman J, Gawad C, Wang P L, et al. Circular RNAs are the predominant transcript isoform from hundreds of human genes in diverse cell types. Plos One, 2012, 7(2): 1-12. |
| [5] |
Guo J U, Agarwal V, Guo H, et al. Expanded identification and characterization of mammalian circular RNAs. Genome Biology, 2014, 15(7): 409-423. DOI:10.1186/s13059-014-0409-z |
| [6] |
Cocquerelle C, Mascrez B, Hétuin D, et al. Mis-splicing yields circular RNA molecules. Faseb Journal Official Publication of the Federation of American Societies for Experimental Biology, 1993, 7(1): 155-160. |
| [7] |
Jeck W R, Sorrentino J A, Wang K, et al. Circular RNAs are abundant, conserved, and associated with ALU repeats. RNA, 2012, 19(2): 141-57. |
| [8] |
Kelly S, Greenman C, Cook P R, et al. Exon skipping is correlated with exon circularization. Journal of Molecular Biology, 2015, 427(15): 2414-2417. DOI:10.1016/j.jmb.2015.02.018 |
| [9] |
Zhang X O, Wang H B, Zhang Y, et al. Complementary sequence-mediated exon circularization. Cell, 2014, 159(1): 134-147. DOI:10.1016/j.cell.2014.09.001 |
| [10] |
Ivanov A, Memczak S, Wyler E, et al. Analysis of Intron Sequences Reveals Hallmarks of Circular RNA Biogenesis in Animals. Cell Reports, 2015, 10(2): 170-177. DOI:10.1016/j.celrep.2014.12.019 |
| [11] |
Suzuki H, Zuo Y, Wang J, et al. Characterization of RNase R-digested cellular RNA source that consists of lariat and circular RNAs from pre-mRNA splicing. Nucleic Acids Research, 2006, 34(8): 63-70. DOI:10.1093/nar/gkl151 |
| [12] |
Zhang Y, Zhang X O, Chen T, et al. Circular Intronic Long Noncoding RNAs. Molecular Cell, 2013, 51(6): 792-806. DOI:10.1016/j.molcel.2013.08.017 |
| [13] |
Salzman J, Chen RE, Olsen M N, et al. Cell-type specific features of circular RNA expression. Plos Genetics, 2013, 9(9): 1-15. |
| [14] |
Xia S, Feng J, Lei L, et al. Comprehensive characterization of tissue-specific circular RNAs in the human and mouse genomes. Briefings in Bioinformatics, 2016, 8: 1-9. |
| [15] |
Suzuki H, Tsukahara T. A view of pre-mRNA splicing from RNase R resistant RNAs. International Journal of Molecular Sciences, 2014, 15(6): 9331-9342. DOI:10.3390/ijms15069331 |
| [16] |
Bahn J H, Zhang Q, Li F, et al. The landscape of microRNA, Piwi-interacting RNA, and circular RNA in human saliva. Clinical Chemistry, 2015, 61(1): 221-230. DOI:10.1373/clinchem.2014.230433 |
| [17] |
Werfel S, Nothjunge S, Schwarzmayr T, et al. Characterization of circular RNAs in human, mouse and rat hearts. Journal of Molecular & Cellular Cardiology, 2016, 98: 103-107. |
| [18] |
Pan X, Xiong K. PredcircRNA:computational classification of circular RNA from other long non-coding RNA using hybrid features. Molecular Biosystems, 2015, 11(8): 2219-2226. DOI:10.1039/C5MB00214A |
| [19] |
Li Z, Huang C, Bao C, et al. Exon-intron circular RNAs regulate transcription in the nucleus. Nature Structural & Molecular Biology, 2015, 22(3): 256-264. |
| [20] |
Zheng Q, Bao C, Guo W, et al. Circular RNA profiling reveals an abundant circHIPK3 that regulates cell growth by sponging multiple miRNAs. Nature Communications, 2016, 7(11215): 1-13. |
| [21] |
Shi X, Sun M, Liu H, et al. Long non-coding RNAs:a new frontier in the study of human diseases. Cancer Letters, 2013, 339(2): 159-166. DOI:10.1016/j.canlet.2013.06.013 |
| [22] |
Hansen T B, Jensen T I, Clausen B H, et al. Natural RNA circles function as efficient microRNA sponges. Nature, 2013, 495(7441): 384-388. DOI:10.1038/nature11993 |
| [23] |
Li F, Zhang L, Li W, et al. Circular RNA ITCH has inhibitory effect on ESCC by suppressing the Wnt/β-catenin pathway. Oncotarget, 2015, 6(8): 6001-6013. DOI:10.18632/oncotarget.v6i8 |
| [24] |
Ashwal-Fluss R, Meyer M, Pamudurti N R, et al. circRNA biogenesis competes with pre-mRNA splicing. Molecular Cell, 2014, 56(1): 55-66. DOI:10.1016/j.molcel.2014.08.019 |
| [25] |
Zhang Y, Zhang X O, Chen T, et al. Circular intronic long noncoding RNAs. Molecular Cell, 2013, 51(6): 792-806. DOI:10.1016/j.molcel.2013.08.017 |
| [26] |
Jeck W R, Sharpless N E. Detecting and characterizing circular RNAs. Nature Biotechnology, 2014, 32(5): 453-461. DOI:10.1038/nbt.2890 |
| [27] |
Fabian M R, Sonenberg N. The mechanics of miRNA-mediated gene silencing:a look under the hood of miRISC. Nature Structural & Molecular Biology, 2012, 19(6): 586-593. |
| [28] |
Shi X, Sun M, Liu H, et al. Long non-coding RNAs:A new frontier in the study of human diseases. Cancer Letters, 2013, 339(2): 159-166. DOI:10.1016/j.canlet.2013.06.013 |
| [29] |
Salmena L, Poliseno L, Tay Y, et al. ceRNA hypothesis:The Rosetta Stone of a hidden RNA language?. Cell, 2011, 146(3): 353-358. DOI:10.1016/j.cell.2011.07.014 |
| [30] |
Jens M. Circular RNAs are a large class of animal RNAs with regulatory potency. Nature, 2013, 495(7441): 333-338. DOI:10.1038/nature11928 |
| [31] |
Chen J, Li Y, Zheng Q, et al. Circular RNA profile identifies circPVT1 as a proliferative factor and prognostic marker in gastric cancer. Cancer Letters, 2017, 388: 208-219. DOI:10.1016/j.canlet.2016.12.006 |
| [32] |
Wang K, Long B, Liu F, et al. A circular RNA protects the heart from pathological hypertrophy and heart failure by targeting miR-223. European Heart Journal, 2016, 37(33): 2602-2611. DOI:10.1093/eurheartj/ehv713 |
| [33] |
Qu S, Yang X, Li X, et al. Circular RNA:A new star of noncoding RNAs. Cancer Letters, 2015, 365(2): 141-148. DOI:10.1016/j.canlet.2015.06.003 |
| [34] |
Xu H, Guo S, Li W, et al. The circular RNA Cdr1as, via miR-7 and its targets, regulates insulin transcription and secretion in islet cells. Scientific Reports, 2015, 5(1): 12453-12465. DOI:10.1038/srep12453 |
| [35] |
Xue J, Liu Y, Luo F, et al. Circ100284, via miR-217 regulation of EZH2, is involved in the arsenite-accelerated cell cycle of human keratinocytes in carcinogenesis. Biochimica et Biophysica Acta, 2017, 1863(3): 753-763. DOI:10.1016/j.bbadis.2016.12.018 |
| [36] |
Peng L, Chen G, Zhu Z, et al. Circular RNA ZNF609 functions as a competitive endogenous RNA to regulate AKT3 expression by sponging miR-150-5p in Hirschsprung's disease. Oncotarget, 2017, 8(1): 808-818. |
| [37] |
Kelemen O, Convertini P, Zhang Z, et al. Function of alternative splicing. Gene, 2013, 514(1): 1-30. DOI:10.1016/j.gene.2012.07.083 |
| [38] |
Chen L L. The biogenesis and emerging roles of circular RNAs. Nature Reviews Molecular Cell Biology, 2016, 17(4): 205-211. DOI:10.1038/nrm.2015.32 |
| [39] |
Chao C W, Chan D C, Kuo A, et al. The mouse formin (Fmn) gene:abundant circular RNA transcripts and gene-targeted deletion analysis. Molecular Medicine, 1998, 4(9): 614-628. |
| [40] |
Abe N, Hiroshima M, Maruyama H, et al. Rolling circle amplification in a prokaryotic translation system using small circular RNA. Angewandte Chemie International Edition, 2013, 52(27): 7004-7008. DOI:10.1002/anie.201302044 |
| [41] |
Wutz A. Gene silencing in X-chromosome inactivation:advances in understanding facultative heterochromatin formation. Nature Reviews Genetics, 2011, 12(8): 542-553. DOI:10.1038/nrg3035 |
| [42] |
Názer E, Lei E P. Modulation of chromatin modifying complexes by noncoding RNAs in trans. Current Opinion in Genetics & Development, 2014, 25(1): 68-73. |
| [43] |
Zhang Y, Yang L, Chen L L. Life without a tail:New formats of long noncoding RNAs. International Journal of Biochemistry & Cell Biology, 2013, 54(1): 338-349. |
| [44] |
Chuan H, Ge S. What happens at or after transcription:Insights into circRNA biogenesis and function. Transcription, 2015, 6(4): 61-64. DOI:10.1080/21541264.2015.1071301 |
| [45] |
Chen L L, Yang L. Regulation of circRNA biogenesis. RNA Biology, 2015, 12(4): 381-388. DOI:10.1080/15476286.2015.1020271 |
| [46] |
Perry R P, Kelley D E. Inhibition of RNA synthesis by actinomycin D:characteristic dose-response of different RNA species. Journal of Cellular Physiology, 1970, 76(2): 127-139. DOI:10.1002/(ISSN)1097-4652 |
| [47] |
Baroni T E, Chittur S V, George A D, et al. Advances in RIP-chip analysis:RNA-binding protein immunoprecipitation-microarray profiling. Methods in Molecular Biology, 2008, 419(419): 93-108. |
| [48] |
Lukiw W. Circular RNA (circRNA) in Alzheimer's disease (AD). Frontiers in Genetics, 2013, 4(4): 307-308. |
| [49] |
Bachmayrheyda A, Reiner A T, Auer K, et al. Correlation of circular RNA abundance with proliferation-exemplified with colorectal and ovarian cancer, idiopathic lung fibrosis, and normal human tissues. Scientific Reports, 2015, 5(8057): 8057-8058. |
| [50] |
Molkentin J D, Lu J R, Antos C L, et al. A calcineurin-dependent transcriptional pathway for cardiac hypertrophy. Cell, 1998, 93(2): 215-228. DOI:10.1016/S0092-8674(00)81573-1 |
| [51] |
Everett A D, Tufro-Mcreddie A, Fisher A, et al. Angiotensin receptor regulates cardiac hypertrophy and transforming growth factor-beta 1 expression. Hypertension, 1994, 23(5): 587-592. DOI:10.1161/01.HYP.23.5.587 |
| [52] |
Molkentin J D, Nd D G. Cytoplasmic signaling pathways that regulate cardiac hypertrophy. Annual Review of Physiology, 2003, 63(1): 391-426. |
| [53] |
Zheng C, Niu H, Li M, et al. Cyclic RNA has-circ-000595 regulates apoptosis of aortic smooth muscle cells. Molecular Medicine Reports, 2015, 12(5): 6656-6662. DOI:10.3892/mmr.2015.4264 |
| [54] |
Burd C E, Jeck W R, Liu Y, et al. Expression of linear and novel circular forms of an INK4/ARF-associated non-coding RNA correlates with atherosclerosis risk. PLoS Genet, 2010, 6(12): 1-15. |
| [55] |
Zhao Z, Li X, Jian D, et al. Hsa_circ_0054633 in peripheral blood can be used as a diagnostic biomarker of pre-diabetes and type 2 diabetes mellitus. Acta Diabetologica, 2017, 54(3): 237-245. DOI:10.1007/s00592-016-0943-0 |
| [56] |
Qu S, Song W, Yang X, et al. Microarray expression profile of circular RNAs in human pancreatic ductal adenocarcinoma. Genomics Data, 2015, 5: 385-387. DOI:10.1016/j.gdata.2015.07.017 |
| [57] |
Yang W, Du W W, Li X, et al. Foxo3 activity promoted by non-coding effects of circular RNA and Foxo3 pseudogene in the inhibition of tumor growth and angiogenesis. Oncogene, 2015, 35(30): 3919-3931. |
| [58] |
Li P, Chen S, Chen H, et al. Using circular RNA as a novel type of biomarker in the screening of gastric cancer. Clinica Chimica Acta, 2015, 444: 132-136. DOI:10.1016/j.cca.2015.02.018 |
| [59] |
Tseng Y Y, Moriarity B S, Gong W, et al. PVT1 dependence in cancer with MYC copy-number increase. Nature, 2014, 512(7512): 82-86. |
| [60] |
Wang F, Yuan J H, Wang S B, et al. Oncofetal long noncoding RNA PVT1 promotes proliferation and stem cell-like property of hepatocellular carcinoma cells by stabilizing NOP2. Hepatology, 2014, 60(4): 1278-1290. DOI:10.1002/hep.27239 |
| [61] |
Wang X, Zhang Y, Huang L, et al. Decreased expression of hsa_circ_001988 in colorectal cancer and its clinical significances. International Journal of Clinical & Experimental Pathology, 2015, 8(12): 16020-16025. |
 2017, Vol. 37
2017, Vol. 37