文章信息
- 杨旭, 黄惟巍, 姚宇峰, 刘存宝, 孙文佳, 白红妹, 马雁冰.
- YANG Xu, HUANG Wei-wei, YAO Yu-feng, LIU Cun-bao, SUN Wen-jia, BAI Hong-mei, MA Yan-bing.
- 人乳头瘤病毒16型L1基因在毕赤酵母中表达的影响因素分析
- Impact Factors on the Expression of Recombinant Human Papillomavirus 16 L1 Protein in Pichia pastoris
- 中国生物工程杂志, 2017, 37(10): 1-7
- China Biotechnology, 2017, 37(10): 1-7
- http://dx.doi.org/DOI:10.13523/j.cb.20171001
-
文章历史
- 收稿日期: 2017-06-14
- 修回日期: 2017-06-27
2. 云南省重大传染病疫苗研发重点实验室 昆明 650118;
3. 云南省重大传染病疫苗工程技术研究中心 昆明 650118;
4. 云南省重大传染病疫苗研发国家地方联合工程研究中心 昆明 650118
甲醇营养型毕赤酵母被广泛应用于重组异源蛋白的表达[1]。作为单细胞真核微生物,它具有真核表达系统的诸多优势,如能实现外源蛋白的分泌表达,具有真核表达系统的翻译后修饰如糖基化和磷酸化等功能,能在基本培养基上快速生长,而且无潜在内毒素和病毒脱氧核糖核酸的污染[2-4]。此外,毕赤酵母能实现高密度生长和发酵,因而适合于大规模的工业化生产[3]。然而,并不是所有的外源基因都能在毕赤酵母成功表达,通常需要对表达进行优化从而提升外源基因在毕赤酵母中的表达水平[1]。在这些优化策略中,密码子优化是最常采用的方法之一。密码子优化的方法基于不同生物对各种密码子使用频率的差异对外源基因进行优化,高表达蛋白质中的高频率密码子相应的tRNA的丰度更高,这种对高频率密码子的偏爱使得蛋白的合成速率更高[5]。有研究报道通过密码子优化方法实现了人乳头瘤病毒主要衣壳蛋白L1的高效表达[6-8]。此外,通过基因重组方法增加基因剂量从而实现基因的高拷贝和通过蛋白酶缺陷菌株提高蛋白质的稳定性也是提高蛋白表达的常用方法[9]。在本研究采取了多策略的优化方法以期实现HPV16L1在毕赤酵母中的高效表达。
1 材料与方法 1.1 材料 1.1.1 质粒、菌株与宿主菌真核毕赤酵母表达系统PichiaPinkTM包括表达质粒pPink HC(高拷贝)、pPink LC(低拷贝)、表达用宿主菌strains 1~4购自美国Invitrogen公司, 根据酵母密码子偏好优化的HPV 16 L1全长基因序列M16、Y16由生工生物工程(上海)股份有限公司合成,并构建质粒PUC57;哺乳动物细胞密码子偏好优化的HPV 16 L1基因P16由质粒p16shell (美国国立癌症研究所Dr. Schiller提供)通过PCR扩增获得,携带野生型HPV16 L1基因的序列W16由本室(中国医学科学院医学生物学研究所分子免疫实验室)保存。E.coli DH5α购自宝生物工程(大连)有限公司。
1.1.2 试剂DNA marker、Taq多聚酶、T4 DNA连接酶、限制性内切酶购自大连TaKaRa公司;质粒提取试剂盒和DNA凝胶回收试剂盒购自北京天根生化科技有限公司;酵母提取物和胰蛋白胨均购自英国OXOID公司;鼠抗HPV 16 L1单克隆抗体及HRP标记兔抗鼠IgG购自美国Santa Cluz公司;显色底物Pierce ECL购自美国Thermo公司;其他试剂均为国产分析纯。酵母生长培养用甘油复合培养基BMGY和诱导用甲醇复合培养基BMMY由本实验室自行配制,培养基配方中均分别含1%的酵母提取物,2%蛋白胨,100mmol/L磷酸盐、1.34% YNB(酵母氮碱),4×10-4%生物素,BMGY中含1%甘油,BMMY中含0.5%甲醇用于蛋白的诱导。
1.2 方法 1.2.1 HPV16L1不同基因序列设计合成和重组质粒构建根据Genebank中登录的氨基酸序列并基于毕赤酵母密码子使用频率设计合成M16[UniProtKB/Swiss-Prot: P03101.2]和Y16[NC_001526.2]的基因序列,设计过程中避免稀有密码子(氨基酸同义密码子的使用频率 < 0.1)和构建过程中使用的内切酶位点,M16和Y16的区别在于M16的密码子分布较为均衡,选用较偏好的密码子,而Y16倾向于挑选同义密码子中使用频率最高的密码子,全基因序列由上海生物工程有限公司合成,同时合成引物序列,见表 1。以携带HPV 16L1不同序列基因的质粒DNA为模板,对HPV16 L1基因进行扩增。反应条件为:95 ℃ 30 s;54 ℃ 30 s,72 ℃ 90 s,共30个循环。PCR产物经1%琼脂糖凝胶电泳鉴定。表达载体pPink HC或LC经Bsa I/EcoR I/Sph I和Kpn I酶切后回收载体片段,和经同样双酶切处理的PCR扩增产物在T4DNA连接酶作用下16℃连接过夜,转化DH5α感受态细胞,经AMP+LB琼脂平板筛选阳性克隆并测序鉴定。
| 引物序列(5′~ 3′) | 酶切位点 | |
| W16HC/LC-F | AAGGTCTCGAATTCGAAACGATGTCTCTTTGGCTGCCTAGTGA | Bsa I |
| W16HC/LC -R | GGGGTACCTTACAGCTTACGTTTTTTGCGTTTA | Kpn I |
| P16HC/LC-F | CGGAATTCGAAACGATGAGCCTGTGGCTGC | EcoR I |
| P16HC/LC-R | GGGGTACCTCACAGCTTCCTCTTCTTCC | Kpn I |
| M16HC/LC-F | CGGAATTCGAAACGATGTCATTGTGGCTACCTTCT | EcoR I |
| M16HC/LC-R | GTGCATGCTTACAGTTTCCTCTGTTTACGTTT | Sph I |
| Y16HC/LC-F | CGGAATTCGAAACGATGTCTTTGTGGTTGCCATCT | EcoR I |
| Y16HC/LC-R | GGGGTACCTTACAACTTTCTCTTCTTTCTCTTAGCA | Kpn I |
1.2.2 HPV16L1重组质粒转化PichiaPinkTM毕赤酵母细胞和表达
不同序列特征HPV16L1基因(M16、Y16、P16和W16)构建pPink HC和LC表达载体,经测序和酶切鉴定正确的重组质粒电转化PichiaPinkTM不同菌株。酵母菌株接种于5ml YPD培养基过夜培养,然后将过夜培养物转接至含500mlYPD的2L三角烧瓶中,30℃280 r/min培养至菌体OD600=1.3~1.5,4℃ 1 500g离心5 min分离菌体,将菌体重悬于冰预冷的灭菌双蒸水中,按上述步骤离心菌体沉淀, 再将菌体重悬于冰预冷的灭菌双蒸水中, 同样条件离心后用1M冰预冷的山梨醇重悬菌体, 再次同样条件离心, 菌体用1.5ml冰预冷的山梨醇重悬,即为酵母感受态细胞。重组质粒在电转化前用Afl II内切酶进行线性化处理,取5~20μg线性化重组质粒,加入80μl上述制备好的酵母感受态细胞,将重组质粒和感受态混悬液加入预冷的2mm电转杯中,在冰上放置5min,将电转杯放入电转仪(GenePulser Xecll, Bio-Rad), 调整参数为电压1.5 kV/cm, 电容50 μF, 电阻129Ω,电击时间为±5 msec。静置1h后将内容物涂布ade-平板,于30℃培养4~7天进行阳性克隆筛选。随机挑选平板上的白色阳性克隆接种至25ml BMGY培养基中,于30℃ 280r/min培养至OD600= 3.0~3.5,1 500 g离心5min,将菌体沉淀重悬于25ml BMMY中,于30℃ 280 r/min诱导24h, 以未诱导的含表达载体的菌为阴性对照。
1.2.3 重组蛋白表达的Western blot分析诱导24h后收获菌体离心,样品经12 %的SDS-PAGE分离后,经Trans-blot®SD(Bio-Rad)恒流150mA 1h转移至0.45μm的PVDF膜上,以含5 %脱脂奶粉的TBST(20 mmol/L Tris·HCl, 0.05% Tween 20, pH 7.2)于室温封闭2 h;加入1:500用TBST稀释的鼠抗HPV16 L1单克隆抗体(sc-2005, Santa Cruz),室温孵育2h;TBST洗涤3次后,加入1:10 000用TBST稀释的HRP标记的兔抗鼠IgG,室温孵育1 h;TBST洗涤3次后,将洗涤后的PVDF膜用ECL(Pierce®ECL, Thermo, USA)显色底物浸染5min,X光底片曝光分析结果。
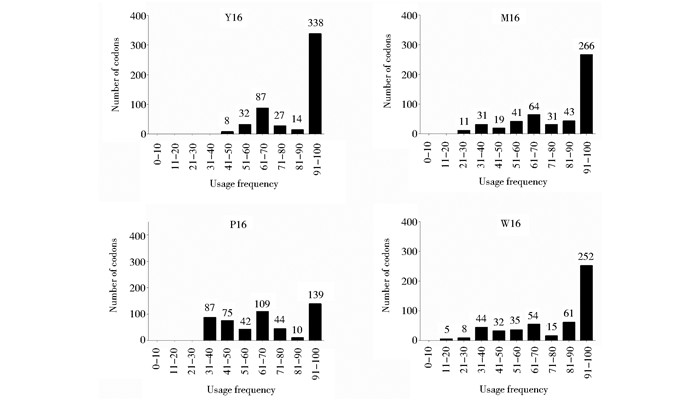
1.2.4 不同HPV16L1基因序列密码子分布情况和序列特征分析为了分析基因序列密码子不同分布情况和蛋白表达水平之间的关系,通过在线网站(http://www.genscript.com/cgi-bin/tools/codon_freq_table)引用毕赤酵母密码子的使用频率表,并通过(http://www.genscript.com/cgi-bin/tools/rare_codon_analysis).分析4个HPV16L1基因序列M16、Y16、P16、W16密码子在毕赤酵母中的使用分布情况,统计结果见图 1。通过(http://rna.tbi.univie.ac.at/)预测mRNA折叠自由能、密码子适应指数CAI(codon adaptation index)并进行GC含量分析,结果见表 2。
|
| 图 1 HPV16 L1不同基因序列在毕赤酵母中的密码子频率分布情况 Figure 1 Codon frequency distribution of different gene versions of HPV16 L1 for expression in P. pastoris |
| Y16 | M16 | P16 | W16 | |
| Folding free energy(Kcal/mol) | -451.50 | -409.40 | -606.50 | -384.70 |
| GC% | 47.826 | 43.853 | 64.097 | 38.011 |
| CAI | 0.87 | 0.76 | 0.58 | 0.73 |
2 结果与分析 2.1 HPV16L1重组质粒的PCR及酶切鉴定
构建后重组质粒进行PCR鉴定,产物经1%琼脂糖凝胶电泳,在分子量约1 500bp处得到与预期分子量大小相符的DNA条带。各重组质粒经双酶切鉴定,目的条带位于约1 500bp左右,与预期相符。
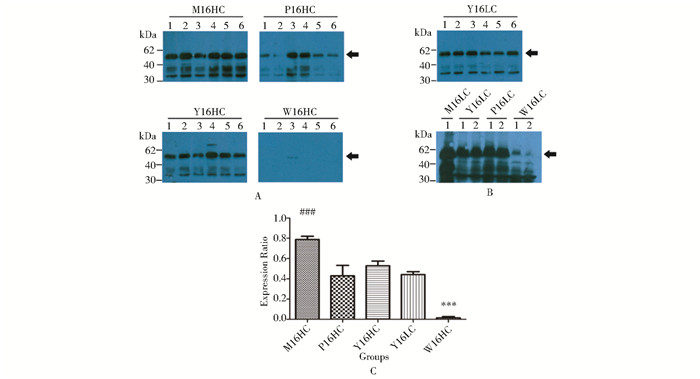
2.2 不同密码子优化HPV16L1基因序列在毕赤酵母中的表达 2.2.1 HPV16L1基因序列M16、Y16、P16、W16在pPink HC中的表达HPV16 L1基因序列M16、Y16、P16、W16分别构建表达载体pPink HC和LC,转化Strain1,蛋白诱导表达24h后收集样品进行Western blot检测,结果显示,毕赤酵母密码子偏好优化的基因序列M16和Y16以及哺乳动物细胞密码子优化序列P16都在Stain 1中获得了有效表达(图 2A和2B),目的蛋白分子量在55kDa左右,在30~50kDa之间也出现反应条带,可能为降解蛋白,因为在无目的基因的宿主菌对照中该分子质量之间并无条带出现。在诱导表达的6个单菌落中,W16只有一个达到可检测水平(图 2A)。通过对WB反应条带的半定量分析发现,在4个序列中,M16的表达水平最高,和P16相比, Y16的表达水平略高,而在P16诱导表达的6个单菌落中,其中2个的表达水平与M16和Y16表达水平相当,而W16的表达水平明显低于其余序列的表达水平(图 2C)。
|
| 图 2 不同HPV16L1基因序列在毕赤酵母中表达的WB结果分析 Figure 2 Immunoblotting analyses on expression of different gene versions of HPV16 L1 in P.pastoris A: Expression of the L1 protein introduced by plasmid pPink HC; B: Expression of the L1 protein introduced by plasmid pPink LC; C: Semi-quantitative analyses of immunoblotting results for the expression of HPV16 L1; Lanes 1~6: different colonies. ### : P < 0.001, compared to P16HC, Y16HC, W16HC and Y16LC; *** : P < 0.001, compared to M16HC, P16HC, Y16HC, and Y16LC |
在异源蛋白优化表达过程中,基因剂量是另一个需要考虑的因素,基因剂量的增加可能会增强mRNA的转录水平和丰度。pPink HC和pPinkLC表达载体通过不同启动子强度来控制选择基因ADE2的表达,pPink HC使用弱的启动子,因此只有多拷贝的质粒整合才会使转化质粒在腺嘌呤缺陷培养基中生长。而pPink LC则相反,其使用强的启动子,只需一个拷贝的基因整合即可使转化质粒在腺嘌呤缺陷的培养基中生长,因此,通过两种不同的质粒可以达到不同拷贝表达克隆的筛选。M16、Y16、P16、W16分别克隆于pPink HC和pPinkLC,诱导表达后经Western blot检测蛋白表达水平,结果显示,M16、Y16、P16、W16在pPink HC和pPinkLC中获得了相似的表达水平(图 2B和图 2C),Y16在pPink HC和pPinkLC中的表达无明显差异(图 2B和图 2C),所有优化基因序列在pPinkLC中获得了有效表达(图 2B),提示基因剂量未对HPV16 L1表达产生显著影响。
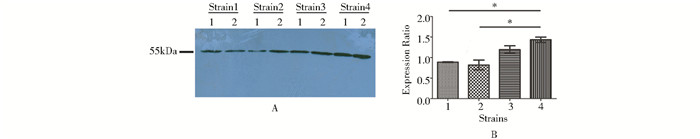
2.3 HPV16 L1基因序列Y16在不同蛋白酶缺陷株中的表达Y16HC重组质粒分别电转化pPink strain 1、2、3、4,strain 1为野生型ade2基因敲除的菌株,其余菌株均从Strain1改造而来。Strain2为pep 4敲除菌株,为蛋白酶A缺陷株,Strain3为prb1基因敲除株,导致其蛋白酶B合成缺陷,而Strain4为蛋白酶A和B的双敲除株,即prb1和pep 4双基因敲除。诱导表达24h后经Western blot和半定量分析,实验重复三次,结果均一致,代表性结果分别见图 3A和3B,结果显示,HPV16L1基因序列Y16在蛋白酶A和B双缺陷的strain 4中获得了最高表达。
|
| 图 3 HPV16 L1(Y16)在pPink不同蛋白酶缺陷株中的表达 Figure 3 Expression of HPV16(Y16)in different pPink strains A: Representative picture for immunoblotting and B: the semi-quantitative analyses for using host strains with different proteinase phenotypes. Lane 1 and 2 represented two different colonies. *: P < 0.05 |
4个来源不同的HPV16 L1基因序列Y16、M16、P16和W16的密码子频率分布统计结果如图 1,它们的最优密码子数分别为338、266、139和252, W16中含有5个频率低于20%的密码子,而在其他3个序列中这一频率的数字为0。序列的自由能预测、GC含量和密码子适应指数(codon adaptation index, CAI)见表 2。
3 讨论遗传密码子的简并性即同一氨基酸对应多个密码子的现象使得同一种蛋白可以由众多的同义基因序列所编码,但这些编码相同氨基酸的不同密码子,在不同物种中使用的频率并非完全地平均分布,绝大多数生物倾向于只利用这些密码子中的一部分,密码子优化方法正是基于其这样的偏爱性。尽管密码子优化策略并不一定总是成功[10-11],但它依然是最为广泛使用的提高外源基因表达水平的方法。本研究通过将4个HPV16L1基因序列构建pPinkHC和LC载体,转化strain 1以考察HPV16 L1蛋白表达水平,结果(图 2A)显示,和其他基因序列相比,M16显示了最高的表达水平,Y16显示了比P16稍高的表达水平,P16中有两个单克隆达到了和M16相类似的表达水平,而W16的表达水平显著低于其他基因序列的表达水平。为了探讨密码子使用频率分布和表达水平之间的关系,对4个HPV16L1基因序列的密码子分布频率进行了统计(图 1),结果显示,表达最差的W16其密码子分布频率和M16及Y 16相比,除了有5个频率小于20%的密码子外,其余密码子和M16和Y16相似。M16和Y16相比,其密码子分布更为均衡,M16的密码子之中,有42个密码子的使用频率在40%以下,而这一数字在Y16为0;M16的最优密码子数是266,而Y16为338,P16为哺乳动物细胞密码子优化基因序列,与M16和Y16相比,其拥有最少的最优密码子,W16的最优密码子数目为252个,而P16的最优密码子数目为139个。Y16拥有最多的最优密码子数目,应该获得最高的理论蛋白表达量,但实验结果显示M16拥有最高的蛋白表达水平,M16与Y16相比,其拥有较少的最优密码子数目, 但其密码子分布更为均衡;P16为哺乳动物细胞密码子优化的基因序列,在四个基因序列中,其拥有最少的最优密码子数目,但其在毕赤酵母中也获得明显高于W16的表达水平。结果显示,在不考虑mRNA结构的前提下,优化序列的过程中并没有必要使用那些频率最优的密码子,而避免使用那些极端的稀有密码子可能对提高蛋白的表达水平更为有利。
密码子适应指数(the codon adaptation bias, CAI)是用于对优化的基因序列进行分析的常用指标之一[12],它用以指示异源基因与目标宿主基因之间最优密码子的接近程度,数值1被认为是最佳的理论值。当一个外源基因的密码子与目标宿主偏爱密码子越接近,即CAI越接近理论值1时,蛋白翻译效率越高,将获得更高的表达水平。在研究中使用的4个HPV16L1基因序列中,拥有最佳CAI指数的基因序列其表达并不是最优的,而CAI指数较低的P16在毕赤酵母却获得了有效表达,提示密码子偏好只是影响表达的一个因素,不能简单地将表达效率高低归结于使用的密码子频率高低,其间因密码子优化而改变的mRNA二级结构可能起着重要的影响。然而以mRNA二级结构预测指导基因表达优化具有难度,缺乏可靠的理论指导。自由能的预测是对mRNA二级结构进行分析时常用的指标,反映了mRNA稳定性特征。有文献报道通过自由能预测基因序列的mRNA二级结构从而增加目的蛋白的表达量[13-15],我们对4个基因序列的自由能进行了预测,结果显示,自由能预测的理论数值与实际的蛋白表达水平并不相符,因此不能从简单的自由能高低获得表达效率高低的预判,这期间可能得考虑mRNA二级结构的序列分布,蛋白因子的结合等等复杂因素。基因剂量也被认为是影响蛋白表达的关键因素,通过基因重组方法在选择压力下挑选高拷贝的基因重组子,提高mRNA的转录水平,有助于提高蛋白的表达量[9]。本研究采用商业化的pPinkTM表达系统简化重组菌的筛选,pPinkHC和pPinkLC表达载体通过启动子的强弱来控制选择基因ADE2(Adenine auxotrophy)的表达,pPinkHC使用弱的启动子,因此只有多拷贝的质粒整合才会使转化质粒在腺嘌呤缺陷培养基中生长,而pPink LC则相反。结果显示,不同HPV16L1基因序列均在pPinkLC中获得了表达,Y16在pPinkHC和pPinkLC中的表达无明显差异。鉴于pPinkHC理论上保证了高拷贝数的插入,因此推论,基因拷贝数对HPV16L1的表达并未产生严重影响。
本研究尝试了不同策略提高HPV16L1在毕赤酵母中的表达,包括密码子使用情况,基因拷贝数,蛋白质在宿主中表达时的稳定性。研究结果显示,密码子优化显著提高了HPV16L1在毕赤酵母中的表达,蛋白酶缺陷株提高了蛋白的稳定性,从而提高了蛋白的表达。值得注意的是,哺乳动物细胞密码子优化的基因序列P16在毕赤酵母中获得了比野生型W16更高的表达,即使它拥有比W16更少的最优密码子和更多的稀有密码子,提示除了密码子因素外,还需要考虑其他的因素,比如mRNA二级结构的改变以及由此导致的对蛋白质功能的影响等,这为后续的研究提供了参考。综上所述,本研究为外源蛋白在毕赤酵母中的优化表达提供了有益的参考。
| [1] |
Ahmad M, Hirz M, Pichler H, et al. Protein expression in Pichia pastoris:recent achievements and perspectives for heterologous protein production. Appl Microbiol Biotechnol, 2014, 98(12): 5301-5317. DOI:10.1007/s00253-014-5732-5 |
| [2] |
Ciarkowska A, Jakubowska A. Pichia pastoris as an expression system for recombinant protein production. Postepy Biochem, 2013, 59(3): 315-321. |
| [3] |
Damasceno L M, Huang C J, Batt C A., et al. Protein secretion in Pichia pastoris and advances in protein production. Appl Microbiol Biotechnol, 2012, 93(1): 31-39. DOI:10.1007/s00253-011-3654-z |
| [4] |
Macauley-Patrick S, Fazenda M L, Mcneil B, et al. Heterologous protein production using the Pichia pastoris expression system. Yeast, 2005, 22(4): 249-270. DOI:10.1002/(ISSN)1097-0061 |
| [5] |
Lanza A M, Curran K A, Rey l G, et al. A condition-specific codon optimization approach for improved heterologous gene expression in Saccharomyces cerevisiae. BMC Syst Biol, 2014, 8: 33. DOI:10.1186/1752-0509-8-33 |
| [6] |
Norkiene M, Gedvilaite A. Influence of codon bias on heterologous production of human papillomavirus type 16 major structural protein L1 in yeast. Scientific World Journal, 2012, 2012: 979218. |
| [7] |
Hanumantha R N, Baji B P, Rajendra L, et al. Expression of codon optimized major capsid protein (L1) of human papillomavirus type 16 and 18 in Pichia pastoris; purification and characterization of the virus-like particles. Vaccine, 2011, 29(43): 7326-7334. DOI:10.1016/j.vaccine.2011.07.071 |
| [8] |
Kim H J, Kwag H L. Codon optimization of the human papillomavirus type 58 L1 gene enhances the expression of soluble L1 protein in Saccharomyces cerevisiae. Biotechnol Lett, 2013, 35(3): 413-421. DOI:10.1007/s10529-012-1097-y |
| [9] |
Shen Q, Wu M, Wang H B, et al. The effect of gene copy number and co-expression of chaperone on production of albumin fusion proteins in Pichia pastoris. Appl Microbiol Biotechnol, 2012, 96(3): 763-772. DOI:10.1007/s00253-012-4337-0 |
| [10] |
Curran K A, Leavitt J M, Karim A S, et al. Metabolic engineering of muconic acid production in Saccharomyces cerevisiae. Metab Eng, 2013, 15: 55-66. DOI:10.1016/j.ymben.2012.10.003 |
| [11] |
Agashe D, Martinez-gomez N C, Drummond D A, et al. Good codons, bad transcript:large reductions in gene expression and fitness arising from synonymous mutations in a key enzyme. Mol Biol Evol, 2013, 30(3): 549-560. DOI:10.1093/molbev/mss273 |
| [12] |
Jansen R, Bussemaker H J, Gerstein M. Revisiting the codon adaptation index from a whole-genome perspective:analyzing the relationship between gene expression and codon occurrence in yeast using a variety of models. Nucleic Acids Res, 2003, 31(8): 2242-2251. DOI:10.1093/nar/gkg306 |
| [13] |
Kim H J, Lee S J. Optimizing the secondary structure of human papillomavirus type 16 L1 mRNA enhances L1 protein expression in Saccharomyces cerevisiae. J Biotechnol, 2010, 150(1): 31-36. DOI:10.1016/j.jbiotec.2010.07.032 |
| [14] |
Bartoszewski R A, Jablonsky M, Bartoszewska S, et al. A synonymous single nucleotide polymorphism in DeltaF508 CFTR alters the secondary structure of the mRNA and the expression of the mutant protein. J Biol Chem, 2010, 285(37): 28741-28748. DOI:10.1074/jbc.M110.154575 |
| [15] |
Ilyinskii P O, Schmidt T, Lukashev D, et al. Importance of mRNA secondary structural elements for the expression of influenza virus genes. OMICS, 2009, 13(5): 421-430. DOI:10.1089/omi.2009.0036 |
 2017, Vol. 37
2017, Vol. 37




