文章信息
- 梅雪昂, 陈艳, 王瑞钊, 肖文海, 王颖, 李霞, 元英进.
- MEI Xue-ang, CHEN Yan, WANG Rui-zhao, XIAO Wen-hai, WANG Ying, LI Xia, YUAN Ying-jin.
- 产玉米黄质的人工酵母细胞的构建
- Engineered Yeast Cell for Producing Zeaxanthin
- 中国生物工程杂志, 2016, 36(8): 64-72
- CHINA BIOTECHNOLOGY, 2016, 36(8): 64-72
- http://dx.doi.org/DOI:10.13523/j.cb.20160809
-
文章历史
- 收稿日期: 2016-01-13
- 修回日期: 2016-03-03
2. 天津化学化工协同创新中心合成生物学平台 天津 300072
2. SynBio Research Platform, Collaborative Innovation Center of Chemical Science and Engineering, School of Chemical Engineering and Technology, Tianjin 300072, China
利用合成生物学的方法构建人工细胞从而获得结构复杂、性质独特、来源稀缺及价值高昂的天然化合物越来越受到研究者的瞩目,其中通过对关键外源基因来源的筛选、竞争路径的敲除及关键外源基因表达强度的调控等已经成为提高目的产物产量的重要手段。首先,不同来源的同功酶在底物选择性、催化活性及与底盘的适配性上都会存在差异,因此筛选与底盘最适配的基因来源对于目的产物的合成有着重要的意义。Ding等[1]通过在酿酒酵母中筛选6种不同来源的紫杉二烯生物合成路径中关键基因香叶基二磷酸合酶,得出东北红豆杉来源的香叶基二磷酸合酶使得紫杉二烯的最高产量为72.8 mg/L。Du等[2]通过在解脂酵母中筛选3种不同来源的麦角甾醇生物合成路径中关键基因7-脱氢胆固醇还原酶,得出蟾蜍来源的C-22还原酶使得菜油甾醇的最高产量为143 mg/L。
其次,在天然化合物的生物合成路径中,都存在许多的分支竞争路径,通过对竞争路径基因的敲除可以达到提高目标产物产量的目的。乙酰辅酶A作为大量天然化合物合成的重要前体,其在酿酒酵母中有限的供给导致了相应产物的低产量,Lian等[3]通过敲除乙酰辅酶A生成中的竞争路径,使得目标产物正丁醇的产量提升了4倍。再次,构建人工细胞生产天然化合物时,引入的外源基因的表达强度势必会影响目标产物的产量,因此对外源基因表达强度的精确调控显得尤为重要。Gregory Stephanopoulos的研究小组在大肠杆菌中合成紫杉二烯时,通过对启动子的强弱和质粒的拷贝数进行组合设计后,得到了最优产量的菌株,其紫杉二烯产量高达1020±80 mg/L [4]。
玉米黄质是自然界中常见的一种橘红色脂溶性萜烯化合物,在抑制肿瘤细胞生长、预防心血管疾病、增强免疫功能、防止白内障等方面发挥着重要的作用[5-7]。然而,人体并不能自身合成玉米黄质,必须通过食物补充获得。目前玉米黄质的主要来源为植物提取、化学合成和微生物合成。由于植物提取高昂的生产方法而造成较低的收益率,而化学合成的玉米黄质虽然可以用于商业化生产,但由于其较低的品质只能作为颜料而不能用作食品添加剂或药物,因此微生物合成以其低成本、高产量、产品安全性和绿色环保的优越性被视为最有前途的生产方法。在微生物中异源合成玉米黄质的研究主要集中在大肠杆菌和恶臭假单胞菌。Li等[8]通过在一株产β-胡萝卜素的大肠杆菌中整合欧文氏菌来源的羟化酶,得到一株玉米黄质摇瓶产量为43.46 mg/L的大肠杆菌。Beuttler等[9]通过在假单胞菌中导入玉米黄质生物合成路径的质粒,经过发酵条件的优化,玉米黄质的摇瓶产量为51 mg/L。
酿酒酵母是公认的安全模式微生物工程菌株,而大肠杆菌和恶臭假单胞菌却是非食品安全菌,大肠杆菌本身含有内毒素和有毒蛋白,恶臭假单胞菌则是人类少见的条件致病菌。酿酒酵母的遗传背景清楚、基因操作简单、可进行大规模发酵,拥有比原核细胞更强大的蛋白表达系统和翻译后修饰系统,更适合于合成结构复杂的化合物。然而,截止目前利用酿酒酵母合成玉米黄质的报道却很少,仅有Sun等[10]在2012年通过整合木糖利用途径及玉米黄质的代谢路径(图 1),得到一株可以直接利用木糖生产0.74±0.02 mg/L玉米黄质的菌株。同年,Liang等[11]通过将组成型启动子换成诱导型启动子,虽然玉米黄质的产量相对于组成型启动子提高了50倍,最终产量为37 mg/L。但是与玉米黄质在大肠杆菌中的产量相比,玉米黄质在酿酒酵母中的产量仍相对较低。因此,亟待开发高产玉米黄质的重组酿酒酵母。
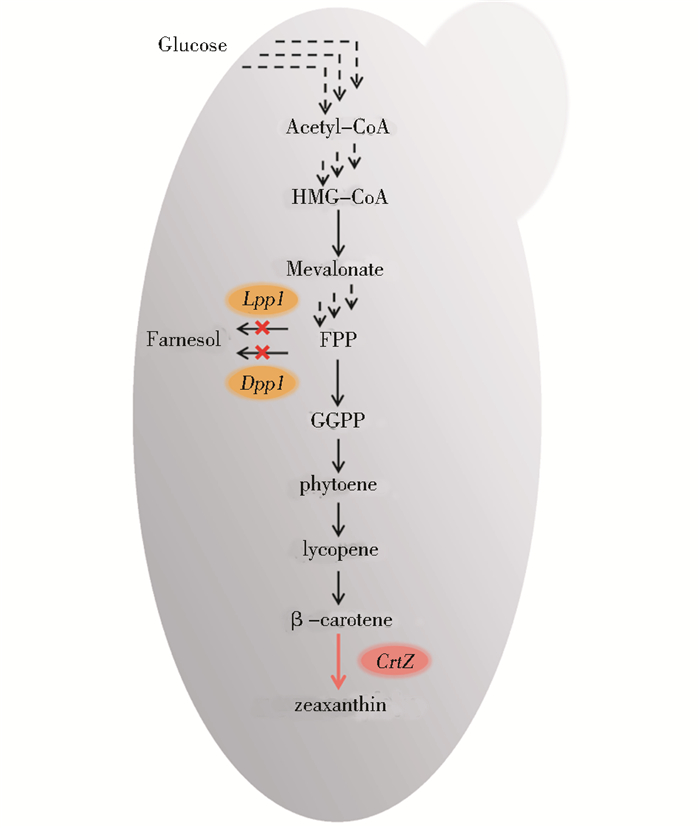
|
| 图 1 工程酿酒酵母中玉米黄质的生物合成路径 Figure 1 Biosynthesis pathway of zeaxanthin in engineered S.cerevisiae |
本研究拟在一株高产β-胡萝卜素的酿酒酵母基础上,构建产玉米黄质的人工酵母。并通过筛选关键酶的基因来源、提高内源FPP的供应(敲除竞争路径)以及对关键酶表达强度的调控,提高玉米黄质的产量,为在微生物中异源表达天然产物提供参考依据。
1 材料与方法 1.1 工具酶及试剂T4 DNA连接酶和限制性内切酶XbaI、SmaI、PmeI购自Fermentas公司;Q5超保真DNA聚合酶和限制性内切酶NotI-HF、BsaI-HF、BsmBI购自NEB公司;质粒小提试剂盒、普通DNA产物纯化试剂盒、琼脂糖凝胶DNA回收试剂盒购自天根公司;玉米黄质标准品(CAS号:144-68-3)购自上海源叶生物科技有限公司;PEG3350、鲑鱼精DNA、各类氨基酸粉末购自鼎国昌盛公司;醋酸锂购自北方天医化学试剂厂;去氨基酸酵母氮源购自GENVIEW公司。
1.2 菌株、质粒和培养基本文涉及的所有质粒和菌株信息详见表 1、表 2。宿主菌E. coli DH5α购自Biomed公司,产β-胡萝卜素的酿酒酵母菌株CY01为本实验室保存。pJET1.2购自赛默飞公司,pRS415K为本实验室保存。
| Plasmid | Description | Source |
| pRS415K | Single copy plasmid in S.cerevisiae with LEU2 and Kan marker | This laboratory |
| pRS425K | Multiple copies plasmid in S.cerevisiae with LEU2 and Kan marker | This laboratory |
| LDL06 | pRS425K harboring cassette Delta1-DRURADR-T CYC1 | This laboratory |
| mxa1 | pJET1.2 harboring cassette ho leftarm-DRURADR-ho rightarm | This study |
| mxa1-GAL1 | pJET1.2 harboring cassette ho leftarm-P GAL1-T HIS5-DRURADR-ho rightarm | This study |
| mxa2 | pJET1.2 harboring cassette ty4 leftarm-DRURADR-ty4 rightarm | This study |
| mxa2-GAL1 | pJET1.2 harboring cassette ty4 leftarm-P GAL1-T HIS5-DRURADR-ty4 rightarm | This study |
| mxa2-GAL3 | pJET1.2 harboring cassette ty4 leftarm-P GAL3-T HIS5-DRURADR-ty4 rightarm | This study |
| mxa2-GAL7 | pJET1.2 harboring cassette ty4 leftarm-P GAL7-T HIS5-DRURADR-ty4 rightarm | This study |
| mxa3 | pJET1.2 harboring cassette YPL062W leftarm-DRURADR-YPL062W rightarm | This study |
| mxa3-GAL1 | pJET1.2 harboring cassette YPL062W leftarm-P GAL1-T HIS5-DRURADR-YPL062W rightarm | This study |
| mxa1-GAL1-Aa | pJET1.2 harboring cassette ho leftarm-P GAL1-Aa crtZ-T HIS5-DRURADR-ho rightarm | This study |
| mxa1-GAL1-As | pJET1.2 harboring cassette ho leftarm-P GAL1-As crtZ-T HIS5-DRURADR-ho rightarm | This study |
| mxa1-GAL1-Eu | pJET1.2 harboring cassette ho leftarm-P GAL1-Eu crtZ-T HIS5-DRURADR-ho rightarm | This study |
| mxa1-GAL1-Pa | pJET1.2 harboring cassette ho leftarm-P GAL1-Pa crtZ-T HIS5-DRURADR-ho rightarm | This study |
| mxa1-GAL1-Ps | pJET1.2 harboring cassette ho leftarm-P GAL1-Ps crtZ-T HIS5-DRURADR-ho rightarm | This study |
| mxa1-GAL1-Ss | pJET1.2 harboring cassette ho leftarm-P GAL1-Ss crtZ-T HIS5-DRURADR-ho rightarm | This study |
| mxa1-GAL1-B.SD | pJET1.2 harboring cassette ho leftarm-P GAL1-B.SD212 crtZ-T HIS5-DRURADR-ho rightarm | This study |
| mxa1-GAL1-B.DC | pJET1.2 harboring cassette ho leftarm-P GAL1-B.DC263 crtZ-T HIS5-DRURADR-ho rightarm | This study |
| mxa1-GAL1-Hp | pJET1.2 harboring cassette ho leftarm-P GAL1-Hp crtZ-T HIS5-DRURADR-ho rightarm | This study |
| mxa2-GAL1-Eu | pJET1.2 harboring cassette ty4 leftarm-P GAL1-Eu crtZ-T HIS5-DRURADR-ty4 rightarm | This study |
| mxa2-GAL3-Eu | pJET1.2 harboring cassette ty4 leftarm-P GAL3-Eu crtZ-T HIS5-DRURADR-ty4 rightarm | This study |
| mxa2-GAL7-Eu | pJET1.2 harboring cassette ty4 leftarm-P GAL7-Eu crtZ-T HIS5-DRURADR-ty4 rightarm | This study |
| mxa30-GAL1-Eu | pJET1.2 harboring cassette YPL062W leftarm-P GAL1-Eu CrtZ-T HIS5-DRURADR-YPL062W rightarm | This study |
| mxa-LPP1 | pRS415K harboring cassette △Lpp1∷DRURADR | This study |
| mxa-DPP1 | pRS415K harboring cassette △Dpp1∷DRURADR | This study |
| Strain | Description | Source |
| CY01 | β-carotene producing strain | This laboratory |
| SyBE_Sc0123Z001 | CY01, △ho∷PGAL1-Aa CrtZ-THIS5-DRURADR | This study |
| SyBE_Sc0123Z002 | CY01, △ho∷ PGAL1-As CrtZ-THIS5-DRURADR | This study |
| SyBE_Sc0123Z003 | CY01, △ho∷ PGAL1-Eu CrtZ-THIS5-DRURADR | This study |
| SyBE_Sc0123Z004 | CY01, △ho∷ PGAL1-Pa CrtZ-THIS5-DRURADR | This study |
| SyBE_Sc0123Z005 | CY01, △ho∷ PGAL1-Ps CrtZ-THIS5-DRURADR | This study |
| SyBE_Sc0123Z006 | CY01, △ho∷ PGAL1-Ss CrtZ-THIS5-DRURADR | This study |
| SyBE_Sc0123Z007 | CY01, △ho∷ PGAL1-B.SD212 CrtZ-THIS5-DRURADR | This study |
| SyBE_Sc0123Z008 | CY01, △ho∷ PGAL1-B.DC263 CrtZ-THIS5-DRURADR | This study |
| SyBE_Sc0123Z009 | CY01, △ho∷ PGAL1-Hp CrtZ-THIS5-DRURADR | This study |
| SyBE_Sc0123Z010 | CY01, △ho∷ PGAL1-Eu CrtZ-THIS5 | This study |
| SyBE_Sc0123Z011 | SyBE_Sc0123Z010, △Lpp1∷DRURADR | This study |
| SyBE_Sc0123Z012 | SyBE_Sc0123Z010, △Dpp1∷DRURADR | This study |
| SyBE_Sc0123Z013 | SyBE_Sc0123Z010, △Lpp1 | This study |
| SyBE_Sc0123Z014 | SyBE_Sc0123Z010, △Lpp1, △Dpp1∷DRURADR | This study |
| SyBE_Sc0123Z015 | SyBE_Sc0123Z010, △Lpp1, △Dpp1 | This study |
| SyBE_Sc0123Z016 | SyBE_Sc0123Z015, △ty4∷ PGAL1-Eu CrtZ-THIS5-DRURADR | This study |
| SyBE_Sc0123Z017 | SyBE_Sc0123Z015, △ty4∷ PGAL3-Eu CrtZ-THIS5-DRURADR | This study |
| SyBE_Sc0123Z018 | SyBE_Sc0123Z015, △ty4∷ PGAL7-Eu CrtZ-THIS5-DRURADR | This study |
| SyBE_Sc0123Z019 | SyBE_Sc0123Z015, △ty4∷ PGAL1-Eu CrtZ-THIS5 | This study |
| SyBE_Sc0123Z020 | SyBE_Sc0123Z015, △ty4∷ PGAL1-Eu CrtZ-THIS5, YPL062W∷ PGAL1-Eu CrtZ-THIS5-DRURADR | This study |
LB培养基(10 g/L氯化钠;10 g/L胰蛋白胨;5 g/L酵母提取物;固体培养基添加15 g/L琼脂粉)用于大肠杆菌培养;YPD培养基(40 g/L葡萄糖;20 g/L胰蛋白胨;10 g/L酵母提取物;固体培养基添加20 g/L琼脂粉)用于酿酒酵母培养;YPDG培养基(40 g/L葡萄糖;20 g/L胰蛋白胨;10 g/L酵母提取物;10 g/L D-半乳糖;)用于酿酒酵母发酵;SD-TRP-LEU-HIS-URA固体培养基(20 g/L葡萄糖;6.7g/L去氨基酸酵母氮源;2g/L缺色氨酸、亮氨酸、组氨酸和尿嘧啶的混合氨基酸粉末;20 g/L琼脂粉)用于酵母转化子筛选和培养;5-氟乳清酸(5-FOA)固体培养基(20 g/L葡萄糖;6.7 g/L去氨基酸酵母氮源;2 g/L混合氨基酸粉末;1 g/L 5-氟乳清酸;20 g/L琼脂粉)用于URA营养标签的回收重复利用。
1.3 整合质粒载体和基因表达盒的构建基因表达盒的构建过程如图 2a、图 2b所示。设计特异性引物(表 3),以酿酒酵母CY01的基因组为模板扩增得到ho左右同源臂、GAL1启动子、HIS5终止子。以质粒LDL06为模板扩增得到DRURADR。将GAL1启动子、HIS5终止子通过OE-PCR方法拼接起来,得到两端包含XbaI和SmaI酶切位点,且在GAL1启动子和HIS5终止子之间包含两个BsmBI酶切位点的片段PGAL1-THIS5。同时将ho左同源臂、DRURADR、ho右同源臂,通过OE-PCR的方法顺次拼接,得到两端包含PmeI酶切位点、且在ho左同源臂与DRURADR、ho右同源臂之间包含XbaI和SmaI酶切位点的片段。然后与平末端载体pJET1.2连接得到ho整合质粒mxa1。之后将片段PGAL1-THIS5通过XbaI和SmaI酶切位点进行连接,得到整合质粒mxa1-GAL1。采用相同方法构建获得整合质粒mxa2-GAL1、mxa2-GAL3、mxa2-GAL7、mxa3-GAL1。
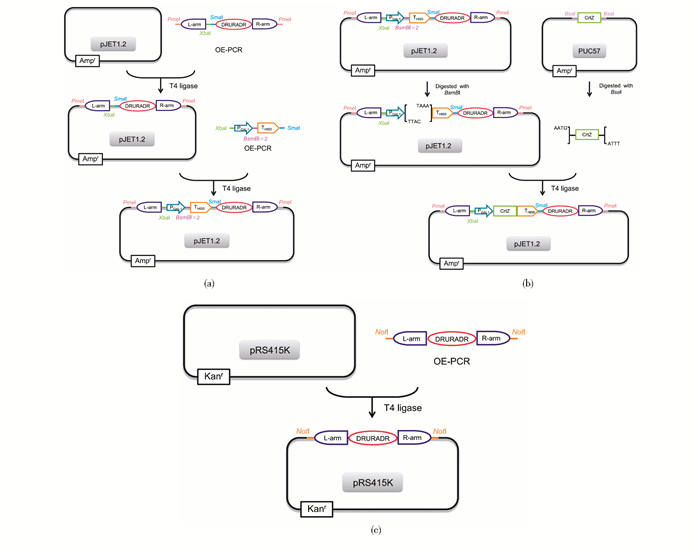
|
| 图 2 基因表达盒和敲除盒的构建 Figure 2 Construction of gene expression and deletion cassette (a) Construction of the expression vector (b) Gene insertion into the expression vector (c) Construction of the gene deletion cassette |
| Primers | Sequence(5′-3′) |
| DRURADR-F | TTCGGCTTCATGGCAATTC |
| DRURADR-R | GGTAACGCCAGGGTTTTCC |
| ho-left-F | GTTTAAACAGAATGAGTGGTATTAAAGAAATAAGG |
| ho-left-R | GAATTGCCATGAAGCCGAATCTAGACGAAATGAACATAAATATACGGTG |
| ho-right-F | GGGAAAACCCTGGCGTTACCTGGGTGTCACATTAAATATATCG |
| ho-right-R | GTTTAAACATTACTGATACCATTTTTTGGTATTAA |
| ty4-left-F | GTTTAAACAAACAAACAAAGACGTATAGTCCA |
| ty4-left-R | GAATTGCCATGAAGCCGAATCTAGAGAGCGGTATGTATTCATGCAC |
| ty4-right-F | GGGAAAACCCTGGCGTTACCAGCGTGTGTTTTATACTTCTCTTATATAG |
| ty4-right-R | GTTTAAACAACATTATATAGAAATATGGGCTTCC |
| YPL062W-left-F | GTTTAAACGAATGGATGATTGTTAGACTAGAAAG |
| YPL062W-left-R | GAATTGCCATGAAGCCGAATCTAGAGAAGTTCACAGAGACCGTGC |
| YPL062W-right-F | GGGAAAACCCTGGCGTTACCACGTCTTTCCTTTTGAACATCTA |
| YPL062W-right-R | GTTTAAACGCCAAAGTACTCTTATAAGAAAAAAAG |
| GAL1p-F | TCTAGAAGTACGGATTAGAAGCCGC |
| GAL1p-R | TTTAGGAGACGCGTCTCCCATTTATAGTTTTTTCTCCTTGACGTTAA |
| HIS5t-F | AATGGGAGACGCGTCTCCTAAAATAGATTAATTTAAACAGTATATGTACAGT |
| HIS5t-R | CCCGGGAAATTCATCCTCTATCATAGAAACAGG |
| GAL3p-F | TCTAGATTGCTAGCCTTTTCTCGGT |
| GAL3p-R | TTTAGGAGACGCGTCTCCCATTACTATGTGTTGCCCTACCTTTT |
| GAL7p-F | TCTAGATTTGCCAGCTTACTATCCTTCT |
| GAL7p-R | TTTAGGAGACGCGTCTCCCATTTTTTGAGGGAATATTCAACTGTT |
| Lpp1-left-F | ATAAGAATGCGGCCGCTAAAAAACAAAAAATAATGGAATCAAC |
| Lpp1-left-R | GAATTGCCATGAAGCCGAACCTTGGTAGAATATGACGAGTTTC |
| Lpp1-right-F | GGGAAAACCCTGGCGTTACCTAACACTTACAGAGTCCTATCAGGAAAG |
| Lpp1-right-R | ATAAGAATGCGGCCGCTCGGTGGAAAAACGCCA |
| Dpp1-left-F | ATAAGAATGCGGCCGCTATATATAGATAGAAACCCAACGTTGG |
| Dpp1-left-R | GAATTGCCATGAAGCCGAATTTGGTCGTTTGCTATGATTTAATTC |
| Dpp1-right-F | GGGAAAACCCTGGCGTTACCGAATAAAAAAGAATATATACTCCACATGAC |
| Dpp1-right-R | ATAAGAATGCGGCCGCTCTAGGCTGTTTATAGATTGATTACACC |
β-胡萝卜素羟化酶基因均由金斯瑞公司依照酿酒酵母密码子偏好性对序列进行优化并适当规避常用限制性酶切位点,且在基因两端添加5′端gcggccgcggtctcca;3′端taaaggagaccgcggccgc后合成。将9种不同来源的β-胡萝卜素羟化酶基因质粒用BsaI酶切位点切割,获得相应基因片段;将整合质粒用BsmBI酶切位点切割,纯化回收后经T4连接酶连接。得到相应的基因表达盒。
1.4 基因敲除盒的构建基因敲除盒的构建过程如图 2c所示。以酵母SyBE_Sc0123Z003基因组为模板,设计上、下游引物(表 3)PCR扩增Lpp1上、下游300bp同源臂,以质粒LDL01为模板,设计上、下游引物PCR扩增带DRURADR营养标签片段,之后将Lpp1上游片段、DRURADR、Lpp1下游片段通过OE-PCR连接起来,得到两端包含NotI酶切位点的片段,然后与pRS415K通过NotI酶切位点连接,得到△Lpp1∷DRURADR敲除盒。△Dpp1∷DRURADR敲除盒采用相同方法构建。
1.5 酵母转化、基因敲除和筛选将9个β-胡萝卜素羟化酶的基因表达盒质粒用PmeI酶切位点切割,获得相应的整合片段,采用醋酸锂法[12]分别单独转化酿酒酵母β-胡萝卜素生产菌株CY01,通过ho左、右同源序列与酵母基因组上ho位点发生重组而整合到基因组上。转化后采用SD-TRP-LEU-HIS-URA固体板进行筛选,于30℃培养2~3天,得到的转化子进行划线分纯培养后提取酵母基因组进行PCR验证,从而确定阳性克隆。
重组菌株SyBE_Sc0123Z003通过涂布5-氟乳清酸(5-FOA)固体板回收重复利用URA营养标签,得到重组菌株SyBE_Sc0123Z010。将质粒mxa-LPP1和mxa-DPP1用NotI酶切位点切割获得相应的整合片段,采用醋酸锂法分别转化酿酒酵母SyBE_Sc0123Z010得到重组菌株SyBE_Sc0123Z011,SyBE_Sc0123Z012。重组菌株SyBE_Sc0123Z012通过涂布5-氟乳清酸(5-FOA)固体板回收重复利用URA营养标签,得到重组菌株SyBE_Sc0123Z013。将质粒mxa-DPP1用NotI酶切位点切割后获得相应的整合片段,采用醋酸锂法转化酿酒酵母SyBE_Sc0123Z013得到重组菌株SyBE_Sc0123Z014。
重组菌株SyBE_Sc0123Z014通过涂布5-氟乳清酸(5-FOA)固体板回收重复利用URA营养标签,得到重组菌株SyBE_Sc0123Z015。将质粒mxa2-GAL1-Eu、mxa2-GAL3-Eu和mxa2-GAL7-Eu用PmeI酶切位点切割获得相应的整合片段,采用醋酸锂法分别转化酿酒酵母SyBE_Sc0123Z015得到重组菌株SyBE_Sc0123Z016,SyBE_Sc0123Z017,SyBE_Sc0123Z018。重组菌株SyBE_Sc0123Z016通过涂布5-氟乳清酸(5-FOA)固体板回收重复利用URA营养标签,得到重组菌株SyBE_Sc0123Z019。将质粒mxa3-GAL1-Eu用PmeI酶切位点切割获得相应的整合片段,采用醋酸锂法分别转化酿酒酵母SyBE_Sc0123Z019得到重组菌株SyBE_Sc0123Z020。
1.6 重组酵母菌株的发酵将正确阳性克隆菌株接种于5ml种子培养基中,在30℃、250r/min培养11~13h,以初始菌体浓度OD600=0.1分别接种于50ml发酵培养基中,于30℃、250r/min条件下培养,以此条件继续培养至72h。
1.7 玉米黄质的提取、检测与定量取两等份的发酵液,4 000g离心2min收集菌体,并水洗两次。将其中一份菌体置于80℃烘干至恒重,称重计算细胞干重;另一份菌体用以产物提取,具体方法为:用3N HCl重悬细胞,置于沸水浴中煮沸2min,然后立即冰浴3min;将破碎的细胞12 000r/min、4℃离心4min弃上清,水洗2次后加入丙酮,并涡旋5min;最后离心收集丙酮相,用2μm滤膜过滤后上紫外液相检测。色谱条件:色谱柱:BDS HYPERSIL C18色谱柱(150mm×4.6mm,5μm);检测器:Waters 2489 UV/Vis;泵:Waters e2695;流动相:乙腈:甲醇=65:35;柱温:25℃;玉米黄质检测波长为450nm。定量方法:使用相同的色谱条件测定玉米黄质标准品的标准溶液(10~200 mg/L),绘制浓度-峰面积标准曲线,对玉米黄质定量。
2 结果与讨论 2.1 β-胡萝卜素羟化酶来源的优选β-胡萝卜素羟化酶(CrtZ)广泛存在于细菌、植物、古细菌等生物中,是玉米黄质合成中的重要基因,它催化底物β-胡萝卜素形成β-隐黄素,β-隐黄素进一步羟化形成玉米黄质。不同来源的外源基因虽然编码同一蛋白,但其表达产物在底物的选择性及底物的催化活性上存在一定的差异,并且经密码子优化后在酿酒酵母中的表达强度、折叠状态也可能存在差异。因此筛选出一个最适合本研究底盘表达系统的基因来源对于玉米黄质的生物合成是极其重要的。本研究选取了9种不同来源的CrtZ,包括水生副球菌(Agrobacterium aurantiacum)、产碱杆菌(Alcaligenes sp.strain PC-1),欧文氏菌(Erwinia uredovora)、成团泛菌(Pantoea agglomerans)、玉米细菌性枯萎病菌(Pantoea stewartii)、硫化叶菌(Sulfolobus solfataricus P2)、短波单胞菌(Brevundimonas sp. SD212)、短波单胞菌(Brevundimonas vesicularis DC263)、雨生红球藻(Haematococcus pluvialis),依次简写为Aa CrtZ、As CrtZ、Eu CrtZ、Pa CrtZ、Ps CrtZ、Ss CrtZ、B.SD CrtZ、B.DC CrtZ、Hp CrtZ。
将9种来源的CrtZ均整合至酿酒酵母的ho位点,并采用GAL1作为启动子。对重组酵母菌株进行50ml摇瓶发酵,用HPLC检测产物。根据在相同测定条件下得到的玉米黄质标准品浓度-峰面积标准曲线,对重组酵母细胞提取的玉米黄质进行定量。由图 3所示,以Eu CrtZ为例,对比初始β-胡萝卜素菌株成功检测到玉米黄质。
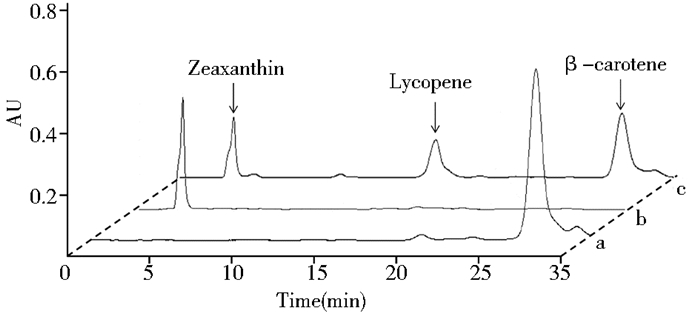
|
| 图 3 提取产物的HPLC检测 Figure 3 HPLC chromagraphy a:β-carotene producing strain; b:Zeaxanthin standard; c:Zeaxanthin producing strain |
实验结果表明,通过在相同的单拷贝位点整合不同来源的CrtZ,并保证发酵条件相同的情况下,玉米黄质的产量存在明显差异。如图 4所示,玉米黄质产量最高的是Eu CrtZ,其次是Ps CrtZ、Ss CrtZ,再次之是Pa CrtZ、B.SD CrtZ、B.DC CrtZ,而Aa CrtZ、As CrtZ、Hp CrtZ玉米黄质积累相对较低。Scaife等[13]通过在积累β-胡萝卜素的大肠杆菌中比较多种来源的CrtZ,发现Ps CrtZ、Ss CrtZ与Pa CrtZ相比能够积累更多的玉米黄质,且B.SD CrtZ和B.DC CrtZ与Pa CrtZ基本相当。Fraser等[14]曾经通过体外实验表征Aa CrtZ,As CrtZ,Hp CrtZ和Eu CrtZ,与Aa CrtZ和As CrtZ相比较,Eu CrtZ在β-胡萝卜素向玉米黄质转化方面具有更好的功能,并发现Aa CrtZ,As CrtZ,Hp CrtZ更倾向于以角黄素为底物进行羟化反应。Li等[8]也发现Eu CrtZ相较于Pa CrtZ和Hp CrtZ对于玉米黄质的生成具有更好的作用。本研究通过在酿酒酵母中整合不同来源的CrtZ,也得到了相同的实验结果,Eu CrtZ具有更好的催化活性。
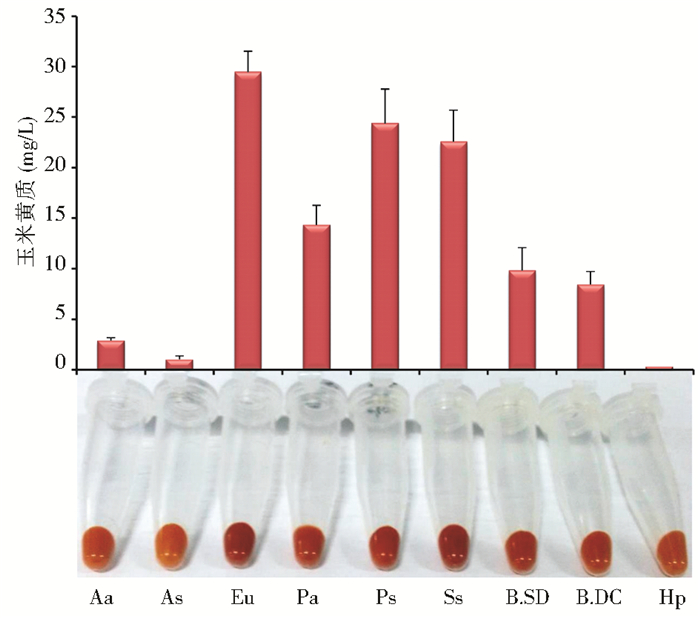
|
| 图 4 不同来源β-胡萝卜素羟化酶对玉米黄质产量的影响 Figure 4 Effect of different β-carotene hydroxylase on zeaxanthin production |
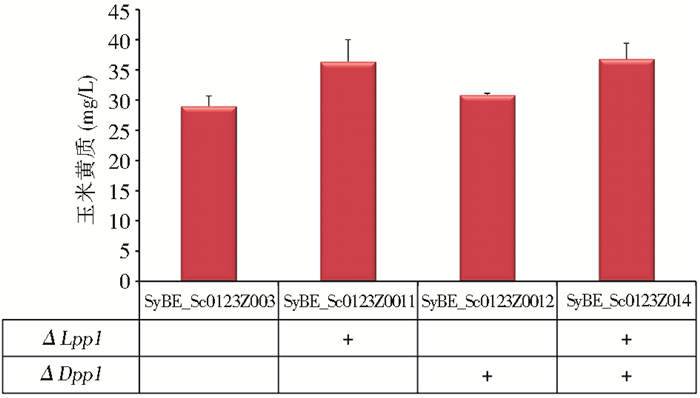
法呢基焦磷酸(FPP)作为异戊二烯途径中的重要中间体,是许多重要代谢产物如甾体、泛醌、萜烯等的重要合成前体。FPP处在类异戊二烯化合物合成的一个分支点上,FPP向法呢醇、鲨烯的转化是FPP合成GGPP(香叶基焦磷酸)的竞争路径,此前的研究多采用下调ERG9,通过将ERG9原本的启动子替换为表达强度更弱的启动子,以削弱FPP向鲨烯的转化达到增强FPP的目的。Westfall等[15]通过将ERG9的原始启动子替换为甲硫氨酸抑制的MET3启动子,使得紫穗槐二烯的产量提高了一倍,但是额外添加抑制剂对于实现产物的工业生产提高了生产成本。因此,本研究拟从阻断FPP向法尼醇的转化角度出发,达到积累更多FPP的目的,为玉米黄质的生物合成提供更多的前体。FPP向法呢醇的转化主要由磷酸脂磷酸酶(Lpp1和Dpp1)这两个基因负责[16],以SyBE_Sc0123Z003为底盘菌,分别构建敲除Lpp1、Dpp1及双敲Lpp1和Dpp1的菌株SyBE_Sc0123Z0011、SyBE_Sc0123Z0012、SyBE_Sc0123Z0014。由图 5的发酵结果可以看出,敲除Lpp1和Dpp1均可以提高玉米黄质的产量,但是提升效果并不显著,尤其是单独敲除Dpp1时,玉米黄质产量的提升很少,与本研究的结果类似,Takahashi等[17]发现单独敲除Dpp1时几乎不能促进萜烯的产量提升。Lpp1与Dpp1的敲除对于玉米黄质产量的提高并不显著,这可能是由于FPP还是其他多种物质的前体,如鲨烯、多萜醇、血红素A和醌等[18],在本研究所用酵母中,FPP可能更多地转化为这些物质。Farhi等[19]研究发现,敲除Lpp1和Dpp1对于提高萜类物质的产量是有限的,这是由于Lpp1和Dpp1分别定位于细胞膜和液泡,它们可能并不直接参与法呢醇的生成。
|
| 图 5 敲除Lpp1和Dpp1对玉米黄质产量的影响 Figure 5 Effect of △Lpp1 and △Dpp1 on zeaxanthin production |
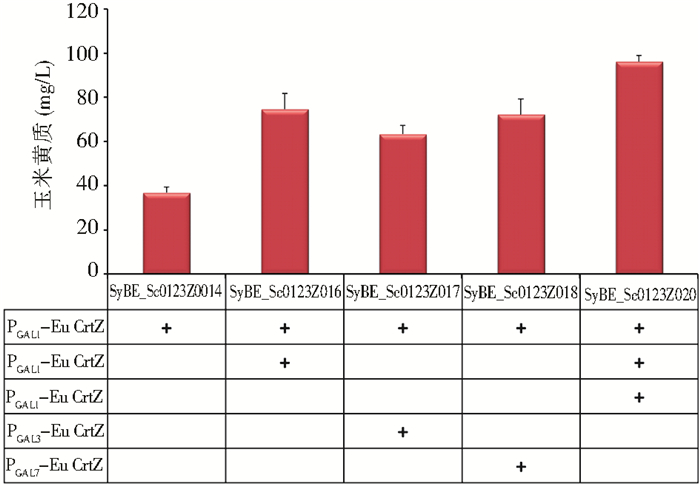
通过在ho位点整合Eu CrtZ并敲除Lpp1和Dpp1得到的玉米黄质产量虽然有所提高,但依然较低。Zhang等[20]研究表明,玉米黄质由一系列酶催化合成,CrtZ所催化的反应是玉米黄质生物合成的关键限速步骤。若基因的表达强度不足则不能完全转化上游中间产物,造成中间代谢物的积累和浪费,若其表达强度过高则多数酶分子不能参与合成反应,加重细胞表达负担[21]。启动子作为基因表达的重要调节因子[22],调节启动子的强弱使得基因表达的微调得以实现[23]。因此拟通过增加基因的拷贝数及改变启动子的强弱来调节基因的表达强度,从而达到精确调控β-胡萝卜素羟化酶以提高玉米黄质产量的目的。本研究在已有一个拷贝并由GAL1启动子调控的基础上,选择了GAL1、GAL3、GAL7三个表达强度存在差异[24]的启动子调控第二个β-胡萝卜素羟化酶的拷贝。通过50ml摇瓶发酵,并HPLC检测产物后发现,如图 6所示,增加基因的拷贝数均提高了玉米黄质的产量,GAL1和GAL7作为表达强度相当的强启动子比GAL3这一表达强度较弱的启动子实现了玉米黄质更多的积累。因此本研究继续在两个GAL1高强度启动子的拷贝下又增加了一个GAL1高强度启动子的拷贝数,如图 6结果显示,玉米黄质的产量提高到96.2mg/L,实现了酿酒酵母中玉米黄质的高产。
|
| 图 6 羟化酶基因不同表达强度对玉米黄质产量的影响 Figure 6 Effect of different expression level of CrtZ on zeaxanthin |
本研究为了克服因不同来源的β-胡萝卜素羟化酶在酿酒酵母中表达差异的不同而造成玉米黄质产量的不同,通过筛选9种来源的羟化酶以得到最适配本研究底盘细胞的基因来源。而后通过基因敲除FPP竞争路径的手段,实现了玉米黄质产量的提高。通过增加β-胡萝卜素羟化酶的拷贝数及调节启动子的强弱实现了玉米黄质产量的进一步提高。
| [1] | Ding M Z, Yan H F, Li L F, et al. Biosynthesis of taxadiene in Saccharomyces cerevisiae:selection of geranylgeranyl diphosphate synthase directed by a computer-aided docking strategy. PloS One , 2014, 9 (10) : e109348. DOI:10.1371/journal.pone.0109348 |
| [2] | Du H X, Xiao W H, Wang Y, et al. Engineering Yarrowia lipolytica for campesterol overproduction. PloS One , 2016, 11 (1) : e0146773. DOI:10.1371/journal.pone.0146773 |
| [3] | Lian J, Si T, Nair N U, et al. Design and construction of acetyl-CoA overproducing Saccharomyces cerevisiae strains. Metabolic Engineering , 2014, 24 : 139–149. DOI:10.1016/j.ymben.2014.05.010 |
| [4] | Ajikumar P K, Xiao W H, Tyo K E, et al. Isoprenoid pathway optimization for Taxol precursor overproduction in Escherichia coli. Science , 2010, 330 (6000) : 70–74. DOI:10.1126/science.1191652 |
| [5] | Sajilata M G, Singhal R S, Kamat M Y. The carotenoid pigment zeaxanthin-a review. Comprehensive Reviews in Food Science and Food Safety , 2008, 7 (1) : 29–49. DOI:10.1111/crfs.2008.7.issue-1 |
| [6] | Nishino H, Murakoshi M, Tokuda H, et al. Cancer prevention by carotenoids. Archives of Biochemistry and Biophysics , 2009, 483 (2) : 165–168. DOI:10.1016/j.abb.2008.09.011 |
| [7] | Moeller S M, Jacques P F, Blumberg J B. The potential role of dietary xanthophylls in cataract and age-related macular degeneration. Journal of the American College of Nutrition , 2000, 19 (5) : 522–527. |
| [8] | Li X R, Tian G Q, Shen H J, et al. Metabolic engineering of Escherichia coli to produce zeaxanthin. Journal of Industrial Microbiology & Biotechnology , 2015, 42 (4) : 627–636. |
| [9] | Beuttler H, Hoffmann J, Jeske M, et al. Biosynthesis of zeaxanthin in recombinant Pseudomonas putida. Applied Microbiology and Biotechnology , 2011, 89 (4) : 1137–1147. DOI:10.1007/s00253-010-2961-0 |
| [10] | Sun J, Shao Z Y, Zhao H M, et al. Cloning and characterization of a panel of constitutive promoters for applications in pathway engineering in Saccharomyces cerevisiae. Biotechnology and Bioengineering , 2012, 109 (8) : 2082–2092. DOI:10.1002/bit.24481 |
| [11] | Liang J, Ning J C, Zhao H M. Coordinated induction of multi-gene pathways in Saccharomyces cerevisiae. Nucleic Acids Research , 2013, 41 (4) : 1–10. |
| [12] | Gietz R D, Schiestl R H. Transforming yeast with DNA. Methods in Molecular and Cellular Biology , 1995, 5 (5) : 255–269. |
| [13] | Scaife M A, Ma C A, Ninlayarn T, et al. Comparative analysis of β-carotene hydroxylase genes for astaxanthin biosynthesis. Journal of Natural Products , 2012, 75 (6) : 1117–1124. DOI:10.1021/np300136t |
| [14] | Fraser P D, Miura Y, Misawa N. In vitro characterization of astaxanthin biosynthetic enzymes. Journal of Biological Chemistry , 1997, 272 (10) : 6128–6135. DOI:10.1074/jbc.272.10.6128 |
| [15] | Westfall P J, Pitera D J, Lenihan J R, et al. Production of amorphadiene in yeast, and its conversion to dihydroartemisinic acid, precursor to the antimalarial agent artemisinin. Proceedings of the National Academy of Sciences , 2012, 109 (3) : e111–e118. DOI:10.1073/pnas.1110740109 |
| [16] | Faulkner A, Chen X, Rush J, et al. The LPP1 and DPP1 gene products account for most of the isoprenoid phosphate phosphatase activities in Saccharomyces cerevisiae. Journal of Biological Chemistry , 1999, 274 (21) : 14831–14837. DOI:10.1074/jbc.274.21.14831 |
| [17] | Takahashi S, Yeo Y, Greenhagen B T, et al. Metabolic engineering of sesquiterpene metabolism in yeast. Biotechnology and Bioengineering , 2007, 97 (1) : 170–181. DOI:10.1002/bit.v97:1 |
| [18] | Xie W P, Ye L D, Lv X M, et al. Sequential control of biosynthetic pathways for balanced utilization of metabolic intermediates in Saccharomyces cerevisiae. Metabolic Engineering , 2015, 28 : 8–18. DOI:10.1016/j.ymben.2014.11.007 |
| [19] | Farhi M, Marhevka E, Masci T, et al. Harnessing yeast subcellular compartments for the production of plant terpenoids. Metabolic Engineering , 2011, 13 (5) : 474–481. DOI:10.1016/j.ymben.2011.05.001 |
| [20] | Zhang L, Gong Y F, Liu X D, et al. Cloning and expression analysis of β-carotene hydroxylase gene(chyb) from Dunaliella salina. Journal of Agricultural Biotechnology , 2013, 21 (8) : 920–930. |
| [21] | Liu D, Du J, Zhao G R, et al. Applications of synthetic biology in medicine and energy. CIESC J , 2011, 62 (9) : 2391–2397. |
| [22] | Hammer K, Mijakovic I, Jensen P R. Synthetic promoter libraries-tuning of gene expression. Trends in Biotechnology , 2006, 24 (2) : 53–55. DOI:10.1016/j.tibtech.2005.12.003 |
| [23] | Siegl T, Tokovenko B, Myronovskyi M, et al. Design construction and characterisation of a synthetic promoter library for fine-tuned gene expression in actinomycetes. Metabolic Engineering , 2013, 19 : 98–106. DOI:10.1016/j.ymben.2013.07.006 |
| [24] | Paddon C J, Westfall P J, Pitera D J, et al. High-level semi-synthetic production of the potent antimalarial artemisinin. Nature , 2013, 496 (7446) : 528–532. DOI:10.1038/nature12051 |
 2016, Vol. 36
2016, Vol. 36




