文章信息
- 张文倩, 周晓, 肖文海, 王颖
- ZHANG Wen-qian, XIAO Wen-hai, ZHOU Xiao, WANG Ying
- 人工酵母后鲨烯路径基因对7-脱氢胆固醇合成的影响
- Effect of Post-squalene Genes on the Synthesis of 7-Dehydrocholesterol in the Artificial Saccharomyces cerevisiae
- 中国生物工程杂志, 2016, 36(6): 39-50
- China Biotechnology, 2016, 36(6): 39-50
- http://dx.doi.org/10.13523/j.cb.20160606
-
文章历史
- 收稿日期: 2015-11-25
- 修回日期: 2015-12-07
2. 天津化学化工协同创新中心合成生物学平台 天津 300072
2. SynBio Research Platform, Collaborative Innovation Center of Chemical Science and Engineering, Tianjin 300072, China
酵母由于具备强大的蛋白表达和修饰能力以及完整的内膜系统,是表达很多结构复杂天然化合物的优良宿主[1, 2, 3, 4, 5]。麦角固醇(Ergosterol)是酵母细胞膜中重要的组成成分[6],而鲨烯(Squalene)则是麦角固醇合成路径上最为关键的节点[7, 8]。以鲨烯为界,可将麦角固醇合成路径分为前鲨烯路径(Pre-squalene Pathway)和后鲨烯路径(Post-squalene Pathway)。对前鲨烯路径的改造基本不会改变后鲨路径代谢流的强弱[8]。Polakowski等[8]报道,在野生型酿酒酵母(Saccharomyces cerevisiae)中过表达前鲨烯路径限速酶HMG-CoA还原酶(HMGR),会造成鲨烯的大量积累,而对后鲨路径中相关固醇类化合物的合成影响微弱。后鲨烯路径是一个复杂的代谢过程,涉及12个酶、13步化学反应[9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19]。其中ERG1(角鲨烯环氧酶) [9]和ERG11(羊毛固醇14α-去甲基化酶)[11]被认为是麦角固醇合成中的关键基因,即只有过表达这两个蛋白才会显著改变后鲨烯路径相关固醇类化合物的含量[7]。值得注意的是,这些固醇类代谢物恰好是合成异源甾体类药物的重要前体[4, 5, 20, 21]。而实现这些甾体类药物在酵母中的异源表达,需引入相应的异源表达模块。而这些异源模块的导入,往往会打破细胞内部原始的代谢平衡[22, 23]。这或许就是人工酵母中异源甾体类化合物产量偏低[4, 5, 24]的原因之一。因此,若要提高甾体类物质在酵母中异源表达的水平,需对后鲨烯路径基因与异源产物合成的关系进行解析,并通过调整后鲨烯路径相关蛋白的表达,对内源路径和外源模块进行适配,使代谢流最大化地顺畅流向目标产物。然而与此相关的研究未见报道。
7-脱氢胆固醇(7-dehydrocholesterol,7-DHC)是合成维生素D3的重要直接前体,即7-DHC可在紫外线照射下直接转化为维生素D3[20, 21]。此外,7-DHC也是制备胆甾相液晶材料的原材料,在生物医学、电磁场检测、显示等方面均有着广泛的应用[25]。构建人工酵母以葡萄糖为底物合成7-DHC,可以弥补传统化学合成[26]或生物转化[27]在生产成本及环境保护等方面的诸多缺陷[28]。Lang等[29]和Hohmann等[30]在阻断竞争性酵母内源麦角固醇合成通路的同时,通过引入异源固醇C24-还原酶(DHCR24)并过表达截短的羟甲基戊二酰辅酶A还原酶(tHMGR),构建7-DHC生产菌株。在此基础上,Su等[24]证明增强前体乙酰辅酶A的供给,可提高人工酵母中7-DHC的产量。此外,通过引入肺炎链球菌(Streptococcus pneumoniae)NADH氧化酶NOX和荚膜组织胞浆菌(Histoplasma capsulatum)选择性氧化酶AOX1,来降低胞内自由态NADH/NAD+的比值,缓解由于麦角固醇缺陷造成的氧化还原不平衡,同样是促进7-DHC合成的有效手段[24]。然而,目前已报道的7-DHC的最高产量[24]尚不能满足实际工业生产的需要,而相关研究尚未涉及后鲨烯路径的改造。
本文以人工酵母合成7-DHC为例,研究后鲨烯路径基因对异源甾体物质合成的影响。首先以ERG6(编码固醇C-24甲基转移酶)[16]缺失的酿酒酵母BY4742为底盘,通过引入人源DHCR24和过表达内源tHMGR,构建7-DHC生产菌株。在此基础上,将后鲨烯路径进行分割并构建成相应的过表达模块,引入到所构建的7-DHC合成菌株中。通过GC-TOF/MS分析并结合主成分分析,研究不同后鲨烯模块的过表达对相关固醇类代谢物乃至7-DHC合成的影响,为后续在人工酵母中提高7-DHC乃至其他甾体类药物的产量,提供潜在靶标。
1 材料与方法 1.1 菌株与培养方法本研究所用所有菌株见表 1。大肠杆菌(Escherichia coli)Trans 1-T1 Phage Resistant感受态细胞购于北京全式金生物有限公司。酿酒酵母BY4742及其ERG6单基因缺失菌株ΔERG6均由本实验室保存。
| Genotype/description | Source | |
| Plasmids | ||
| pYES2.0 | Shuttle plasmid for E. coli and S. cerevisiae, Amp r, URA3 | This laboratory |
| pRS425K | Shuttle plasmid for E. coli and S. cerevisiae, Kan r, LEU2 | This laboratory |
| pDHCR24 | pYES2.0 harboring cassette PGK1p-DHCR24-PGK1t | This study |
| ptHMGR | pRS425K harboring cassette PGK1t-TPI1p-tHMGR-CYC1t | This study |
| pC24-tH | pYES2.0 harboring cassette PGK1p-DHCR24-PGK1t-TPI1p- thmg1-CYC1t | This study |
| pERG1 | pRS425K harboring cassette ENO2t-TEF1p-ERG1-GPM1t | This study |
| pERG7 | pRS425K harboring cassette GPM1t-PDC1p-ERG7-GPDt | This study |
| pERG11 | pRS425K harboring cassette GPDt-PGK1p-ERG11-FBA1t | This study |
| pERG24-25-26-27 | pRS425K harboring cassette FBA1t-TPI1p-ERG24-PGK1t -HXT7p-ERG25-CYC1t-TDH3p-ERG26-TEF1t-GPM1p-ERG27-TPI1t | This study |
| pERG2-3 | pRS425K harboring cassette TPI1t-TDH2p-ERG2-PGI1t -HXK2p-ERG3-TEF2t | This study |
| S. cerevisiae Strains | ||
| BY4742 | MATα, his3Δ1,leu2Δ0,lys2Δ0,ura3Δ0 | This laboratory |
| ΔERG6 | BY4742, ERG6∷KanMX | This laboratory |
| SyBE0019-Sc-002 | ΔERG6 with plasmid pC24-tH | This study |
| SyBE0019-Sc-003 | ΔERG6 with plasmid pC24-tH and pERG1 | This study |
| SyBE0019-Sc-004 | ΔERG6 with plasmid pC24-tH and pERG7 | This study |
| SyBE0019-Sc-005 | ΔERG6 with plasmid pC24-tH and pERG11 | This study |
| SyBE0019-Sc-006 | ΔERG6 with plasmid pC24-tH and pERG24-25-26-27 | This study |
| SyBE0019-Sc-007 | ΔERG6 with plasmid pC24-tH and pERG2-3 | This study |
大肠杆菌37℃培养于LB培养基[24]。根据需要,接菌前向LB培养基中添加100 μg/ml的氨苄青霉素或100 μg/ml的卡纳霉素。酿酒酵母30℃培养于YPD培养基[24]。酵母菌株转化子筛选和产物发酵使用SD培养基[24]。
1.2 基因元件的获得与菌株构建本文所用引物(表 2)均由北京奥科鼎盛生物科技有限公司合成。本文所用大肠杆菌-酿酒酵母穿梭质粒pRS425K和pYES2.0均为本实验室保藏。本文中涉及的所有内源基因(tHMG1、ERG1、ERG7、ERG11、ERG24、ERG25、ERG26、ERG27、ERG2、ERG3)、启动子(PGK1p、TPI1p、TEF1p、PDC1p、TPI1p、HXT7p、TDH3p、GPM1p、TDH2p、HXK2p)和终止子(CYC1t、GPM1t、GPDt、FBA1t、PGK1t、TEF1t、TPI1t、PGI1t、TEF2t),均以酿酒酵母BY4742基因组为模版,通过PCR扩增获得。
| Primer name | Sequence(5′~3′) |
| TEF1p-EGR1-F | AAAACTGCAGGCGGCCGCAATGTTTCTACTCCTTTTTT |
| TEF1p-EGR1-R | GTGCAACGTTAACAGCAGACATTTTGTAATTAAAACTTAGAT |
| ERG1-F | ATCTAAGTTTTAATTACAAAATGTCTGCTGTTAACGTTGCAC |
| ERG1-R | CATCAAATCATTCATTCTTCAGACTTAACCAATCAACTCACCAAAC |
| ERG1-GPM1t-F | GTTTGGTGAGTTGATTGGTTAAGTCTGAAGAATGAATGATTTGATG |
| ERG1-GPM1t-R | CGCGGATCCGCGGCCGCTATTCGAACTGCCCATTCAG |
| PDC1p-ERG7--F | AAAACTGCAGGCGGCCGC ATGCGACTGGGTGAGCAT |
| PDC1p-ERG7-R | GTCAGAATAAAATTCTGTCATTTTGATTGATTTGACTGTGTTATTTTG |
| EGR7-F | CAAAATAACACAGTCAAATCAATCAAAATGACAGAATTTTATTCTGAC |
| ERG7-R | AATGCAAGATTTAAAGTAAATTCACTTAGATCTTTTGTTCTGGATTTC |
| ERG7-GPDt-F | GAAATCCAGAACAAAAGATCTAA GTGAATTTACTTTAAATCTTGCATT |
| ERG7-GPDt-R | CGCGGATCCGCGGCCGCGGAATCTGTGTATATTACTGCATCTAG |
| PGK1p-ERG11-F | AAAACTGCAGGCGGCCGC TATTTTAGATTCCTGACTTCAACTCAAG |
| PGK1p-ERG11-R | CGATTGACTTGGTAGCAGACATTGTTTTATATTTGTTGTAAAAAGTAGATAATTAC |
| EGR11-F | GTAATTATCTACTTTTTACAACAAATATAAAACATGTCTGCTACCAAGTCAATCG |
| ERG11-R | CATTAAAAAACTATATCAATTAATTTGAATTAACTTAGATCTTTTGTTCTGGATTTC |
| ERG11-FBA1t-F | GAAATCCAGAACAAAAGATCTAAGTTAATTCAAATTAATTGATATAGTTTTTTAATG |
| ERG11-FBA1t-R | CGCGGATCCGCGGCCGCAAAGATGAGCTAGGCTTTTGTAAAAATATC |
| TPI1p-ERG24-F | AAAACTGCAGGCGGCCGC TATATCTAGGAACCCATCAGGTTG |
| TPI1p-ERG24-R | GGGATTCAAAGCTGATACCATTTTTAGTTTATGTATGTGTTTTTTGTAGT |
| ERG24-F | ACTACAAAAAACACATACATAAACTAAAA ATGGTATCAGCTTTGAATCCC |
| ERG24-R | CTATCGATTTCAATTCAATTCAATCGAACGCAGAATTTTCGAGT |
| ERG24-PGK1t-F | ACTCGAAAATTCTGCGTTCG ATTGAATTGAATTGAAATCGATAG |
| ERG24-PGK1t-R | CGCGGATCCGCGGCCGCAACGAACGCAGAATTTTCG |
| PGK1t-HXT7p-F | AAAACTGCAGGCGGCCGCATTGAATTGAATTGAAATCGATAG |
| PGK1t-HXT7p-R | CGAAATTGTTCCTACGAGAAGTAACGAACGCAGAATTTTCG |
| HXT7p-ERG25-F | CGAAAATTCTGCGTTCGTTACTTCTCGTAGGAACAATTTCG |
| HXT7p-ERG25-R | GTTGTTGAAAACGGCAGACATTTTTTGATTAAAATTAAAAAAACTTTTTG |
| ERG25-F | CAAAAAGTTTTTTTAATTTTAATCAAAAAATGTCTGCCGTTTTCAACAAC |
| ERG25-R | GTAAGCGTGACATAACTAATTACATGTTAGTTAGTCTTCTTTTGAGC |
| ERG25-CYC1t-F | GCTCAAAAGAAGACTAACTAA CATGTAATTAGTTATGTCACGCTTAC |
| ERG25-CYC1t-R | CGCGGATCCGCGGCCGCAAAGCCTTCGAGCGTCCC |
| CYC1t-TDH3p-F | AAAACTGCAGGCGGCCGCCATGTAATTAGTTATGTCACGCTTAC |
| CYC1t-TDH3p-R | GTGATATAGAGTGTAAATGAGCATATACAAAAGCCTTCGAGCGTCCC |
| TDH3p-ERG26-F | GGGACGCTCGAAGGCTTTTGTATATGCTCATTTACACTCTATATCAC |
| TDH3p-ERG26-R | AACTGAATCTATCTTTGACATTTTGTTTTGTGTGTAAATTTAGTGAAG |
| ERG26-F | CTTCACTAAATTTACACACAAAACAAA ATGTCAAAGATAGATTCAGTT |
| ERG26-R | GAAAAGTCTTATCAATCTCCTTATTTGATAGCGCCGATCAAAGTATTTG |
| ERG26-TEF1t-F | CAAATACTTTGATCGGCGCTATCAAATAAGGAGATTGATAAGACTTTTC |
| ERG26-TEF1t-F | CGCGGATCCGCGGCCGCGATAGCGCCGATCAAAGTA |
| TEF1t-GPM1p-F | AAAACTGCAGGCGGCCGCAAATAAGGAGATTGATAAGACTTTTC |
| TEF1t-GPM1p-R | CTTAAAGTCATACATTGCACGACTAGATAGCGCCGATCAAAGTA |
| GPM1p-ERG27-F | TACTTTGATCGGCGCTATCTAGTCGTGCAATGTATGACTTTAAG |
| GPM1p-ERG27-R | GTTACGATAGCTACTTTCCTGTTCATTATTGTAATATGTGTGTTTGTTTGG |
| ERG27-F | CCAAACAAACACACATATTACAATA ATGAACAGGAAAGTAGCTATCGTAAC |
| ERG27-R | GAAGATAATATTTTTATATAATTATATTAATCTTAAATGGGGGTTCTAGTTTC |
| ERG27-TPIt-F | GAAACTAGAACCCCCATTTAA GATTAATATAATTATATAAAAATATTATCTTC |
| ERG27-TPIt-R | CGCGGATCCGCGGCCGCTATATAACAGTTGAAATTTGGATAAGAAC |
| TDH2p-ERG2-F | AAAACTGCAGGCGGCCGCATTGGTTTTTCCAGTGAATGA |
| TDH2p-ERG2-R | CAAAAGGAGTGGGAAAAACTTCATTTTGTTTTGTTTGTTTGTGTGATG |
| ERG2-F | CATCACACAAACAAACAAAACAAAATGAAGTTTTTCCCACTCCTTTTG |
| ERG2-R | TTAGAACTTTTTGTTTTGCAACAAGTAGGTATATATTTAAGAGCGATTTGT |
| ERG2-PGIt-F | ACAAATCGCTCTTAAATATATACCTACTTGTTGCAAAACAAAAAGTTCTAA |
| ERG2-PGIt-R | CGCGGATCCGCGGCCGCGTAGTTTAGTGTTTTTCTTCCAGTG |
| PGIt-HXK2p-F | AAAACTGCAGGCGGCCGCACAAATCGCTCTTAAATATATACCTA |
| PGIt-HXK2p-R | TGCTCTTCTATGGCGTTCAGTAGTTTAGTGTTTTTCTTCCAGTG |
| HXK2p-ERG3-F | CACTGGAAGAAAAACACTAAACTACTGAACGCCATAGAAGAGCA |
| HXK2p-ERG3-R | CAGCGACTTCTAAGACCAAATCCATTTTATTTAATTAGCGTACTTATTATGTG |
| ERG3-F | CACATAATAAGTACGCTAATTAAATAAA ATGGATTTGGTCTTAGAAGTCGCTG |
| ERG3-R | ATTATATGGAAGCAATAATTATTACTCTCAGTTGTTCTTCTTGGTATTTGG |
| ERG3-TEF2t-F | CCAAATACCAAGAAGAACAACTGAGAGTAATAATTATTGCTTCCATATAAT |
| ERG3-TEF2t-R | CGCGGATCCGCGGCCGCGGGGTAGCGACGGATTAATG |
| PGK1t-TPI1p-F | AAAACTGCAGGCGGCCGCATTGAATTGAATTGAAATCGATAG |
| PGK1t-TPIp-R | CAACCTGATGGGTTCCTAGATATAAACGAACGCAGAATTTTCG |
| TPI1p-tHMGR-F | CGAAAATTCTGCGTTCGTTTATATCTAGGAACCCATCAGGTTG |
| TPI1p-tHMGR-R | CAGTTTTCACCAATTGGTCCATTTTTAGTTTATGTATGTGTTTTTTGTAGT |
| tHMGR-F | ACTACAAAAAACACATACATAAACTAAAAATGGACCAATTGGTGAAAACTG |
| tHMGR-R | GTAAGCGTGACATAACTAATTACATGTTAGGATTTAATGCAGGTGACG |
| tHMGR-CYC1t-F | CGTCACCTGCATTAAATCCTAACATGTAATTAGTTATGTCACGCTTAC |
| tHMGR-CYC1t-R | CGCGGATCCGCGGCCGCAAAGCCTTCGAGCGTCCC |
| Note: The restriction sites were underlined | |
在Genbank中查找相应人源DHCR24氨基酸序列(GI:114155130),根据酿酒酵母密码子偏好性对该基因编码序列进行优化,并交由金唯智公司合成。合成时该基因序列3′端依次添加PGK1t终止子序列和BsrGⅠ、SpeⅠ限制性酶切位点,5′端引入HindⅢ、EcoRⅠ、XbaⅠ限制性酶切位点。DHCR24全基因合成产物用HindⅢ和BsrGⅠ酶切,然后连接到大肠杆菌-酵母穿梭质粒pYES2.0相应的酶切位点上。所获得的质粒用EcoRⅠ和XbaⅠ酶切后,与使用EcoRⅠ和SpeⅠ酶处理过的PGK1p PCR产物相连,构成质粒pDHCR24(含有表达盒PGK1p-DHCR24-PGK1t,即DHCR24表达盒)。将PGK1t、TPI1p、tHMG1、CYC1t的PCR产物用OE-PCR拼接在一起。拼接产物用PstⅠ与BamHⅠ酶切后,连接到质粒pRS425K的相应位点上,构成质粒ptHMGR(含有表达盒PGK1t-TPI1p-tHMG1-CYC1t)。该表达盒与DHCR24表达盒之间,可利用终止子PGK1t和CYC1t作为前后同源臂,通过同源重组拼接在一起。即将tHMGR片段用PstⅠ和BamHⅠ从载体ptHMGR上切下,通过RADOM法[31],与使用SpeI处理过的质粒pDHCR24拼接在一起,构成质粒pC24-tH(含有表达盒PGK1t-TPI1p-tHMGR-CYC1t,即7-DHC合成模块,图 1c)。

|
| 图 1 人工酿酒酵母中7-脱氢胆固醇合成路径及相应遗传改造 Fig. 1 Overview of 7-DHC biosynthesis pathway and the corresponding genetic modification in S. cerevisiae (a) Overview of 7-DHC biosynthesis pathway in S. cerevisiae. 7-DHC was produced in S. cerevisiae BY4742 via disruption of ergosterol synthesis pathway (deleting gene ERG6), introduction of DHCR24 and over-expression of tHMGR (b) Post-squalene gene(s) overexpression module. The Post-squalene genes were comprised 5 modules, i.e. ERG1, ERG7, ERG11, ERG24-ERG25-ERG26-ERG27 and ERG2-ERG3. The promoters and terminators for each gene within these modules were presented by grey squares and golden arrows, respectively (c) 7-DHC synthesis module (d) 7-DHC titers for post-squalene gene(s) overexpression strains |
将后鲨烯路径分割成ERG1、ERG7、ERG11、ERG24-25-26-27和ERG2-3这5个模块(图 1b)。表达盒中每个相邻的片段以及片段与载体pRS425K之间有40 bp同源序列,可通过RADOM法[31]将相应片段拼接在一起,构成每个基因表达盒,即质粒pERG1、pERG7、pERG11、pERG24-25-26-27和pERG2-3。
通过醋酸锂法[32],将pC24-tH导入野生型酿酒酵母BY4742和ERG6单基因缺失菌株ΔERG6中,得到人工7-DHC合成酵母SyBE0019-Sc-002。再将质粒pERG1、pERG7、pERG11、pERG24-25-26-27和pERG2-3分别导入SyBE0019-Sc-002,获得后鲨烯路径不同模块过表达的菌株SyBE0019-Sc-003、SyBE0019-Sc-004、SyBE0019-Sc-005、SyBE0019-Sc-006、SyBE0019-Sc-007(表 1)。
1.3 发酵过程及产物检测挑取酵母单菌落,接种于4 ml SD培养基中,30℃、220 r/min摇床振荡培养过夜。随后转接到50 ml SD培养基中继续培养至OD600为4。取二级种子转接到100 ml SD培养基中。发酵初始OD600为0.2,30℃、220 r/min摇床振荡培养36 h后,4 000 r/min离心2 min收集菌体。发酵过程中,通过紫外可见分光光度计测量不同时间发酵液在波长600 nm处的吸光度,来测定发酵液中菌株的生物量。
所收集的菌体用无菌水洗涤2次后,用液氮冷冻并研磨。将细胞粉末收集到10 ml离心管中并称重后,向离心管内加入5 ml 1.5 mol/L KOH-甲醇溶液,60℃皂化4 h。然后加入2 ml正己烷萃取,漩涡振荡使之混合均匀后,1 000 r/min离心5 min,取上层清液至新的离心管中进行真空冷冻干燥。之后向离心管内加入100 μl N-甲基-N-三甲基硅基三氟乙酰胺(MSTFA),30℃衍生化反应3 h。
将产物用正己烷稀释10倍,使用气相色谱-飞行时间质谱(GC-TOF/MS,Waters Corp. USA)对发酵产物中后鲨烯路径相关固醇类化合物进行分析检测。检测条件除柱温变化外与苏皖等[24]所报道的7-DHC检测方法相同。本文所使用的柱温变化为:首先在70 ℃保持 1 min;之后以20 ℃/min的速度升至250 ℃,在250 ℃维持2 min;然后以15 ℃/min的速度升到280 ℃,在280 ℃维持15 min。将质谱的碎片峰和NIST 数据库(2006)进行比较,对色谱图中各个峰进行鉴定。
1.4 统计学分析将后鲨烯模块过表达菌株中各种固醇类化合物的含量,利用 Matlab(The MathWorks,R2008a)软件,进行主成分分析(principal components analysis,PCA)分别得到 PCA 得分图(PCA Score Plot)和载荷图(PCA Loading Plot)。
2 结果与分析 2.1 酿酒酵母中7-DHC的合成要在酵母中实现7-DHC的合成,必须阻断其内源麦角固醇合成路径的表达[24]。本文选择缺失了基因ERG6的酿酒酵母ΔERG6作为出发菌株,通过导入7-DHC合成模块(即引入人源DHCR24,并过表达tHMGR,图 1a、图 1c),获得可生成7-DHC的人工酵母SyBE0019-Sc-002。将酿酒酵母BY4742、ΔERG6和SyBE0019-Sc-002在摇瓶中培养36 h后收集菌体细胞,提取胞内的总固醇进行GC-TOF/MS分析。如图 2所示,与野生型酿酒酵母BY4742相比,菌株ΔERG6和SyBE0019-Sc-002缺少保留时间为21.02的峰,经质谱鉴定该物质为麦角固醇(图 2)。这表明由于ERG6缺失,菌株ΔERG6和SyBE0019-Sc-002中麦角固醇合成通路被成功阻断。同时菌株ΔERG6中大量积累酵母固醇(zymosterol,保留时间为20.68,图 2)。与菌株野生型BY4742和ΔERG6相比,菌株SyBE0019-Sc-002在保留时间20.39出现一个峰,质谱碎片有325、351和366三个特征峰,与7-DHC标准品碎片特征峰相符(图 2)。这表明该菌株可以以葡萄糖为底物合成7-DHC,其产量为5.20 mg/g细胞干重。值得注意的是,菌株SyBE0019-Sc-002中大量积累鲨烯(保留时间为16.23),这很可能是由于过表达tHMGR且后鲨烯路径蛋白表达强度相对较弱造成的。因此,需对后鲨烯路径基因的表达量进行优化。
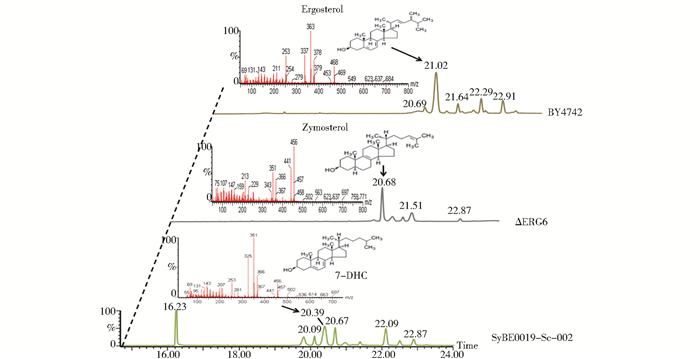
|
| 图 2 酿酒酵母BY4742、ΔERG6和7-DHC生产菌株SyBE0019-Sc-002发酵产物GC-TOF/MS分析 Fig. 2 GC-TOF/MS analysis of S.cerevisiae BY4742, ΔERG6 and 7-DHC producing strain SyBE0019-Sc-002 |
将后鲨烯路径分割成ERG1、ERG7、ERG11、ERG24-25-26-27和ERG2-3这5个模块(图 1b),分别在7-DHC合成菌株SyBE0019-Sc-002中进行过表达。将后鲨烯模块过表达菌株连同出发菌株SyBE0019-Sc-002在摇瓶中进行发酵,并利用GC-TOF/MS测定胞内各类固醇类代谢物的含量。其中菌株SyBE0019-Sc-007(模块ERG2-3过表达)中7-DHC的产量为6.23 mg/g细胞干重,较出发菌株SyBE0019-Sc-002提高了19.8%,是迄今报道的微生物异源表达中摇瓶水平的最高产量[24]。而该菌株中总固醇的含量略有增加(图 1d、图 3b);而其他后鲨烯路径过表达菌株中无论是7-DHC的产量还是总固醇的含量,都较出发菌株有所下降(图 1d、图 3b)。这一结果表明,模块ERG2-3的过表达很可能通过增强整体后鲨烯路径代谢流来提高7-DHC的产量,而模块ERG1、ERG7、ERG11和ERG24-25-26-27表达量的增加则不利于7-DHC的合成。

|
| 图 3 发酵产物中后鲨烯路径相关固醇类代谢物的鉴定 Fig. 3 Identification of sterols in the production for post-squalene gene(s) overexpression strains (a) GC-TOF/MS profile of the production for post-squalene gene(s) overexpression strains (b) Sterol contents in peak area per gram dry matter of post-squalene gene(s) overexpression strains |
利用GC-TOF/MS对后鲨烯模块过表达菌株发酵产物中相关固醇类代谢物进行分析。在色谱检测到的化合物中,鲨烯(保留时间16.21)、羊毛固醇(lanosterol,保留时间22.86)、14-去甲基羊毛固醇(14-demethylanosterol,保留时间23.23)和酵母固醇(保留时间20.66)均位于酵母内源后鲨烯路径上(图 1a、图 3a)。ERG6敲除后,酵母固醇在ERG2(编码固醇C-8异构酶)[17]和ERG3(固醇C-5脱氢酶)[18]的催化下,形成7-脱氢链固醇(7-Dehydrodesmosterol,保留时间19.79,图 1a、图 3a)从羊毛固醇到7-脱氢链固醇的路径与哺乳动物胆固醇合成通路中的Bloch路径(Bloch Pathway,图 1a)[33]相一致。所引入的人源DHCR24,不仅可以催化7-脱氢链固醇生成所需终产物7-DHC(保留时间20.36),还可催化Bloch路径上其他中间代谢产物[34],形成24,25-双脱氢羊毛固醇(24,25-Dihydrolanosterol,保留时间22.08)、4,4-Dimethyl-cholesta-8-en-3β-ol(保留时间22.46)、4α-甲基酵母固烯醇(4α-Methylzymostenol,保留时间21.36)、Cholesta-8-en-3β-ol(保留时间20.09)等化合物(图 1a、图 3a)。这些化合物也可互为后鲨烯路径蛋白的底物和产物,形成一条非酵母内源的路径(图 1)。该路径与哺乳动物细胞内另一条胆固醇合成通路Kandutsch-Russell路径(Kandutsch-Russell Pathway,图 1a)[33]相符。因此,人工7-DHC合成酵母的后鲨烯路径同时包含内源的Bloch路径和伴随异源DHCR24的引入所新形成的Kandutsch-Russell路径。这两条路径通过DHCR24相互交叉(图 1a)。此外,酵母固醇还可直接被ERG3催化形成5-脱氢酵母固醇(5-dehydrozymosterol,保留时间20.95,图 1a、图 3a)。推测该化合物难以被ERG2催化形成7-DHC的直接前体7-脱氢链固醇,因此以副产物的形式积累在酵母体内。
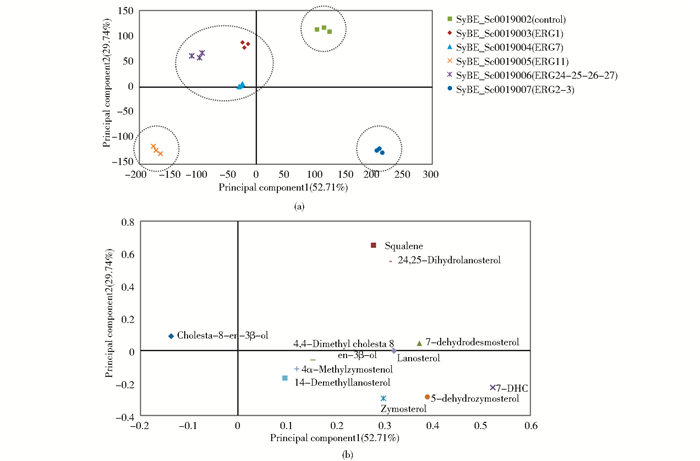
|
| 图 4 后鲨烯路径过表达菌株固醇类代谢物的PCA分析 Fig. 4 PCA analysis of sterols for post-squalene gene(s) overexpression strains (a) PCA score plot for sterols of post-squalene gene(s) overexpression strains(b) PCA loading plot for sterols of post-squalene gene(s) overexpression |
如图 3a所示,各固醇类代谢物在不同后鲨烯模块过表达菌株中丰度不同(图 3a)。对这些GC-TOF/MS检测到的固醇类化合物进行PCA分析(图 4)。由得分图(图 4a)可以看出,后鲨烯模块过表达菌株聚为4类:过表达模块ERG1、ERG7和ERG24-25-26-27分别进行的菌株SyBE_Sc0019003、SyBE_Sc0019004与SyBE_Sc0019006聚成一类;而未进行后鲨烯模块过表达的出发菌株SyBE_Sc0019002、过表达模块ERG11菌株SyBE_Sc0019005以及过表达模块ERG2-3菌株SyBE_Sc0019007分别各成一类。同时,从PCA载荷图(图 4b)可以看出,对PCA得分图中聚类贡献较大的代谢物,即潜在的生物标志物主要包含鲨烯、羊毛固醇、Bloch路径上的酵母固醇和7-脱氢链固醇、Kandutsch-Russell路径上的24,25-双脱氢羊毛固醇、终产物7-DHC、以及副产物5-脱氢酵母固醇。其中,7-DHC、7-脱氢链固醇和5-脱氢酵母固醇在不同后鲨烯模块过表达菌株中的变化最为显著(图 3b、图 4b)。
ERG2-3配合DHCR24以酵母固醇为底物合成目标产物7-DHC(图 5)。对麦角固醇合成来说,ERG2-3并不是后鲨烯路径中的关键基因,其表达量的上升对胞内固醇组分影响微弱[7]。而在本研究中,过表达ERG2-3模块则会在降低胞内鲨烯含量的同时,显著提升胞内羊毛固醇及其下游固醇组分,特别是7-DHC、7-脱氢链固醇和5-脱氢酵母固醇这3个最为关键的生物标志物的含量(图 5)。因此,现阶段对ERG2-3表达强度的调整,在7-DHC的表达及其在总固醇中所占比例方面,都有明显的促进作用(图 3b)。这说明该调整对整个后鲨烯路径的整体强化起到了显著的作用。此外,由于模块ERG2-3的过表达,菌株SyBE_Sc0019007中大量积累副产物5-脱氢酵母固醇(图 5)。在后续的实验中,希望通过酶的定向进化,改变ERG2和ERG3的底物选择性。以期通过减少ERG3对酵母固醇的识别,或增强ERG2对5-脱氢酵母固醇的催化能力,减少副产物的积累。

|
| 图 5 7-DHC合成路径中后鲨烯部分各关键代谢物产量变化 Fig. 5 The change of critical metabolites in post-squalene section of 7-DHC synthesis pathway |
羊毛固醇及其被DHCR24催化后形成的24,25-双脱氢羊毛固醇,是ERG11的直接底物(图 5)。Veen等[7]报道在过表达tHMGR的酿酒酵母中,ERG11控制整个后鲨烯路径代谢流。而本研究中,过表达模块ERG11,会显著降低后鲨烯路径中酵母固醇上游的生物标志物—鲨烯、羊毛固醇以及24,25-双脱氢羊毛固醇的含量,而对下游酵母固醇的含量影响微弱(图 5)。因此,现阶段对ERG11表达强度的调整,会显著强化其他固醇物质到酵母固醇的转化。在后续的研究中,还可以在过表达模块ERG11的同时,尝试过表达模块ERG24-25-26-27或ERG2-3,以此增强整体后鲨烯路径的代谢流,以达到提高7-DHC产量的目的。
3 结论本研究将后鲨烯路径进行分割并模块化,分别在所构建的人工7-DHC合成酵母中进行过表达。通过GC-TOF/MS分析并结合PCA分析,发现不同后鲨烯路径模块的过表达,会不同程度地改变后鲨烯路径中各反应步骤代谢流的大小,表现为相关固醇类代谢物含量上的差异,并最终影响7-DHC的合成。其中,对模块ERG11表达的优化,主要增强后鲨烯路径中酵母固醇上游的代谢流;而对模块ERG2-3的表达优化,则显著强化整体后鲨烯路径代谢流,从而提高7-DHC的产量。该产量为迄今所报道的摇瓶水平的最高产量。因此,ERG11和ERG2-3是后续优化人工7-DHC合成酵母的潜在靶标。而本文为在酵母中异源表达甾体类化合物,进行后鲨烯路径与异源模块的适配,提供了一个可供借鉴的示例。
| [1] | Zhou K,Qiao K,Edgar S,et al. Distributing a metabolic pathway among a microbial consortium enhances production of natural products. Nat Biotechnol,2015,33(4):377-383. |
| [2] | Xie W,Lv X,Ye L,et al. Construction of lycopene-overproducing Saccharomyces cerevisiae by combining directed evolution and metabolic engineering. Metab Eng,2015,30:69-78. |
| [3] | Paddon C J,Keasling J D. Semi-synthetic artemisinin: a model for the use of synthetic biology in pharmaceutical development. Nat Rev Microbiol,2014,12(5):355-367. |
| [4] | Szczebara F M,Chandelier C,Villeret C,et al. Total biosynthesis of hydrocortisone from a simple carbon source in yeast. Nat Biotechnol,2003,21(2):143-149. |
| [5] | Duport C,Spagnoli R,Degryse E,et al. Self-sufficient biosynthesis of pregnenolone and progesterone in engineered yeast. Nat Biotechnol,1998,16(2):186-189. |
| [6] | Parks L W,Casey W M. Physiological implications of sterol biosynthesis in yeast. Annu Rev Microbiol,1995,49:95-116. |
| [7] | Veen M,Stahl U,Lang C. Combined overexpression of genes of the ergosterol biosynthetic pathway leads to accumulation of sterols in Saccharomyces cerevisiae. FEMS Yeast Res,2003,4(1):87-95. |
| [8] | Polakowski T,Stahl U,Lang C. Overexpression of a cytosolic hydroxymethylglutaryl-CoA reductase leads to squalene accumulation in yeast. Appl Microbiol Biotechnol,1998,49(1):66-71. |
| [9] | Hull C M,Loveridge E J,Rolley N J,et al. Co-production of ethanol and squalene using a Saccharomyces cerevisiae ERG1 (squalene epoxidase) mutant and agro-industrial feedstock. Biotechnol Biofuels,2014,7(1):133. |
| [10] | Milla P,Athenstaedt K,Viola F,et al. Yeast oxidosqualene cyclase (Erg7p) is a major component of lipid particles. J Biol Chem,2002,277(4):2406-2412. |
| [11] | Martel C M,Parker J E,Warrilow A G,et al. Complementation of a Saccharomyces cerevisiae ERG11/CYP51 (sterol 14alpha-demethylase) doxycycline-regulated mutant and screening of the azole sensitivity of Aspergillus fumigatus isoenzymes CYP51A and CYP51B. Antimicrob Agents Chemother,2010,54(11):4920-4923. |
| [12] | Shah-Alam-Bhuiyan M,Eckstein J,Barbuch R,et al. Synthetically lethal interactions involving loss of the yeast ERG24: the sterol C-14 reductase gene. Lipids,2007,42(1):69-76. |
| [13] | Gachotte D,Sen S E,Eckstein J,et al. Characterization of the Saccharomyces cerevisiae ERG27 gene encoding the 3-keto reductase involved in C-4 sterol demethylation. Proc Natl Acad Sci USA,1999,96(22):12655-12660. |
| [14] | Baudry K,Swain E,Rahier A,et al. The effect of the erg26-1 mutation on the regulation of lipid metabolism in Saccharomyces cerevisiae. J Biol Chem,2001,276(16):12702-12711. |
| [15] | Nose H,Miyara T,Kushida N,et al. Isolation of temperature-sensitive Saccharomyces cerevisiae with a mutation in erg25 for C-4 sterol methyl oxidase. J Antibiot (Tokyo),2002,55(11):962-968. |
| [16] | Kaneshiro E S,Johnston L Q,Nkinin S W,et al. Sterols of Saccharomyces cerevisiae erg6 knockout mutant expressing the Pneumocystis carinii S-adenosylmethionine: sterol C-24 methyltransferase. J Eukaryot Microbiol,2015,62(3):298-306. |
| [17] | Ruan B,Lai P S,Yeh C W,et al. Alternative pathways of sterol synthesis in yeast. Use of C(27) sterol tracers to study aberrant double-bond migrations and evaluate their relative importance. Steroids,2002,67(13-14):1109-1119. |
| [18] | Morio F,Pagniez F,Lacroix C,et al. Amino acid substitutions in the Candida albicans sterol Delta5,6-desaturase (Erg3p) confer azole resistance:characterization of two novel mutants with impaired virulence. J Antimicrob Chemother,2012,67(9):2131-2138. |
| [19] | Zweytick D,Hrastnik C,Kohlwein S D,et al. Biochemical characterization and subcellular localization of the sterol C-24(28) reductase,erg4p,from the yeast Saccharomyces cerevisiae. FEBS Lett,2000,470(1):83-87. |
| [20] | Carlberg C. Genome-wide (over)view on the actions of vitamin D. Front Physiol,2014,5:167. |
| [21] | Lehmann U,Hirche F,Stangl G I,et al. Bioavailability of vitamin D(2) and D(3) in healthy volunteers,a randomized placebo-controlled trial. J Clin Endocrinol Metab,2013,98(11):4339-4345. |
| [22] | Keasling J D. Manufacturing molecules through metabolic engineering. Science,2010,330(6009):1355-1358. |
| [23] | Dahl R H,Zhang F,Alonso-Gutierrez J,et al. Engineering dynamic pathway regulation using stress-response promoters. Nat Biotechnol,2013,31(11):1039-1046. |
| [24] | Su W,Xiao W H,Wang Y,et al. Alleviating redox imbalance enhances 7-Dehydrocholesterol production in engineered Saccharomyces cerevisiae. PloS One,2015,10(6):e0130840. |
| [25] | Gray G W,McDonnell D G. Synthesis and liquid crystal properties of chiral alkyl-cyano-biphenyls (and -p-terphenyls) and of some related chiral compounds derived from biphenyl. Mol Cryst Liq Cryst,1976,37(1):189-211. |
| [26] | Confalone P N,Kulesha I D,Uskokovic M R. A new synthesis of 7-dehydrocholesterol. J Org Chem,1981,46(5):1030-1032. |
| [27] | Bell M C,Schmidt-Grimminger D C,Connor M G,et al. A cervical teratoma with invasive squamous cell carcinoma in an HIV-infected patient: a case report. Gynecol Oncol,1996,60(3):475-479. |
| [28] | Lian J,Si T,Nair N U,et al. Design and construction of acetyl-CoA overproducing Saccharomyces cerevisiae strains. Metab Eng,2014,24:139-149. |
| [29] | Lang C,Markus V. Preparation of 7-dehydrocholesterol and/or the biosynthetic intermediates and/or secondary products thereof in transgenic oganisms. US Patent,12607017,2011-07-23. |
| [30] | Hohmann H P,Lehmann M,Merkamm M. Production of non-yeast sterols by yeast. US Patent,20120231495,2012-09-13. |
| [31] | Lin Q,Jia B,Mitchell L A,et al. RADOM,an efficient in vivo method for assembling designed DNA fragments up to 10 kb long in Saccharomyces cerevisiae. ACS Synth Biol,2015,4(3):213-220. |
| [32] | Gietz R D,Schiestl R H. High-efficiency yeast transformation using the LiAc/SS carrier DNA/PEG method. Nat Protoc,2007,2(1):31-34. |
| [33] | Acimovic J,Rozman D. Steroidal triterpenes of cholesterol synthesis. Molecules,2013,18(4):4002-4017. |
| [34] | Zerenturk E J,Sharpe L J,Ikonen E,et al. Desmosterol and DHCR24: unexpected new directions for a terminal step in cholesterol synthesis. Prog Lipid Res,2013,52(4):666-680. |
 2016, Vol. 36
2016, Vol. 36




