文章信息
- 陈敏, 陈慧, 包海, 黄鹏, 王延伟.
- CHEN Min, CHEN Hui, BAO Hai, HUANG Peng, WANG Yan-wei.
- 植物miRNA启动子研究进展
- Advances in the Research of miRNA Promoters in Plants
- 中国生物工程杂志, 2016, 36(5): 125-131
- China Biotechnology, 2016, 36(5): 125-131
- http://dx.doi.org/DOI:10.13523/j.cb.20160518
-
文章历史
- 收稿日期: 2015-12-11
- 修回日期: 2016-01-25
植物通过调控基因在特定组织、发育时期和外界刺激下的差异表达,以满足自身生长的需要和适应复杂多变的外界环境。植物基因表达可在多个水平实施调控,其中,转录水平的调控是植物基因表达调控的重要环节之一,主要受到多种顺式作用元件和反式作用因子(转录因子)的协同调控。植物基因的启动子区域包含多种重要的顺式作用元件,是基因表达调控的关键。启动子可对下游基因进行特异性调控,以实现基因的有序表达,进而满足植物正常生长发育的需求,并帮助植物体抵御外界逆境胁迫。
microRNA(miRNA,微RNA)是近年来发现的一类短链非编码小分子RNA,目前,在多种生物体中通过克隆或生物信息学预测的方法均发现了miRNA的存在[1]。miRNA的鉴定及其功能研究已成为近年来研究的前沿和热点,在植物中,其首先在拟南芥[2]中得以鉴别,随后在水稻[3]、小麦[4]、大豆[5]、苜蓿[6]和杨树[7]等多种植物中都开展了研究。miRNA作为一类广泛存在的调控分子,通过与靶mRNA完全或部分互补配对,来介导靶mRNA的切割或翻译抑制,从而在调控植物形态建成、发育进程和环境适应性方面扮演着重要的角色[8]。迄今,在部分植物中已开展了miRNA启动子的研究,鉴定了植物中的一批miRNA启动子,揭示出其调控元件的保守性和多样性。因此,有必要对植物miRNA启动子研究的进展作一综述,以加深人们对miRNA基因的转录调控和表达特征的认识,并有助于进一步探索miRNA调控植物基因表达的分子机制。
1 miRNA的合成及其表达特点 1.1 miRNA的合成特点随着植物基因组中一系列非编码RNA的发现,具有调控功能的RNA日益受到研究的重视。根据RNA转录本的长度,非编码RNA通常被分为两类:长链非编码RNA(long non-coding RNA,>200nt)和非编码小分子RNA(small non-coding RNA,<50nt),包括siRNA、miRNA及其它种类的小分子RNA。miRNA基因在核内由RNA Polymerase II(Pol II)转录形成miRNA primary precursor(pri-miRNA)的转录物,其自身可回折形成不完全匹配的茎环结构,这是miRNA区别于其它RNA分子的重要结构特征。pri-miRNA在核内由DCL-1加工成pre-miRNA,在植物中其长度一般为64~303nt[9],pre-miRNA进一步被DCL-1加工成miRNA:miRNA*双链复合体。其后,双链复合体被转运出核,在细胞质中形成成熟的miRNA,进一步整合进RNA诱导的沉默复合物,并进行miRNA和相应靶mRNA之间的互补配对(图 1)。如果靶mRNA与miRNA之间匹配完全,则miRNA能够诱导mRNA的专一性切割;如果靶mRNA与miRNA之间匹配松散,则miRNA抑制mRNA的翻译[10-11]。随着植物中发现的miRNA数目不断增多,越来越多的证据表明miRNA对其目标基因的调节是植物正常发育所必需的[12]。
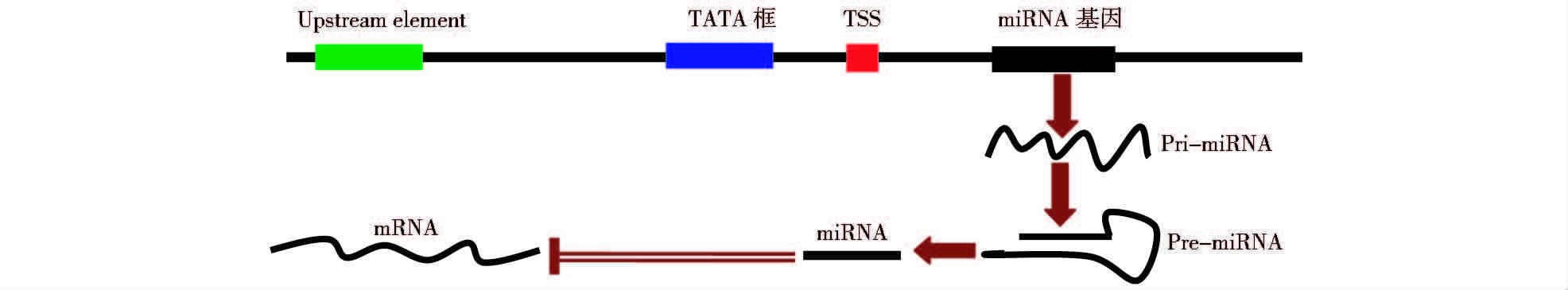
|
| 图 1 miRNA启动子元件及作用机制 Figure 1 The components of miRNA promoter and the regulation mechanism |
miRNA的表达通常具有时序性和组织特异性,研究显示,miRNA在植物的生长发育、形态建成及逆境应答反应中起着多种作用[13-16]。拟南芥dcl突变体表征表现为分裂组织的过度增殖、花的时序发生延迟等发育异常。对miRNA发挥作用必需的其它基因如ago-1进行突变,同样发现了类似dcl突变的表征及其它的一些形态变化,如叶子的极性改变、花分生组织的过度增殖等现象[17]。此外,其它与miRNA相关的基因突变体,如henl和hy11同样可以引起与dcl类似的表型[18]。
miRNA在调控植物对胁迫的反应中也发挥着重要作用。在植物中,首先是在拟南芥中发现了对逆境应答的miRNA[19]。miR393经低温、干旱、盐或激素(ABA)处理后表达显著上调,miR319在低温胁迫后表达上调,但不对于旱、NaCl 响应;miR389a在以上胁迫后下调[14]。受低温诱导的miRNA有miR393、miR397和miR402[14]、miR165/miR166、miR169和miR172[20]。研究发现,对低温响应的miRNA的启动子区存在着与胁迫相关的顺式作用元件。其中,占比例最高的是W-box。已知W-box是WRKY(TF)结合的必需元件,暗示WRKY可能在诱导miRNA的表达中起着重要作用[20]。在对拟南芥的研究中发现,miR399的表达受培养基中磷供应水平的调控[16, 21]。最近,在杨树中报道了19个miRNA成员对低温胁迫响应[22]。miR169g是miR169家族中唯一证实受干旱诱导的基因,在根中的诱导要比地上部分显著[23]。干旱胁迫6h后,miR169g在根中的表达最高,而在地上部分的表达仅在胁迫24h后达到最高。而且,在miR169g的启动子区鉴定到两个对干旱胁迫响应的元件,这与水稻中其前体对干旱胁迫的响应是一致的[23]。
病原物,如细菌、真菌、病毒及线虫,可对植物的正常生长产生严重的伤害。为了对抗病原物的损害,植物中可激发相应的防御机制。最近的研究发现,miRNA介导的转录后调控通过靶向转运抑制物反应1(TIR1)(一个生长素受体),从而在植物防御病原物反应中起着重要作用[24]。用病原菌接种拟南芥后,miR393的表达上调[25]。TIR1是miR393的靶基因。过表达miR393a的转基因拟南芥对病菌的抗性增强,miR393呈高水平表达而TIR1的表达很低。在林木中,开展了病菌侵染松树后miRNA响应的研究,研究者以松树树干木质部为材料克隆到了26个miRNA,发现miRNA在树瘤发育中起着重要的调控作用[26]。
2 miRNA启动子特点与蛋白质编码基因类似,植物miRNA基因的启动子也位于其结构基因5′上游区域,是活化RNA聚合酶、与转录因子特异结合、调控基因转录起始的一段DNA序列[27]。植物启动子由核心启动元件(core promoter)和上游顺式作用元件(upstream cis-acting elements)构成,其中核心启动元件包括转录起始位点(transcription start site,TSS,标记为+1)和上游约-30bp处的TATA框(Hogness box,一段富含AT碱基的DNA序列[28]);上游顺式作用元件一般位于上游-80~-220bp处,分为一般上游启动元件(general upstream element)和特异上游启动元件(specific upstream elements)[29](图 1)。转录因子TFⅡD通过与TATA框的识别结合来介导Pol II识别转录起始位点,形成转录前复合物,并且与其它转录因子和相关蛋白质作用而启始转录[30]。
3 miRNA启动子的鉴定方法目前,研究者通常应用生物信息学的方法并结合基因组数据库进行启动子的预测。鉴定miRNA启动子的第一个步骤是利用基因组序列找到并下载相应植物miRNA的基因序列,其后是鉴定miRNA的TSS。一般认为,启动子区域位于TSS上游序列,然而在不同研究中,研究者选取了TSS上游不同长度的序列进行启动子的生物信息学预测[31]。最初,在水稻中采用5′RACE(rapid amplification of cDNA ends)的方法鉴定到52个miRNA基因的TSS,此外,还利用BioProspector[32]分析了TSS上游-20到-50的TATA框的分布规律,以解释不同miRNA的转录调控的差异性[33]。
在拟南芥中,利用数据库AGRIS(Arabidopsis gene regulatory information server)和AtProbe(Arabidopsis thaliana promoter binding element database)对其miRNA的启动子进行了预测,该研究首先选取了TSS -5 000到+5 000的区域进行初步预测,其后将范围缩小至TSS -800区域,鉴定到了顺式作用元件富集的DNA序列[34]。Devi等[35]鉴定水稻非生物胁迫响应的miRNA启动子时,先从RAP-DB (http://rapdb.dna.affrc.go.jp)下载miRNA基因TSS上游2kb的序列,再用TSSP数据库(http://linux1.softberry.com)对启动子进行预测。类似地,Zeng等探索了大豆中与低磷胁迫相关miRNA的启动子[36],首先从大豆基因组(http://www.phytozome.net)[37]中下载了24个miRNA的TSS上游2kb的序列,再利用TSSP预测TSS和TATA框[36]。Han等[38]进一步利用降解组文库对大豆miRNA的启动子进行了全面的预测和分析。整体来说,目前对于miRNA基因启动子的鉴定主要依靠生物信息学的预测,研究者根据现有植物启动子的结果,整理形成启动子序列和功能的数据库,通过与数据库比对,研究者可找到同源的启动子元件序列。
4 植物中miRNA启动子的研究在拟南芥中采用5′RACE的方法鉴定了52个miRNA基因(如MIR156,MIR157、MIR159、MIR160、MIR166、MIR169和MIR172等)的核心启动元件、TSS和TATA框,为分析植物miRNA基因的差异表达提供了分子基础[33]。其后,研究者进一步探索了拟南芥的63个miRNA启动子,并鉴定了与4个转录因子(AtMYC2、ARF、SORLREP3和LFY)结合的启动子元件序列,初步鉴定了miRNA偏好的启动子序列,显示出miRNA不同于蛋白质编码基因的转录特性[34]。Zhao等[39]采用ChIP(chromatin immunoprecipitation)方法在拟南芥中预测了167个miRNA基因的启动子,并与2 000个蛋白质编码基因的启动子序列和2 000个随机的基因组序列的上游1 000bp进行了比较,对miRNA基因的启动子和蛋白编码基因的启动子进行了平均自由能(average free energy,AFE)分析。
近期报道了水稻miRNA基因启动子的研究。研究者系统地探索了水稻在低温、H2O2、重金属、干旱、ABA和盐胁迫下差异表达的miRNA的启动子,分析了不同环境胁迫下miRNA基因启动子区域上TFBM(transcription factor binding motif)的特点,将特异性表达的miRNA、TFBM与特定的非生物胁迫联系起来[40]。另外,研究者[41]对比分析了拟南芥62个miRNA和水稻55个miRNA的启动子,讨论了两个物种中TSS的AFE并进行了PWM (position weight matrices)分析,进而在拟南芥和水稻中分别找到了4个(AtMYC2、G-box、AtMYB2 和LTRE)和10个(LFY、RAV1-A、CAAT-box、MYB4、W-box、GCC-box等)富集的启动子元件,为研究植物miRNA的差异表达提供了新的线索[41]。
除了拟南芥、水稻等模式植物之外,在木本植物中近年来也开展了miRNA启动子的研究。例如,在橡胶树中[42],研究者利用PlantCARE数据库(http://bioinformatics.psb.ugent.be/webtools/plantcare/html)对乙烯响应的miRNA(miR2118、miR159、miR164和miR166)启动子区域的顺式作用元件进行了预测,鉴定了一些激素响应的元件,如ERE、AuxRR-core、AuxRR-core、GARE-motif和P-box等,有助于进一步理解和探索miRNA转录表达的差异性。
5 miRNA启动子顺式作用元件鉴定顺式作用元件,又称为转录因子结合域(TFBM)。目前,研究者寻找启动子区域顺式作用元件的方法一般是将植物miRNA基因的启动子序列与已知数据库中的顺式元件进行同源比对,找出保守的启动子元件。PLACE(http://www.dna.affrc.go.jp/htdocs/PLACE)和PlantCARE是2个关于植物基因顺式作用元件序列的数据库[43-44],包括顺式作用元件(cis-acting elements)、增强子(enhancer)和阻遏因子(inhibitor)等。在水稻中,研究者利用PLACE鉴定已知的顺式作用元件,并用MELINA-II(http://melina2.hgc.jp/public/index.html)[45]来比较序列预测的算法。在大豆中利用PlantCARE数据库[46]来分析大豆miRNA基因的顺式作用元件。另外,对于预测到的新特异性启动元件,通常用MELINA II分析DNA序列,找到一些具有显著性特征的元件。
在拟南芥中,除了一些通用启动子元件(如TATA框、AtMYC2、ARF和SORLREP3)之外,研究者还鉴定了一些新的特殊的上游顺式作用元件,如CArG、CCA1、Ibox、GCC-box、T-box、LFY motif、Bellringer BS3和RAV1-B等。这些结果为进一步揭示植物miRNA基因表达调控的分子机制提供了重要线索。在水稻中,研究者鉴定了16个与环境胁迫相关的启动子元件,包括9个已知的顺式作用元件(MYC recognition site、MYB recognition site、WRKY71OS和ASF1MOTIFCAMV等)、2个DNA域(CGCCGCCG、CGGCGGCG)和5个特异性的元件[40]。这些结果进一步丰富了人们对水稻胁迫相关的miRNA启动子的了解,为研究水稻对环境胁迫的响应和miRNA的表达特异性提供了新的依据。
Zhou等[47]利用TSSP-TCM软件系统分析了4种植物(包括拟南芥、毛果杨、水稻和高粱)miRNA基因启动子的结构,比较了基因间和基因内miRNA;高保守性、低保守性及非保守性miRNA启动子的区别,鉴定了6个物种特异性的启动子,如在拟南芥中富集的启动序列(GGCCTGAGCC和TTAGGGTTTC)、毛果杨中的GCAGCAGAAG和CGGGTCAAAC、高粱中的GCATGCATGC和GAACTAAACA,为进一步理解植物miRNA的表达提供了新的视角[47]。
另外,通过对miRNA基因及其启动子序列的分析,可为理解通过重复和插入而产生新miRNA提供线索。在拟南芥中,研究者利用MIPS A.thaliana database (MAtDB)[48]对miRNA基因(MIR157、MIR158、MIR163、MIR165、MIR405和MIR447)上游1 000bp序列进行了分析,再利用DIALIGN和Motif Sampler对这6个miRNA家族各个成员的序列进行了比对和相似性分析,进一步证明了miRNA基因的插入重复进化模型[49]。
Han等用生物信息学的方法系统预测了大豆中440个miRNA的核心启动元件(TSS和TATA框),并分析了369个miRNA的顺式作用元件,如LRE(light responsive element)、SREs(stress responsive element)、GRE(cis-acting element regulating plant growth)、HRE(hormone-regulated cis-acting element)和TSE(tissue-specific cis-acting element)等[38]。此外,在大豆中发现了由miRNA的靶标蛋白(转录因子)、ARF(auxin response factor)和GARF(gibberellin response factor)、顺式作用元件(ARF recognition motif和gibberellin(GA) response (GAR) elements)和miRNA基因(MIR160d/f、MIR167a/g)构成的负反馈调控环路,即miRNA的靶基因是编码与自身启动子的顺式作用元件结合的转录因子,那么,miRNA可以通过切割降解其靶基因来影响自身启动元件的激活或抑制,进而调控自身的转录[38]。这些结果说明转录因子结合顺式作用元件是调控基因表达的一个重要方式,进一步证实了启动子对miRNA表达的调控作用。
6 结 语近年来,在多种植物中已开展了miRNA启动子的研究,然而仍存在许多问题。首先,迄今报道的植物miRNA启动子的研究大部分是生物信息学预测的结果,未进行实验验证和功能求证,因而多数miRNA启动子元件功能及作用机制并不十分确切,需要后续进行miRNA基因启动子和miRNA表达的关联分析及实验探索。再者,除了一般启动子元件之外,植物miRNA特有的新型启动子序列和功能尚有待发现。而且,植物基因表达受到严格的调控,启动子是转录水平调控的关键环节之一,现在对启动子的研究和调控大都集中在转录因子对顺式作用元件的结合调控,然而,研究者对miRNA启动元件受到的其它方面的调控仍然知之甚少,如miRNA启动子在表观遗传学上的调控(甲基化、糖基化、DNA序列折叠等)。
植物启动子的研究还存在着许多空白,需要研究者进一步深入探索。随着生物信息学预测手段和分子生物学实验技术的不断完善,更多植物启动子及其功能将会逐渐明确。miRNA对植物的发育和逆境胁迫的应答起着重要调控作用,鉴定植物miRNA的启动子将有助于人们深入理解miRNA调控植物基因表达的分子机制。随着小RNA测序技术的广泛应用和测序深度的不断提高,植物中鉴定到的miRNA的数目将会日益增多。系统鉴定这些miRNA的启动子,将会大大加速人们对miRNA差异表达分子机制的理解,其序列中特异调控元件的鉴定也将为基因工程改良植物性状提供新的分子元件。
| [1] | Bartel D P. MicroRNAs: genomics, biogenesis, mechanism, and function. Cell,2004, 116 (2) : 281 –297. |
| [2] | German M A, Pillay M, Jeong D H, et al. Global identification of microRNA-target RNA pairs by parallel analysis of RNA ends. Nature Biotechnology,2008, 26 (8) : 941 –946. |
| [3] | Lu T, Cui L, Zhou Y, et al. Transcriptome-wide investigation of circular RNAs in rice. RNA,2015, 21 (12) : 2076 –2087. |
| [4] | Han R, Jian C, Lv J, et al. Identification and characterization of microRNAs in the flag leaf and developing seed of wheat (Triticum aestivum L.).. BMC Genomics,2014, 15 : 289 . |
| [5] | Barros-Carvalho G A, Paschoal A R, Marcelino-Guimaraes F C, et al. Prediction of potential novel microRNAs in soybean when in symbiosis. Genetics and Molecular Research,2014, 13 (4) : 8519 –8529. |
| [6] | Long R C, Li M N, Kang J M, et al. Small RNA deep sequencing identifies novel and salt-stress-regulated microRNAs from roots of Medicago sativa and Medicago truncatula. Physiologia Plantarum,2015, 154 (1) : 13 –27. |
| [7] | Ren Y, Sun F, Hou J, et al. Differential profiling analysis of miRNAs reveals a regulatory role in low N stress response of Populus. Functional & Integrative Genomics,2015, 15 (1) : 93 –105. |
| [8] | Chen L N, Dong H P, Huang K X, et al. Transcriptomic analyses of nitrogen assimilation processes in a Chinese strain of Aureococcus anophagefferens. Genomics Data,2015, 5 : 344 –345. |
| [9] | Lee Y, Ahn C, Han J, et al. The nuclear RNase III Drosha initiates microRNA processing. Nature,2003, 425 (6956) : 415 –419. |
| [10] | Zeng Y, Wagner E J, Cullen B R. Both natural and designed microRNAs can inhibit the expression of cognate mRNAs when expressed in human cells. Molecular Cell,2002, 9 (6) : 1327 –1333. |
| [11] | Zeng Y, Yi R, Cullen B R. MicroRNAs and small interfering RNAs can inhibit mRNA expression by similar mechanisms. Proceedings of the National Academy of Sciences of the United States of America,2003, 100 (17) : 9779 –9784. |
| [12] | Carrington J C, Ambros V. Role of microRNAs in plant and animal development. Science,2003, 301 (5631) : 336 –338. |
| [13] | Hirsch J, Lefort V, Vankersschaver M, et al. Characterization of 43 non-protein-coding mRNA genes in Arabidopsis, including the MIR162a-derived transcripts. Plant Physiology,2006, 140 (4) : 1192 –1204. |
| [14] | Sunkar R, Zhu J K. Novel and stress-regulated microRNAs and other small RNAs from Arabidopsis. The Plant Cell,2004, 16 (8) : 2001 –2019. |
| [15] | Wang X J, Reyes J L, Chua N H, et al. Prediction and identification of Arabidopsis thaliana microRNAs and their mRNA targets. Genome Biology,2004, 5 (9) : R65 . |
| [16] | Chiou T J, Aung K, Lin S I, et al. Regulation of phosphate homeostasis by MicroRNA in Arabidopsis. The Plant Cell,2006, 18 (2) : 412 –421. |
| [17] | Schauer S E, Jacobsen S E, Meinke D W, et al. DICER-LIKE1: blind men and elephants in Arabidopsis development. Trends in Plant Science,2002, 7 (11) : 487 –491. |
| [18] | Chen S, Lesnik E A, Hall T A, et al. A bioinformatics based approach to discover small RNA genes in the Escherichia coli genome. Biosystems,2002, 65 (2-3) : 157 –177. |
| [19] | Jones-Rhoades M W, Bartel D P. Computational identification of plant microRNAs and their targets, including a stress-induced miRNA. Molecular Cell,2004, 14 (6) : 787 –799. |
| [20] | Zhou X, Wang G, Sutoh K, et al. Identification of cold-inducible microRNAs in plants by transcriptome analysis. Biochimica et Biophysica Acta (BBA)-Gene Regulatory Mechanisms,2008, 1779 (11) : 780 –788. |
| [21] | Fujii H, Chiou T J, Lin S I, et al. A miRNA involved in phosphate-starvation response in Arabidopsis. Current Biology,2005, 15 (22) : 2038 –2043. |
| [22] | Lu S, Sun Y H, Chiang V L. Stress-responsive microRNAs in Populus. The Plant Journal,2008, 55 (1) : 131 –151. |
| [23] | Zhao B, Liang R, Ge L, et al. Identification of drought-induced microRNAs in rice. Biochemical and Biophysical Research Communications,2007, 354 (2) : 585 –590. |
| [24] | Navarro L, Dunoyer P, Jay F, et al. A plant miRNA contributes to antibacterial resistance by repressing auxin signaling. Science,2006, 312 (5772) : 436 –439. |
| [25] | Fahlgren N, Howell M D, Kasschau K D, et al. High-throughput sequencing of Arabidopsis microRNAs: evidence for frequent birth and death of MIRNA genes. PLoS One,2007, 2 (2) : e219 . |
| [26] | Lu S, Sun Y H, Amerson H, et al. MicroRNAs in loblolly pine (Pinus taeda L.) and their association with fusiform rust gall development. The Plant Journal,2007, 51 (6) : 1077 –1098. |
| [27] | Usha S, Jyothi M N, Sharadamma N, et al. Identification of microRNAs and their targets in Finger millet by high throughput sequencing. Gene,2015, 574 (2) : 210 –216. |
| [28] | Morton T, Petricka J, Corcoran D L, et al. Paired-end analysis of transcription start sites in Arabidopsis reveals plant-specific promoter signatures. The Plant Cell,2014, 26 (7) : 2746 –2760. |
| [29] | Hieno A, Naznin H A, Hyakumachi M, et al. ppdb: plant promoter database version 3.0. Nucleic Acids Research,2014, 42 (Database issue) : D1188 –D1192. |
| [30] | Yamamoto Y Y, Yoshitsugu T, Sakurai T, et al. Heterogeneity of Arabidopsis core promoters revealed by high-density TSS analysis. The Plant Journal,2009, 60 (2) : 350 –362. |
| [31] | Kumari S, Ware D. Genome-wide computational prediction and analysis of core promoter elements across plant monocots and dicots. PLoS One,2013, 8 (10) : e79011 . |
| [32] | Liu Y, Wei L, Batzoglou S, et al. A suite of web-based programs to search for transcriptional regulatory motifs. Nucleic Acids Research,2004, 32 (Web Server issue) : W204 –W207. |
| [33] | Xie Z, Allen E, Fahlgren N, et al. Expression of Arabidopsis MIRNA genes. Plant Physiology,2005, 138 (4) : 2145 –2154. |
| [34] | Megraw M, Baev V, Rusinov V, et al. MicroRNA promoter element discovery in Arabidopsis. RNA,2006, 12 (9) : 1612 –1619. |
| [35] | Devi S J, Madhav M S, Kumar G R, et al. Identification of abiotic stress miRNA transcription factor binding motifs (TFBMs) in rice. Gene,2013, 531 (1) : 15 –22. |
| [36] | Zeng H Q, Zhu Y Y, Huang S Q, et al. Analysis of phosphorus-deficient responsive miRNAs and cis-elements from soybean (Glycine max L.). Journal of Plant Physiology,2010, 167 (15) : 1289 –1297. |
| [37] | Goodstein D M, Shu S, Howson R, et al. Phytozome: a comparative platform for green plant genomics. Nucleic Acids Research,2012, 40 (Database issue) : D1178 –D1186. |
| [38] | Han Y Q, Hu Z, Zheng D F, et al. Analysis of promoters of microRNAs from a Glycine max degradome library. Journal of Zhejiang University Science B,2014, 15 (2) : 125 –132. |
| [39] | Zhao X, Zhang H, Li L. Identification and analysis of the proximal promoters of microRNA genes in Arabidopsis. Genomics,2013, 101 (3) : 187 –194. |
| [40] | Devi S J, Madhav M S, Kumar G R, et al. Identification of abiotic stress miRNA transcription factor binding motifs (TFBMs) in rice. Gene,2013, 531 (1) : 15 –22. |
| [41] | Zhao X, Li L. Comparative analysis of microRNA promoters in Arabidopsis and rice. Genomics,. Proteomics & Bioinformatics,2013, 11 (1) : 56 –60. |
| [42] | Kanjanawattanawong S, Tangphatsornruang S, Triwitayakorn K, et al. Characterization of rubber tree microRNA in phytohormone response using large genomic DNA libraries, promoter sequence and gene expression analysis. Molecular Genetics and Genomics,2014, 289 (5) : 921 –933. |
| [43] | Higo K, Ugawa Y, Iwamoto M, et al. PLACE: a database of plant cis-acting regulatory DNA elements. Nucleic Acids Research,1998, 26 (1) : 358 –359. |
| [44] | Rombauts S, Dehais P, Van Montagu M, et al. PlantCARE, a plant cis-acting regulatory element database. Nucleic Acids Research,1999, 27 (1) : 295 –296. |
| [45] | Okumura T, Makiguchi H, Makita Y, et al. Melina II: a web tool for comparisons among several predictive algorithms to find potential motifs from promoter regions. Nucleic Acids Research,2007, 35 (Web Server issue) : W227 –W231. |
| [46] | Lescot M, Dehais P, Thijs G, et al. PlantCARE, a database of plant cis-acting regulatory elements and a portal to tools for in silico analysis of promoter sequences. Nucleic Acids Research,2002, 30 (1) : 325 –327. |
| [47] | Zhou M, Sun J, Wang Q H, et al. Genome-wide analysis of clustering patterns and flanking characteristics for plant microRNA genes. FEBS Journal,2011, 278 (6) : 929 –940. |
| [48] | Schoof H, Ernst R, Nazarov V, et al. MIPS Arabidopsis thaliana Database (MAtDB): an integrated biological knowledge resource for plant genomics. Nucleic Acids Research,2004, 32 (Database issue) : D373 –D376. |
| [49] | Wang Y, Hindemitt T, Mayer K F. Significant sequence similarities in promoters and precursors of Arabidopsis thaliana non-conserved microRNAs. Bioinformatics,2006, 22 (21) : 2585 –2589. |
 2016, Vol. 36
2016, Vol. 36




