文章信息
- 梁栋, 邢永强, 蔡禄
- LIANG Dong, XING Yong-qiang, CAI Lu
- 肾肿瘤相关基因的共表达网络构建与分析
- The Construction and Analysis of the Coexpression Network for the Gene Related to Renal Tumor
- 中国生物工程杂志, 2016, 36(2): 30-37
- China Biotechnology, 2016, 36(2): 30-37
- http://dx.doi.org/10.13523/j.cb.20160205
-
文章历史
- 收稿日期: 2015-11-23
- 修回日期: 2015-11-30
肾肿瘤是泌尿系统常见的肿瘤之一,近年来的发病率趋势呈明显上升,成为威胁人类健康的最重要肿瘤之一[1],在我国,肾部恶性肿瘤已经占到成人恶性肿瘤的2%~3%。肿瘤致病机理涉及多个基因间的相互作用,因此,仅关注单个或某几个基因的研究方法已经不能满足当前的研究需要。近年来,随着基因芯片、RNA-Seq等高通量测序技术的兴起,使得同时研究多个基因表达情况成为可能。目前,针对肾肿瘤基因芯片的研究相对较少,因此,根据基因芯片数据来研究肾肿瘤发生的相关基因具有明显意义。鉴于基因芯片数据的数量较大,本文采用基因共表达网络构建的方法进行研究,从大量基因中筛选肾肿瘤密切相关的枢纽基因。
1 数据和方法 1.1 数 据本文数据下载自NCBI中的GEO数据库(Gene Expression Omnibus,http://www.ncbi.nlm.nih.gov/geo)。GEO数据库包括原始数据和预处理后的数据,由于研究目的等因素的差异,不同研究小组对原始数据的预处理方法并不一致,因此,本文下载并使用原始数据。数据获取的平台编号是GPL97,选取的数据集编号是GSE6280,芯片类型为hgu133b。该数据集来自2009年Yusenko等[2]的工作。从该数据集中选取20个样本,其中14个样本取自肾部肿瘤组织,6个样本取自肾部正常组织(表 1)。
| 正常组织样本 | 肿瘤组织样本 |
| GSM144481 GSM144482 GSM144483 GSM144492 GSM144493 GSM144494 | GSM144484 GSM144485 GSM144486 GSM144487 GSM144488 GSM144489 GSM144490 GSM144491 GSM144495 GSM144496 GSM144497 GSM144498 GSM144499 GSM144450 |
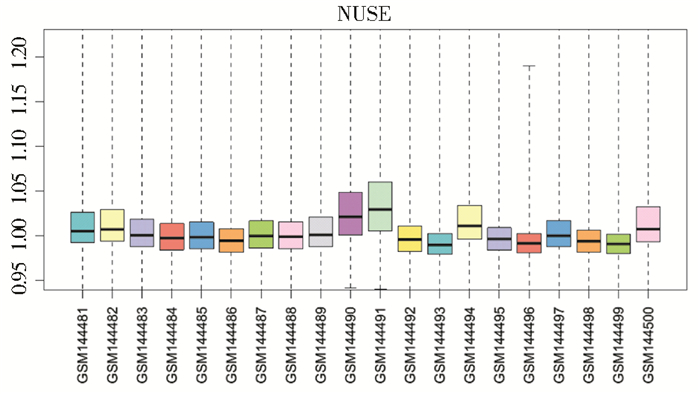
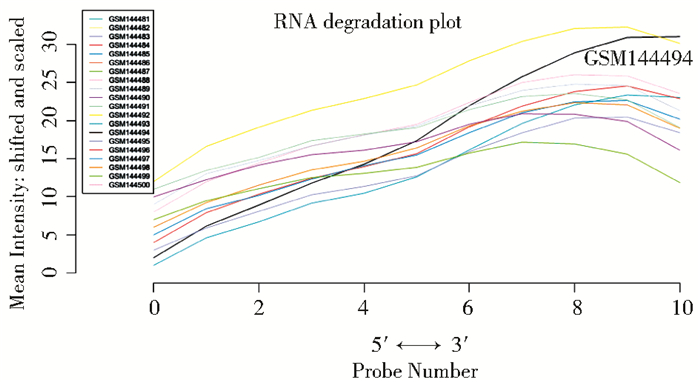
芯片数据预处理的主要流程包括:数据过滤、标准化、对数化。本文通过NUSE箱线图和RNA降解曲线完成数据过滤。NUSE定义为一个探针组在某个样品的PM值(匹配探针)的标准差除以该探针组在各样品中PM值标准差的中位数。若NUSE值在1附近,说明芯片质量比较可靠;若NUSE值超过1.05,则芯片质量就有问题。由于RNA从5′端开始降解,所以理论上5′端荧光强度应当低于3′端荧光强度,利用RNA降解曲线可对降解率进行评估,斜率越小,说明降解程度越小,样本芯片数据越可信。
通过标准化调整由于基因芯片技术引起的误差。对数化主要是对样本基因和参照基因做比值,可在一定程度上减小芯片之间、荧光染色和图像扫描引起的系统误差。通过GCRMA一体化算法可实现数据标准化和对数化,该过程可使用R语言gcrma包完成。
1.2.2 筛选表达差异基因方法首先将预处理后的数据转化为基因表达矩阵,其中行表示基因,列表示各样本中所有基因的表达值。再令正常样本为“0”,肿瘤样本为“1”,构建20维的样本特征(正常/肿瘤)向量,基于样本特征向量对基因表达矩阵中各行的基因表达向量进行线性拟合,对拟合结果进行经验贝叶斯检验。设定相关P值作为阈值筛选表达差异基因。可使用R语言Limma完成该过程。
1.2.3 共表达网络构建方法2005年,Zhang等[3]提出的加权基因共表达网络分析(weighted gene co-expression network analysis,WGCNA)算法,由于其高效准确性,已经广泛用于基因共表达网络的构建,因此,采用WGCNA算法作为网络构建的算法。该网络构建流程见图 1。

|
| 图 1 共表达网络构建流程 Fig. 1 The pipeline of co-expression network construction |
将表达差异基因的表达值从表达矩阵中提取出来作为共表达网络构建的输入数据。网络构建主要包括以下几步:(1)定义基因表达相关矩阵S=[Smn=|cor(m,n)|],Smn 代表基因m和n的表达相关系数;(2)选择加权系数β,将表达相关矩阵转化为邻接矩阵A=[amn=|smn|β],amn代表基因m和n的邻接系数。(3)邻接矩阵转化为拓扑重叠矩阵TOM=TOMmn=lmn+amnmin{km,kn}+1-amn,其中lmn=∑u amuanu,其中u代表同时和基因m与n相邻接的基因集合,km=∑M amM,其中M代表和基因m相邻接的基因集合[4];(4)基因间相异度矩阵dissTOM=1-TOM,对dissTOM层次聚类得到系统聚类树;(5)动态混合切割算法,从系统聚类树中识别网络模块[5];(6)计算各模块本征向量基因(Module eigengene,ME)。ME代表模块的整体表达水平,计算模块ME间的皮尔森相关系数,定义模块ME间的平均距离为1-皮尔森相关系数。采用average-linkage层次聚类法对模块ME进行聚类,合并相似程度较高的模块,得到共表达网络。
1.2.4 筛选共表达网络关键模块和枢纽基因方法采用2种方法筛选网络关键模块。方法一,计算各模块ME与样本特征向量的皮尔森相关系数。方法二,引入基因显著性(Gene significance,GS)反映模块与肿瘤发生的关联[6]。具体做法:计算各基因表达量与样本特征向量的皮尔森相关系数,即为GS;再对模块内的基因求GS绝对值均值,绝对值均值越大,说明基因模块与肿瘤发生越相关。
枢纽基因筛选需引入模块身份概念(module membership,MM),用来衡量基因在模块内的重要性,定义如下:MMM(i)=cor(xi,MEM),表示基因i在模块M中的模块身份,xj代表基因i的表达向量,MMM(i)的绝对值越大,基因i在模块M中越重要[6]。筛选方法主要有两种:(1)对GS和MM设定阈值,一般令GS>0.7(P值<0.01),MM>0.9(P值<0.01),则该基因可识别为枢纽基因;(2)利用WGCNA包中的函数networkScreeing直接完成对枢纽基因的识别[6] 。
2 结 果 2.1 芯片数据预处理获取基因芯片原始数据后,需对原始数据预处理,消除由于实验技术导致的表达量变化。采用NUSE箱线图和RNA降解曲线的数据处理结果分别见图 2和图 3。
|
| 图 2 NUSE箱线图 Fig. 2 NUSE Box plot |
|
| 图 3 RNA降解曲线 Fig. 3 RNA degradation curve The horizontal axis represents fragments of RNA from 5′end to 3′ end,the vertical axis represents the fluorescence intensity |
图 2显示,所有样本的NUSE值均低于1.05,说明通过NUSE箱线图数据过滤方法确认全部样本可用于后续分析,并没有筛查掉不合格样本。图 3显示,正常样本GSM144494的RNA降解曲线斜率明显偏大,说明该样本的RNA降解较为严重,芯片数据质量较差。为保证后续分析计算结果的可信度,综合图 2和图 3 的数据过滤结果,删除正常样本GSM144494,最终获得的有效样本包括5个正常样本和14个肿瘤样本。基于GCRMA一体化算法,使用R语言gcrma包对19个样本的芯片数据进行标准化和对数化。
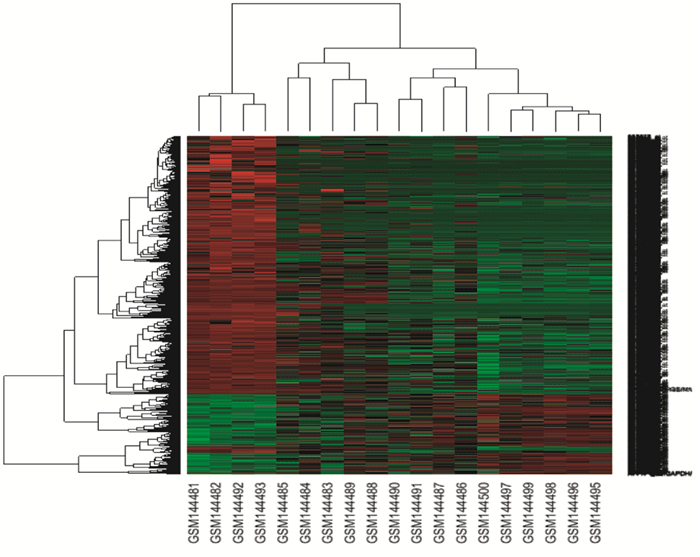
2.2 表达差异基因筛选为获取与肿瘤发生的相关基因,需计算正常样本与肿瘤样本之间的基因表达变化,从而筛选表达差异基因。通过样本特征向量和基因表达向量线性拟合的经验贝叶斯检验方法,从22 645个基因中筛选出表达变化倍数(Fold change)大于2,P值小于0.01的1 180个基因作为表达差异基因。通过分析正常样本和肿瘤样本的表达差异,发现肿瘤样本中有928个基因呈现表达下调,258个基因呈现表达上调。针对1 180个基因进行表达差异聚类分析,分析结果见图 4。可以看出,正常样本与肿瘤样本的表达水平具有明显差异。正常样本GSM144481,GSM144482,GSM144492,GSM144493的多数基因表达上调,而正常样本GSM144483与肿瘤样本的基因表达模式差异相对较小。
|
| 图 4 表达差异基因聚类 Fig. 4 A cluster of differential expression gene Red bars denote genes in which expression is up-regulated. Green bars genes in which expression is down-regulated |
从预处理后的芯片数据中筛选出1 180个表达差异基因,并提取这些基因的表达矩阵,以此矩阵作为输入数据,通过1.2.3 节中的网络构建方法将这些基因分成若干模块,形成所要构建的共表达网络。
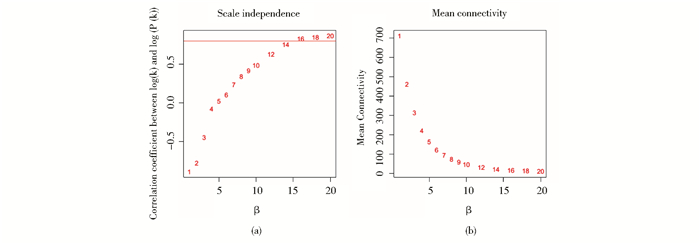
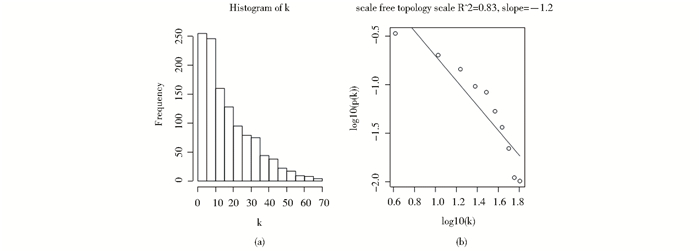
2.3.1加权系数β的选择研究表明共表达网络具有无尺度网络(scale-free network)特征,即P(k)~k-1,k表示节点连接度[7]。因此,根据无尺度网络规则,加权系数β需满足log(k)与log(P(k))呈负相关的条件,且相关系数越大,网络的无尺度特征越显著。本文基于不同β条件下的log(k)与log(P(k))的相关系数进行加权系数选择(图 5)。考虑到计算的复杂性和运行时间,选择相关系数大于0.8时,β=16构建共表达网络较为合理(图 5a)。加权系数与网络的平均连接度关系图显示,当β=16时网络的平均连接度等于16.88103(图 5b)。加权系数β确定后,即可将1 180个表达差异基因的表达矩阵转化为邻接矩阵、拓扑矩阵以及基因间相异度矩阵,并构建表达差异基因的共表达网络关系图。计算网络图中每个基因节点的连接度,得到各基因节点的连接度频率分布特征(图 6a)。β=16时,log(k)与log(P(k))呈线性负相关关系(图 6b)。通过分析图 6a、图 6b可得出结论,当β=16时,共表达网络属于无尺度网络。
|
| 图 5 确定加权系数β Fig. 5 The decision of weighting coefficients β (a)The horizontal axis represents different weighting coefficient β. The vertical axis represents the correlation coefficient between log(k) and log(P(k)). The red line represents that the correlation coefficient is equal to 0.8.(b)The average network connectivity under different weighting coefficients |
|
| 图 6 无尺度网络特性检验 Fig. 6 The test of property of scale-free network (a) The distribution of connection degree k of nodes (b) The correlation coefficient of log(k) and log(P(k)) |
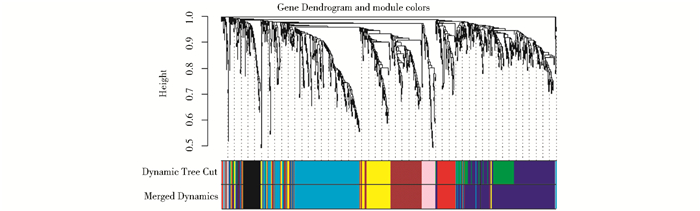
加权系数β确定后,得到1 180个表达差异基因的基因间相异度矩阵dissTOM,对dissTOM进行层次聚类得到1 180个基因的系统聚类树(图 7)。应用动态混合切割算法从该系统聚类树中识别出9个网络模块(图 7)。
|
| 图 7 动态混合剪切算法对基于dissTOM的基因系统聚类树的识别结果 Fig. 7 Identifying result of the system clustering tree by dynamic hybrid algorithm based on dissTOM The top figure represents the system clustering tree based on dissTOM. The under figure of Tree Cut Merged and Merged Dynamic represent network modules before and after merging modules(each color represents one module) |
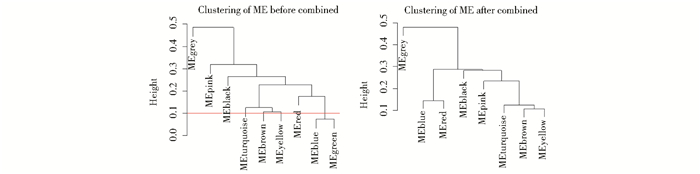
通过求得表示模块整体表达水平的本征向量基因ME,可计算模块ME间的皮尔森相关系数和模块ME间的平均距离。基于该平均距离,采用average-linkage层次聚类法对已识别的9个网络模块进行聚类分析(图 8)。考虑到green模块与blue模块间的相关系数大于0.9,相似程度最高,将green模块合并到blue模块(图 7、图 8)。最终,将1 180个基因划分为8个共表达网络模块。每个共表达网络模块包含的基因数量见表 2。需要强调的是,grey模块是指无法聚集到其他7个模块的基因集合。
|
| 图 8 合并模块前后的ME聚类 Fig. 8 The clustering based on ME before and after merging modules |
| 模块 | black | Blue | Brown | Pink | Red | Turquoise | Yellow | grey |
| 基因数 | 69 | 355 | 172 | 49 | 79 | 308 | 132 | 16 |
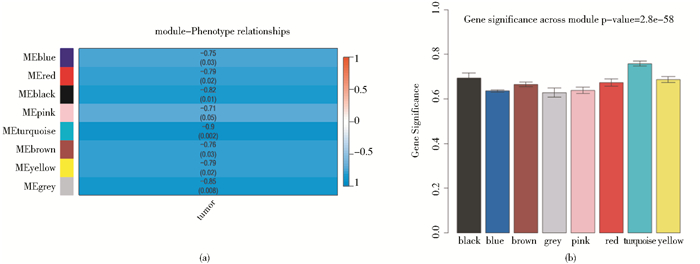
对各模块与样本特征向量进行关联分析,可选出与肿瘤发生显著相关的模块。再从选出的模块中挑选出枢纽基因(hub gene),得到影响肾肿瘤发生的枢纽基因。首先,通过计算网络模块的本征向量基因ME与样本特征向量的皮尔森相关系数,寻找与肿瘤发生显著相关的关键网络模块,计算结果见图 9a。分析图 9a发现:(1)各模块的整体表达水平均与肿瘤发生呈负相关关系,即肿瘤样本的基因表达水平整体呈下调趋势。(2)turquoise模块的整体表达水平与肿瘤发生的负相关系数程度显著高于其他7个模块(相关系数=-0.90,P值=0.002)。为保证关键网络模块识别结果的可靠性,通过计算模块内基因的GS绝对值的方法再次识别肿瘤发生的关键网络模块(图 9b)。可以看出,turquoise网络模块内基因的GS绝对值最大(P值=2.8e-58),即该模块与肿瘤发生的关联程度最大。用上述两种不同的关键模块筛选方法,同时发现turquoise网络模块与肿瘤发生的关联程度最大。因此,枢纽基因的筛选将在turquoise网络模块内进行。
|
| 图 9 各模块与肿瘤发生相关性 Fig. 9 Correlation between each module and tumor occurrence (a) Pearson correlation coefficient between the module eigengene of modules and the sample feature vector. Numbers in rectangular represent the correlation coefficients and numbers in brackets indicate the corresponding P values (b) The distribution of GS absolute values of genes in each module The horizontal axis represents each module and the vertical axis represents the GS value |
要筛选模块内的枢纽基因,需要计算每个基因的模块身份MM值。为检验MM值的大小是否与肿瘤发生有密切关系,需对各模块内基因的GS和MM进行相关性分析,分析结果见图 10。结果表明,turquoise模块内的基因GS与MM相关系数最大,即turquoise模块与肿瘤发生的关联程度最大,与上文结论一致,证明MM值能够反映基因与肿瘤发生的相关性,即基因的MM值越大,基因在模块中越重要,与肿瘤的发生的关联性也越大[8]。
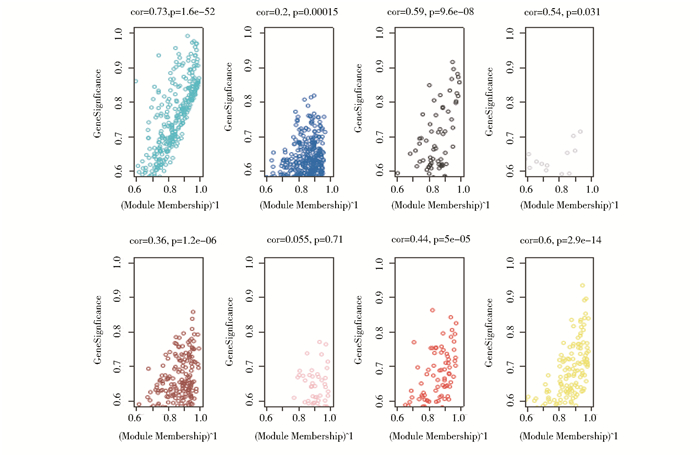
|
| 图 10 各模块内基因的GS与MM相关性散点图 Fig. 10 Scatter diagram of the correlation between GS and MM of all genes in each module |
验证MM识别的枢纽基因的有效性之后,采用2.2.4 节中提到的第1种枢纽基因筛选方法,从turquoise模块内的308个基因中筛选出98 个枢纽基因;采用2.2.4节中提到的第2种枢纽基因筛选方法,从turquoise模块内筛选出30个枢纽基因。考虑到结果的假阳性,选取2种筛选方法获得的共有基因作为有效枢纽基因。比较两种方法后,获得23个枢纽基因。去除其中4 个没有注释的基因,最终得到19个有注释的枢纽基因(表 3)。
| Probe ID | Gene symbols | Entrez ID | Probe ID | Gene symbols | Entrez ID |
| 227662_at | SYNPO2 | 171024 | 233498_at | ERBB4 | 2066 |
| 235746_s_at | PLA2R1 | 22925 | 228507_at | PDE3A | 5139 |
| 236300_at | PDE3A | 5139 | 227361_at | HS3ST3B1 | 9953 |
| 228640_at | PCDH7 | 5099 | 227727_at | MRGPRF | 116535 |
| 223869_at | SOST | 50964 | 228875_at | FAM162B | 221303 |
| 244289_at | ZNF300P1 | 134466 | 225544_at | TBX3 | 6926 |
| 227702_at | CYP4X1 | 260293 | 228827_at | RUNX1T1 | 862 |
| 244313_at | CR1 | 1378 | 222722_at | OGN | 4969 |
| 243792_x_at | PTPN13 | 5783 | 227204_at | PARD6G | 84552 |
| 229529_at | TCF21 | 6943 |
本文通过人类的肾部组织的基因芯片原始实验数据的预处理,得到19个标准化的样本芯片的数据。通过计算正常组织与肿瘤组织的基因表达变化,筛选出1 180个正常组织与肿瘤组织差异表达的基因。应用加权基因共表达网络分析算法对1 180个表达差异基因层次聚类后共识别出8个网络模块。通过分析网络模块与样本本征向量的关联性,确定turquoise网络模块与肿瘤发生的相关性最大。最终,从turquoise网络模块内识别出19个可能对肾肿瘤发生有关键作用的枢纽基因。为进一步解析枢纽基因的生物学功能,使用R语言的GOstats包对19个枢纽基因进行了GO富集分析。结果显示,基因PLA2R1和TBX3的GO富集程度最高,且与细胞衰老相关。细胞衰老是抑制肿瘤发生的主要机制之一。可以推测,枢纽基因PLA2R1和TBX3可能对肾肿瘤的形成具有重要影响。已有研究发现PLA2R1基因的肿瘤抑制作用,与本项理论工作的结论一致。例如,Vindrieux等[9]的研究表明PLA2R1基因有肿瘤抑制的作用。Bernard等[10]认为PLA2R1的表达下调会导致包括肾部肿瘤在内的一系列疾病的发生。GO富集分析仅识别出PLA2R1和TBX3这两个有生物学意义的枢纽基因,其余的17个肾肿瘤枢纽基因将是我们下一步工作的重点研究内容。应用分子生物学实验方法验证枢纽基因对肾肿瘤的调控作用也将是今后工作的重要内容。
致谢 感谢内蒙古科技大学创新基金面上项目(No.2014QDL012)对本研究提供的支持。
| [1] | Mankoff D A,Thompson J A,Gold P,et al. Identification of interleukin-2-induced complete response in metastatic renal cell carcinoma by FDG PET despite radiographic evidence suggesting persistent tumor. Am J Sci,1997,169(4):1049-1050. |
| [2] | Yusenko M V,Zubakov D,Kovacs G. Gene expression profiling of chromophobe renal cell carcinomas and renal oncocytomas by Affymetrix Gene Chip using pooled and individual tumours. Int J Biol Sci,2009,5(6):517-527. |
| [3] | Zhang B,Horvath S A. General framework for weighted gene co-expression network analysis. Stat Appl Genet Mol Biol,2005,4(2):1-45. |
| [4] | Ravasz E,Somera A L,Mongru D A,et al. Hierarchical organization of modularity in metabolic networks. Science,2002,297(5586):1551-1555. |
| [5] | Langfelder P,Zhang B,Horvath S. Defining clusters from a hierarchical cluster tree:the Dynamic Tree Cut package for R. Bioinformatics,2008,24(5):719-720. |
| [6] | Langfelder P,Horvath S. WGCNA:an R package for weighted correlation network analysis. BMC Bioinformatics,2008,9(25):559-559. |
| [7] | Barabasi A L,Albert R. Emergence of scaling in random networks science. Science,1999,286(5439):509-512. |
| [8] | Goh K I,Cusick M E,Valle D,et al. The human disease network. PNAS,2007,104(21):8685-8690. |
| [9] | Vindrieux D,Augert A,Girard C A,et al. PLA2R1 mediates tumor suppression by activating JAK2. Cancer Res,2013,73(20):6334-6345. |
| [10] | Bernard D,Vindrieux D. PLA2R1:expression and function in cancer. BBA-REV Cancer,2014,1846(1):40-44. |
 2016, Vol. 36
2016, Vol. 36




