文章信息
- 马晨露, 唐存多, 史红玲, 王瑞, 岳超, 夏敏, 邬敏辰, 阚云超
- MA Chen-lu, TANG Cun-duo, SHI Hong-ling, WANG Rui, YUE Chao, XIA Min, WU Min-chen, KAN Yun-chao
- 头孢菌素C乙酰化酶的半理性改造及7-ACA的生物合成
- Semi-rational Modification of Cephalosporin C Acylase and Biosynthesis of 7-ACA
- 中国生物工程杂志, 2015, 35(12): 65-71
- China Biotechnology, 2015, 35(12): 65-71
- http://dx.doi.org/10.13523/j.cb.20151210
-
文章历史
- 收稿日期: 2015-07-06
- 修回日期: 2015-09-01
2. 江南大学无锡医学院 无锡 214122
2. Wuxi Medical School, Jiangnan University, Wuxi 214122, China
头孢类抗生素(Cephalosporins)是一类广谱抗生素,由于其抗菌作用强且有较好的β-内酰胺酶耐受性,全球对其的需求也呈上升的趋势[1]。7-氨基头孢烷酸(7-Amino cephalosporanic acid,7-ACA)是合成半合成头孢类抗生素的重要中间体,其传统的制备途径是将头孢菌素C(Cephalosporin C,CPC)通过化学方法的脱乙酰化处理而成,化学法反应条件苛刻、对环境污染大[2, 3]。与传统的化学方法相比,新型的生物催化法具有安全性高、环境友好、选择性强及设备投入低等优势[2]。生物催化法常见的主要有两步酶法和一步酶法[3]。两步酶法所使用的关键酶是D-氨基酸氧化酶和戊二酰-7-氨基头孢烷酸乙酰化酶[4]。尽管两步酶法的工艺已经日趋成熟,但两步酶法副产物多、酶的制备及生产工艺相对复杂的缺陷依然存在[5]。一步酶法更能简化生产工艺、降低生产成本,在工业生产中更具吸引力[6, 7]。
乙酰化酶可以归为五大类型,同一类型中各个成员氨基酸的序列相似度在90%以上,它们的功能特性也比较相似[8],其中仅有第Ⅰ和第Ⅲ类的乙酰化酶具有微弱的CPC乙酰化酶活性[9],而Aeromonas sp. ACY95 来源的一个乙酰化酶具有专一的催化CPC的活性[10],但是自然界中天然的CPC乙酰化酶的活性均较低,不能直接满足工业生产的需求[2]。迄今为止,工业上应用的几个CPC乙酰化酶均是由戊二酰-7-氨基头孢烷酸乙酰化酶改造而来[11, 12, 13, 14]。尽管国内的清华大学[6, 7, 15, 16]、上海医药工业研究院[17, 18]等单位针对CPC乙酰化酶的改造做了大量的工作,也取得了良好的成果,但是国内CPC乙酰化酶的市场仍然被韩国、德国等国的企业所垄断。CPC较戊二酰-7-氨基头孢烷酸的结构多了一个侧链基团,不利于与酶的活性口袋结合,已有文献报道,拓宽乙酰化酶活性口袋的开口大小可以明显提高乙酰化酶对CPC的催化活性[14]。因此,本研究团队试图借助半理性设计的手段,以拓宽戊二酰-7-氨基头孢烷酸乙酰化酶的底物结合的活性口袋为宗旨来改变它的底物特异性,提高其对CPC的催化活性。
本文以Pseudomonas sp SE 83来源的Ⅲ型CPC乙酰化酶(CAⅢ)的基因(ENA号:M18278.1)为研究对象,根据大肠杆菌密码子的偏爱性,利用OPTIMIZER服务器对密码子进行优化,然后进行全基因的合成,并借助pET32a质粒在大肠杆菌BL21中进行表达研究。与此同时,将CAⅢ与CPC的三维结构进行分子对接,找出底物结合位点附近活性口袋的氨基酸残基,然后以拓宽活性口袋的开口大小为宗旨进行半理性的分子改造,以提高CAⅢ对CPC的催化活性,获得真正意义上的CPC乙酰化酶。
1 材料与方法 1.1 材 料 1.1.1 菌株与质粒Escherichia coli JM109和E. coli BL21(DE3)菌株由本研究室保藏;pMD19-T-caⅢ质粒购自大连宝生物公司;pET32a质粒由本研究室保藏。
1.1.2 试剂与试剂盒QuikChange Lightning Multi Site-Directed Mutagenesis Kit购自Agilent Technologies公司,用于多点定点突变;限制性内切酶SacⅠ和XhoⅠ以及PageRuler Prestained Protein Ladder购自Thermo Scientific公司;DNA Ligation Kit Ver.2.1和DNA Marker购自TaKaRa公司;一站式His标记蛋白质微量纯化套装购于北京天恩泽基因科技有限公司,用于重组酶的快速亲和纯化;对二甲氨基苯甲醛(p-Dimethylaminobenzaldehyde,p-DAB)和7-ACA标准品购自上海生工生物工程有限公司;CPC-Na盐和头孢菌素C乙酰化酶标样由南阳衡淯制药有限公司馈赠;其它试剂均为国产或进口分析纯。
1.2 方 法 1.2.1 头孢菌素C乙酰化酶基因的合成基于对已报道的文献分析[12, 13, 14],本文选择以Pseudomonas sp SE 83来源的Ⅲ型头孢菌素C乙酰化酶(CAⅢ)的基因(ENA号:M18278.1)为研究对象。基于Codon Usage Database中E. coli K12的密码子使用频率表,利用OPTIMIZER(http://genomes.urv.cat/OPTIMIZER/)服务器进行密码子优化,然后委托TaKaRa进行全基因合成,在基因两端分别加上SacⅠ和XhoⅠ酶切位点后连接至pMD19-T质粒,获得pMD19-T-caⅢ供后续的研究。
1.2.2 头孢菌素C乙酰化酶突变位点的理性选择利用AutoDock 4.2软件将CAⅢ的三维结构(PDB号:4HSR)与优化后的CPC的三维结构进行分子对接模拟,然后利用Pymol软件对获得的结果进行观察和分析,找出底物结合相关的活性口袋上的关键氨基酸残基,然后将一些侧链基团较大的氨基酸残基拟突变为侧链基团较小的氨基酸残基,以降低活性口袋对CPC与酶结合的空间障碍。
1.2.3 引物的设计、合成及多点突变基于上述理性分析的结果,利用Agilent公司提供的QuikChange Primer Design Program(www.agilent.com/genomics/qcpd)设计多点定点突变的引物,并委托Invitrogen公司进行引物合成。以pMD19-T-caⅢ为亲本,参照QuikChange Lightning Multi Site-Directed Mutagenesis Kit的说明书进行多点突变操作,获得含CAⅢ突变体编码基因的重组质粒pMD19-T-caⅢM。
1.2.4 头孢菌素C乙酰化酶及其突变的可溶性表达分别将pMD19-T-caⅢ和pMD19-T-caⅢM质粒用SacⅠ和XhoⅠ进行双酶切,然后将目的基因连接至经同样双酶切的pET32a质粒上,转化E. coli BL21(DE3)菌株,经抗性筛选和菌落PCR检测分别筛选出重组子BL21/pET32a-caⅢ和BL21/pET32a-caⅢM,将得到的重组子送上海生工进行测序分析。分别将经测序鉴定后的BL21/pET32a-caⅢ和BL21/pET32a-caⅢM菌落接种至4 ml的LB液体培养基中,200 r/min、37℃培养14 h,然后以4%的接种量转接至100 ml新鲜的LB培养基中,200 r/min、37℃培养2.5 h,加入终浓度为0.1 mmol/L的IPTG,于30℃诱导8 h。诱导表达以后参照Pollegioni等[13]的方法,用低浓度的NaOH进行菌体裂解,用磷酸二氢钠中和后收集裂解上清。裂解上清液经一站式His标记蛋白质微量纯化套装纯化后获得电泳纯的重组CPC乙酰化酶reCAⅢ和reCAⅢM。
1.2.5 酶活性及蛋白分析对传统的p-DAB比色法作了一定的改进[13]。用0.1mol/L、pH8.0的磷酸缓冲液配制终浓度为1.5 mg/ml的7-ACA,用1 mol/L的NaOH调pH至8.0,分别取0、1、2、4、6、8、12、16和20 μl的7-ACA于离心管中,再用0.1 mol/L、pH8.0的磷酸缓冲液补足到20 μl。向每管中加入20 μl 25℃保温3 min的CPC,混匀,25℃水浴10 min后加入5倍体积的终止液(20%乙酸与0.05 mol/L NaOH按2∶1的比例混匀),混匀后12 000 r/min离心3 min,取200 μl的上清与40 μl的显色剂(用甲醇配制的0.5%的p-DAB)混匀,室温静置10 min后取200 μl测415 nm的吸光值,以0号为空白对照。以7-ACA的质量为纵坐标、OD415为横坐标制作标准曲线,并求出回归方程。用0.1 mol/L的磷酸缓冲液配制20 g/L的CPC底物,用1mol/L的NaOH调节pH至8.0。取20 μl底物与20 μl稀释适当倍数的酶液混匀,25℃保温10 min,加入5倍体积的终止液终止反应,12 000 r/min离心3 min,取200 μl的上清与40 μl的显色剂混匀,室温反应10 min后取200 μl测415 nm的吸光值。酶活定义为在测定条件下每分钟产生1 μmol 7-ACA所需的酶量。采用Bradford法[19]测定蛋白的浓度,以牛血清白蛋白为参照标准。用12.5%的分离胶进行SDS-PAGE分析[20],并用Quantity One软件计算目的蛋白的表观分子量。
1.2.6 7-ACA的生物合成以40 IU/g CPC的加酶量分别将50 ml浓度为20 g/L的CPC于25℃下转化1、2、4、6、8、10和12 h,用p-DAB比色法测定7-ACA的产量,进而计算出CPC的转化率和7-ACA的得率,初步评价头孢菌素C乙酰化酶的转化效率。本研究中CPC的转化率定义为参与转化的CPC的质量与总CPC的质量之比,7-ACA的得率定义为生成7-ACA的质量与总CPC的质量之比。
2 结 果 2.1 分子对接及突变位点的选择用AutoDock 4.2软件将CAⅢ的三维结构(PDB号:4HSR)与优化后的CPC的三维结构进行分子对接,选取结合自由能最低的对接构型,然后利用pymol软件找出距离底物分子CPC 3范围内的氨基酸残基,结果如图1所示。对这些残基做进一步观测分析发现,His249、Arg250、His283和His296 4个氨基酸残基(图中用红色标出)恰好位于CAⅢ活性口袋开口的位置,且与CPC的距离最近。已有研究报道,减少活性口袋附近氨基酸残基侧链基团对底物结合的空间障碍,可以改变乙酰化酶的底物特异性,能够显著提高其对大分子CPC的活性[11, 12, 13, 14]。为了减少活性口袋附近氨基酸残基侧链基团对底物结合的空间障碍,结合20种天然氨基酸残基各自的特征,本研究中选择分别将三个选定的His突变为Ala,而将Arg突变为Ser。
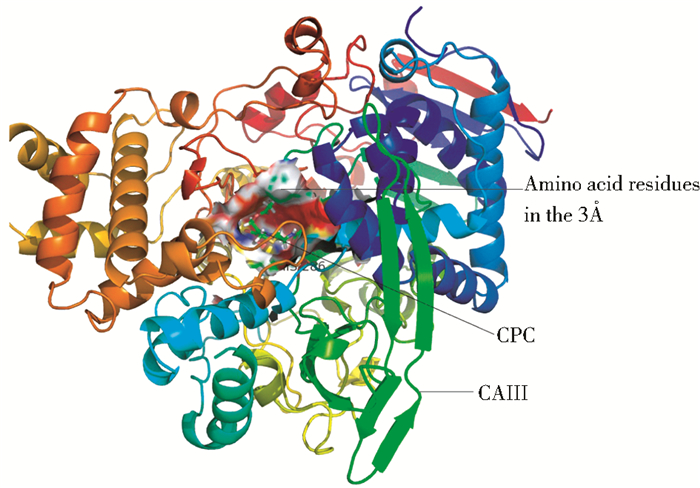
|
| 图 1 CPC与CAⅢ三维结构的分子对接模型 Fig. 1 The molecule-docked conformation between 3-D structures of a CPC and CAⅢ |
基于上述理性分析的结果,利用Agilent公司提供的QuikChange Primer Design Program设计多点定点突变的3条引物并委托Invitrogen公司进行合成,引物的序列如表1所示。以TaKaRa公司代构建的pMD19-T-caⅢ为亲本,参照QuikChange Lightning Multi Site-Directed Mutagenesis Kit的说明书进行多点突变操作,挑取5个重组子送上海生工公司进行测序,从中筛选到了1个4个位点均完成了突变的重组子,将该质粒命名为pMD19-T-caⅢM。
| Name | Amino acid position | Primer sequence (5′-3′) | Tm (℃) |
| H 249-A 249 | His 249 | ctggcgggtgacccg gccagtgttttcgaaatcccg | 78.85 |
| R 250-S 250 | Arg 250 | ||
| H 283-A 283 | His 283 | tccgggtttcccg gccttcgcgcacaacg | 78.19 |
| H 296-A 296 | His 296 | gcgtactgcgttacc gccgcgttcatggacatc | 78.59 |
分别将pET32a、pMD19-T-caⅢM和pMD19-T-caⅢ质粒用SacⅠ和XhoⅠ进行双酶切,酶切产物用1%琼脂糖凝胶电泳检测,结果如图2a所示。pMD19-T-caⅢM和pMD19-T-caⅢ的酶切产物均有两条清晰的条带,一条为pMD19-T质粒的片段,另一条为2 300 bp左右的目的基因片段。将pET32a及目的基因条带割胶回收后用DNA Ligation Kit Ver.2.1试剂盒进行连接,转化BL21感受态细胞,经氨苄青霉素抗性筛选后再进行菌落PCR筛选,结果如图2b所示。该图显示利用T7-promoter和T7-terminator引物均能扩增出3 000 bp左右的条带,其中包括2300 bp目的基因的片段和700 bp左右质粒上的片段,表明挑取的菌落均为阳性重组子,送上海生工公司进行测序鉴定,结果与预期的相符。将测序鉴定的重组子分别命名为BL21/pET32a-caⅢ和BL21/pET32a-caⅢM。
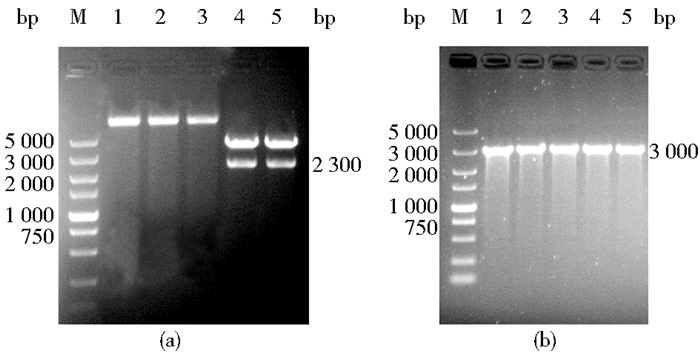
|
| 图 2 重组质粒的构建与鉴定 Fig. 2 The electrophoretogram of plasmid double digestion and individual bacterial colonies PC (a) Plasmid double digestion M: 5 000 bp DNA Ladder marker; 1~3: Digested products of pET32a; 4: Digested products of pMD19-T-caIII; 5: Digested products of pMD19-T-caIIIM (b) Individual bacterial colonies PCR identification M: 5 000 bp DNA Ladder marker; 1~2: PCR products of BL21/ pET32a-caIII; 3~5: PCR products of BL21/ pET32a-caIIIM |
分别将挑取阳性重组子BL21/pET32a-caIII和BL21/pET32a-caIIIM进行低诱导剂浓度、低温诱导表达,BL21/pET32a作为阴性对照。各收集100 ml发酵液中的菌体经碱裂解及中和后收集裂解上清,用一站式His标记蛋白质微量纯化套装进行纯化,并用3 kDa截留分子量的超滤离心管进行咪唑的去除和产物的浓缩,最终分别定容至3 ml。将裂解上清液和纯化后的产物一并进行SDS-PAGE分析,结果如图3所示。图3中显示,重组子BL21/pET32a-caIII和BL21/pET32a-caIIIM均为可溶性表达,且形成了明显的大小亚基,Quantity One软件估算出大亚基的表观分子量为60 kDa,小亚基的表观分子量为44 kDa。同时,结果也表明经过His标签亲和层析后,获得了电泳纯的重组酶。利用考马斯亮蓝法测得纯化、浓缩后的reCAIII的浓度为4.11 mg/ml,reCAIIIM的浓度为4.37 mg/ml。

|
| 图 3 reCAIII和reCAIIIM的SDS-PAGE分析 Fig. 3 The SDS-PAGE analysis of the expressed reCAIII and reCAIIIM M: PageRuler Prestained Protein Ladder; 1: Expressed products of BL21/pET32a; 2: Expressed products of BL21/pET32a-caIII; 3: Expressed products of BL21/pET32a-caIIIM; 4: The purified reCAIII; 5: The purified reCAIIIM |
按照1.2.5节中所述的方法制作7-ACA的标准曲线,并得出回归方程和相关系数。利用p-DAB比色法测出纯化、浓缩后的reCAIII对CPC的酶活为25 IU/ml,其比酶活为6.02 IU/mg;而reCAIIIM对CPC的酶活为117 IU/ml,比酶活为26.7 IU/mg。结果表明,突变酶reCAIIIM对CPC的比酶活明显高于原酶,较原酶提高了3.44倍。
2.5 CPC的生物转化按照1.2.6 节中的方法初步研究了突变酶催化CPC合成7-ACA的工艺,CPC转化率及7-ACA得率随时间的变化情况如图4所示。随着反应时间的延长,CPC的转化率和7-ACA的得率均会增加,当反应进行到12 h时CPC的转化率和7-ACA的得率分别达到96.3%和63.4%,且趋于稳定。
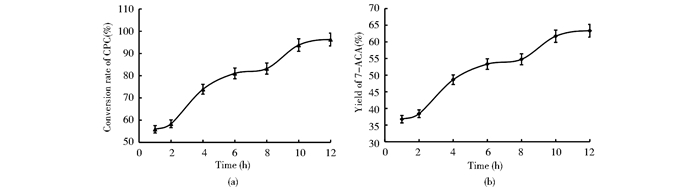
|
| 图 4 底物转化率和产物得率 Fig. 4 The substrate conversion rate and product yield |
7-ACA作为当前应用最广的一线抗菌药物——头孢类抗生素重要的合成中间体,研究它的生产制备具有十分重要的意义。在7-ACA的制备方法中,一步酶法制备最具吸引力[18]。目前,7-ACA一步酶法的工业化主要受制于现有的乙酰化酶对CPC的催化效率较低。国内的科研工作者对乙酰化酶的改造也做了大量的工作,取得了不错的成果,但是目前国内一步酶法生产7-ACA所需的头孢菌素C乙酰化酶仍然被韩国、德国等国的企业垄断。本研究选择的来源于Pseudomonassp SE 83的Ⅲ型头孢菌素C乙酰化酶(CAⅢ)与Pseudomonas sp N176来源的序列高度相似,也具有相似的催化机理[8]。本文将CAⅢ的编码基因根据E. coli K12的密码子偏爱性利用OPTIMIZER进行了密码子优化,利用带有硫氧还蛋白标签的pET32a质粒在低温度、低诱导剂浓度的条件下实现了CAⅢ基因的可溶性表达。与笔者前期利用pET28a质粒表达失败的结果对比分析,表明硫氧还蛋白能够显著提高这类高分子量蛋白的可溶性表达,可为其它类似蛋白的可溶性表达提供参考。
分子对接模拟能够快速地预测酶与底物的最适结合位点,在酶的理性分子改造中是一种强大的辅助手段,迄今为止也有了许多成功应用的案例[21, 22]。本研究中利用分子对接的手段确定了CPC乙酰化酶与底物结合的关键氨基酸残基,然后通过将其替换为侧链基团更小的残基,突变体对CPC的催化活性有了显著的提高。进一步证明了拓宽活性口袋的开口,减小酶与底物结合的空间阻碍能够提高乙酰化酶对CPC的催化活性,这也与Shin等[14]报道的结果一致。本研究获得的突变酶reCAIIIM的催化活性较原酶有了显著的提高,但与前人报道的催化活性相比仍然较低[13, 14, 18],这主要归因于本研究表达出的重组酶α亚基的N端还留有一段较大的硫氧还蛋白标签以及本研究选定的酶活测定温度较低。在后续的研究中,笔者也将进一步研究硫氧还蛋白标签及反应温度对CPC乙酰化酶活性的影响。
目前,常用的头孢菌素C乙酰化酶活性的测定方法主要有碱滴定法、p-DAB比色法[13]和HPLC法[23],其中碱滴定法主要是用在工业生产中简单、粗糙、快速地评估头孢菌素C乙酰化酶的活性,在此不做讨论。HPLC法能够非常准确地测定7-ACA的量进而准确地计算出头孢菌素C乙酰化酶的活性,但受仪器所限,测定速度较慢而且很难实现高通量的测定。传统的p-DAB比色法,由于前人所述的终止液对反应的终止效果有限,也不太适合高通量的测定。本研究中,笔者对传统的p-DAB比色法做了一定的改进,利于后续高通量的测定。本研究确定的四个关键氨基酸残基以及建立的能够实现高通量筛选的p-DAB比色法有利于我们对该酶做进一步的组合饱和突变,为我们后续深入的改造奠定了坚实的基础,也为提高我国CPC乙酰化酶的市场占有率奠定了坚实的基础。
| [1] | Biocatalytic production of semi-synthetic cephalosporins:process technology and integration. Tramper J, Beeftink HH, Janssen AEM, Ooijkaas LP, van Roon JL, Strubel M, Schron CGPH Synthesis of β-Lactam Antibiotics. Springer Netherlands, 2001:206-249. |
| [2] | Pollegioni L, Rosini E, Molla G. Cephalosporin C acylase:dream and(/or) reality. Appl Microbiol Biot, 2013, 97(6):2341-2355. |
| [3] | Sonawane V C. Enzymatic modifications of cephalosporins by cephalosporin ccylase and other enzymes. Crit Rev Biotechnol, 2006, 26(2):95-120. |
| [4] | Pollegioni L, Molla G, Sacchi S, et al. Properties and applications of microbial D-amino acid oxidases:current state and perspectives. Appl Microbiol Biot, 2008, 78(1):1-16. |
| [5] | 姚舜,罗晖,常雁红,等.一步酶法生产7-氨基头孢烷酸的研究进展.现代化工, 2013, 33(2):11-14. Yao S, Luo H, Chang Y H, et al. Research progress in one-step enzymatic conversion of cephalosporin C to 7-aminocephalosporanic acid. Modern Chemical Industry, 2013, 33(2):11-14. |
| [6] | Wang Y, Yu H, Song W, et al. Overexpression of synthesized cephalosporin C acylase containing mutations in the substrate transport tunnel. J Biosci Bioeng, 2012, 113(1):36-41. |
| [7] | Zhu X, Luo H, Chang Y, et al. Characteristic of immobilized cephalosporin C acylase and its application in one-step enzymatic conversion of cephalosporin C to 7-aminocephalosporanic acid. World J Microb Biot, 2011, 27(4):823-829. |
| [8] | Li Y, Chen J, Jiang W, et al. In vivo post-translational processing and subunit reconstitution of cephalosporin acylase from Pseudomonas sp. 130. Eur J Biochem, 1999, 262(3):713-719. |
| [9] | Aramori I, Fukagawa M, Tsumura M, et al. Cloning and nucleotide sequencing of new glutaryl 7-ACA and cephalosporin C acylase genes from Pseudomonas strains. J Ferment Bioeng, 1991, 72(4):232-243. |
| [10] | Deshpande B S, Ambedkar S S, Shewale J G. Cephalosporin C acylase and penicillin V acylase formation by Aeromonas sp. ACY 95. World J Microb Biot, 1996, 12(4):373-378. |
| [11] | Conti G, Pollegioni L, Molla G, et al. Strategic manipulation of an industrial biocatalyst-evolution of a cephalosporin C acylase. FEBS J, 2014, 281(10):2443-2455. |
| [12] | Golden E, Paterson R, Tie W J, et al. Structure of a class Ⅲ engineered cephalosporin acylase:comparisons with class I acylase and implications for differences in substrate specificity and catalytic activity. Biochem J, 2013, 451(2):217-226. |
| [13] | Pollegioni L, Lorenzi S, Rosini E, et al. Evolution of an acylase active on cephalosporin C. Protein Sci, 2005, 14(12):3064-3076. |
| [14] | Shin Y C, Jeon J Y, Jung K H, et al. Cephalosporin C acylase mutant and method for preparing 7-ACA using same. US Patent, 0207519A1, 2007. |
| [15] | Li Q, Huang X, Zhu Y. Evaluation of active designs of cephalosporin C acylase by molecular dynamics simulation and molecular docking. J Mol Model, 2014, 20(7):2314. |
| [16] | Xiao Y, Huo X, Qian Y, et al. Engineering of a CPC acylase using a facile pH indicator assay. J Ind Microbiol Biot, 2014, 41(11):1617-1625. |
| [17] | 王颖秋,郑林冲,谢丽萍,等.基于易错PCR的头孢菌素C酰化酶的定向进化.中国医药工业杂志, 2013,4:344-347. Wang Y Q, Zhen L C, Xie L P, et al. Directed evolution of cephalosporin C acylase activity by error-prone PCR. Chinese Journal of Pharmaceuticals, 2013,4:344-347. |
| [18] | 徐雪丽,张伟,刘艳,等.头孢菌素C酰化酶突变位点的研究.中国生物工程杂志, 2015,2:59-65. Xu X L, Zhang W, Liu Y, et al. Study on mutaions of cephalosporin C acylase. China Biotechnology, 2015,2:59-65. |
| [19] | Bradford M. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Anal Biochem, 1976, 72:248-254. |
| [20] | Laemmli U K. Cleavage of structural proteins during the assembly of the head of bacteriophage T4. Nature, 1970, 227:680-685. |
| [21] | Perez C, Pastor M, Ortiz A R, et al. Comparative binding energy analysis of HIV-1 protease inhibitors:incorporation of solvent effects and validation as a powerful tool in receptor-based drug design. J Med Chem, 1998, 41:836-852. |
| [22] | Timmers L F, Ducati R G, Sanchez-Quitian Z A, et al. Combining molecular dynamics and docking simulations of the cytidine deaminase from Mycobacterium tuberculosis H37Rv. J Mol Model, 2012, 18(2):467-479. |
| [23] | Liu Y, Gong G, Zhu C, et al. Environmentally safe production of 7-ACA by recombinant Acremonium chrysogenum. Curr Microbiol, 2010, 61(6):609-614. |
 2015, Vol. 35
2015, Vol. 35




