文章信息
- 陈彦猛, 黄瑶, 胡杰, 胡源
- CHEN Yan-meng, HUANG Yao, HU Jie, HU Yuan
- APOBEC3A通过脱氨基作用抑制HBV复制
- APOBEC3A Inhibited HBV Replication Dependent Its Deamination
- 中国生物工程杂志, 2015, 35(12): 8-14
- China Biotechnology, 2015, 35(12): 8-14
- http://dx.doi.org/10.13523/j.cb.20151202
-
文章历史
- 收稿日期: 2015-08-06
- 修回日期: 2015-11-09
乙型肝炎病毒(Hepatitis B virus,HBV)感染是一个严重的公共问题,全球大约有350万左右的慢性乙肝患者[1]。HBV慢性感染可引起慢性乙型肝炎(chronic hepatitis B,CHB)、肝硬化(liver cirrhosis,Lc)和原发性肝细胞癌(hepatocellulor carcinoma,HCC)等相关疾病[2],而治疗慢性乙型肝炎的首要目标是持久抑制病毒复制。目前治疗慢性乙型肝炎的抗病毒药物主要有两种:干扰素(interferon,IFN)和核苷酸类似物(Nucleotide analogs,NAs)疗法[3],但是由于副作用、应答效果不佳、停药后反弹等因素,导致治疗失败。因此寻找新的靶点分子成为抗病毒研究的热点和重点。
HBV的复制周期主要包括:病毒入胞后释放出松弛环状DNA(relaxed circular DNA,RcDNA),入核转变成共价闭合环状DNA(covalently closed circular DNA,cccDNA);以cccDNA转录的前基因组RNA(pregenomic RNA,pgRNA)为模板,在病毒核心颗粒中逆转录合成病毒基因组;含有RcDNA的病毒成熟颗粒可以被病毒包膜蛋白分泌到胞外,也可以内循环进入细胞核,转变成cccDNA,从而维持cccDNA pool size (10~50拷贝/细胞)。
APOBEC3(apolipoprotein B mRNA editing enzyme,catalytic polypeptide-like 3)是一类天然免疫分子,包含7个家族成员(APOBEC3A-H),能够将胞嘧啶核苷脱氨基生成尿嘧啶核苷,对于抑制HIV、HBV等病毒的复制起着重要作用[4, 5]。其中APOBEC3A是由199个氨基残基组成,编码23kDa的蛋白质,具有单一的依赖于锌指结构的胞苷脱氨酶功能区(cytidine deaminase active sites,CDA)。目前,对于APOBEC3A抑制HBV的效应及机制的报道很少,最近德国的Lucifora等[6]报道IFN-α可以上调APOBEC3A表达,在细胞核内与HBV的核心蛋白相互作用,直接作用于cccDNA发生脱氨基作用,从而被修复途径降解。由于APOBEC3A主要作用于DNA单链,因此尚不清楚APOBEC3A能否被包装进入病毒核心颗粒中,在逆转录过程中作用于HBV的单链,从而抑制HBV的复制。因此,本文主要从病毒的逆转录环节研究了APOBEC3A对病毒复制的抑制作用,其研究结果有助于深入阐明APOBEC3A对HBV复制抑制的分子机制,并有助于研制出针对APOBEC3A靶点的新抗病毒药物。
1 材料与方法 1.1 材 料肝癌细胞系HuH7细胞为本实验室保存;DMEM培养基、胰酶、胎牛血清均购自美国Gibco公司;转染试剂(TransIT®-LT1)购自美国Mirus公司;细胞增殖检测试剂盒(CellTiter 96®AQueous Non-radioactive Cell Proliferation Assay)、荧光定量PCR试剂盒(GoTaq® Probe qPCR Master Mix)、海肾荧光素酶检测试剂盒(Renilla Luciferase Assay System)购自美国Promega公司;Taq酶购自日本TaKaRa公司;抗Flag鼠抗、抗GAPDH鼠抗、免疫荧光染色试剂盒(抗小鼠Alexa Fluor 488)均购自上海碧云天生物技术公司;抗HBc抗体购自美国Santa Cruz公司;Protein G Agarose磁珠购自美国Millpore公司;引物合成与测序由上海Invitrogen公司完成。APOBEC3A表达质粒由广州复能基因公司构建。HBV复制质粒pCH9/3091为第三军医大学林兰博士赠予。
1.2 方 法 1.2.1 质粒构建和细胞培养APOBEC3A表达质粒是其cDNA克隆序列(NCBI收录号:NM_001270406.1)连接到pReceiver-M12载体,氨基端带有Flag标签。HBV复制质粒pCH9/3091为CMV启动子,下端带有HBV基因组全长1.1倍体的序列。HuH7细胞采用DMEM培养基,37℃,5% CO2条件下培养。6孔板接种1.2×105个HuH7细胞,转染步骤按照Mirus公司的说明书进行。其中HBV复制质粒pCH9/3091和APOBEC3A表达质粒的转染比例为1∶3。
1.2.2 MTT法检测过表达APOBEC3A对细胞毒性的影响96孔板中接种3.65×103个HuH7细胞,转染不同浓度的APOBEC3A表达质粒。48h后加入MTS/PML混合液,37℃作用2h后用酶标仪检测490nm的吸光度。
1.2.3 免疫荧光检测APOBEC3A在细胞中的定位六孔板中放入10×10mm玻片后转染HBV复制质粒pCH9/3091或/与APOBEC3A共转染48h后,4%多聚甲醛固定25min,0.2% Triton X-100通透处理10min,山羊血清封闭后滴加小鼠抗Flag抗体(1∶1 000),4℃湿盒中孵育过夜,Alexa Fluor 488标记的山羊抗小鼠IgG抗体(1∶10 000)室温孵育2h,DAPI染核,抗荧光淬灭封片液封片后在荧光显微镜下观察。
1.2.4 IP试验检测APOBEC3A与病毒颗粒的相互作用转染3天后,提取细胞裂解液进行免疫沉淀试验(Immunoprecipitation,IP)。Protein G Agarose磁珠清洗2次后加入500μg蛋白,4℃作用1h,离心去除磁珠以除去非特异性结合的杂蛋白,加入抗HBc抗体孵育过夜,再将抗原抗体混合物加入磁珠中4℃旋转作用2h,煮沸变性后收集上清进行Western blot。
1.2.5 Western blot检测APOBEC3A的表达转染3天后分别提取细胞裂解液和病毒颗粒中的蛋白,蛋白上样量为30μg。15% SDS-PAGE电泳后转移至PVDF膜上,5%脱脂奶粉封闭2h,加入抗Flag小鼠单克隆抗体(1∶1 000)于4℃孵育过夜,加入山羊抗小鼠二抗(1∶10 000)室温孵育1h,加入底物发光剂ECL进行显色。
1.2.6 病毒核心颗粒DNA和cccDNA的提取采用蔗糖密度梯度离心法提取病毒核心颗粒中HBV DNA[7]:转染3天后,细胞用CPLB裂解液(10mmol/L Tris-HCl,1mmol/L EDTA,1% NP-40,2%蔗糖)裂解后,10 000r/min离心5min,上清液用微管酶(Micrococcal Nuclease,M’Nase)37℃消化1h,然后在30%蔗糖密度介质中,45 000r/min超速离心2h提取病毒颗粒,蛋白酶K消化过夜后用酚/氯仿抽提,乙醇沉淀得到HBV DNA。
采用Hirt法提取HBV cccDNA[8]:细胞转染7天后,用Hirt裂解液(50mmol/L Tris-HCl,10mmol/L EDTA,1% SDS,150mmol/L NaCl)裂解细胞,加入5mol/L NaCl 4℃过夜沉淀基因组DNA,12 000g离心30min后得到的上清液用酚/氯仿抽提,乙醇沉淀。抽提得到的DNA再用Plasmid-safeTM ATP-Dependent DNase (PSAD酶)消化处理HBV dlDNA和ssDNA,纯化后的cccDNA用作荧光定量PCR(fluorescent quantitative PCR,FQ-PCR)检测分析。
1.2.7 FQ-PCR检测病毒核心颗粒DNA和cccDNA运用特异性Taqman探针法检测cccDNA和病毒核心颗粒DNA,并采用线粒体DNA做内参[9]。克隆和扩增线粒体DNA的引物见表 1,克隆条件为95℃ 3min,95℃ 30s,54℃ 30s,72℃ 60s,35个循环,72℃ 10min。扩增条件为95℃ 10min,95℃ 10s,55℃ 15s,72℃ 20s,收集荧光信号,35个循环。检测cccDNA和病毒核心颗粒DNA的引物和探针另文发表。
| Primer | Sequence (5′to 3′) | Position |
| Primer of mitochondrial DNA | ||
| MitoFP1(for clone) | TGAGATTAGTAGTATGGGAG | 467-486 |
| MitoRP1(for clone) | CACCCTATTAACCACTCACG | 15-34 |
| MitoFP2(for FQ-PCR) | AGGCTGGTGTTAGGGTTC | 368-385 |
| MitoRP2(for FQ-PCR) | GATAGCATTGCGAGACG | 81-97 |
| 3D PCR primer of X region | ||
| HBVXOFP | CGCAAATATACATCGTATCCAT | 1354-1375 |
| HBVXORP | AAGAGTYYTYTTATGTAAGACYTT | 1644-1667 |
| HBVXIFP | ATGGCTGCTARGCTGTGCTGCCAA | 1374-1397 |
| HBVXIRP | AAGTGCACACGGTYYGGCAGAT | 1565-1586 |
| 3D PCR primer of Prec/C region | ||
| HBVPrec/COFP | GTACTAGGAGGCTGTAGGCATA | 1770-1791 |
| HBVPrec/CORP | AGAGCTGAGGCGGTATCTAGAA | 1989-2010 |
| HBVPrec/CIFP | TAAATTGGTCTGCGCACCAGCA | 1790-1811 |
| HBVPrec/CIRP | GATCTCGTACTGAAGGAAAGAA | 1967-1988 |
| 3D PCR primer of S region | ||
| HBVSOFP | CGGCGTTTTATCATCTTCCTCTTCAT | 274-299 |
| HBVSORP | CATCCATATAACTGAAAGCCAAACAGT | 719-745 |
| HBVSIFP | TCTTCATCCTGCTGCTATGCCTCAT | 405-429 |
| HBVSIRP | AAAGCCCTACGAACCACTGAACAAAT | 576-601 |
3D PCR(Differential DNA denaturation PCR)是一种根据DNA链变性温度的不同检测碱基突变的技术。病毒基因组发生G-A或C-T突变后,可以通过3D PCR在较低的变性温度下扩增出来,所以被广泛应用于检测病毒DNA的脱氨基作用。巢式PCR扩增HBV X区段,Prec/C区段和S区段的引物序列参考文献[10] (表 1)。X区段扩增条件为:第一轮为94℃ 5min,94℃ 30s,55℃ 30s,72℃ 30s,35个循环,72℃ 10min。第二轮为82.6~90℃ 5min,82.6~90℃ 60s,45℃ 30s,72℃ 30s,35个循环,72℃ 10min。 Prec/C区段扩增为:第一轮为95℃ 5min,95℃ 30s,55℃ 30s,72℃ 30s,35个循环,72℃10min。第二轮为82.6~90℃ 5min,82.6~90℃ 60s,55℃ 30s,72℃ 60s,35个循环,72℃ 10min。 S区段扩增条件为:第一轮为95℃ 5min,95℃ 30s,60℃ 30s,72℃ 60s,35个循环,72℃ 10min。第二轮为82.6~90℃ 5min,82.6~90℃ 60s,60℃ 30s,72℃ 20s,35个循环,72℃ 10min。
1.3 统计学分析采用GraphPad Prism 5进行统计分析。结果采用均数±标准差(x±s)表示。两组数据之间的比较采用两独立样本均数t检验,以P<0.05作为有显著性差异。
2 结 果 2.1 过表达APOBEC3A对细胞毒性的影响在HuH7肝癌细胞中过表达不同浓度的外源性APOBEC3A,通过MTT法检测细胞的相对活力,结果表明外源性表达的APOBEC3A对HuH7细胞没有引起明显的损失变化(P>0.05)(图 1)。
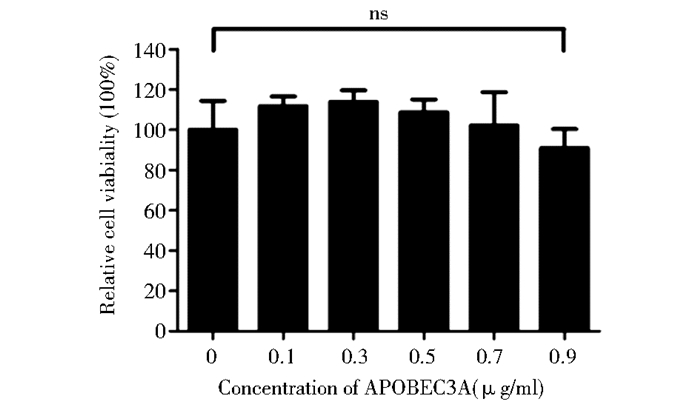
|
| 图 1 过表达APOBEC3A对HuH7细胞毒性的影响 Fig. 1 Effects of exogenous of APOBEC3A on cell viability in HuH7 cells ns: No significant difference (P>0.05) |
通过免疫荧光,我们发现APOBEC3A主要位于细胞核中(图 2a),这与Aynaud等[11]的结果一致。与HBV复制质粒共转染HuH7细胞,APOBEC3A大部分仍然位于细胞核内,同时少量位于细胞质中(图 2a)。IP试验进一步证实APOBEC3A与病毒颗粒相互作用,表明在HBV感染后,APOBEC3A可与病毒颗粒结合,在逆转录环节对病毒的复制发生作用(图 2b)。
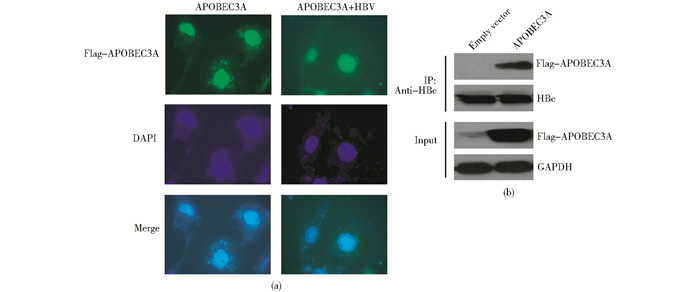
|
| 图 2 免疫荧光和IP试验检测APOBEC3A的分布和结合 Fig. 2 Distribution and combination of APOBEC3A were detected by immunofluorescence and IP (a) Cellular localization of Flag-tagged APOBEC3A in the absence or presence of HBV replication plasmid by immunofluorescence microscopy using an anti-Flag antibody (b) Binding of APOBEC3A to HBV core protein Cytoplasmatic lysates were incubated with an anti-HBc protein antibody, and the immunoprecipitated proteins were analyzed by Western blot with anti-Flag or anti-HBc antibodies. The expression of APOBEC3A and GAPDH in cell lysates of HuH7 cells were analyzed by Western blotting using anti-Flag or anti-GAPDH antibodies as input control |
接着我们分析了过表达外源性的APOBEC3A对HBV复制的影响:转染3天后收集细胞总蛋白,Western blot表明APOBEC3A在HuH7细胞中过表达成功(图 3a);同时收集细胞上清,ELISA检测HBsAg的表达水平,发现与对照相比,上清中HBsAg的表达水平显著下降了80%(P=0.0036,图 3b);FQ-PCR表明核心颗粒中HBV DNA显著下降了64%(P=0.040,图 3c)。
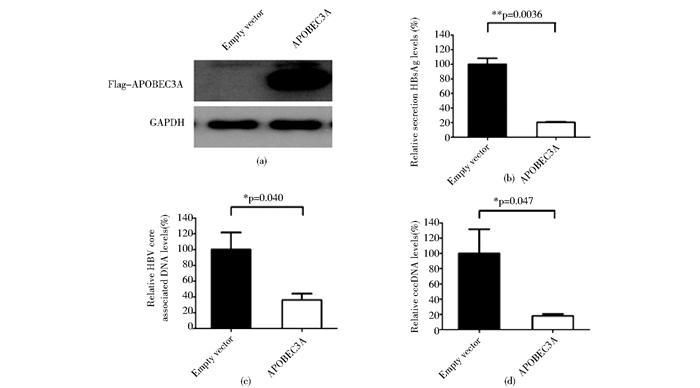
|
| 图 3 APOBEC3A对HBV复制的抑制作用 Fig. 3 The effects of APOBEC3A on HBV viral DNA replication (a) Expression of exogenous APOEBEC3A was confirmed by Western blot using anti-Flag antibody (b) Relative levels of HBsAg in the supernatant (c) Relative levels of HBV DNA in core particles (d) Relative levels of cccDNA All experiments were performed at least three independent experiments. The error bars were shown as mean ± SD. * P< 0.05; ** P< 0.01 |
HBV cccDNA是病毒持续感染的关键因子,因此我们进一步检测了cccDNA表达水平的变化。转染7天后,用Hirt法提取cccDNA,并采用线粒体DNA作内参。为了精确定量各个细胞样本中线粒体DNA的含量,我们首先克隆了479bp的线粒体DNA片段作为标准质粒,并通过FQ-PCR检测了每个样本中线粒体DNA的水平作为内参。其次我们用Taqman探针法检测了样本中cccDNA的表达水平。结果表明:与对照相比,过表达APOBEC3A引起cccDNA表达的水平显著下降了81% (P=0.047,图 3d)。
2.4 APOBEC3A对核心颗粒中的HBV DNA的脱氨基作用为了进一步阐明APOBEC3A对HBV复制的抑制可能通过对核心颗粒中的HBV DNA进行脱氨基作用,我们采用3D PCR对HBV X区段、Prec/C区段和S区段的DNA片段进行扩增。结果表明:与对照相比,HBV DNA的X区在较低的变性温度(85.2℃)有较强的扩增条带(图 4a),表明X区发生了更多的G-A突变或C-T突变;此外,与对照相比,Prec/C和S区段的变性温度没有明显的差异(图 4a)。这一结果表明APOBEC3A对HBV基因组的脱氨基作用具有区域性,主要发生在X区。
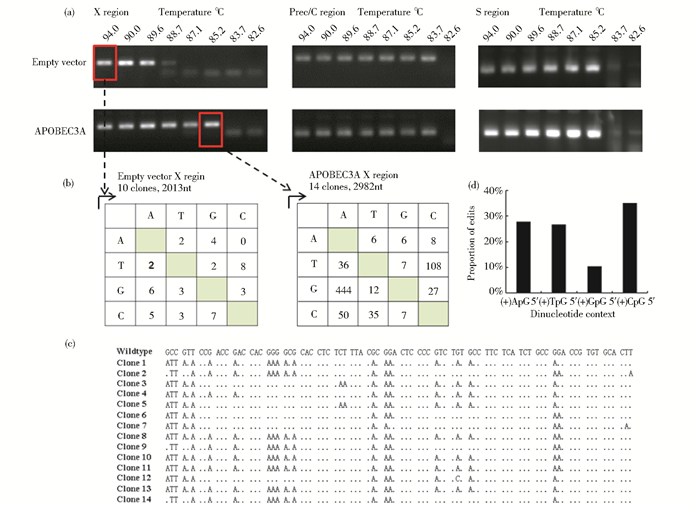
|
| 图 4 3D PCR和测序分析APOBEC3A对病毒核心颗粒DNA的脱氨基作用 Fig. 4 APOBEC3A hyperedits HBV DNA in the core particles (a) 3D PCR analysis of HBV X, Prec/C and S regions (b) Mutation matrices of HBV DNA DNA fragments amplified at 94℃ in the presence of empty vector and DNA fragments recoveded at 85.2℃ in the presence of APOBEC3B were cloned into pMD19-T vectors, and 14 clones were randomly selected (c) Sequence alignment of HBV X region from 14 clones in the presence of APOBEC3A (d) Dinucleotide editing profile of G to A mutations of HBV plus-polarity strand DNA |
为了进一步验证APOBEC3A对X区的脱氨基作用,我们对APOBEC3A作用后的HBV DNA在85.2℃扩增的产物进行了克隆测序。测序结果表明:与对照相比,过表达APOBEC3A后,核心颗粒DNA发生了大量的G-A突变,这种G-A突变是由于HBV负链脱氨基作用引起;同时存在着少量的C-T突变,而这种突变是由于APOBEC3A直接作用于HBV正链引起(图 4b和图 4c)。在这些突变中,T-C突变可能是由于rTaq酶缺少高保真性引起[12]。最后我们分析了APOBEC3A对HBV DNA脱氨基作用的特征,二联核苷酸统计结果显示G-A的高突变更多发生在GpC位点(图 4d)。
3 讨 论HBV感染与HCC关系非常密切[13],HBV慢性感染使患HCC危险性增加5~49倍,超过50%的HCC与HBV感染相关[14]。因此控制HBV感染在HCC发生发展过程中的作用非常重要。
早期文献报道APOBEC3能够抑制HBV复制,其中APOBEC3B和APOBEC3G能通过对病毒基因组进行脱氨基作用而抑制病毒复制[10]。最近Lucifora等[6]报道认为干扰素能上调APOBEC3A,在细胞核内对闭合环状的cccDNA发生脱氨基作用,从而抑制病毒的复制。为了研究APOBEC3A是否能在逆转录过程对HBV发生作用,我们通过在肝癌细胞HuH7中过表达APOBEC3A,检测了病毒颗粒中APOBEC3A的定位和APOBEC3A对病毒基因组的脱氨基作用,结果表明:(1)APOBEC3A位于细胞核内,当HBV感染后可与病毒颗粒结合,从而在HBV逆转录过程发生作用,下一步的研究将阐明这种结合作用是否依赖于HBV聚合酶和/或pgRNA;(2) 我们的研究结果表明:APOBEC3A对HBV的负链和正链都能进行脱氨酸作用,其中G-A的突变远远高于C-T突变,表明APOBEC3A对HBV负链的脱氨基作用更为明显,这可能是APOBEC3A对单链(负链)DNA的作用远远高于不完整的双链DNA; (3)APOBEC3A对HBV基因组DNA的脱氨基作用具有明显的区域性。通过3D PCR我们分别检测了X区、Prec/C区和S区DNA的脱氨基作用,发现X区明显发生脱氨基作用,而Prec/C区和S区几乎没有发生。这可能是由于X区是HBV逆转录时负链刚好完成的区段,而正链DNA尚未合成,更容易受到APOBEC3A的脱氨基作用。
HBV cccDNA的检测一直是难点,传统的Southern blot比较繁琐,而常见的采用跨缺口的荧光定量法不能完全避免RcDNA的干扰,因此在研究中我们一方面采用了PSAD酶处理提取的cccDNA,以降解其中的RcDNA,提高检测的准确性;同时采用Hirt法提取cccDNA后,由于线粒体DNA可与cccDNA共纯化[9],因此我们采用了线粒体DNA作为内参。为了准确检测样本中线粒体DNA的量,我们首先克隆了线粒体DNA的部分片段(470bp)作为标准质粒,并通过FQ-PCR检测了样本中线粒体DNA的含量,从而可以较好地矫正样本的差异。
本研究主要研究了APOBEC3A在HBV感染后可与病毒颗粒结合,在病毒复制的逆转录环节作用于HBV的单链,发生脱氨基作用,从而抑制病毒的复制,对其机制的深入研究有助于将APOBEC3A作为新的靶点分子来筛选抗病毒药物。
| [1] | Jules L, Dienstag M D. Drug therapy hepatitis B virus infection. N Engl J Med, 2008, 359:1486-1500. |
| [2] | Wei Y, Neuveut C, Tiollais P, et al. Molecular biology of the hepatitis B virus and role of the X gene. Pathol Biol, 2010, 58(4):267-272. |
| [3] | European Association for the Study of the Liver. EASL clinical practice guidelines:management of chronic hepatitis B virus infection. J Hepatol, 2012, 57(1):167-185. |
| [4] | Vieira V C, Soares M A. The role of cytidinedeaminases on innate immune responses against human viral infections. Biomed Res Int, 2013:1-18. |
| [5] | Smith H C, Bennett R P, Kizilyer A, et al. Functions and regulation of the APOBEC family of proteins. Semin Cell Dev Biol, 2012, 23(3):258-268. |
| [6] | Lucifora J, Xia Y, Reisinger F, et al. Specific and nonhepatotoxic degradation of nuclear hepatitis B virus cccDNA. Science, 2014, 343(6176):1221-1228. |
| [7] | Hu Y, Cheng X, Cao F, et al. beta-Thujaplicinol inhibits hepatitis B virus replication by blocking the viral ribonuclease H activity. Antiviral Res, 2013, 99(3):221-229. |
| [8] | Gao W, Hu J. Formation of hepatitis B virus covalently closed circular DNA:removal of genome-linked protein. J Virol, 2007, 81(12):6164-6174. |
| [9] | Seeger C, Sohn J A. Targeting hepatitis B virus with CRISPR/Cas9. Mol Ther Nucleic Acids, 2014, 3:e216. |
| [10] | Vartanian J P, Henry M, Marchio A, et al. Massive APOBEC3 editing of hepatitis B viral DNA in cirrhosis. PLoS Pathog, 2010, 6(5):e1000928. |
| [11] | Aynaud M M, Suspène R, Vidalain P O, et al. Human tribbles 3 protects nuclear DNA from cytidine deamination by APOBEC3A. J Biol Chem, 2012, 287(46):39182-39192. |
| [12] | Keohavong P, Thilly W G. Fidelity of DNA polymerases in DNA amplification. Proc Natl Acad Sci U S A, 1989, 86(23):9253-9257. |
| [13] | Shlomai A, de Jong Y P, Rice C M. Virus associated malignancies:the role of viral hepatitis inhepatocellular carcinoma. Semin Cancer Biol, 2014, 26:78-88. |
| [14] | Nguyen V T, Law M G, Dore G J. Hepatitis B-related hepatocellular carcinoma:epidemiological characteristics and disease burden. J Viral Hepat, 2009, 16(7):453-463. |
 2015, Vol. 35
2015, Vol. 35




