文章信息
- 张晨晨, 孟志忠, 陆远芳, 陈新, 李杉
- ZHANG Chen-chen, MENG Zhi-zhong, LU Yuan-fang, CHEN Xin, LI Shan
- 脱氮硫杆菌硫化合物载体SoxYZ蛋白的同源建模和结构分析
- Homology Modeling and Structure Analysis of SoxYZ: A Carrier of Sulfur Compounds from Thiobacillus denitrificans
- 中国生物工程杂志, 2015, 35(7): 68-75
- China Biotechnology, 2015, 35(7): 68-75
- http://dx.doi.org/10.13523/j.cb.20150710
-
文章历史
- 收稿日期:2015-01-15
- 修回日期:2015-04-28
随着现代工业的迅猛发展,煤的低温焦化、天然气开采、含硫矿石的冶炼、制药、食品发酵、造纸等生产过程中会产生大量的含硫废水和尾气,严重影响生态环境,危害人体健康[1]。与传统的物理、化学脱硫方法相比,生物脱硫的反应条件温和、能耗低、投资运行成本低、对环境不易造成二次污染,因此成为近些年来研究脱硫技术的热点。目前工业应用较广的是硫杆菌,已经证实的硫杆菌属有8种,其中脱氮硫杆菌(Thiobacillus denitrificans)是较为特殊的一种,在有氧条件下它可以氧化硫化物,在胞外聚集单质硫;而在厌氧条件下则能以硫代硫酸盐为电子供体,以硝酸盐或其他氧化态氮为电子受体进行生长,同时起到脱硫反硝化的作用[2, 3]。1993年荷兰Paques与Shell公司利用脱氮硫杆菌自身代谢的特异性,联合开发了一种名为Shell-Paques的生物脱硫技术并实现商业化,成功地将其应用于多个工业领域的废气净化过程[4]。近年来,在地下水和饮用水的脱硫、脱硝方法中,其自养反硝化特点也得到广泛的研究和应用[5]。Sox蛋白体系是硫氧化细菌代谢途径中极其重要的组成成分,其核心是SoxYZ蛋白二聚体,因底物硫代硫酸盐、硫化物、亚硫酸盐等必须共价结合在SoxY亚基C端附近的半胱氨酸残基上才能与其他酶发生相互作用进而完成代谢过程[6, 7],但目前为止PDB数据库中尚没有脱氮硫杆菌SoxYZ蛋白的晶体结构。
蛋白质的结构决定其生物功能,因而对蛋白质结构的构建和分析对了解未知蛋白质的生物功能具有重要意义。研究蛋白质结构常用的方法有X射线衍射法、核磁共振、圆二色谱法等,但由于技术手段的限制,采用实验法来测定蛋白质结构受到极大的局限[8]。以计算机技术为依托的分子模拟技术的产生,为解决上述难题提供了一定的理论依据。蛋白质空间结构预测方法有三种,即从头预测法、同源建模法(homology modeling)和折叠识别法,其中同源建模法是最主要的手段,也是目前为止蛋白质结构预测中最成功、准确率最高的方法[9]。本研究利用同源建模法,对SoxYZ蛋白的二聚体结构进行了预测及分析,以期深入了解该蛋白质在代谢途径中所起的作用,能够为以后Sox家族蛋白质间相互作用机制的研究和工程菌性能的改造提供理论依据。
1 材料与方法 1.1 实验材料脱氮硫杆菌SoxY 和SoxZ蛋白的氨基酸序列[10]在NCBI数据库中的登录号分别为YP_314324.1和YP_314323.1
实验过程所需的生物信息数据库、服务器和软件如下:基因和蛋白质序列检索数据库NCBI(http://www.ncbi.nlm.nih.gov/),蛋白质数据库UniProt(http://www.uniprot.org/),蛋白质结构数据RCSB PDB(http://www.rcsb.org/pdb/home/home.do),蛋白质结构分析与评估在线服务器(https://www.came.sbg.ac.at/prosa.php),Discovery Studio(DS)3.5分子模拟平台。
1.2 实验方法 1.2.1 蛋白质序列分析和同源建模将SoxY 和SoxZ蛋白的氨基酸序列分别导入Discovery Studio,利用Macromolecules模块下的BLAST Search(NCBI Server)在PDB数据库中搜索合适的蛋白质结构作为模板。通常情况下,建模时使用多个模板结构会比只用一个模板结构的效果更好[9],所以根据搜索结果分别选取PDB序列号为2NNC_A、2OXG_Y、2OX5_Y和2OXH_Y的蛋白质[6]作为SoxY的模板,其与SoxY的序列一致性依次是45%、43%、41%、41%;选取PDB序列号为2OXG_Z、2OXH_Z、2OX5_Z和1V8H_A的蛋白质作为SoxZ的模板,其与SoxZ的序列一致性依次是39%、39%、37%、35%。Align Sequences to Templates用于进行目标序列和模板序列的多重比对,结果见图 1,红色标注的是完全保守的氨基酸残基。由于SoxY的模板N端结构解析不完全,造成SoxY蛋白N端有38个氨基酸无法形成对应的空间结构,除此之外其结构完整,但是缺失的部分并不影响我们后续研究的进行。最后利用Bulid Homology Models进行多模板序列对目标序列的同源建模,模型数设置为20个。最终根据模型的能量评估值分别选取PDF Total Energy值最低的构象作为模型初始结构。

|
| 图 1 目标序列和模板序列的多重比对结果 Fig. 1 Sequence alignments between target sequences and template sequences |
在分辨率为1.4Å的条件下,Véronique Sauvé等解析得到Paracoccus pantotrophus的SoxYZ蛋白的天然结构(PDB:2OXG)。在晶体中,SoxYZ能形成更复杂的异质四聚体结构,但是这种四聚体是没有生物学意义的,在溶液中,SoxYZ只能以二聚体形式存在。在其相互作用界面处有20个氢键和1个盐桥参与维持SoxYZ二聚体空间结构的稳定,相互作用导致SoxYZ的溶剂可及表面积(solvent accessible surface,SAS)(1 121±18)Å2,分别占SoxY和SoxZ表面积的20%和18%。这里我们选择2OXG为模板来搭建SoxYZ二聚体模型。将同源建模所得SoxY 和SoxZ蛋白初始结构分别与2OXG的Y链和Z链拟合,最终得到SoxYZ二聚体初始结构[11]。为消除初始模型中不利的分子接触与缺陷,需要利用分子力学的方法来进行优化。这里我们利用Simulation模块中的Minimization在CHARMm力场下固定碳骨架连续用最陡下降法(steepest descent)和共轭梯度法(conjugate gradient)对初始模型进行能量最小化处理,以0.001kcal/mol为收敛依据,直至RMS<0.001kcal/mol,最后得到稳定的二聚体结构[12, 13]。
1.2.3 SoxYZ二聚体模型三维结构质量评估为检验同源建模所得二聚体模型的合理性,本文采用三种方法对优化后的模型进行评估。PROCHECK是用来评价模型三维结构的立体化学参数,评价结果生成的Ramachandran图中横坐标表示蛋白质或多肽链立体结构中肽键内α碳原子和羰基碳原子间的键的旋转度psi(ψ),纵坐标指α碳原子和氮原子间的键的旋转度phi(φ),理论上psi和phi都可自由变化,但键的转动会带动其他原子一起转动,所以在实际中由于分子各个基团的空间障碍和作用力的影响,Ramachandran图中就出现了氨基酸的允许和不允许构象;VERIFY_3D用来确定模型三维结构和它自身氨基酸序列之间的兼容性;PROSA用来判断蛋白质氨基酸残基之间的相互作用能[14]。
1.2.4 SoxYZ二聚体相互作用分析利用DS分子模拟平台中的Macromolecules模块分析了SoxY和SoxZ亚基间的疏水相互作用、氢键作用,而对两亚基间静电相互作用和相互作用能的分析由Simulation模块完成。
2 结果与分析 2.1 SoxYZ蛋白三维结构特征描述将预测所得SoxYZ二聚体模型和模板2OXG进行Cα骨架叠加(图 2),均方根偏差(RMSD)为0.728Å。SoxY和SoxZ亚基所含氨基酸残基数分别为117和 104,它们都拥有类似免疫球蛋白的β折叠片,属于典型的Greek key拓扑结构,每个亚基所含7个β折叠片呈反向平行方式排列,分别位于两个平面内,一侧3个,另一侧4个。SoxYZ二聚体中两亚基空间结构的明显差别是SoxZ亚基N端连接第2个和第3个β折叠片的长loop环(氨基酸残基序列号23~44)、SoxY亚基N端的右手螺旋结构(氨基酸残基序列号42~52)及C端高度保守的氨基酸残基链-G-G-C-G-G-,其所含半胱氨酸残基正是硫化合物的结合位点[6]。
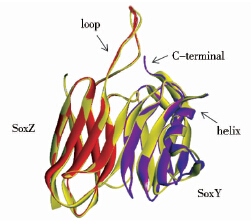
|
| 图 2 SoxYZ和2OXG(黄色)的Cα骨架叠加图,红色为SoxZ亚基,紫色为SoxY亚基 Fig. 2 Superimposition of α-carbon backbones of SoxYZ on 2OXG (yellow),SoxZ subunit was shown in red,SoxY subunit was shown in purple |
利用Discovery Studio 分子模拟平台,SoxYZ二聚体经PROCHECK程序评估后产生的Ramachandran图如图 3(a)所示,由统计分析可知氨基酸残基在允许区域的分布为98.9%,这充分说明蛋白质三维结构中各个氨基酸残基之间所形成的二面角是合理的。对不同区域的统计划分不包括甘氨酸(图 3中三角形)和脯氨酸(图 3中正方形)在内[15],所以位于不合理区域的氨基酸残基仅为SoxZ亚基的ALA42,它位于远离SoxY亚基C端的loop区,对活性中心的影响较小,故这个不合理角度是可以接受的。Verify Protein(Profiles-3D)评估程序给出了二聚体中各个氨基酸残基的Verify Score值[图 3(b)],仅有SoxY的PRO102、SOXZ的ARG6和ILE7这3个氨基酸残基的得分小于0,结果分析显示SoxYZ二聚体的Verify Score值为92.19,接近Verify Expected High Score值100.32,且明显大于Verify Expected Low Score值45.14,这说明蛋白质模型三维结构与自身氨基酸序列的兼容性是合理的。
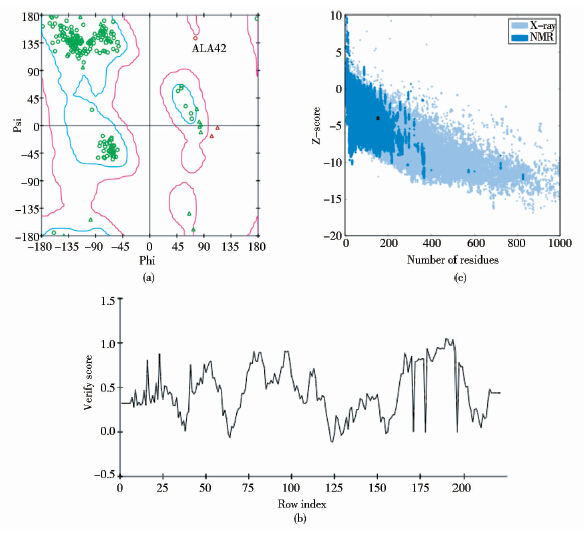
|
| 图 3 SoxYZ蛋白模型质量评估结果图 Fig. 3 The quality assessment and results of SoxYZ protein model (a) The ramachandran result (b) The Verify-3D evaluatioin result (c) The PROSA evaluatioin result |
经PROSA服务器在线评估后产生的结果如图 3(c)所示,图中阴影部分是已被实验确定的RCSB PDB 数据库中所有与SoxYZ蛋白大小相似的蛋白质的Z-score值,SoxYZ蛋白在图中用黑色圆点表示,其Z-score值为-3.98,处于已知结构合理的蛋白质的Z-score分布区域中,这说明所我们构建的SoxYZ蛋白模型在能量上是合理的。
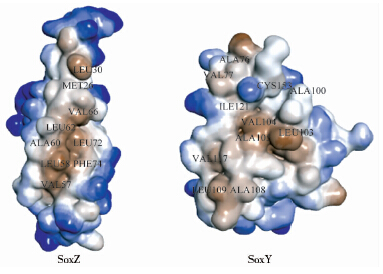
2.3 SoxYZ二聚体相互作用分析 2.3.1 疏水相互作用分析利用DS模拟平台中的Calculate Solvent Accessibility程序包分别计算SoxYZ二聚体和两单体的溶剂可及表面积,结果显示SoxYZ二聚体的SAS值为10 922.9Å2,因二聚体间的相互作用被包埋在内部的SAS值为1 738.14Å2,分别是SoxY和SoxZ蛋白SAS值的13.9%和13.5%。图 4是SoxYZ二聚体界面处疏水性氨基酸残基的分布图,蓝色为亲水性氨基酸残基,棕色为疏水性氨基酸残基,颜色深浅和亲疏水性大小有关,亲水性到疏水性的区间与模板相同,均为-4.5~4.5。SoxZ亚基的MET26、LEU30、VAL57、LEU58、ALA60、LEU62、VAL66、LEU72和PHE74,SoxY亚基的ALA76、VAL77、ALA100、LEU103、VAL104、ALA105、ALA108、LEU109、VAL117、ILE121和CYS153残基表现出了典型的疏水性质,两亚基间可形成较强的疏水界面。蛋白质界面分析发现SoxY和SoxZ亚基的Polar Contact Surface Area值分别为474.35Å2、380Å2;Nonpolar Contact Surface Area值分别为415.32Å2和468.46Å2,利用半经验法[16]对SoxYZ二聚体的疏水率(hydrophobicity)进行定量描述
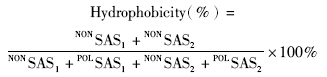
|
| 图 4 SoxYZ 二聚体界面处的疏水分布蓝色为亲水残基,棕色为疏水残基 Fig. 4 Hydrophobicity distribution of SoxYZ heterodimer at interface,hydrophilic residues were shown in blue, hydrophobic residues were shown in brown |
SoxYZ二聚体界面处共含有12个氢键和1个Π键来维持其三维结构的稳定,该Π键由SoxY:TYR116的苯环和SoxZ:LYS75:NZ形成,属于Pi-Cation作用类型。界面处各氨基酸残基间的氢键相互作用如图 5所示,紫色和绿色线性分子分别表示SoxY和SoxZ亚基的界面氨基酸残基,黑色的虚线表示氢键,黑色的实线代表Π键,表 1列出了二聚体结合界面处所有形成氢键的氨基酸残基及其氢键键长,它们主要是由一侧亚基上氨基酸残基的羰基氧和另一侧亚基氨基酸残基的酰胺氢相互作用所形成。由表 1可知结合界面处氨基酸残基间的氢键键长均小于2.7Å,属于短强氢键,它们是维持蛋白质二聚体三维结构稳定性的主要作用力。
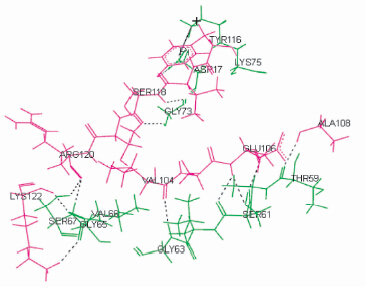
|
| 图 5 SoxYZ 二聚体结合界面处氨基酸残基间的氢键 Fig. 5 The hydrogen bonds of residues at the interface of SoxYZ heterodimer SoxY was shown in purple,SoxZ were shown in green,dotted line was the hydrogen bond,solid line was the pi bond |
| SoxY | SoxZ | Distance (Å) |
| GLU106:HN | SER61:OG | 2.498 4 |
| GLU106:HN | SER61:O | 2.309 8 |
| ALA108:HN | THR59:O | 2.354 8 |
| TYR116:HH | ASP17:OD2 | 2.111 7 |
| SER118:HN | GLY73:O | 2.359 8 |
| LYS122:HN | GLY65:O | 1.781 8 |
| LYS122:HZ3 | VAL66:O | 2.126 |
| GLU106:O | SER61:HN | 2.134 1 |
| VAL104:O | GLY63:HN | 1.988 9 |
| ARG120:O | SER67:HN | 1.971 8 |
| ARG120:O | SER67:HG | 1.909 5 |
| SER118:O | GLY73:HN | 2.222 3 |
利用Simulation模块下的Calculate Electrostatics程序包来计算SoxYZ二聚体的静电势,原子半径和电荷的分配选择Delphi Default为模板,结果如图 6所示。以SoxZ亚基在左侧SoxY亚基在右侧为正面结构,SoxYZ二聚体表面存在大面积的负电势区域,而水平旋转180°后SoxYZ二聚体表面则呈现大量连续的正电势区域。图 7给出了二聚体中各氨基酸残基的平均电势,其区间范围为-57.48~45.86KT/e (K为Boltzmann常数,T为热力学温度,对应298K,e为单位电荷)。半胱氨酸残基5Å范围内SoxY亚基的氨基酸残基GLY75、ALA76、ASN99、LYS122、MET123、GLY124、GLY152、CYS153和SoxZ亚基的THR28、LEU30、ARG31、GLY65形成的正电势区域与SoxY亚基C端区域的ALA100、GLY151、GLY154、GLY155形成的负电势区域形成静电互补[图 6(c) ]。
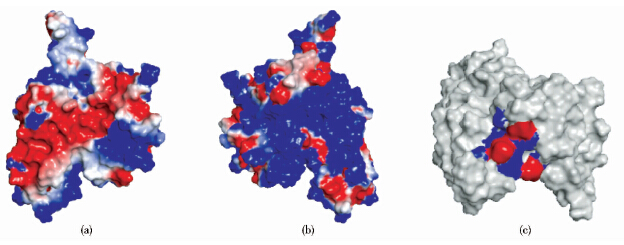
|
| 图 6 SoxYZ 二聚体的静电势分布图,蓝色为正电势区,红色为负电势区 Fig. 6 The electrostatic potential distribution of SoxYZ heterodimer,positive potential was shown in blue,negative potential was shown in red (a) The front view (b) The back view (c) The overhead view |
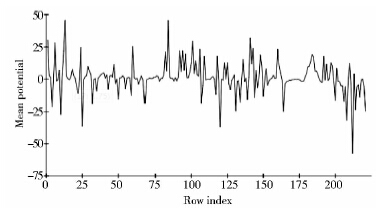
|
| 图 7 SoxYZ二聚体各氨基酸残基的的平均电势 Fig. 7 The mean potential of residues in SoxYZ heterodimer |
为了确定SoxYZ二聚体相互作用过程中的重要氨基酸残基,在CHARMm力场下我们计算了亚基SoxZ和SoxY在界面处各个氨基酸残基的相互作用能。计算结果显示亚基SoxZ和SoxY的总相互作用能为-404.781 72kcal/mol,其中VDW作用能和静电作用能分别为-80.925 13kcal/mol和-323.856 57kcal/mol,静电作用能明显大于VDW作用能,说明静电作用是促使二聚体结构形成的主要驱动力。表 2列出了相互作用能小于-1kcal/mol的重要氨基酸残基,与SoxY亚基C端有强相互作用能的氨基酸残基包括THR28、ARG31、LYS32、SER64、GLY65、VAL66、SER67,它们对SoxYZ二聚体活性区域结构的稳定起着重要作用。
| Residue | VDW interaction energy (kcal/mol) | Electrostatic interaction energy(kcal/mol) | Interaction energy (kcal/mol) |
| SoxZ_LYS19 | -0.824 118 | -31.513 | -32.337 101 |
| SoxZ_PRO25 | -0.135 21 | -12.752 5 | -12.887 7 |
| SoxZ_MET26 | -1.026 6 | -10.167 6 | -11.194 2 |
| SoxZ_GLU27 | -0.465 773 | -33.150 398 | -33.616 199 |
| SoxZ_THR28 | -2.345 91 | -7.174 84 | -9.520 75 |
| SoxZ_ARG31 | -3.398 | -12.815 | -16.212 999 |
| SoxZ_LYS32 | -0.643 3 | -40.405 3 | -41.048 599 |
| SoxZ_ASN56 | -0.370 578 | -2.939 41 | -3.3099 9 |
| SoxZ_VAL57 | -0.288 606 | -10.432 3 | -10.720 9 |
| SoxZ_THR59 | -3.169 59 | -9.709 61 | -12.879 2 |
| SoxZ_ALA60 | -1.450 45 | -4.305 4 | -5.755 85 |
| SoxZ_SER61 | -4.603 52 | -18.835 199 | -23.438 7 |
| SoxZ_LEU62 | -4.661 43 | 3.641 14 | -1.020 29 |
| SoxZ_GLY63 | -2.238 38 | -19.810 101 | -22.048 5 |
| SoxZ_SER64 | -2.202 58 | -24.550 699 | -26.753 3 |
| SoxZ_GLY65 | -2.387 16 | -17.396 5 | -19.783 701 |
| SoxZ_VAL66 | -8.402 96 | -19.755 699 | -28.158 701 |
| SoxZ_SER67 | -5.200 77 | -11.766 6 | -16.967 4 |
| SoxZ_ASN69 | -0.357 426 | -14.538 7 | -14.896 1 |
| SoxZ_TYR71 | -6.182 28 | -5.632 23 | -11.814 5 |
| SoxZ_LEU72 | -4.617 73 | -12.834 1 | -17.451 799 |
| SoxZ_GLY73 | -5.473 72 | -1.288 4 | -6.762 12 |
| SoxZ_PHE74 | -2.867 24 | -9.487 58 | -12.354 8 |
| SoxZ _LYS75 | -3.815 98 | -58.547 901 | -62.363 899 |
蛋白质的一级结构决定其三级结构,一般认为序列同源性大于30%的蛋白质即为同源蛋白,它们有着相似的结构和功能[14]。本研究所选蛋白质模板与未知蛋白质的同源性均在35%以上,保证了建模初始结构的准确性。分子力学常采用一次求导法来寻找能量处于极小值状态时的分子构象,最常用的两种方法为SD和CG。SD法适用于初始构象远离能量最低点时,此时优化能量下降快,方向变化和优化的幅度都较大,但是当构象处于能量最低点时,势能倒数接近0,导致收敛较慢;而CG法在体系接近能量最低点时收敛速度很快,但容易陷入局部势阱,对初始结构要求较高[17],因此我们连续使用SD和CG法对初始结构进行能量最小处理,最终得到稳定的SoxYZ二聚体结构。
二聚作用是蛋白质-蛋白质相互作用的一种简单形式,在分子水平上定量地阐明蛋白质分子间的亲和力和相互识别机制,是正确认识蛋白质聚集体结构和功能之间关系并合理地设计出有用的蛋白质聚集体的关键[18]。研究表明,模板2OXG中因相互作用而减少的SAS值与模型SoxYZ相比略小,但是减少的SAS值分别占模板SoxY和SoxZ的比值却比模型SoxYZ的大,这可能与SoxYZ模型和模板2OXG界面处的半保守及非保守的氨基酸残基分布有关;疏水相互作用分析发现该二聚体作用界面处氨基酸残基表现出典型的疏水性特征,而蛋白质表面以亲水性氨基酸为主,属亲水性蛋白,与Sauvé等[6]的实验吻合。由于SoxYZ蛋白中仅有一个半胱氨酸残基,即硫化合物共价结合位点,所以两亚基间无二硫键形成,氢键成为稳定二聚体构象的主要作用力。静电相互作用分析显示,SoxY亚基C端被SoxZ亚基的9个氨基酸残基形成的正电中心环绕,这可能有助于含硫阴离子与半胱氨酸残基的Sγ硫原子共价结合。二聚作用是一种有效而灵活的调节机制,相互作用能分析说明静电作用是SoxYZ二聚体形成的主要驱动力,不稳定的含硫化合物离子与二聚体的结合可能是对自身的一种保护作用。
利用同源建模法我们首次构建了脱氮硫杆菌的SoxYZ二聚体模型并进行了结构分析,这为今后进行SoxYZ蛋白晶体结构的研究提供了依据。虽然评价结果证明该预测模型是合理的,但是仍需要进一步的试验来验证。
4 结 论以多结构为模板利用同源建模法构建了稳定的SoxYZ二聚体结构;采用PROCHECK、VERIFY_3D和PROSA三种方法从结构和能量两方面验证了所建模型的合理性;SoxYZ二聚体相互作用分析发现SoxYZ的疏水率为50.85%,界面处有12个短强氢键和1个Π键参与维持二聚体结构的稳定,两亚基表面呈现明显的正负电势互补;界面处氨基酸残基作用能的分析揭示了两亚基的结合模式和重要残基,静电作用是促使二聚体结构形成的主要驱动力,其中SoxZ亚基的残基Thr28、Arg31、Lys32、Ser64、Gly65、Val66、Ser67对SoxY亚基活性位点构象的稳定有重要作用。
致谢 本文的工作受到华南理工大学生物科学与工程学院本科生创新基金重点项目的资助与支持,感谢生物科学与工程学院信息室孟志忠副教授和各位同学的支持与帮助。
| [1] | 贡俊,张肇铭.脱氮硫杆菌氧化硫化氢过程中的生物氧化和化学氧化.环境科学学报,2006,26(3):477-482. Gong J, Zhang Z M. Biological and chemical oxidation during oxidation of hydrogen sulfide by Thiobacillus denitrificans. Acta Scientiae Circumstantiae, 2006,26(3):477-482. |
| [2] | 李绪.脱氮硫杆菌在工业废气和废水脱硫脱氮中的应用研究.天津:天津大学,化工学院,2008. Li X. Study on treatment of Thiobacillus denitrificans in flue gas and waste water denitrification and desulfurization processes. Tianjin: Tianjin University, School of Chemical Engineering and Technology,2008. |
| [3] | Friedrich C G, Bardischewsky F, Rother D, et al. Prokaryotic sulfur oxidation. Current Opinion in Microbiology, 2005,8(3): 253-259. |
| [4] | 郑彦彬,王威.生物脱硫技术在煤化工领域应用的可能性.煤化工,2006,34(2):54-56. Zheng Y B, Wang W. Possible applications of biological desuiphurization technology in coal chemical Industry. Coal Chemical Industry, 2006,34(2):54-56. |
| [5] | 张忠智,鲁莽,魏小芳,等.脱氮硫杆菌的生态特性及其应用.化学与生物工程,2005, 22(2):52-54. Zhang Z Z, Lu M, Wei X F, et al. Ecological characters and application of Thiobacillus denitrificans. Chemistry & Bioengineering, 2005,22(2):52-54. |
| [6] | Sauvé V, Bruno S, Berks B C, et al. The SoxYZ complex carries sulfur cycle intermediates on a peptide swinging arm. The Journal of Biological Chemistry, 2007,282(32):23194-23204. |
| [7] | Mohapatra B R, Gould W D, Dinardo O, et al. An overview of the biochemical and molecular aspects of microbial oxidation of inorganic sulfur compounds. CLEAN-Soil Air Water, 2008, 36(10-11): 823-829. |
| [8] | 张志刚.耐热β-半乳糖苷酶的结构分析及同源建模.现代食品科技,2013,29(4):706-709. Zhang Z G. Structure analysis and homology modeling of thermostable β-galactosidase. Modern Food Science and Technology, 2013,29(4):706-709. |
| [9] | 李军.纤维素酶E4的同源建模和分子对接研究.广州:华南理工大学,生物科学与工程学院,2012. Li J. Study on Homology Modeling and Molecular-docking Simulation of Cellulase E4. Guangzhou: South China University of Technology, School of Bioscience & Bioengineering, 2012. |
| [10] | Beller H R, Chain PS G, Letain T E, et al. The genome sequence of the obligately chemolithoautotrophic, facultatively anaerobic bacterium Thiobacillus denitrificans. Journal of Bacteriology, 2006,188(4): 1473-1488. |
| [11] | 徐钰.几类重要蛋白质的分子动力学模拟研究.长春:吉林大学,化学学院,2012. Xu Y. Theoretical Studies on the Catalytic Mechanisms of Several Important Enzymes. Changchun: Jilin University, College of Chemistry,2012. |
| [12] | Bagchi A, Ghosh T C. A structural study towards the understanding of the interactions of SoxY, SoxZ, and SoxB, leading to the oxidation of sulfur anions via the novel global sulfur oxidizing (sox) operon. Biochemical and Biophysical Research Communications, 2005,335(2):609-615. |
| [13] | Ray S, Bagchi A. Structural analysis of the mode of interactions of SoxB protein with SoxYZ complex from Allochromatium vinosum in the global sulfur oxidation cycle. Computational Molecular Biology, 2013,3(1):1-5. |
| [14] | 李书祥.柑橘绿霉菌CYP51的同源模建与分子对接模拟研究.武汉:华中师范大学,生命科学学院,2012. Li S X. Homology Modeling and Molecular Docking Studies of CYP51 from Penicillium digitatum. Wuhan: Central China Normal University, School of Life Sciences,2012. |
| [15] | Laskowski R A, MacArthur M W, Moss D S, et al. PROCHECK-a program to check the stereochemical quality of protein structures. Journal of Applied Crystallography,1993,26(2):283-291. |
| [16] | 李霞.克雷伯杆菌中FNR蛋白三级结构模建及二聚作用研究.青岛:青岛科技大学,化工学院,2012. Li X. Tertiary Structure Modelling and Dimerization Study of the FNR Protein from Klebsiella pneumoniae. Qingdao: Qingdao University of Science & Technology, College of Chemical Engineering,2012. |
| [17] | 陈正隆,徐为人,汤立达.分子模拟的理论与实践.北京:化学工业出版社,2007. Chen Z L, Xu W R, Tang L D. Theory and Practice of Molecular Modeling. Beijing: Chemical Industry Press,2007. |
| [18] | 马晓慧.蛋白质二聚体相互作用和识别的计算机模拟.北京:北京工业大学,生命科学与生物工程学院,2012. Ma X H. Computer Simulation of the Interaction and Recognition of Protein Dimmers. Beijing: Beijing University of Technology, School of Life Sciences & Bioengineering,2012. |
 2015, Vol. 35
2015, Vol. 35




