文章信息
- 李佳鑫,冯炜,王志钢,王彦凤
- LI Jia-xin,FENG Wei,WANG Zhi-gang,WANG Yan-feng
- CRISPR/Cas9技术及其在转基因动物中的应用
- CRISPR/Cas9 System and Its Applications in Transgenic Animals
- 中国生物工程杂志,2015,35(6): 109-115
- China Biotechnology,2015,35(6): 109-115
- http://dx.doi.org/10.13523/j.cb.20150616
-
文章历史
- 收稿日期:2015-3-3
- 修回日期:
在基因组上敲除或者靶向修饰特定基因一直是基因功能研究中的热点问题,20世纪80年代建立起来的利用同源重组的方式进行基因敲除技术由于重组效率较低未能得以广泛应用。以后的研究发现生物体细胞的基因组DNA在发生双链断裂时会通过DNA同源重组途径和非同源末端连接修复途径修复断裂的双链,在这个过程中会发生较高几率的基因插入、缺失和改变,由此出现了基因组定点编辑技术(Targeted genome editing technology),即利用各种方法对染色体DNA双链进行切割,进而诱发DNA损伤修复,在靶位点引入特定的基因突变[1, 2]。利用该技术可以更加方便地探究基因功能,深入准确了解某些遗传疾病的发病机理,也可以通过对目标生物的基因组进行特异性的基因编辑得到需要的性状。
目前基因组定点编辑技术主要包括锌指核酸内切酶(zinc-finger nucleases、ZFNs)、类转录激活因子效应物核酸酶(transcription activator-like effector nucleases、TALEN)以及RNA引导的CRISPR/Cas9核酸内切酶系统。其中ZFNs和TALEN人工内切酶的作用原理是一致的,都是由DNA结合蛋白与FokⅠ核酸内切酶融合而成,在DNA靶位点诱导形成双链断裂损伤(double-stranded break、DSBs)[3]。最近出现的CRISPR/Cas9技术则是通过一段短的RNA序列与DNA靶序列通过碱基互补原则形成双链,结合Cas9蛋白诱导形成DNA双链断裂DSBs。由于CRISPR/Cas9技术与其他技术相比具有操作简单、重组效率高的特点,故被《Science》杂志评为2013年全球十大科技突破之一[4]。本文主要从CRISPR/Cas9技术的建立、作用机理以及在动物基因功能研究中的应用等几方面综述了CRISPR/Cas9技术的发展及在转基因动物中的应用概况。
1 CRISPR/Cas基因组定点编辑技术的建立基因组定点编辑技术一般需具备两方面的功能,一是能够特异性识别基因组上的目标位点,二是能够在目标位点对DNA双链进行切割诱发DNA损伤修复机制[5]。由于CRISPR/Cas系统在这两方面表现出了较强的功能,故自其被发现至今,作为新型的基因组定点编辑技术被广泛应用并且取得了较大的进展。
1.1 CRISPR/Cas系统的发现与分类CRISPR/Cas系统是在多数古生菌和细菌中存在的一种获得性免疫系统,1987年在K12大肠杆菌的碱性磷酸酶基因位点附近首次发现串联间隔重复序列,于2002年被正式命名为成簇规律间隔的短回文重复序列(clustered regularly interspaced short palindromic repeats,CRISPR)[5]。2005年三个研究小组均发现CRISPR的间隔序列(spacer)和宿主菌的染色体之外的遗传物质高度同源,推测其具有对外源遗传物质的免疫作用[6, 7, 8]。2007年CRISPR/Cas系统首次被证明在细菌中可用于抵抗噬菌体入侵[9],之后利用噬菌体侵染嗜热链球菌(Streptococcus thermophilus)的实验证明了CRISPR/Cas系统具有对外源遗传物质进行免疫的功能,由此对CRISPR/Cas系统的研究进入了新的阶段[10]。CRISPR/Cas系统具有一系列的Cas基因,这些基因在宿主应对外源遗传物质入侵时发挥着不同的作用,而且在不同的物种中,Cas基因的种类也各不相同,具有丰富的多样性,会表达出多种同源但功能并不相关的Cas蛋白。综合保守性的Cas蛋白之间的进化关系及Cas基因操纵子的组成方式等因素,通常将CRISPR/Cas系统分为TypeⅠ、Type II 和TypeⅢ这三种不同的类型[11],其中TypeII(CRISPR/Cas9)系统研究得最为彻底,应用也最为广泛。
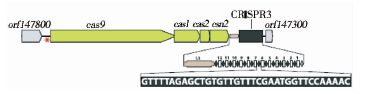
1.2 CRISPR/Cas9系统的作用机制TypeII CRISPR/Cas9系统是最为简单的CRISPR系统,它主要由编码Cas蛋白的基因、一个短的前导区以及一个由间隔序列和重复序列构成的CRISPR的基因座构成。前导区是由一段富含AT碱基的非编码序列构成,作为CRISPR的启动子发挥作用。它的长度一般少于500bp。嗜热链球菌具有最为典型的TypeII CRISPR/Cas9系统,其编码Cas蛋白的基因以操纵子的形式存在,在基因组上的顺序一般为“cas9-cas1-cas2-csn2”,在编码Cas9蛋白基因的上游还存在一个编码tracrRNA的基因。其中csn2基因与CRISPR/Cas9系统获取间隔序列的过程有关(图 1)[12]。Cas9蛋白是TypeII CRISPR/Cas系统的标志性蛋白,由1409个氨基酸构成,含有两个核酸酶结构域,即氨基端的RuvC结构域及位于蛋白质中间位置的HNH结构域。当这两个核酸酶结构域同时具有活性时,Cas9蛋白具有核酸内切酶活性,当其中的一个结构域失活时,Cas9蛋白具有切口酶活性[13]。
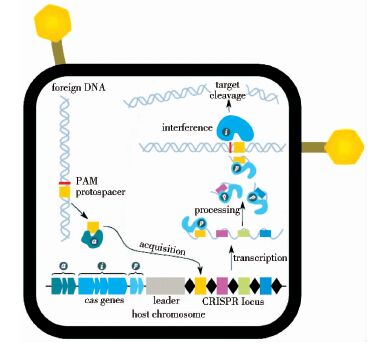
虽然CRISPR/Cas9的作用机制目前并不完全清楚,但其作用过程大致分为三个阶段:首先是间隔序列(spacer)的获取,当外源基因侵入宿主时,激活CRISPR/Cas9系统并发挥免疫活性,靶向分解外源的DNA序列并将原型间隔序列(protospacer)整合到宿主基因组CRISPR位点的5′端;其次是功能基因的表达,此时CRISPR基因座基因被转录为小干扰CRISPR RNAs(crRNA)并且Cas9蛋白被同时合成;最后是对外源基因的干扰,此时crRNA和Cas9蛋白在tracrRNA的指导下形成核蛋白复合物,特异性识别同源外源序列并发挥核酸内切酶活性,分解外源遗传物质,其中间隔序列的获得是关键步骤[12]。间隔序列的获得是指外来DNA序列(protospacer)整合到宿主基因组,整合的位置位于CRISPR的5′端的两个重复序列之间。通常protospacer的5′端或者3′端延伸的几个碱基序列很保守,被称为间隔序列临近基序(protospacer adjacent motifs,PAM),它的长度一般为2~5个碱基,一般与protospacer相隔1~4个碱基。间隔序列的获得一般分为三个阶段:首先识别入侵的核酸和扫描外源DNA潜在的PAM,将临近PAM的序列作为候选protospacer;然后在CRISPR基因座的5′端合成重复序列;最后新的间隔序列整合到两个重复序列之间(图 2)[12, 14, 15]。
由于CRISPR基因座中spacer序列具有多态性,之前整合到CRISPR基因座的spacer基因可以作为标签用于识别不同的细菌株系,所以CRISPR技术最早是作为一种菌种分类的技术用来在遗传背景下对细菌菌株进行命名。随后由于基因定点编辑技术及人工核酸酶的出现,CRISPR/Cas系统开始广泛应用于基因结构及功能的研究中。
基于CRISPR/Cas9系统工作原理,有研究者将其应用于真核细胞的基因组编辑[16, 17],并将tracrRNA 和间隔 RNA(spacer RNA)组合起来,形成一个“向导 RNA”分子(single-guide RNA,sgRNA),然后将其与 Cas9 蛋白混合在一起形成“RNA指导的Cas9核酸酶(RNA-guided Cas9 nuclease)”,实现了在哺乳动物细胞中对特定DNA 位点进行编辑[18]。sgRNA 是根据目标基因的核苷酸序列设计而成的20个寡核苷酸特异识别序列,非常方便设计,由此CRISPR 技术得到了迅猛的发展。
sgRNA序列与基因组上的靶序列通过碱基配对原则结合,然后与Cas9蛋白形成sgRNA/Cas9复合物。通过靶位点附近存在PAM(protospacer adjacent motifs)序列,Cas9蛋白实现与靶位点的成功结合,野生型的Cas9蛋白可以在基因组靶位点诱导形成DNA双链断裂(DSBs)。一个DSBs可以通过两种不同的基因修复途径之一进行修复,一是非同源末端连接(Non-homologous end joining,NHEJ)修复途径,另一个是同源直接修复(Homology directed repair,HDR)途径。NHEJ修复途径是一个易错的修复途径,经常会引入插入/缺失突变导致移码突变或无义突变,有效地破坏靶基因的开放阅读框(Open reading frame,ORF),导致基因功能失活;HDR修复途径则需要有修复模板的存在,该途径用于DSB的修复时可以完整地将修复模板的序列整合到基因组靶位点上,所以经常用于在基因组靶位点引入特殊的基因突变[19]。这两种修复途径通常分别用于基因敲出和定点敲入。
2 CRISPR/Cas9技术在转基因动物中的应用随着对CRISPR/Cas9系统的研究深入,该技术现已广泛地应用于生物领域的各个方面,在动物基因功能和转基因动物研究中包括了knock-out特定基因、定点knock-in目的基因和knock-down靶基因等方面,为转基因动物研究提供了简易、实用的基因组定点编辑技术。
2.1 在基因敲除中的应用CRISPR/Cas9技术由两个基本的成分组成,sgRNA和Cas9蛋白,sgRNA用于识别靶基因,Cas9蛋白用于诱发DSBs。当修复模板序列不存在时,基因组将启动NHEJ修复途径,由于这种修复途径中往往会引入插入/缺失突变,导致DSB位点下游序列发生移码突变或无义突变,从而破坏ORF[20],直接导致目标基因功能的减弱或者完全丧失。但是需要特别指出的是,这些由NHEJ修复途径所造成的突变种类是随机的,所以需要通过进一步的实验验证。
利用CRISPR/Cas9技术构建靶基因konck-out细胞系,首先需要明确靶基因的功能及表达情况,其次需要确定该细胞系单克隆生长情况及生长条件,最后需要对靶基因进行同源序列分析。一般选择在单克隆细胞生长状况良好的细胞系中对同原序列较少且表达状况良好的基因进行突变。sgRNA序列的设计则首先要明确靶基因的编码序列(Coding sequence,CDS)区域,分析相应的基因组结构,明确其外显子区域并在外显子区域上确定待敲除位点。为了最大化地破坏基因的生理功能,一般待敲除位点尽可能选择靠近起始密码子的位置。确定待敲除位点后,可以通过在线的sgRNA设计软件设计sgRNA。常用的sgRNA设计软件包括麻省理工学院的CRISPR Design(http://crispr.mit.edu/)和德国癌症研究中心的E-Crisp(http;//www.e-crisp.org/E-CRISP/designcrispr.html)。根据设计好的sgRNA序列构建与Cas9蛋白共表达的表达载体,转染靶细胞,利用有限稀释法筛选单克隆,验证基因突变。
近期,中国学者利用CRISPR/Cas9技术成功构建了TYR等位基因突变的猪胎儿成纤维细胞系和PARK2和PINK1双基因knock-out的细胞系,并且利用体细胞核移植技术(somatic cell nuclear transfer,SNCT)成功培育出基因突变的猪个体,证实了CRISPR/Cas9技术引入的基因修饰可以通过生殖细胞稳定传递到下一代[21]。同时,韩国学者利用该技术在牛的eGFP转基因体细胞系中成功地实现了对eGFP基因的敲除,敲除效率超过40%[22]。在食蟹猴的单细胞胚胎中注射Cas9 mRNA和sgRNA,同时损坏了Ppar-γ和Rag1两个基因的结构和表达,并且没有脱靶现象发生[23]。这些研究成果说明利用CRISPR/Cas9技术对大型动物进行基因组定点编辑是高效、可靠的,并且可以产生特定基因修饰的个体。
2.2 在基因定点整合中的应用利用CRISPR/Cas9技术在基因组的目标位点产生DSBs后,当修复模板存在的情况下,细胞会激活另外一种DNA损伤修复途径即基因同源重组修复途径(HDR途径)。这种途径会严格按照修复模板来修复断裂的DNA双链,可以在目标基因的突变位点产生精确的特定突变,常用来在基因组目标位点引入外源基因[24]。该途径用于基因组定点编辑的效率比利用NHEJ途径进行基因敲除的效率低,根据细胞类型和状态以及基因位点和修复模板的不同,引入外源基因的效率也有所差别[25]。利用 HDR修复途径对DSB进行修复,修复模板必须与DSB位点上下游的序列高度同源,为此在目标位点引入外源基因时通常需要在外源基因的上下游分别设计一段与DSB位点上下游序列高度同源的同源臂序列[26]。
修复模板既可以是传统的构建在载体质粒上的双链DNA序列也可以是单链的寡聚核苷酸序列(single-strand DNA oligo nucleotides,ssODNs)。对于小片段(<50bp)的外源基因的靶向整合,一般会利用ssODNs 作为修复模板,它的同源臂的长度一般在50~80 bp之间,而且它的整合效率要比质粒作为供体模板进行基因整合的效率高。对于大片段(>100 bp)的外源基因的靶向整合,一般会利用供体质粒(donor plasmid)作为修复模板,它的同源臂的长度一般在800 bp左右[27]。利用HDR修复途径在目标基因的靶位点造成的基因插入突变同样需要利用实验去验证。
利用CRISPR/Cas9技术构建knock-in细胞系其操作流程与构建knock-out细胞系的操作流程基本相同,区别是需要构建一个引入外源基因的修复模板。将设计好的sgRNA和Cas9蛋白的表达载体与修复模板共转染靶细胞,利用有限稀释法筛选单克隆,测序验证基因整合。最近,韩国学者和中国学者合作利用CRISPR/Cas9技术成功地对牛的诱导多功能干细胞(induced pluripotent stem cell,IPSCs)基因组进行了基因定点编辑。他们选择NANOG基因座作为靶向修饰的位点,通过在IPSCs细胞中转染特异于NANOG基因的sgRNA和Cas9蛋白的共表达载体对NANOG基因进行修饰。他们设计了一个可以表达外源基因的供体质粒EFla-eGFP-IRES-Puro,将该质粒与sgRNA/Cas9蛋白的表达载体共转染牛IPSCs细胞,测序验证表明CRISPR/Cas9人工核酸内切酶诱发基因组同源重组,成功地将外源基因整合到NANOG位点并得到表达。进一步研究表明通过显微注射的方法在受精卵中导入NANOG-targeting sgRNAs/Cas9 和供体质粒,GFP基因可以在囊胚中得到表达[28]。这一结果显示出CRISPR/Cas9技术可以在牛的基因组中进行高效特异的基因编辑,为培育转基因家畜提供了有效手段。日本学者利用同样的方法对斑马鱼的基因进行了基因组定点编辑,他们选择evx2基因位点和eng1b基因位点作为靶向修饰的位点。通过将表达外源基因的供体质粒与sgRNA,Cas9 mRNA同时显微注射到斑马鱼的胚胎中,成功地将外源基因整合到目标位点并得到表达[29]。美国学者利用CRISPR/Cas9技术在鼠的胚胎干细胞中进行了外源基因的靶向整合,将外源FLAG-BIO标记抗原和外源GFP基因分别靶向整合到鼠的基因组中的mef2c位点的C端和Oct4位点,发现同源臂的长度与外源基因靶向整合效率有关[30]。
2.3 在基因转录调控中的应用对于特定基因的表达调控是研究基因功能的重要工具,自然产生或者人工合成的某些特异性的DNA结合蛋白可以通过与转录调控因子结合来改变目标基因的表达[31 ,32]。 当Cas9的两个核酸结构域同时失活,这样的Cas9蛋白称之为dCas9蛋白,它不具有对DNA链进行切割的活性,但依旧具有基于sgRNA特异性与靶位点进行结合的能力。dCas9蛋白可以作为一个可编辑的DNA结合蛋白在sgRNA的诱导下通过与目标基因的功能区域结合,调控该基因的转录过程[33]。dCas9系统调控目标基因转录在不同物种中发挥作用的方式也不相同。在原核细胞中,dCas9蛋白基于sgRNA的特异性与基因组中的编码区域结合并阻碍RNA聚合酶的结合与延伸,能够有效抑制目标基因的转录。但是在真核生物体内,利用这种方式调控目标基因转录的效果并不理想[34]。在真核生物体内,转录调控机制相对复杂,基因的表达调控通常是多个转录调控因子的共同作用实现的。所以在真核细胞中,需要建立一个转录调控影响因子与dCas9蛋白融合表达的机制[35]。通过将dCas9蛋白与转录抑制影响因子与转录激活影响因子在体内融合表达,可以高效特异地对目标基因的转录进行调控,也可以通过dCas9技术构建knock-down细胞系来确定目标基因转录的调控区域。Gilbert等[36]在含有GFP基因的293T细胞中同时转染dCas9蛋白和转录调控因子的共表达载体和特异于GFP基因的sgRNA的表达载体。随后的研究表明,KRAB转录抑制子与dCas9蛋白融合表达可以高效抑制目标基因的转录,VP64转录激活子与dCas9蛋白融合表达可以高效增强目标基因的转录。在这种调控方式中,由于转录调控因子是与dCas9蛋白融合表达来调控目标基因的转录,所以对于基因组多个基因的同时转录调控因其调控方向的单一性,限制了CRISPRi系统的大规模应用。为了解决这一问题,近期又发展了相关技术,美国学者将靶位点的特异性识别和转录调控功能综合到一个分子中,通过将CRISPR sgRNA序列和能够招募RNA结合蛋白(该蛋白可以作为转录调控因子)的RNA序列结合起来,形成一个能够同时具有特异性识别目标位点和转录调控功能的RNA分子,称之为支架RNA(scaffolds RNA,scRNA)[37]。实验证明将sgRNA与MS2发卡结构结合能够在人的细胞中招募转录激活因子来调控基因的表达[38]。利用这种方式可以实现同时在基因组的多个基因位点进行不同方向的转录调控,进而调控生物体的代谢途径,展示出CRISPR/Cas9技术在基因功能研究领域的应用前景。
2.4 CRISPR/Cas9技术相对其他基因编辑技术的应用优势在CRISPR/Cas9系统出现之前,ZFNs人工核酸酶与TALEN人工核酸酶是另外两种用于基因定点编辑的技术,CRISPR/Cas9系统与这两种技术相比有明显的技术优势,包括更加容易设计sgRNA、构建与Cas9共表达的表达载体、较高的打靶效率以及在靶位点形成多种突变,更加适用于在多种细胞中进行大规模基因编辑。
在实验设计方面,CRISPR/Cas9系统具有简易性特点,只要根据靶基因序列需要设计一对寡核苷酸(oligo)序列用来编码特异的sgRNA,与Cas9基因蛋白的表达载体连接,几乎可以在基因组的任意位点实现基因定点突变;在对DNA双链进行切割方式方面,野生型的CRISPR/Cas9系统的Cas9蛋白已经证明可以在靶序列的第17个碱基对和第18个碱基对之间(PAM序列5′端的第三个碱基对)进行DNA双链的切割,切割位点十分保守;在基因定点突变的效率方面,CRISPR/Cas9系统与以前的人工核酸酶技术相比具有高效率诱导突变的特点,这一特点在不同物种和细胞都得到了体现[39]。同时,利用CRISPR/Cas9系统还可以实现在同一基因组的多个基因位点进行突变和编辑[40],这极大地提高了基因突变的效率也缩短了整个突变型细胞系构建的时间。
3 CRISPR/Cas9的脱靶效应及改进措施的研究同其他核酸内切酶类似,Cas9也会在目标位点外的其他位点即脱靶位点(off-target)对DNA双链进行切割,产生脱靶效应。由于sgRNA与基因结合时会允许3个以下的碱基的错配,而且sgRNA对靶位点的识别主要是通过14 bp序列(PAM序列及其3′端的11 bp)配对实现,这个长度的序列会在基因组中有较多的相似序列,是造成脱靶现象出现的主要原因。同时脱靶效应还受到多种因素的影响,如Cas9蛋白的浓度以及靶位点序列在基因组中相似序列的数量等[41]。利用CRISPR/Cas9技术对靶基因进行定点编辑,如何去降低脱靶作用是一个必须事先考虑的问题。
在转基因动物细胞系中利用CRISPR/Cas9技术进行基因定点编辑,一般通过两步去降低脱靶作用。首先可以利用一些在线的CRISPR/Cas9 设计软件对sgRNA进行分析,确定其脱靶位点出现的可能性,选取脱靶位点最少的序列作为靶位点进行基因定点编辑;其次是利用滴定的方式转染Cas9和sgRNA的表达质粒,脱靶作用与转染的Cas9质粒的量密切相关,可以通过滴定的方式来保证转染效率的同时降低脱靶作用。2013年zhang等建立的“双缺口CRISPR/Cas9技术”为进一步降低脱靶作用提供了有效的解决办法[42]。这一技术通过突变使得Cas9蛋白的两个核酸结构域RuvC和HNH中的一个失活,可以将野生型的Cas9蛋白转变为只能在DNA双链中的一条链进行切割并形成缺口的缺口酶(Cas9n),同时在靶位点的上下游互补序列上设计一对相邻且位于不同DNA链上的sgRNA,这样Cas9n可以在DNA的两条链上同时形成缺口,产生一种特殊的DSB。由于这种特殊的DSB只有当突变位点两端互补的DNA单链同时出现缺口时才会形成,诱导形成基因突变,所以极大降低了脱靶作用,提高了CRISPR/Cas9对于目标基因靶序列的特异性。利用Cas9n对靶位点进行基因突变的特异性要比野生型的Cas9介导的基因突变要高200到1500倍。
4 结语与展望利用CRISPR/Cas9基因组定点编辑技术可以快速构建转基因细胞系,进而获得性状优良的转基因动物品种。除了对单个基因的定点修饰产生特定的性状改变,CRISPR/Cas9技术还可以对同一基因组中的不同基因进行同时编辑,利用该技术可以研究多个基因的功能及基因间的相互作用机制,甚至可以实现重新构建一条代谢通路的目的,这对于通过动物转基因技术实现对多基因调控性状的改良并培育高产优质新品系具有重要的意义,CRISPR/Cas9技术在转基因动物研究方面具有广阔的发展前景。
作为一种新兴的技术,CRISPR/Cas9系统还有许多未知的方面需要进一步的研究,例如,CRISPR/Cas系统的精细作用机制,Cas9蛋白的催化机制和分子结构,对TypeⅠ和TypeⅢ两型的开发利用,作为一种细菌和古生菌的免疫系统在动物中发挥作用会不会产生副作用,如何进一步提高它的特异性等。毋庸置疑,CRISPR/Cas技术的出现为基因功能和基因组调控研究提供了一个简单、高效的技术,已经成为基因组修饰和动物品种改良的重要工具,将给这一领域的研究带来革命性的变化。
| [1] | Urnov F D, Rebar E J, Holmes M C, et al. Genome editing with engineered zinc finger nucleases. Nat Rev Genet, 2010, 11(9):636-646. |
| [2] | Davis D, Stokoe D. Zinc finger nucleases as tools to understand and treat human diseases. BMC Med, 2010, 8(1):42 doi: 10.1186/1741-7015-8-42. |
| [3] | Walsh R M, Hochedlinger K. A variant CRISPR-Cas9 system adds versatility to genome engineering. Proc Natl Acad Sci U S A, 2013, 110(39):15514-15515. |
| [4] | News 2013 RUNNERS-UP Genetic Microsurgery for the Masses Science, 2013, 342(6165):1434-1435. |
| [5] | Jansen R, Embden J D, Gaastra W,et al. Identification of genes that are associated with DNA l: repeats in prokaryotes. Mol Microbiol, 2002, 43(6):1565-1575. |
| [6] | Bolotin A, Quinquis B, Sorokin A,et al. Clustered regularly interspaced short palindrome repeats (CRISPRs) have spacers of extrachromosomal origin. Microbiology, 2005, 151(8):2551-2561. |
| [7] | Mojica F J, Díez-Villaseñor C, García-Martínez J, et al. Intervening sequences of regularly spaced prokaryotic repeats derive from foreign genetic elements. J Mol Evol, 2005, 60(2):174-182. |
| [8] | Pourcel C, Salvignol G, Vergnaud G,et al. CRISPR elements in Yersinia pestis acquire new repeats by preferential uptake of bacteriophage DNA, and provide additional tools for evolutionary studies. Microbiology, 2005, 151(3):653-663. |
| [9] | Barrangou R, Fremaux C, Deveau H,et al. CRISPR provides acquired resistance against viruses in prokaryotes. Science, 2007, 315(5819):1709-1712. |
| [10] | Marraffini L A, Sontheimer E J. CRISPR interference limits horizontal gene transfer in Staphylococci by targeting DNA. Science, 2008, 322(5909):1843-1845. |
| [11] | Jinek M, Chylinski K, Fonfara I,et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science, 2012, 337(6096):816-821 |
| [12] | Barrangou, Rodolphe, Oost, et al, CRISPR-Cas Systems: RNA-mediated Adaptive Immunity in Bacteria and Archaea. Heidelberg: Springer, 2013.176-196. |
| [13] | Gasiunas G, Barrangou R, Horvath P,et al. Cas9-crRNA ribonucleoprotein complex mediates specific DNA cleavage for adaptive immunity in bacteria. Proc Natl Acad Sci U S A, 2012, 109(39):2579-2586. |
| [14] | Deveau H, Barrangou R, Garneau J E, et al. Phage response to CRISPR-encoded resistance in Streptococcus thermophilus. J Bacteriol, 2008, 190(4):1390-1400. |
| [15] | Horvath P, Barrangou R. CRISPR/Cas, the immune system of bacteria and archaea. Science, 2010, 327(5962):167-170. |
| [16] | Cong L, Ran F A, Cox D,et al.Multiplex genome engineering using CRISPR/Cas systems.Science,2013, 339(6121):819-823. |
| [17] | Mali P, Yang L, Esvelt K M, et al. RNA-guided human genome engineering via Cas9. Science, 2013,339(6121):823-826. |
| [18] | Pyzocha N K, Ran F A, Hsu P D, et al. Rna-guided genome editing of mammalian.Cell, 2014, 1114:269-277. |
| [19] | Wagner J C, Platt R J, Goldfless S J, et al.Efficient CRISPR-Cas9-mediated genome editing in Plasmodium falciparum. Nat Methods, 2014, 11(9):915-918. |
| [20] | Ran F A, Hsu P D, Wright J, et al. Genome engineering using the CRISPR-Cas9 system. Nat Protoc, 2013, 8(11):2281-2308. |
| [21] | Zhou X, Xin J, Fan N, et al. Generation of CRISPR/Cas9-mediated gene-targeted pigs via somatic cell nuclear transfer. Cell Mol Life Sci, 2015, 72(6):1175-1184. |
| [22] | Choi W, Yum S, Lee S, et al. Disruption of exogenous eGFP gene using RNA-guided endonuclease in bovine transgenic somatic cells. Zygote, 2014, 26:1-8. |
| [23] | Niu Y, Shen B, Cui Y, et al. Generation of gene-modified cynomolgus monkey via Cas9/RNA-mediated gene targeting in one-cell embryos. Cell, 2014,156(4):836-843. |
| [24] | Platt R J, Chen S, Zhou Y, et al. CRISPR-Cas9 knock in mice for genome editing and cancer modeling. Cell, 2014, 159(2):440-455. |
| [25] | Ma Y, Ma J, Zhang X, et al. Generation of eGFP and Cre knock in rats by CRISPR/Cas9, FEBS J, 2014, 281(17):3779-3790. |
| [26] | Auer T O, Duroure K, De Cian A, et al. Highly efficient CRISPR/Cas9-mediated knock-in in zebrafish by homology-independent DNA repair. Genome Res, 2014, 24(1):142-153. |
| [27] | Yang H, Wang H, Shivalila C S, et al. One-step generation of mice carrying reporter and conditional alleles by CRISPR/Cas-mediated genome engineering. Cell, 2013, 154(6):1370-1379. |
| [28] | Heo Y T, Quan X, Xu Y N,et al. CRISPR/Cas9 nuclease-mediated gene knock-in in bovine-induced pluripotent cells. Stem Cells Dev, 2015, 24(3):393-402. |
| [29] | Kimura Y, Hisano Y, Kawahara A,et al. Efficient generation of knock-in transgenic zebrafish carrying reporter/driver genes by CRISPR/Cas9-mediated genome engineering. Sci Rep, 2014, 4:6545. |
| [30] | Li K, Wang G, Andersen T, et al. Optimization of genome engineering approaches with the CRISPR/Cas9 system. PLoS One, 2014, 9(8):e105779. |
| [31] | Perez-Pinera P, Kocak D D, Vockley C M, et al. RNA-guided gene activation by CRISPR-Cas9-based transcription factors. Nat Methods, 2013, 10(10):973-976. |
| [32] | Deltcheva E, Chylinski K, Sharma C M, et al. CRISPR RNA maturation by trans-encoded small RNA and host factor RNase III. Nature, 2011, 471(7340):602-607. |
| [33] | Qi L S, Larson M H, Gilbert L A, et al. Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression. Cell, 2013, 152(5):1173-1183. |
| [34] | Gilbert L A, Horlbeck M A, Adamson B,et al. Genome-scale CRISPR-mediated control of gene repression and activation. Cell, 2014, 159(3):647-661. |
| [35] | Cong L, Zhou R, Kuo Y C, et al. Comprehensive interrogation of natural TALE DNA-binding modules and transcriptional repressor domains. Nat Commun, 2012, 24(3):968. |
| [36] | Gilbert L A, Larson M H, Morsut L, et al. CRISPR-mediated modular RNA-guided regulation of transcription in eukaryotes. Cell, 2013, 4(2):442-441. |
| [37] | Zalatan J G, Lee M E, Almeida R, et al. Engineering complex synthetic transcriptional programs with CRISPR RNA scaffolds. Cell, 2015, 160(1-2):339-350. |
| [38] | Mali P, Aach J, Stranges P B, et al. CAS9 transcriptional activators for target specificity screening and paired nickases for cooperative genome engineering. Nat Biotechnol, 2013, 31(9):833-838. |
| [39] | Ikmi A, McKinney S A, Delventhal K M, et al. TALEN and CRISPR/Cas9-mediated genome editing in the early-branching metazoan Nematostella vectensis. Nat Commun, 2014, 5:5486. |
| [40] | Larson M H, Gilbert L A, Wang X, et al. CRISPR interference (CRISPRi) for sequence-specific control of gene expression. Nat Protoc, 2013, 8(11):2180-2196. |
| [41] | Cradick T J, Fine E J, Antico C J, et al. CRISPR/Cas9 systems targeting β-globin and CCR5 genes have substantial off-target activity. Nucleic Acids Res, 2014, 41(20):9584-9592. |
| [42] | Ran F A, Hsu P D, Lin CY, et al. Double nicking by RNA-guided CRISPR Cas9 for enhanced genome editing specificity. Cell, 2013, 154(6):1380-1389. |
 2015,Vol. 35
2015,Vol. 35