文章信息
- 崔成成, 毕艳红, 王应明, 李攀, 杨思达, 黄芬, 曾韦锟, 井申荣
- CUI Cheng-cheng, BI Yan-hong, WANG Ying-ming, LI Pan, YANG Si-da, HUANG Fen, ZENG Wei-kun, JING Shen-rong
- 具有增强蛋白质表达活性序列ER3的筛选及其功能区域的初步鉴定
- Screening of ER3 Sequences Enhanced Protein Expression Activity and Its Functional Regions Identification
- 中国生物工程杂志, 2015, 35(3): 18-24
- China Biotechnology, 2015, 35(3): 18-24
- http://dx.doi.org/10.13523/j.cb.20150303
-
文章历史
- 收稿日期:2014-12-11
- 修回日期:2014-12-30
增强子(enhancer)是基因表达调控过程中的一段DNA序列,通过与启动子相互作用增强目的基因的转录。该序列可位于启动子上游或下游,且不受距离影响,可对异源性启动子发挥作用。自Banerji等[1] 于1981年在真核病毒SV40基因组中首次发现此类增强子序列后,相继在其他病毒及真核基因组中也发现有大量增强子的存在[2, 3]。近年来,由于原核表达系统的应用越来越广泛,对其基因表达调控的研究也更加深入,发现在原核表达系统中也存在类似于真核转录增强的作用机制[4, 5, 6]。韩峰等[7]从大肠杆菌基因组中筛选到一个原核增强子样序列3A,可提高干扰素基因表达水平;从痘苗病毒天坛株基因组中筛选到两个原核增强子样序列VV1和VV16,在大肠杆菌表达系统中能够在转录水平提高lacZ基因的活性;同时,携带有痘病毒增强子样序列的表达载体也能够提高干扰素基因表达水平[8]。
本研究从土壤和污水中富集微生物菌体,提取并酶切基因组,将小于500bp的DNA片段插入检测载体,构建基因文库,通过氯霉素抗性筛选含增强子样序列的菌株。通过检测报告基因的表达量,验证了增强子样序列增强外源基因表达的作用。应用序列测定、分析及突变体研究等手段,初步确定了其功能区域,为该增强子样序列的研究和应用奠定了基础。
1 材料与方法 1.1 材 料 1.1.1 主要试剂限制性内切核酸酶Nde Ⅰ、BamH Ⅰ、Bgl II和T4 DNA连接酶购自TaKaRa公司; Taq DNA聚合酶、dNTP、DNA Marker购自天根生化科技(北京)有限公司;质粒小量快速提取试剂盒、DNA胶回收试剂盒购自北京庄盟生技术有限公司;异丙醇,无水乙醇,IPTG等其他试剂均为国产分析纯试剂。
1.1.2 菌株及质粒大肠杆菌(Escherichia coli,E.coli)DH5α、E.coli BL21(DE3)、表达载体pET-21a、克隆载体pMD-18T、人乳头瘤病毒(human papilloma virus,HPV)主要衣壳蛋白基因L1重组菌株L1-pMD18-T/DH5α、氯霉素乙酰转移酶(chloramphenicol acetyltransferase,CAT)基因重组菌株CAT-pET21a/DH5α增强子样序列检测载体L11-CAT-pET-21a均由本实验室构建并保存。
1.1.3 引物设计及合成HPV 16L1截短序列L11扩增引物:上游:5′-GGAATTCCATATGTCTGTTTGGAGACCTTC-3′(下划线部分为Nde I酶切位点);下游:5′-CGCGGATCCCAGTGAGTCTCCATAAGGTTC-3′(下划线部分为BamHI酶切位点)。增强子样序列测序引物ENHP2 5′- GAGGGGAATTGTTATCCGCTCAC -3′。引物由上海捷瑞生物工程有限公司合成。
ER3功能区域研究引物:扩增ER3的全长引物为正向引物ER3P1和反向引物ER3P2,5′端缺失突变体反向引物为ER3P2,正向引物分别为ER33P1、 ER32P1、ER31P1; 3′端缺失突变体正向引物为ER3P1,而反向引物分别为ER34P2、ER35P2、ER36P2,见表 1。
| Primer names | Primer sequence(BglⅡenzyme site was underlined) | Size(bp) |
| ER3P1 | 5′- GAAGATCTGATCCATTTATTTGAACCAT-3′ | ER3(371bp) |
| ER3P2 | 5′- GAAGATCTATCTGTTGCAGCAGATAACC-3′ | |
| ER31P1 | 5′-GA AGATCTAGCAGCATGAGACCAGCC-3′ | ER31(106bp) |
| ER32P1 | 5′-GAAGATCTTATGTACGATAACAAGCGT-3′ | ER32(171bp) |
| ER33P1 | 5′-GAAGATCTGCATTTGTACGAAGTGCGGT-3′ | ER33(265bp) |
| ER34P2 | 5′-GA AGATCTGCTGCTGGATCAGCTTGG-3′ | ER34(265bp) |
| ER35P2 | 5′-GA AGATCTAATTAAATGCGGCGGTAGCA-3′ | ER35(171bp) |
| ER36P2 | 5′-GA AGATCTCAAATGCATGCAGGAGAAG-3′ | ER17(106bp) |
以实验室保存的HPV 16L1-pMD18T质粒为模板,使用特异性引物扩增L1截短序列L11片段(747bp),胶回收目的片段,并使用引物序列设计的酶切位点(NdeⅠ和BamHⅠ)分别对L11片段和CAT-pET21a进行酶切,再进行胶回收,T4连接酶进行连接,转化DH5α,筛选阳性克隆,进行酶切及测序鉴定,构建正确的命名为L11-CAT-pET-21a。
1.2.2 含增强子样序列菌株的筛选及其酶切鉴定采集土壤和污水样品,加入牛肉膏蛋白胨液体培养基中,37℃振荡培养过夜,富集微生物菌体并抽提基因组,使用Sau3AⅠ对基因组进行酶切,并将小于500bp的片段插入增强子样序列检测载体L11-CAT-pET-21a,转化BL21感受态细胞,构建基因文库;之后,使用不同浓度梯度的氯霉素对文库进行含增强子样序列的菌株筛选,得到重组菌株并命名为ER3-L11-CAT-pET21a/BL21,对重组菌氯霉素抗性进行检测,酶切鉴定。
1.2.3 含增强子样序列菌株报告基因表达检测及序列分析IPTG诱导表达筛选到的含增强子样序列重组菌株,SDS-PAGE检测增强蛋白质表达作用(3次独立重复实验)。通过Image J图像处理软件分析融合基因蛋白表达百分含量,采用GraphPad Prism5对所得数据进行统计学分析,统计结果使用X±SD表示,P<0.05认为具有显著性差异,具有统计学意义。
抽提含增强子样序列重组质粒,将质粒与测序引物ENHP2送至北京六合华大基因科技股份有限公司进行测序。测定所含的增强子样序列:将测得序列输入NCBI 数据库Blastn(http://blast.ncbi.nlm.nih.gov)进行序列同源性比对分析。
1.2.4 增强子样序列的功能区域研究为了确定ER3的功能区域,参考已有的研究方法[9, 10],构建ER3缺失突变体(引物见表 1),插入L11-pET-21a检测载体,测定不同突变体对L11基因蛋白表达的作用情况,确定其功能区域,并通过TFsitescan(http://www.ifti.org/cgi-bin/ifti/Tfsitescan.pl)在线程序对其功能区域的转录因子及其结合位点进行分析。
2 结 果 2.1 L11基因扩增产物的鉴定琼脂糖凝胶电泳分析L11基因扩增产物,可见大小与预期(747bp)一致的目的条带,见图 1。
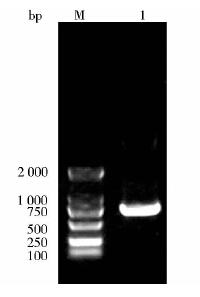
|
| 图 1 琼脂糖凝胶电泳检测L11基因扩增产物 Fig. 1 Analysis of PCR product of L11 with agarose gel electrophoresis M: DNA marker DL2000; 1:The PCR product of L11 gene |
琼脂糖凝胶电泳分析显示,重组质粒L11-CAT-pET21a经Nde I和BamH I双酶切鉴定,可见747bp的L11基因,见图 2。测序结果表明,重组质粒L11-CAT-pET21a构建正确。
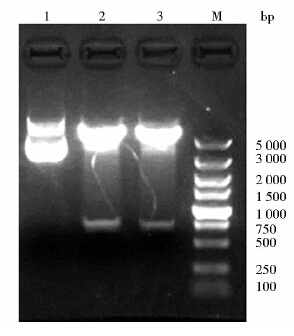
|
| 图 2 重组质粒L11-CAT-pET21a的酶切鉴定 Fig. 2 Identification of recombinant plasmid L11-CAT-pET21a 1: L11-CAT-pET21a plasmid; 2,3: L11-CAT-pET21a was disgested with Nde I/BamH I: M:DNA marker DL5000 |
增强子样序列检测菌株L11-CAT-pET-21a/ BL21的氯霉素抗性生长终浓度为34μg/ml,所以将构建好的基因文库在Amp+(50μg/ml)和氯霉素(34μg/ml、102μg/ml、170μg/ml等)双抗选择培养基上进行筛选,成功筛选到了一株可能含有增强子样序列的菌株ER3-L11-CAT-pET21a/BL21,其氯霉素生长浓度提高了11倍(374μg/ml)。
2.4 增强子样序列增强蛋白表达检测氯霉素抗性筛选到的增强子样菌株ER3-L11-CAT-pET21a/BL21,分析可能是增强子样序列,所以对其进行诱导表达。结果在55kDa处检测到明显条带,与融合蛋白大小相符,而且含有增强子样序列菌株融合蛋白质表达量提高至2.26倍,见图 3,说明ER3序列具有增强蛋白质表达的作用。
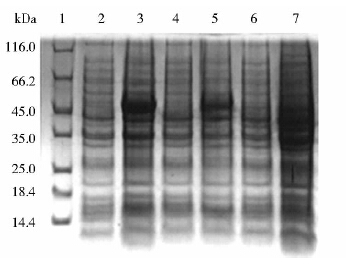
|
| 图 3 SDS-PAGE检测在ER1-L11-pET21a/BL21中L11蛋白的表达 Fig. 3 Analysis of L11 expression in ER1-L11-pET21a/BL21 by SDS-PAGE 1: Protein molecular weight marker; 2: Uninduced ER3-L11-CAT-pET21a/ BL21; 3: Induced ER3-L11-CAT-pET21a/BL21; 4: Uninduced L11-CAT-pET21a/BL21; 5: Induced L11-CAT-pET21a/BL21; 6: Uninduced BL21; 7: Induced BL21 |
对ER3-L11-CAT-pET-21a进行测序,获得ER3核苷酸序列,长度为371bp,序列如下:

对ER3序列进行序列同源性检索发现,该序列与蜡状芽孢杆菌(Bacillus cereus)NC7401的基因序列同源性最高,达到71%;将ER3序列通过NCBI数据库NCBI Conserved Domains Search检测其是否与目前发现的某些基因保守区域具有相似性,发现ER3序列有一个与编码N-乙酰谷氨酸合成酶蛋白相似保守区域,位于200~250bp。
2.6 增强子样序列的功能区域研究为了研究ER3的功能区域,通过DNA序列缺失突变对其进行功能区分析。将ER3片段按100bp大小从5′端和3′端定向缩减,得到6个长度不等的突变体,分别命名为:5′端定向突变体:ER33(Δ1~100bp)、ER32(Δ1~200bp)和ER31(Δ1~265bp);3′端定向突变体:ER34(Δ265~371bp)、ER35(Δ171~371bp)和ER36(Δ107~371bp),见图 4。
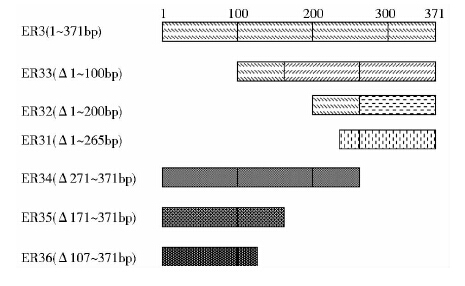
|
| 图 4 ER3序列突变体长度 Fig. 4 The length of ER3 sequence deleted mutants |
将突变体插入载体L11-pET21a中,酶切和测序鉴定正确后,IPTG诱导检测L11蛋白表达水平。实验重复3次,部分SDS-PAGE结果见图 5。
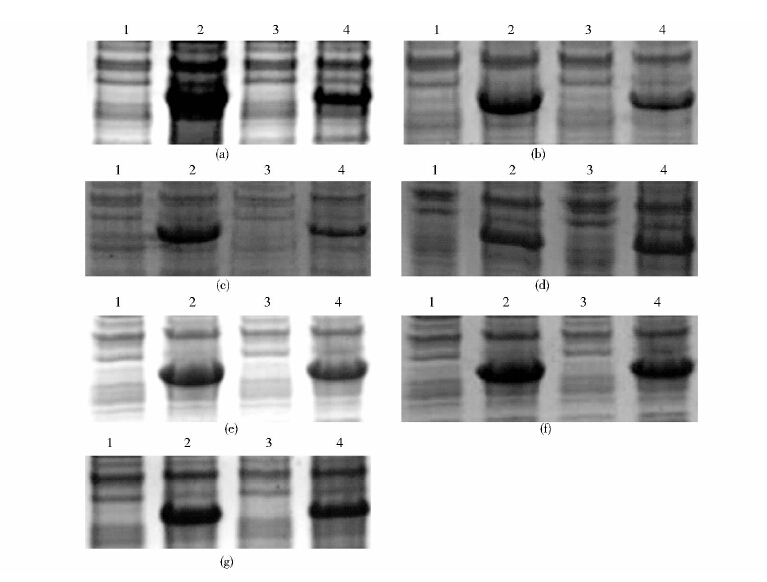
|
| 图 5 SDS-PAGE检测ER3序列突变体菌株外源蛋白L11的表达 Fig. 5 Analysis of L11 protein expressed in ER3 mutational strains by SDS-PAGE (a) ER3 (b)ER33 (c)ER32 (d)ER31 (e)ER34 (f)ER35 (g)ER36;1: Uninduced mutant recombinant strain; 2: Induced mutant recombinant strain; 3: Uninduced L11-pET21a/ BL21; 4: Induced L11-pET21a/ BL21 |
通过Image J分析L11蛋白表达占全菌蛋白质的百分含量,GraphPad Prism5进行统计学分析。由图 6可知,ER3从5′端缺失200bp时,增强蛋白质表达作用与完整序列没有显著性差异改变,剩下106bp时没有增强L11蛋白表达,;而3′端缺失100bp,它的增强活性基本没有改变,这与5′端缺失研究的结果相同,当缺失200bp时,它的增强活性仍然没有改变,只是增强活性稍微降低,结合5′端缺失研究结果,说明171~265bp具有增强活性,而当缺失265bp时,即ER3序列的1~106bp,DNA片段增强活性略微降低,但是仍然与对照有显著性差异,说明这一区域也具有功能活性。因此,ER3序列功能活性区域在1~265bp。
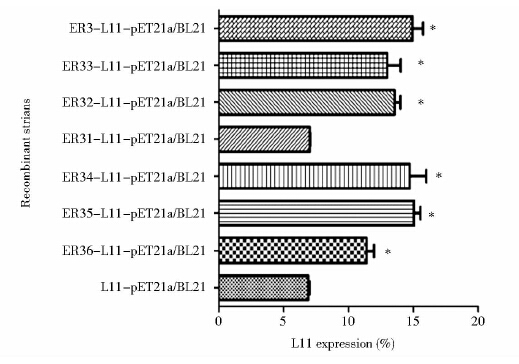
|
| 图 6 ER3突变菌株L11蛋白水平分析 Fig. 6 Quantitative analysis on the levels of L11 in ER3 mutational strains by histogram with L11 levels |
通过TFsitescan在线软件对ER3序列的功能区域进行原核转录因子及其结合位点预测,实线箭头为预测到的结合位点,箭头下标注了相应的转录因子。由图 7可知,ER31~265有多种原核增强样的转录因子结合位点,并且其转录因子除属于Escherichia coli外,还有其他物种(表 2),如Enterococcus faecalis、Pseudomonas aeruginosa、Bacillus subtilis等。由此说明该序列极可能属于转录因子的结合区。
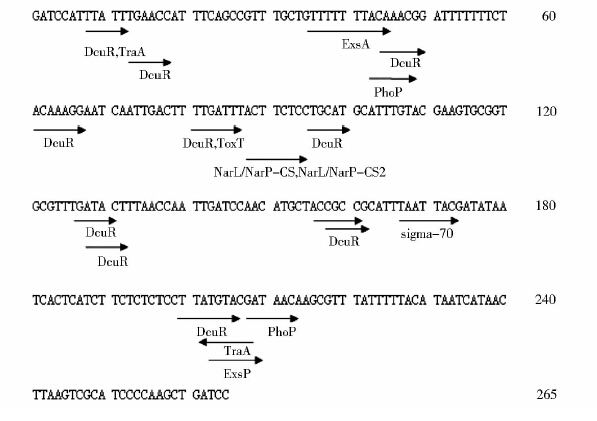
|
| 图 7 ER3功能区原核转录因子结合位点预测 Fig. 7 Putative binding sites of ER3 functional region prokaryotic trascription factors |
| Functional regions | Transcription factor binding sites(TFBS) | Transcription factor (TF) | Promoter (P) | Species |
| ER31~265 | WWYWAA | DcuR | dcuB promoter,dcta promoter,frdA promoter | Escherichia coli |
| TTTTGAT | ToxT | ctx promter,tcpA promoter | Escherichia coli | |
| AAATAAA | TraA[11] | traE1 promoter,iad promoter | Enterococcus faecalis | |
| TNAAAANA | ExsA[12] | pC promoter,pD promoter,pS promoter | Pseudomonas aeruginosa | |
| TTHACA | PhoP[13] | PhoD promoter,PhoA promoter,PstS promoter | Bacillus subtilis | |
| TACYNMT | NarL/NarP[14] | σ70promoter,tac promoter | Neisseria gonorrhoeae | |
| TACYYMT | NarL/NarP[15] | nrf promoter | Escherichia coli K-12 | |
| TATAAT | sigma-70[16] | σ70promoter | Escherichia coli | |
| Note:W=A or T; H=A or C or T; Y=C or T; M=A or C; N=A orG or C or T | ||||
增强子样序列作为一种重要的顺式作用元件,存在于许多生物的基因组中[17],吴淑华等[18]发现SV40病毒DNA Hind III B片段可使IFN-β表达量增加5.9倍。此外,呼吸道合胞病毒(RSV) BamH I G片段、大肠杆菌(JM103-M)约1.0kb的DNA片段在原核表达系统中均能够增强某些原核和真核基因的表达。因此,筛选增强子样序列应用于蛋白质表达具有重要的实用价值。本研究从微生物基因组中筛选到一条原核增强 子样序列ER3,插入到T7启动子上游,可使氯霉素抗性基因的表达水平提高11倍,L11蛋白表达提高2.26倍。接着采用缺失突变的方法对ER3的功能区进行分析,确定了发挥增强作用的区域为1~265bp。这些研究提示,利用原核增强子样序列优化原核表达系统以提高外源基因的表达水平,具有潜在的应用优势。
L11基因属于HPV58型主要衣壳蛋白L1截短序列,L1可在体外表达形成病毒样颗粒(VLPs),为多数HPV预防性疫苗的靶抗原,能够诱导机体产生特异性中和抗体[19, 20]。选择L11为报告基因研究ER3对其表达的增强作用,缘于L1基因上游的poly(A)加尾信号能提前终止L1编码区的转录,并且L1的mRNA序列中含有的特殊信号可抑制其出核或加速降解;此外,L1 的mRNA中AT富含区也能促使翻译终止,造成L1蛋白表达缺失[21]。提高L11-CAT融合蛋白表达水平,对筛选有效的增强子样序列提供了报告基因模型选择的参考依据。此外,HPV主要衣壳蛋白L1在大肠杆菌中的高水平表达,对研发L1蛋白的亚单位疫苗也有一定的意义。
| [1] | Banerji J, Rusconi S, Schaffner W. Expression of a beta-globin gene is enhanced by remote SV40 DNA sequences. Cell, 1981, 27(2 Pt 1): 299-308. |
| [2] | Geyer P K, Green M M, Corces V G. Tissue-specific transcriptional enhancers may act in trans on the gene located in the homologous chromosome: the molecular basis of transvection in Drosophila. EMBO J, 1990, 9(7): 2247-2256. |
| [3] | Heintzman N D, Stuart R K, Hon G, et al. Distinct and predictive chromatin signatures of transcriptional promoters and enhancers in the human genome. Nat Genet, 2007, 39(3): 311-318. |
| [4] | Schnetz K, Rak B. IS5: A mobile enhancer of transcription in Escherichia coli. Proc Natl Acad Sci USA, 1992, 89(4):1244-1248. |
| [5] | Reitzer L J, Magasanik B. Transcription of glnA in E. coli is stimulated by activator bound to sites far from the promoter. Cell, 1986, 45(6): 785-792. |
| [6] | Cenatiempo Y. Prokaryotic gene expression in vitro: transcription-translation coupled systems. Biochimie, 1986, 68(4): 505-515. |
| [7] | 韩峰, 何建新, 廖小慧,等. 原核增强子样序列的筛选及其应用研究. 中华实验和临床病毒学杂志, 2010, 24(3): 175-177. Han F, He J X, Liao X H, et al. Screening and application of enhancer-like sequences 3A. Chinese Journal of Experimental and Clinical Virology, 2010, 24 (3): 175-177. |
| [8] | 韩峰, 秘晓林, 曹茹,等. 痘苗病毒增强子样序列的筛选及应用研究. 中华实验和临床病毒学杂志, 2007, 21(4): 301-303. Han F, Mi X L, Cao R, et al. Screening and application of enhancer-like sequences from vaccinia virus. Chinese Journal of Experimental and Clinical Virology, 2007, 21 (4): 301-303. |
| [9] | Henikoff S. Unidirectional digestion with exonuclease III creates targeted breakpoints for DNA sequencing. Gene, 1984, 28(3): 351-359. |
| [10] | 何建新, 廖小慧, 秘晓林,等. 原核增强子样序列3A的克隆及其结构与功能的研究. 中国生物化学与分子生物学报, 2001, 17(2): 189-193. He J X, Liao X H, Mi X L, et al. Cloning and structure-function of prokaryotic enhancer-like sequence 3A. Chinese Journal of Biochemistry and Molecular Biology, 2001, 17(2): 189-193. |
| [11] | Tanimoto K, Clewell D B. Regulation of the pAD1-encoded sex pheromone response in Enterococcus faecalis: expression of the positive regulator TraE1. J Bacteriol, 1993, 175(4): 1008-1018. |
| [12] | Hovey A K, Frank D W. Analyses of the DNA-binding and transcriptional activation properties of ExsA, the transcriptional activator of the Pseudomonas aeruginosa exoenzyme S regulon. J Bacteriol, 1995, 177(15): 4427-4436. |
| [13] | Eder S, Liu W, Hulett F M. Mutational analysis of the phoD promoter in Bacillus subtilis: implications for Pho Pbinding and promoter activation of Pho regulon promoters. J Bacteriol, 1999, 181(7): 2017-2025. |
| [14] | Householder T C, Belli W A, Lissenden S, et al. cis-and trans-acting elements involved in regulation of aniA, the gene encoding the major anaerobically induced outer membrane protein in Neisseria gonorrhoeae. J Bacteriol, 1999, 181(2): 541-551. |
| [15] | Darwin A J, Tyson K L, Busby S J, et al. Differential regulation by the homologous response regulators NarL and Nar Pof Escherichia coli K-12 depends on DNA binding site arrangement. Mol Microbiol, 1997, 25(3): 583-595. |
| [16] | Siegele D A, Hu J C, Walter W A, et al. Altered promoter recognition by mutant forms of the sigma 70 subunit of Escherichia coli RNA polymerase. J Mol Biol, 1989, 206(4): 591-603. |
| [17] | Kim G Y, Moon J M, Han J H, et al. The sCMV IE enhancer/promoter system for high-level expression and efficient functional studies of target genes in mammalian cells and zebrafish. Biotechnol Lett, 2011, 33(7): 1319-1326. |
| [18] | 吴淑华, 张丽兰, 张秀珍,等. SV40 DNA Hind Ⅲ B 片段增强人 β 干扰素基因在大肠杆菌中的表达. 病毒学报, 1985, 1(4):385-388. Wu S H, Zhang L L, Zhang X Z, et al. SV40 DNA Hind III B fragments increased the expression of human beta interferon gene in Escherichia coli. Chinese Journal of Virology, 1985,1(4):385-388. |
| [19] | Park M A, Kim H J. Optimum conditions for production and purification of human papillomavirus type 16 L1 protein from Saccharomyces cerevisiae. Protein Expr Purif, 2008, 59(1): 175-181. |
| [20] | Kim S N, Jeong H S, Park S N, et al. Purification and immunogenicity study of human papillomavirus type 16 L1 protein in Saccharomyces cerevisiae. J Virol Methods, 2007, 139(1): 24-30. |
| [21] | Leder C, Kleinschmidt J A, Wiethe C, et al. Enhancement of capsid gene expression: preparing the human papillomavirus type 16 major structural gene L1 for DNA vaccination purposes. J Virol, 2001, 75(19): 9201-9209. |
 2015, Vol. 35
2015, Vol. 35




