文章信息
- 曹锡梅, 罗旭光, 梁俊红, 张潮, 白丽娟, 郭大玮
- CAO Xi-mei, LUO Xu-guang, LIANG Jun-hong, ZHANG Chao, BAI Li-juan, GUO Da-wei
- 利用ChIP技术研究SAF基因编码区组蛋白修饰变化
- Determining Histone Modifications on Encoded Region of Serum Amyloid A Activating Transcription Factor Gene in Inflammatory Stimulus Conditions by Chromatin Immunoprecipitation
- 中国生物工程杂志, 2015, 35(3): 8-17
- China Biotechnology, 2015, 35(3): 8-17
- http://dx.doi.org/10.13523/j.cb.20150302
-
文章历史
- 收稿日期:2014-09-17
- 修回日期:2014-12-13
2. 山西医科大学微生物免疫学教研室 太原 030001;
3. 山西医科大学法医学院 太原 030001
2. Department of Microbiology and Immunology, Shanxi Medical University, Taiyuan 030001, China;
3. Department of Forensic Science, Shanxi Medical University, Taiyuan 030001, China
染色体结构通过组蛋白翻译后修饰参与调控基因的表达,这个过程包括乙酰化、甲基化和二磷酸腺苷(ADP)糖基化。以上这些组蛋白的翻译后修饰令核小体形成独特的标记,从而具有调节与DNA和组蛋白相互作用以及与其他蛋白质结合的能力[1, 2]。识别这些特定修饰以及这些修饰在调节染色体重塑中相互作用的方法,将有助于揭示依靠表观遗传性修饰所调节基因表达作用的分子机制。目前测定染色体重塑的主要方法是染色体免疫共沉淀实验(chromatin immunoprecipitation,ChIP),这项技术用于检测和评估DNA与蛋白质之间的相互作用,推定特定DNA序列的染色体结构[3]。该项技术已经广泛应用于研究DNA结合蛋白质、染色体重塑、高分辨基因定位相关的普通转录因子和蛋白质复合物。ChIP技术同DNA基因芯片或克隆技术结合起来可以鉴定新的目的基因,甚至整个基因组环境中目的蛋白的DNA结合位点[2, 4]。
炎症反应基因转录调控过程中,血清淀粉样蛋白A 转录激活因子(serum amyloid A-activating transcription factor,SAF)发挥着重要作用。已知基质金属蛋白酶-1和血清淀粉样蛋白A等基因在该过程中被转录调控,而且后者与淀粉样变性病、风湿性关节炎、胆固醇及高密度脂蛋白代谢障碍等密切相关[5]。本研究拟运用ChIP技术检测SAF基因编码区外显子e4A和e4B区域的组蛋白表观遗传学信息,相关信息将有助于进一步研究SAF自身的转录调控机制和该基因转录与前体mRNA剪接的关系。
1 材料与方法 1.1 THP-1细胞培养和炎性刺激实验选购中国科学院上海生命科学研究院细胞资源中心提供的人单核白血病细胞系(THP-1细胞,TCHu57),按照中心推荐培养条件培养。稳定培养传2~3代后部分细胞给予Lipopolysaccharide(LPS,Sigma,L2880,10μg/ml)刺激,收集LPS刺激不同时间的THP-1细胞用于后续实验。
1.2 ChIPChIP实验步骤大致可以分为三部分:①准备细胞的可溶性染色体,甲醛交联蛋白质-DNA;②免疫沉淀交联的染色体;③半定量聚合酶链反应(PCR)或定量多聚酶链反应(quantitative polymerase chain reaction,qPCR),分析免疫沉淀得到的DNA。
1.2.1 细胞可溶性染色体的准备按照实验设计,细胞计数后离心机4℃、低速800r/min离心、5min收集约2×107个细胞。50ml锥形离心管中用20ml 冰PBS重悬清洗细胞1次,悬液中同时加入200μl 100mmol/L蛋白酶抑制剂PMSF(Cat NO. P8340,北京索莱宝科技有限公司),离心机4℃、低速800r/min离心、5min收集细胞;细胞沉淀用20ml 冰PBS(加200μl 100mmol/L PMSF)重悬后加入547μl 37%甲醛溶液(Sigma,F8775)至终浓度为1%,室温孵育10min;之后悬液中加入2.9ml 1mol/L甘氨酸(Glycine,北京索莱宝科技有限公司),使其终浓度达到0.125mol/L,室温孵育5min终止甲醛的交联作用,离心机4℃、低速800r/min离心、5min收集细胞;20ml冰PBS重悬清洗细胞2次,悬液中分别加入200μl 100mmol/L PMSF、200μl 1μg/ml pepstain(胃酶抑素,F735-1;P-4265)和200μl 1mg/ml aprotinin(抑肽酶,Batch 981532)防止蛋白质降解,离心收集细胞,-20℃冻存备用。
1.2.2 超声波破碎可溶性染色体将上述细胞用含PMSF的SDS nuclei-lysis buffer (上海碧云天生物技术有限公司,P0013G)温和的重悬(2×107个细胞加1ml buffer),冰上孵育10min后置冰上超声裂解细胞。调节细胞超声破碎仪(Sonics&Materials)输出功率为30W,通过不同的超声破碎时间,将可溶性DNA切割至大小为200~1 000bp。离心机4℃、10 000g高速离心10min,收集含有可溶性染色体的上清液于新的1.5ml离心管中,并分装为100μl/管。取50μl上清液65℃去交联,酚-氯仿抽提DNA,并取适量样品电泳检测超声破碎DNA的片段大小。
1.2.3 免疫沉淀交联的染色体蛋白质定量试剂盒2D-Quant Kit定量蛋白浓度(0811-04;GE Healthcare;美国),OD260测吸光度计算DNA浓度。不同的实验样本中加ChIP Dilution Buffer使液体总量为1 000μl(加10μl 100mmol/L PMSF防止蛋白质降解),加60μl 50% Protein A/G PLUS-Agarose(Santa Cruz Biotechnology,Inc.),4℃颠转孵育1h以消除非特异性背景。离心机4℃、3 000g离心1min,收集上清液。另预留50μl上清液作为阳性对照Input,4℃暂存。根据实验设计,不同的实验样本分别加入ChIP级别的特异性抗体免疫沉淀交联的染色体,同时设阴性对照组(加normal mouse IgG),4℃颠转过夜孵育。本实验中所选用的ChIP级别特异性抗体分别为:兔多克隆抗组蛋白H3抗体(rabbit polyclonal to Histone H3,ab1791;abcam Ltd.)、兔多克隆抗组蛋白H3K36 三甲基化抗体[rabbit polyclonal to histone H3(tri methyl K36),ab9050;abcam Ltd.]、兔多克隆抗组蛋白H3K9单甲基化抗体[rabbit polyclonal to histone H3(mono methyl K9),ab9045;abcam Ltd.]和兔多克隆抗RNA聚合酶II CTD重复区5号丝氨酸磷酸化抗体[rabbit polyclonal to RNA polymerase II CTD repeat YSPTSPS(phospho S5),ab5131;abcam Ltd.]。次日不同实验样本分别加80μl 50% Protein A/G,4℃颠转孵育1h,离心机4℃、3 000g离心1min,弃上清液,小心收集免疫沉淀的复合物。依次低盐、高盐、LiCl洗脱缓冲液小心洗涤免疫沉淀复合物各1次,TE缓冲液洗涤2次,小心收集免疫沉淀的复合物。免疫沉淀复合物中加入新鲜配制的Elution buffer 250μl(配制5ml Elution buffer需10%SDS 500μl、1mol/L NaHCO3 500μl、去离子水4ml),室温颠转15min,离心机4℃、3 000g离心1min,收集上清洗脱液于新的1.5ml离心管中。重复1次,合并上清洗脱液,每个实验样本共收集洗脱液约500μl。500μl洗脱液中加入20μl 5mol/L NaCl,65℃水浴过夜逆转甲醛交联(4℃暂存的阳性对照Input同时65℃水浴过夜)。
1.2.4 半定量聚合酶链反应(PCR)分析染色体免疫共沉淀得到的DNA染色体免疫共沉淀得到的DNA作模板进行半定量PCR分析,20μl PCR反应体系中包括2μl 10×Ex Taq Buffer(Mg2+ Plus)、1.6μl dNTP Mixture、0.4μl 10μmol/L上下游引物、2.5μl 溶解沉淀得到的DNA作为模板、0.1μl TaKaRa Ex Taq HS(5U/μl)(DRR006A,TaKaRa),去离子水补足余体积,仔细混匀。加入模板的量要确保信号位于线性剂量反应范围内。PCR扩增仪运行,反应条件如下:
PCR扩增阶段:94℃变性30s;预实验获得最佳退火温度,该温度下退火30s;72℃延伸1min;共35次循环。
产物延伸阶段:72℃延伸5min。
扩增产物经1%琼脂糖凝胶电泳检测后凝胶成像系统扫描保存。具体操作如下:5μl PCR扩增产物加1μl 6×溴酚蓝上样缓冲液,充分混匀后加入加样孔,以5μl DNA Marker(DL5 000bp)作参照,电压为7V/cm,电泳30min。
1.2.5 定量多聚酶链反应(qPCR)分析染色体免疫共沉淀得到的DNA根据半定量PCR获得的最佳退火温度和实验条件设计定量qPCR反应体系和反应条件。总反应体系为20μl,包括2×SYBR Premix Ex Taq (TaKaRa) 10μl、ROX 0.2μl、10μmol/L 上下游引物各0.4μl、模板DNA 1μl、去离子水补足余体积。Stratagene实时荧光定量仪Mx3000p运行,条件为:95℃预变性30s、95℃变性5s、59℃退火30s、72℃延伸30s并收集荧光,40个循环,55~95℃生成溶解曲线,每个样品重复3 次。经过Mx3000p软件分析,得到目的基因和内参GAPDH的循环阈值(Ct)。
2 结 果 2.1 甲醛固定后超声波破碎可溶性染色体为了得到希望长度范围内的DNA片段,首先校准细胞超声破碎仪(Sonics&Materials)输出功率为30W、超声破碎时间为10s,间隔60s,共重复6次,同时调整探头浸入深度,在超声破碎过程中避免液体表面剧烈波动和产生泡沫,图 1 A泳道可溶性DNA被切割至大小为200~1 000bp。实验过程中细胞经甲醛交联固定少于10min或固定10min后延长超声破碎时间至20s(其余条件不变),均没有得到理想片段大小的可溶性DNA(图 1 B、C泳道)。
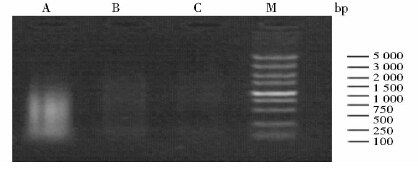
|
| 图 1 超声波破碎可溶性染色体检测电泳图 Fig. 1 Examination of ultrasonic broken soluble chromosome by 1% agarose gel electrophoresis and imaging A,B,C:Different lanes; M.DL5 000 DNA maker |
已知SAF基因产物至少有三种:SAF-1、SAF-2和SAF-3[6, 7],设计适合于外显子e4A、e4B和启动子区Stat3的特异性引物(引物序列见表 1)。以人基因组DNA为模板、PCR扩增,产物符合预期大小、无引物二聚体等,证明引物特异性好。同时以ChIP Input为模板,同样条件PCR扩增产物与预期一致(图 2),证明ChIP过程中试剂及实验步骤有效,免疫沉淀样本成功可用。
| 名称 | 上、下游引物序列 | 位置 | 产物大小(bp) |
| e4A | 5′- GCTCGGCTTATATTTCGG-3′ | 5 621~5 638 | 218 |
| 5′- GATGTTCATTCAGGTGTCC-3′ | 5 820~5 838 | ||
| e4B | 5′-CATGGAGAATGAGTTCTGTAG-3′ | 6 251~6 271 | 237 |
| 5′- CGTGGTGAAGCCTATTGG-3′ | 6 470~6 487 | ||
| Stat3 | 5′-AACGAACTGGATGATGGC-3′ | 1 109~1 126 | 216 |
| 5′-TCTTGCTTGACTGCTTAGG-3′ | 1 306~1 324 |
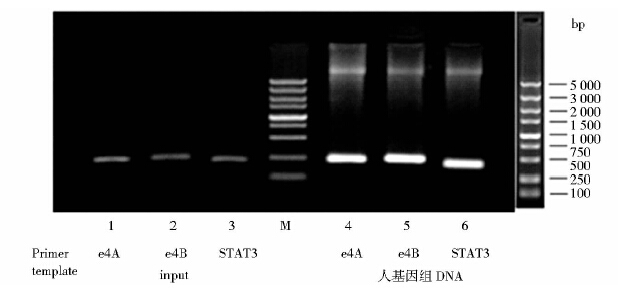
|
| 图 2 引物特异性检测及ChIP Input电泳检测结果 Fig. 2 Detection of primers specificity by PCR amplification |
每个实验样本收集约2×107个细胞,蛋白质定量试剂盒定量不同实验样本蛋白质浓度,OD260测吸光度计算DNA浓度,ChIP实验之前平齐不同样本之间的蛋白质使初始样本之间的DNA量基本一致。
2.4 染色体免疫共沉淀分析SAF基因编码区(e4A和e4B)组蛋白H3、组蛋白H3K36me3、组蛋白H3K9me1和RNA polII CTD 5号丝氨酸磷酸化水平
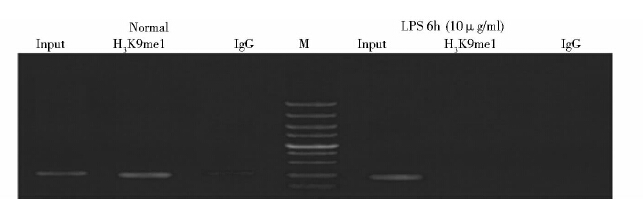
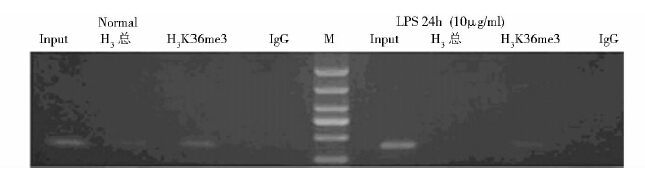
核小体是染色体的基本结构功能单位,由DNA和组蛋白(Histone)构成。组蛋白分子共有5种,分别是H1、H2A、H2B、H3和H4,各两分子的H2A、H2B、H3和H4共同构成核小体的核心,称为组蛋白八聚体(又称为核心组蛋白)。根据实验设计选用抗组蛋白H3抗体、抗H3K9me1和抗H3K36me3抗体观察LPS刺激后SAF基因编码区组蛋白的翻译后修饰变化,以富集得到的DNA为模板进行PCR扩增,由图 3~图 6可见扩增产物符合预期大小,但是随着LPS刺激时间的不同,产物量有明显差异。上述结果间接反映出SAF基因编码区组蛋白修饰的变化,表明LPS刺激后SAF基因编码区(e4A和e4B)组蛋白H3总的信号明显减弱(图 3、图 4、图 6),组蛋白H3第9号赖氨酸单甲基化修饰(H3K9me1)信号减弱(图 5)。SAF基因编码区e4A区域H3K36me3的修饰在LPS刺激前后没有大幅度波动(图 4)。编码区e4B区域组蛋白H3总信号在LPS刺激前就比较弱,LPS刺激24h后明显减弱不易观察到修饰;H3K36me3的修饰在炎性刺激前后均比较低下,LPS刺激24h后修饰水平又有降低(图 6)。Input为阳性对照,IgG为阴性对照。
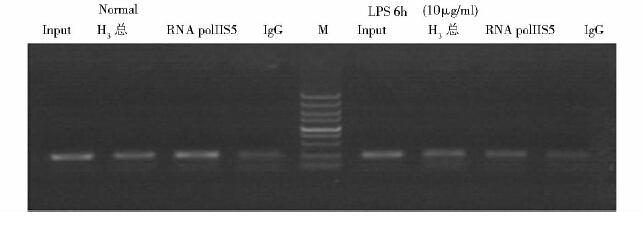
|
| 图 3 抗组蛋白H3和抗RNA polII CTD phS5抗体ChIP沉淀得到的DNA为模板、特异性引物e4A扩增电泳结果 Fig. 3 PCR amplification results of ChIP DNA by specific primers e4A The LPS-stimulated or unstimulated THP-1 cells were colletcted as experimental samples. Antibodies included rabbit polyclonal to Histone H3 and rabbit polyclonal to RNA polymerase II CTD repeat YSPTSPS (phospho S5). LPS: 10μg/ml; Maker: DL5 000 |
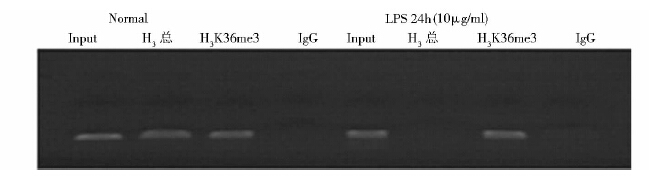
|
| 图 4 抗组蛋白H3和抗组蛋白H3K36me3抗体ChIP沉淀得到的DNA为模板、特异性引物e4A扩增电泳结果 Fig. 4 PCR amplification results of ChIP DNA by specific primers e4A The LPS-stimulated or unstimulated THP-1 cells were colletcted as experimental samples. Antibodies included rabbit polyclonal to Histone H3 and rabbit polyclonal to Histone H3 (tri methyl K36). LPS: 10μg/ml |
|
| 图 5 抗组蛋白H3K9me1抗体ChIP沉淀得到的DNA为模板、特异性引物e4A扩增电泳结果 Fig. 5 PCR amplification results of ChIP DNA by specific primers e4A The LPS-stimulated or unstimulated THP-1 cells were colletcted as experimental samples. Antibodies included rabbit polyclonal to Histone H3 (mono methyl K9). LPS: 10μg/ml; Maker: DL5 000 |
|
| 图 6 抗组蛋白H3和抗组蛋白H3K36me3抗体ChIP沉淀得到的DNA为模板、特异性引物e4B扩增电泳结果 Fig. 6 PCR amplification results of ChIP DNA by specific primers e4B The LPS-stimulated or unstimulated THP-1 cells were colletcted as experimental samples. Antibodies included rabbit polyclonal to Histone H3 and rabbit polyclonal to Histone H3 (tri methyl K36). LPS: 10μg/ml; Maker: DL5 000 |
为反映SAF基因编码区(e4A和e4B)单个核小体上组蛋白H3总信号、H3K36me3和H3K9me1富集程度的变化,我们采用AlphaImager 2200凝胶成像软件分析凝胶谱带。相关实验结果显示,LPS的刺激促使组蛋白H3总信号减弱,软件分析过程中我们将组蛋白H3翻译后修饰信号与组蛋白H3总的信号相比,得出单个核小体组蛋白修饰状况。未受LPS刺激时SAF基因编码区单个核小体组蛋白修饰状况的结果分别为0.93(e4A区域H3K36me3富集程度)和0.45(e4A区域H3K9me1富集程度)。LPS刺激6h后SAF基因编码区e4A单个核小体上H3K9me1的信号明显减弱(图 5),AlphaImager 2200凝胶成像软件未能检出,表明LPS的刺激促使SAF基因编码区e4A单个核小体上H3K9me1的信号减弱。随着LPS刺激作用时间的延长SAF基因编码区e4A组蛋白H3信号随之减弱,至LPS刺激24h AlphaImager 2200凝胶成像软件不易检出组蛋白H3总的信号,但是此时组蛋白H3K36me3的修饰在LPS刺激前后并没有大幅度波动,表明LPS刺激后SAF基因编码区e4A单个核小体上H3K36me3的信号无明显变化。选用抗RNA polII CTD phS5抗体观察LPS刺激前和刺激6h后SAF基因编码区招募RNA polII CTD重复序列5号丝氨酸磷酸化水平的变化,由图 3凝胶电泳结果可见LPS刺激6h后PCR扩增产物量减少,表明LPS刺激6h后SAF基因编码区(e4A 区)RNA polII CTD 5号丝氨酸磷酸化水平降低。为保证实验结果的可信度,ChIP实验均设立对照避免非特异性结合,上述结果可见阳性对照Input有特异性扩增产物,阴性对照IgG未见特异性扩增产物,说明PCR扩增体系工作、实验方法可行,染色体免疫共沉淀结果可靠。
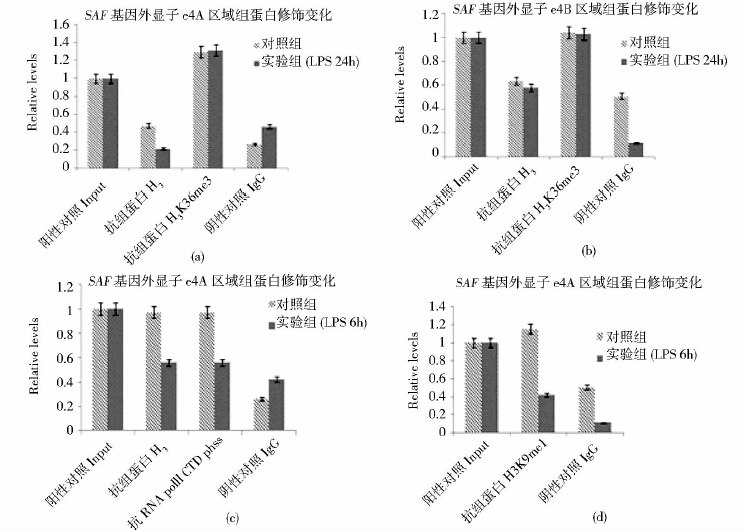
半定量PCR是在扩增终了分析反应平台期的产物,定量qPCR则可以实时检测扩增产物的积累,精确测定与特定DNA区域结合的蛋白质量。相应定量qPCR的结果显示,不同实验点对应的溶解曲线和内参基因GAPDH的溶解曲线均为单峰,且溶解温度大于80℃,没有出现引物二聚体峰和其他非特异性扩增峰(图未示)。相应的扩增曲线比较平坦(图未示),且MxPro软件可以读出其Ct值。根据荧光定量分析仪自动采集的阳性结果的Ct值计算基因表达量,基因表达量根据实验组/对照组=2^-△△Ct计算,其中△△Ct=(Ct目的-Ct内参)实验-(Ct目的-Ct内参)对照。结果显示,SAF基因外显子e4A和e4B区域组蛋白修饰的变化趋势和半定量PCR电泳检测结果基本一致(图 7)。
|
| 图 7 SAF基因外显子e4A和e4B区域组蛋白修饰的变化 Fig. 7 Histone modifications in SAF exons e4A and e4B Expression levels of SAF exons e4A and e4B relative to GAPDH in THP-1 cells determined by qPCR. GAPDH expression was used as an internal control |
染色体免疫共沉淀实验(ChIP)是目前测定染色体重塑的主要方法,这项技术提供了一个强有力的体内研究工具来检测和评估DNA与蛋白质之间的相互作用,可以推定特定DNA序列的染色体结构、真实反映体内基因表达调控的情况。这项技术已经广泛应用于研究DNA结合蛋白质、染色质重塑、高分辨基因定位相关的普通转录因子和蛋白质复合物。本研究运用ChIP技术检测炎性刺激条件下SAF转录激活因子基因编码区外显子e4A和e4B区域的组蛋白表观遗传信息。
3.1 可溶性染色体的交联和超声破碎ChIP实验的基础是得到最优超声条件、获得合适的DNA片段,合适的DNA片段对高分辨率分析来说至关重要。染色体-DNA的交联程度与甲醛的浓度、交联的温度有关,如果染色体-DNA的交联不充分将导致不完全固定,超声破碎时DNA片段的平均长度会小于500bp。如果交联过度会阻碍超声破碎DNA小片段的生成[8]。为了得到希望长度范围的DNA片段首先需将37%甲醛溶液加入细胞悬液中至终浓度为1%,室温孵育固定THP-1细胞10min。超声波破碎可溶性染色体过程中,超声可变性强,因其效率取决于超声声频、声能、处理时间、细胞浓度和细胞类型等,实验优化超声破碎条件为超声破碎仪输出功率30W、超声破碎时间10s、间隔60s,共重复6次,获得200~1 000bp理想大小的DNA片段。
3.2 染色体免疫共沉淀(ChIP)的建立及结果分析ChIP实验过程中的试剂决定实验结果成败,包括特异性抗体的选择。首先,免疫沉淀预实验确定阳性对照Input工作,检测实验体系和实验方法的可行性。其次,确定抗体的合适用量,5μg抗体可以在300~400μg染色体中沉淀出足够的DNA,如果加入抗体量过多会导致整体DNA产量过高。同时设立阴性对照避免非特异性结合[9]。特异性抗体免疫沉淀交联的染色体之前平齐不同实验样本初始DNA量,有学者推荐每个ChIP实验约需3×108个细胞[10],我们实验中所选THP-1细胞约2×107个能够保证实验结果。
分析染色体免疫共沉淀得到的DNA可以用半定量PCR或定量qPCR。随着定量qPCR(quantitative polymerase chain reaction,qPCR)技术的不断发展,其敏感性和高效性不断提高,大大减少了实验过程中的误差、缩短实验时间、避免假阳性或阴性结果。正因为定量qPCR技术的优点吸引着更多的学者选择该方法解决科研问题,如鉴定差异片段的真伪、分析ChIP实验结果等[11]。定量qPCR通过实时检测扩增产物的积累,精确和迅速地测定与特定DNA区域结合的蛋白质量。该评价方法操作步骤少,但是为了准确计算不同条件下实验样本基因表达量需要用内参基因校准,否则会产生假阳性,影响整个实验结果。某些条件下常用内参基因ACTB或GAPDH的表达会变得不稳定,针对本实验选择最适合的内参基因为GAPDH或PGK1[12]。本实验将半定量PCR和定量qPCR结合,分析、检测扩增产物的积累。
3.2.1 组蛋白H3离散有利于RNA polII转录延伸小体由DNA和组蛋白(Histone)构成,是染色体的基本结构功能单位。其中组蛋白分子共有5种,分别是H1、H2A、H2B、H3和H4,各两分子的H2A、H2B、H3和H4共同构成核小体的核心,称为组蛋白八聚体(又称为核心组蛋白)。DNA双螺旋分子缠绕在这一核心上构成核小体的核心颗粒。核小体的核心颗粒之间再由DNA和组蛋白H1构成的连接区连接成串珠样结构。在此基础上核小体进一步旋转折叠,形成纤维状结构及襻状结构,最后形成棒状的染色体容纳于直径仅有数微米的细胞核中。已有研究证实基因的启动子区和增强子附近染色体结构对基因表达有很大影响,即一个凝聚结构阻碍转录因子达到DNA,从而阻止基因表达;一个更为开放的构象和转录因子结合反应相关,介导基因表达[13]。转录因子为了能够结合基因特定的转录识别位点,凝聚的染色体必须首先重塑成一个“开放”构象,从而“装配”转录起始复合物[14]。异染色质蛋白1(heterochromatin protein,HP1)和组蛋白第9号赖氨酸(H3K9)的甲基化调控模板DNA处染色体构象,介导基因转录抑制[15]。我们在实验过程中发现,随着LPS炎性刺激作用时间的延长,SAF基因编码区外显子e4A和e4B区域组蛋白H3的信号逐渐减弱。提示在炎性刺激作用下该区域的组蛋白H3发生离散(eviction),核小体解散、凝聚的染色体重塑呈开放构象、含SAF编码区基因信息的DNA暴露,有利于RNA polII转录延伸。同时,我们观察到外显子e4A区域组蛋白H3K9单甲基化水平在炎性刺激后明显降低,说明炎性刺激作用下不利于HP1结合到组蛋白第9号赖氨酸甲基化位点,HP1不能发挥转录抑制[15]。组蛋白H3K9单甲基化水平的降低和组蛋白H3离散协同促使凝集的染色体重塑呈开放构象,利于RNA polII转录延伸。
果蝇热休克蛋白Hsp70基因是研究RNA polII转录延伸机制的最佳模型。相关研究表明,在Hsp70基因转录激活过程中存在核小体的解散和对转录因子的招募[16],RNA polII的转录过程始于热休克蛋白因子(HSF)、GAGA因子结合位点(GAF)与相应转录元件的结合,核小体的解散对多聚腺苷二磷酸核糖聚合酶(PARP)有依赖性[16]。对酿酒酵母的研究表明,诱导型基因GAL1启动子在转录激活状态下,编码区组蛋白H3同样有离散现象,并且单个核小体H3K9/K14的乙酰化水平升高;若敲除负责H3K9/K14乙酰化的蛋白质Gcn5基因则组蛋白H3的离散现象被抑制[14]。Luebben等通过体外实验证明组蛋白H3的离散是具有乙酰化活性的p300蛋白对组蛋白H3K14乙酰化的结果,体外变异组蛋白H3第14号赖氨酸为精氨酸后人工核小体H3的离散现象消失[17]。染色体免疫共沉淀/DNA基因芯片进行高通量检测人类3 692个基因启动子区核小体的分布情况,显示在转录激活已经表达mRNA的基因转录起始点处会形成无核小体区。部分已经形成转录前起始复合物(pre-initiation complex)但尚未表达mRNA的基因,转录起始点也可形成无核小体区[18]。提示组蛋白H3在SAF基因编码区的离散可能是组蛋白乙酰化的结果,组蛋白的乙酰化可能对聚集在该区域的RNA polII转录延伸有正效应。组蛋白氨基末端区域的不同修饰会影响组蛋白和DNA、组蛋白之间以及染色体重塑蛋白复合体之间的相互作用,组蛋白基因表达控制区域乙酰化增加与转录活性相关。
3.2.2 RNA polII CTD重复序列第5号丝氨酸磷酸化对SAF基因转录活性的影响真核细胞转录过程中,细胞周期蛋白依赖性蛋白激酶CDK7和CDK9参与RNA polII羧基末端 CTD结构域重复多肽序列的磷酸化[19]。CDK7磷酸化转录前起始复合物中RNA polII CTD重复序列第5号丝氨酸,促使RNA polII脱离转录起始复合物进入转录阶段,合成前体mRNA[20]。新生的前体mRNA稳定性差,需进行加帽修饰,之后转录继续向下进行[20]。CDK9磷酸化RNA polII CTD重复序列第2号丝氨酸,同时还磷酸化结合在转录延伸复合物上的负性转录延伸因子DSIF和NELF,解除二者对转录的抑制,促使转录延伸复合物继续转录合成mRNA的全长[21]。实验中我们观察到SAF基因编码区(e4A)RNA polII CTD重复序列第5号丝氨酸磷酸化的聚集水平降低。提示LPS刺激条件下,SAF基因编码区非延伸状态的RNA polII聚集水平减少;我们推断在SAF基因编码区延伸状态的RNA polII聚集增加,有利于合成更多的SAF mRNA剪接/转录子。SAF基因的功能与其磷酸化水平密切相关[22, 23],是否在炎性刺激条件下THP-1细胞中SAF基因产物调控其下游基因的方式是以翻译后修饰方式为主尚需进一步实验证明。
RNA polII CTD重复序列的磷酸化状态和其他因子之间的相互作用影响转录的延伸,而且该区域磷酸化状态的动态变化影响组蛋白的修饰。例如,第5号丝氨酸的磷酸化可以招募Set1,该蛋白质催化组蛋白H3第4号赖氨酸残基三甲基化(H3K4me3)[24];Set2 催化组蛋白H3第36号赖氨酸残基三甲基化(H3K36me3),延伸状态的CTD第2号丝氨酸磷酸化的RNA polII可以稳定Set2,从而催化组蛋白H3第36号赖氨酸三甲基化[25],H3K36me3修饰则是活跃转录的标志[26]。我们观察到LPS刺激后H3K36me3在编码区的修饰水平并无降低,说明此时SAF基因具有转录活性。
致谢 山西医科大学博士启动基金(03201301),山西医科大学科技创新基金(01201401),山西医科大学校青年基金(Q02201207 ),山西医科大学基础医学院“331”科技培植基金计划(201253,201413)。
| [1] | Hebbes T R, Thorne A W, Crane-Robinson C. A direct link between core histone acetylation and transcriptionally active chromatin. EMBO J,1988,7(5):1395-1402. |
| [2] | Statham A L, Robinson M D, Song J Z, et al. Bisulfite sequencing of chromatin immunoprecipitated DNA (BisChIP-seq) directly informs methylation status of histone-modified DNA. Genome Res,2012,22(6):1120-1127. |
| [3] | Weinmann A S, Farnham P J. Identification of unknown target genes of human transcription factors using chromatin immunoprecipitation. Methods,2002,26(1):37-47. |
| [4] | Li Y Y, Tollefsbol T O. Combined chromatin immunoprecipitation and bisulfite methylation sequencing analysis. Methods Mol Biol,2011,791(8):239-251. |
| [5] | Rygg M, Uhlar C M, Thorn C, et al. In vitro evaluation of an enhanced human serum amyloid A (SAA2) promoter-regulated soluble TNF receptor fusion protein for anti-inflammatory gene therapy. Scand J Immunol,2001,53(6):588-595. |
| [6] | Ray B K, Murphy R, Ray P, et al. SAF-2, a splice variant of SAF-1,acts as a negative regulation of transcription. J Biol Chem,2002,277(48):46822-46830. |
| [7] | Ray A, Dhar S, Shakya A, et al. SAF-3, a novel splice variant of the SAF-1/MAZ/Pur-1 family, is expressed during inflammation. FEBS J,2009,276(15):4276-4286. |
| [8] | Kuo M H, Allis C D. In vivo cross-linking and immunoprecipitation for studying dynamic protein: DNA associations in a chromatin environment. Methods,1999,19(3):425-433. |
| [9] | Hainer S J, Pruneski J A, Mitchell R D, et al. Intergenic transcription causes repression by directing nucleosome assembly. Genes Dev,2011,25(1):29-40. |
| [10] | Chen H, Lin R J, Xie W,et al. Regulation of hormone-induced histone hyperacetylation and gene activation via acetylation of an acetylase. Cell,1999,98(5):675-686. |
| [11] | Chen M, Herring B P. Regulation of microRNAs by Brahma-related Gene 1 (Brg1) in smooth muscle cells. J Biol Chem,2013,288(9):6397-6408. |
| [12] | Cao X M, Luo X G, Liang J H, et al. Critical selection of internal control genes for quantitative real-time RT-PCR studies in lipopolysaccharide-stimulated human THP-1 and K562 cells. Biochem Biophys Res Commun,2012,427(2):366-372. |
| [13] | King I F, Francis N J, Kingston R E. Native and recombinant polycomb group complexes establish a selective block to template accessibility to repress transcription in vitro. Mol Cell Biol,2002,22(22):7919-7928. |
| [14] | Govind C K, Zhang F, Qiu H, et al. Gcn5 promotes acetylation, eviction and methylation of nucleosomes in transcribed coding regions. Cell,2007,25(1):31-42. |
| [15] | Lachner M, O'Carroll D, Rea S, et al. Methylation of histone H3 lysine 9 creates a binding site for HP1 proteins. Nature,2001,410(6824):116-120. |
| [16] | Petesch S J, Lis J T. Rapid, transcription-independent loss of nucleosomes over a large chromatin domain at Hsp70 loci. Cell,2008,134(1):74-84. |
| [17] | Luebben W R, Sharma N, Nyborg J K. Nucleosome eviction and activated transcription require p300 acetylation of histone H3 lysine 14. Proc Natl Acad Sci USA,2010,107(45):19254-19259. |
| [18] | Ozsolak F, Song J S, Liu X S, et al. High-throughput mapping of the chromatin structure of human promoters. Nat Biotechnol,2007,25(2):244-248. |
| [19] | Prelich G. RNA polymerase II carboxy-terminal domain kinases: emerging clues to their function. Eukaryot Cell,2002,1(2):153-162. |
| [20] | Rodriguez C R, Cho E J, Keogh M C, et al. Kin28, the TFIIH-associated carboxy-terminal domain kinase, facilitates the recruitment of mRNA processing machinery to RNA polymerase II. Mol Cell Biol,2000,20(1):104-112. |
| [21] | Yamaguchi Y, Takagi T, Wada T, et al. NEL, a multisubunit complex containing RD, cooperates with DSIF to repress RNA polymerase II elongation. Cell,1999,97(1):41-51. |
| [22] | Ray A, Fields A P, Ray B K. Activation of transcription factor SAF involves its phosphorylation by protein kinase C. J Biol Chem,2000,275(50):39727-39733. |
| [23] | Ray A, Yu G Y, Ray B K. Cytokine-responsive induction of SAF-1 activity is mediated by a mitogen-activated protein kinase signaling pathway. Mol Cell Biol, 2002,22(4):1027-1035. |
| [24] | Ng H H, Robert F, Young R A, et al. Targeted recruitment of Set1 histone methylase by elongating PolII provides a localized mark and memory of recent transcriptional activity. Mol Cell,2003,11(3):709-719. |
| [25] | Fuchs S M, Kizer K O, Braberg H, et al. RNA polymerase II carboxyl-terminal domain phosphorylation regulates protein stability of the Set2 methyltransferase and histone H3 di-and trimethylation at lysine 36. J Biol Chem,2012,287(5):3249-3256. |
| [26] | Hossain M A, Chung C, Pradhan S K, et al. The yeast cap binding complex modulates transcription factor recruitment and establishes proper Histone H3K36 trimethylation during active transcription. Mol Cell Biol,2013,33(4):785-799. |
 2015, Vol. 35
2015, Vol. 35




