文章信息
- 王皓, 吴丽, 朱小花, 刘旺旺, 杨公明
- WANG Hao, WU Li, ZHU Xiao-hua, LIU Wang-wang, YANG Gong-ming
- 甲壳素脱乙酰酶的研究概况及展望
- Progress and Prospect of Chitin Deacetylase
- 中国生物工程杂志, 2015, 35(1): 96-103
- China Biotechnology, 2015, 35(1): 96-103
- http://dx.doi.org/10.13523/j.cb.20150114
-
文章历史
- 收稿日期:2014-09-02
- 修回日期:2014-11-25
甲壳素(chitin)又名几丁质,学名为(1,4)-2-乙酰氨基-2-脱氧-β-D-葡聚糖,是自然界生物所含有的一种含量仅次于纤维素的氨基多糖,主要存在于无脊椎动物,如虾、昆虫、海藻、真菌及酵母中。但是甲壳素不溶于水、酸、碱和有机溶剂,所以其没有太大的商业价值,而其脱去乙酰基后的产物壳聚糖则可以溶于酸中并具有抗癌、降低胆固醇、降血压等生物活性,其广泛应用于医药、食品、化工、化妆品等行业[1]。目前生产壳聚糖主要使用的化学法(40%以上氢氧化钠)存在诸多问题。例如,反应时间长、能耗大、产品质量(主要是指平均分子质量和脱乙酰度)不稳定,尤为严重的是排放物造成了巨大的环境污染,对周边生态破坏严重。甲壳素脱乙酰酶(chitin deacetylase,CDA,E.C.3.5.1 .41 )可以水解脱掉甲壳素上的乙酰基。其为解决壳聚糖生产中的环境污染问题提供了一条新的途径,将其与几丁质酶(可将几丁质水解为甲壳寡糖)联用,可以生产出具有特定乙酰化位置的壳聚寡糖,其也可以用来生产脱乙酰度稳定、分子质量分布范围窄的壳聚糖产品等,而这些都是用化学法不能生产的壳聚糖产品。因此,该酶具有重要的工业应用的潜在价值。本文主要从CDA的来源、分离纯化和酶学性质、结构和催化机制、基因的克隆表达及应用前景等方面进行综述展望。 1 CDA的来源
CDA是一种催化甲壳素中N-乙酰基-D-葡糖胺的乙酰胺基水解产生壳聚糖的酶。CDA 来源广泛,包括部分真菌、植物病原体和昆虫等,以真菌最多。该酶在1974年被首次报道,是日本北海道大学的Araki和Ito[2]从接合菌门(Zygomycota)的鲁氏毛霉(Mucor rouxii)中发现的,Araki发现该酶是Mucor rouxii产生壳聚糖的一种方法并对该酶进行了部分纯化和研究。1982年,Kauss等[3]又从子囊菌门(Ascomycota)的植物病原体真菌豆刺盘孢(Colletotrichum lindemuthianum)中发现该酶存在并对其进行了分离纯化,这是首次从非接合菌中发现该酶。以后在这2门中又多次分离得到该酶[4],如Absidia coerulea[4]、Aspergillus nidulans[5]、Penicillium oxalicum[6]、Fusarium proliferatum[7]、Colletotrichum gloeosporioides[8]等。近年来,有人又在担子菌门(Basidiomycota)的Flammulina velutipes[9]、Cryptococcus neoformans[10]中发现CDA的存在。1998年的一个美国专利[11]报道,从城市污水中分离到的一种产碱杆菌属细菌可以胞外大量分泌CDA,且无需纯化,与甲壳素共同培养即可产出壳聚糖,它比真菌更易生长,速率更快,但该菌株的作用模式及其对结晶态的甲壳素是否有更高的活力,没有进一步研究。
2004年,国内的黄惠莉等[12]从海洋泥土中分离出一种产碱属芽孢杆菌,可以分泌CDA,且对其酶学性质进行了研究。此外,国内也有多人从各种真菌中分离出CDA并对其进行酶学性质的研究。例如,2005年,Cai等[13]从42种真菌中筛选出26种有CDA活性,并确定了产酶活力高的构巢曲霉(Aspergillus nidulans)和蓝色犁头霉(Absidia coerulea)所产CDA的酶学性质。2006年,Jiang等[14]比较了几种霉菌(毛霉、根霉、曲霉和青霉)在对数生长末期和稳定期末期的胞内和胞外甲壳素脱乙酰酶的活力。2013年,Sun等[15]从土壤中分离出一种产CDA的红串红球菌(Rhodococcus erythropolis),并对其发酵生产工艺进行了优化。表 1总结了近年来国内外CDA产生菌的部分研究现状。
| 年份 | 研究人 | 主要事件 |
| 1974 | Araki和Ito[2] | 从接合菌纲的双相型真菌Mucor rouxii AHU 6019中发现了CDA |
| 1982 | Kauss等[3] | 从植物病原体Colletotrichum lindemuthianum中提取到CDA,并进行了部分纯化 |
| 1994 | Gao等[4] | 发现Absidia coerulea IFO 5301也可以产生CDA |
| 1995 | Alfonso和Oscar[5] | 从Aspergillus nidulans CECT 2544的菌株自溶培养基中得到一种CDA |
| 1998 | Srinivasan[11] | 一株来自城市污水的产碱杆菌Alcaligenes sp.ATCC 55938能够产生CDA |
| 2005 | Amorim等[16] | 对比了毛霉属中不同菌株产CDA的差异 |
| 2007 | Baker等[10] | 从新型隐球菌(Cryptococcus neoformans)中分离鉴定出4种CDA,并确定了其中3种的产生机制和对真菌细胞壁的作用 |
| 2008 | Yamada等[9] | 从毛柄金钱菌(Flammulina velutipes MH 092086)中分离出CDA,并确定出其基因序列,在毕赤酵母中进行了表达 |
| 2012 | Pareek等[6] | 从草酸青霉(Penicillium oxalicum SAEM-51)中分离纯化出CDA |
| 2013 | Elmekawy等[17] | 研究了一种产碱菌(Alcaligenes sp. ATCC 55938)所产生CDA的催化动力学,及在甲壳素生物转化中的应用 |
| 2013 | Neith等[8] | 研究了源于盘长孢状刺盘孢(Colletotrichum gloeosporioides strain CF-6)的CDA的甲壳素底物特异性 |
| 2004 | 黄惠莉等[12] | 从海洋泥土中分离出一种产碱属芽孢杆菌,可以分泌CDA |
| 2005 | 蔡俊等[13] | 筛选产生CDA的真菌 |
| 2006 | 蒋霞云等[14] | 比较了毛霉、根霉、曲霉和青霉4株霉菌在对数生长末期和稳定期末期的胞内和胞外CDA活力 |
| 2009 | 李朝丽等[18] | 对构巢曲霉(Aspergillus nidulans)产CDA进行活力优化 |
| 2012 | 万芳芳等[7] | 从湘江河岸边的土壤筛选出层生镰孢菌(Fusarium proliferatum) |
| 2012 | 张菁菁等[19] | 从土壤中筛选出一种放线菌中的红球菌属(Rhodococcus)菌 |
| 2013 | Sun等[15] | 从土壤中分离出一种红串红球菌(Rhodococcus erythropolis),并对其发酵生产工艺进行了优化 |
CDA同样在一些昆虫中被发现,如Anopheles gambiae、Apis mellifera、Drosophila melanogaster、Helicoverpa armigera、Mamestra configurata、Tribolium castaneum、Trichoplusia ni等,其中昆虫体内CDA的存在主要与昆虫的中肠围食膜有关[20]。 2 CDA的分离纯化和酶学性质 2.1 CDA的分离纯化
目前,自然界中发现的CDA都是糖蛋白,许多分离纯化蛋白质的方法可用于CDA的纯化。CDA粗酶通常通过离心或(NH4)2SO4溶液沉淀得到。已报道的纯化方法主要为层析法,其具体方法随着分离技术的发展而不断简化。例如,Alfonso等[5]于1995年将构巢曲霉(Aspergillus nidulans)的粗酶提物进行纯化,通过3步层析法,分别将粗酶通过PD-10脱盐柱除去棕色素、通过Mono Q HR 5/5阴离子交换柱与其他干扰酶分离、通过Superose 12 HR 10/30凝胶色谱柱除去受污染的蛋白质,达到电泳纯,纯化倍数为17.4、得率为3.5%、电泳纯的酶的比活力为0.025 U/mg(以几丁质胶体为底物)。随后Ken等[21]于1996年通过饱和(NH4)2SO4溶液沉淀得到豆刺盘孢(Colletotrichum lindemuthianum)的粗酶提取物,采用3步层析法对CDA进行了纯化,达到电泳纯,纯化倍数为944、得率为4.05%、比活力为18.4 U/mg(以乙二醇几丁质为底物),有很大提高。Martinou等[22]于2001年通过1.8 mol/L(NH4)2SO4溶液沉淀得到酿酒酵母(Saccharomyces cerevisiae)的粗酶提取物,采用2步层析法对CDA进行纯化,达到电泳纯,纯化倍数为593、得率为24.5%、比活力为771 U/mg(以六乙酰壳六糖为底物)。Jeraj等[23]于2006年将黑根霉(Rhizopus nigricans)离心后的粗酶提取物通过连续(NH4)2SO4溶液沉淀,再用2步层析法过CM凝胶色谱柱和 DEAE-纤维色谱柱,对CDA进行了纯化,达到电泳纯,纯化倍数为21.2,得率为5.2%、比活力为40.3 U/mg(以乙二醇几丁质为底物)。Nidhi等[24]于2011年将草酸青霉(Penicillium oxalicum)离心后的粗酶液通过超滤,再用2步层析法过CM凝胶色谱柱和 DEAE-纤维色谱柱,对CDA进行了纯化,达到电泳纯,纯化倍数为88.25、得率为11.06%、比活力为55.38 U/mg(以乙二醇几丁质为底物)。
总体来说,随着纯化提取技术的发展,CDA的纯化提取方法也越来越简化,这有利于未来CDA的工业化生产。 2.2 CDA的酶学性质
虽然目前发现的天然CDA都是糖蛋白,有一定的相似性,但由于来源众多,不同来源CDA 的存在位置、最适作用 pH、最适作用温度、碳水化合物含量、底物特异性、分子质量及离子影响等性质存在着较大的差异。根据现有研究结果[2,4,5,9,12,21,24,25,26],大多数细胞外分泌CDA的最佳pH为中性或碱性,范围从7~12,而大多数细胞内几丁质去乙酰酶的最佳pH为4.5~6。几乎所有CDA的最佳温度为50~60℃。
表 2给出了不同来源CDA的性质比较。
| 菌株 | 分子质量(ku) | 糖含量(%) | 酶的位置 | pI | 最适 pH/温度(℃) | 是否被乙酸抑制 | 其他抑制剂 | 激活剂 |
| Bacilus[12] | NA | NA | NA | NA | 4.5~5.0/50 | NA | Zn2+、N-乙酰葡萄糖 | Ca2+ |
| Mucor Rouxii AHU6019[2] | NA | NA | 可溶性细胞质 | NA | 5.5/30 | 乙酸抑制 | 乙二醇壳聚糖、Co2+、Mn2+、Na+、EDTA | 2-巯基乙醇、Zn2+ |
| Mucor Rouxii ATCC24905[25] | 75~80 | 30 | 周质空间 | 3 | 4.5/50 | 羧酸尤其是乙酸抑制 | Co2+、Mn2+、Na+、EDTA | NA |
| Absidicoerulea IFO5301[4] | 75 | NA | 周质空间 | NA | 5/50 | 乙酸抑制 | Fe3+ | NA |
| Aspergillus Nidulans CECT2544[5] | 27.5 | 28 | 胞外 | 2.75 | 7/50 | 不被乙酸抑制 | Ag+、Pb2+、Zn2+、Ca2+、Mn2+、Sn2+、CN-、EDTA 等 | 乙酸根 |
| Colletotrichum lindemuthianum ATCC56676[21] | 31.5~33 | NA | 胞外 | 3.7 | 11.5/60 | 不被乙酸抑制 | Cu2+、Mn2+、Zn2+、Ni2+、Fe2+ | Co2+、EDTA |
| Colletotrichum lindemuthianum DSM 63144[26] | 150 | 67 | 胞外 | 3~5 | 8.5/50 | 不被乙酸抑制 | Mn2+、Zn2+、Cu2+ | Co2+ |
| Penicillium Oxalicum SAEM-51[24] | 53 | NA | 胞外 | 5.2 | 9/50 | 否 | Mo2+、Zn2+、Pb2+、Bi2+、EDTA | Cu2+、Co2+、Fe2+、Cd2+、Mg2+、Ca2+ |
| Flammulina velutipes MH092086[9] | 31 | NA | NA | NA | 7/60 | 是 | Cu2+、Ni2+ EDTA | Co2+、Ca2+、 Zn2+ |
CDA 因微生物种属不同可分为胞内酶和胞外酶,胞内酶存在于细胞质中,胞外酶在提取时相对方便,因而有着更好的应用前景。例如,鲁氏毛霉 (Mucor rouxii)AHU6019 CDA存在于可溶性细胞质中,而豆刺盘孢(Colletotrichum lindemuthianum)ATCC56676和DSM63144以及构巢曲霉(Aspergillus nidulans)CECT2544是胞外酶,发酵液中杂蛋白质少,易于进行纯化,具有潜在生物技术用途的优势[27]。CDA 活性受到多种金属离子和底物等的影响,产物乙酰葡萄糖胺和乙酸对部分CDA 活性有抑制性作用。目前,大多数分离得到的CDA都不需要特殊的金属离子激活。但金属离子对不同来源CDA的活性影响不同,10 mmol/L的Ca2+、Mn2+、Zn2+、Co2+、Fe2+、Cu2+对鲁氏毛霉(Mucor rouxii)来源的CDA均有抑制作用;而豆刺盘孢(Colletotrichum lindemuthianum)来源的CDA则有不同的结果,其中Ca2+,Mg2+对其几乎没有影响,Mn2+使其失去活性的24%,Zn2+、Fe2+使其失去活性的40%~70%,Co2+却能使其完全失去活性。另外,乙酸对前者有强烈的抑制作用,而后者的活性则完全不受影响[18]。
研究表明CDA对不同底物作用的效率不同。Martinou等[28]经过对比发现CDA脱乙酰效率的顺序为:羧甲基甲壳素>甲壳素的脂肪族二元醇>无定型甲壳素>晶态甲壳素。CDA的活性一般对甲壳寡糖高于长链多糖,对可溶性甲壳素衍生物高于结晶态甲壳素。对此,Kafetzopoulos等解释为:当甲壳素分子刚形成时,分子间未完全键合,因此CDA易接近新生甲壳素分子的乙酰基而起作用;一旦甲壳素结晶成微纤维状,CDA就难以进入分子内部,故难以脱除乙酰基[18]。而目前自然界提取的甲壳素均为结晶态甲壳素,这也是目前甲壳素酶法脱乙酰的难点所在。
多数CDA具有较强的专一性,只作用于线性结构的N-乙酰葡糖胺多聚物,但不同酶的最适底物却各不相同。例如,豆刺盘孢的CDA可以高效率的使甲壳四糖、甲壳五糖、甲壳六糖水解脱去乙酰基;鲁氏毛霉和蓝色犁头霉的CDA则可使更长的几丁多糖有高的脱乙酰率;构巢曲霉(Aspergllus niduulans)的CDA当底物为甲壳五糖时显示最高活力,但对甲壳六糖显示低活力[21]。CDA对不同脱乙酰度和不同物理形态的甲壳质的敏感性相差颇大,有一定脱乙酰度的多孔的或无定形的甲壳质比结晶型的甲壳质更容易被酶水解脱去乙酰基[29]。
研究表明,CDA仅能脱固体晶态甲壳素上5%~10%的乙酰氨基葡萄糖残基[30]。Win和Stevens[30]发现在晶体甲壳素处理成超细甲壳素后,反应得到改善,当超细甲壳素用18%甲酸处理后,CDA[来源于蓝犁头霉(Absidia coerulea)]可以将10%脱乙酰度(DD)的甲壳素水解为90%脱乙酰度(DD)的壳聚糖。原因在于甲酸处理可以将甲壳素从相对分子质量2×105降至1.2×104。 3 CDA的结构和催化机制 3.1 CDA的结构
已有的研究表明,CDA属于糖酯酶家族4(carbohydrate esterase family 4,CE-4),其结构特性具有很多相似性。Blair等[31]在2006年首次对CDA(来源于Colletotrichum lindemuthianum)的结构进行了深入分析,发现CDA结构与其他CE-4成员很相似,其采用了一种近似的残缺的(β/α)8折叠的结构。事实上,目前所确定出结构的CE-4成员的结构[30]都是在典型的CE-4(β/α)8折叠基础上的一定程度的“二级结构变换”。CDA与其他CE-4成员的不同处在于其β3股与α2螺旋之间存在一个扩展的环,这对其酸碱催化时形成中间物具有重要影响。此外,CDA存在两个稳定酶蛋白结构的二硫键,一个位于Cys38-Cys237(C38-C237),连接着N端与C端;另一个位于Cys148-Cys152(C148-C152),连接着氨基酸序列MT3与α4螺旋,并使MT3成了活性位点的一侧。
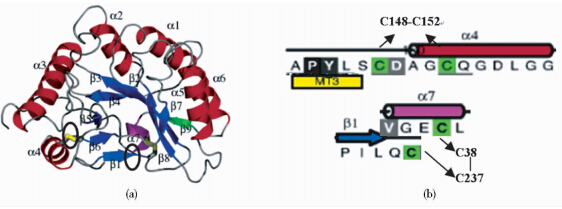
|
| 图 1 CDA的结构 Fig. 1 CDA structure (a) CDA structure,the elliptic part is the disulfide (b) The acid sequences of disulfide bond |
Blair等[31]结合结构数据和生物化学数据发现,CDA(来源于Colletotrachum landemuthaanum)是由一个结构域组成的单核金属酶。已有的研究表明[32]大多数CE-4的催化活性依赖于二价阳离子的存在,如Co2+与Zn2+。而从毕赤酵母和大肠杆菌中表达出的CDA却不受金属螯合剂EDTA的抑制,而且Co2+、Zn2+等多种二价金属离子对其催化活性没有明显效果。故而可推测酶中含有金属离子。Blair等[31]经过电感耦合等离子体-质谱联用仪(ICP-MS)和石墨炉原子吸收光谱(GF-AAS)测定CDA中的金属原子含量,结果发现CDA平均一个酶分子含有0.7~0.8个锌原子。CDA含有一个保守的组氨酸-组氨酸-天冬氨酸三元锌合组,它们相互联系,共同产生酸碱催化作用。
更进一步的结构数据,及其他来源的CDA结构知之甚少,仍有待于以后的研究。 3.2 CDA的催化机制
Tokuyasu等[33]的研究发现CDA(来源于Colletotrachum landemuthaanum)可以强烈地识别出4个底物的乙酰氨基葡萄糖残基,将这4个取代位点从非还原端到还原端分别定义为取代位点-2、-1、0、+1,其中,处于0位点的底物乙酰基被完全脱去,其是催化位点的代表。在此基础上,Blair等[31]进行了进一步的研究,发现,CDA至少使(GlcNAc)2占据0与+1位点才能保持活性。
CDA对壳聚多糖[28]和甲壳寡糖[34]的作用方式也已经有了较多的研究。Martinou等[28]以来源于毛霉菌(Mucor rouxii)的CDA对水溶性壳聚多糖(平均聚合度30)进行随机脱去约32%乙酰的反应。使用1H-NMR和13C-NMR 光谱检测,发现CDA水解乙酰基的反应是多点进攻模式,有3个复合作用点,被水解的底物具有至少3个脱乙酰作用点,而CDA进行一次脱乙酰反应的最大位点数也是3。
Tsigos等[34]对来源于M. rouxii的CDA对甲壳寡糖(2~7个聚合度)作用机制的研究同样表明反应是一种多点进攻模式。脱乙酰后的甲壳寡糖经交替使用两种特别的外切糖苷酶作用,再用HPLC测其序列,再以1H-NMR光谱进一步验证。结果发现寡糖链的长度对酶促作用有很大影响。当甲壳寡糖聚合度少于3时,CDA无酶活性;而N-乙酰壳四糖、N-乙酰壳五糖,CDA能完全脱除其乙酰基;对于N-乙酰壳三糖、N-乙酰壳六糖和N-乙酰壳七糖,虽然它们也能被CDA脱去乙酰基,但还原端残基仍保持完整。
已有的研究表明,酶的水解是一个多点进攻模式,即同时脱去多个位点上的乙酰基,其过程如下:CDA 先与一条底物链结合,然后从结合位置的非还原端开始,脱下数个乙酰基,接着CDA与底物解离,重新与另一条底物结合进行新一轮的脱乙酰反应。这一作用方式也与其他CE-4成员相近[31]。 4 CDA基因的克隆表达
来源于M.rouxii、酿酒酵母S.cerevisiae、豆刺盘孢C.lindemuthianum的CDA基因均已被克隆和测序[34],其所表达的酶蛋白氨基酸序列具有高度同源性,而且都有一个保守域与根瘤蛋白(NodB蛋白),微生物乙酰木聚糖酯酶和木聚糖酶的编码基因中的开放阅读框高度相似。
Caufrier等[35]的研究发现,NodB同源域对于同属CE-4的多种脱乙酰酶具有重要作用,其属于基因的表达区,对包括CDA在内的脱乙酰酶的专一性和脱乙酰效率有关。
国内的蒋云霞等[36]的用保守的特异引物进行 PCR 扩增,结合 RACE 技术,对总状毛霉(Mucor racemosus)甲壳素脱乙酰酶全长cCDA进行了克隆和序列分析,并进行相近物种的比对及三级结构预测,发现总状毛霉的CDA基因共编码 448 个氨基酸,在该基因中部还包含一个含144个氨基酸的多糖脱乙酰酶结构域,约占CDA基因全长的32%,其与米根霉来源的CDA基因的相似度为75%,所预测三级结构经验证具有CDA的脱乙酰功能及多糖脱乙酰酶结构域。
使用不同方法对不同微生物来源的CDA进行克隆表达的结果有较大差异。Shrestha等[37]从C.lindemuthianum UPS9中分离出CDA基因,并使用质粒载体pPIC9K在P. pastoris中进行表达,最后得到分子质量为25 kDa的去糖基化CDA,比活力达到71.67 U/mg,但酶的得率仅为7.2 mg/L。后来Kang等[38]根据现有的一些研究结果,选择pHBM905A作为克隆载体,也将C.lindemuthianum UPS9的CDA基因在P. pastoris中进行了克隆表达,最后得到分子质量为25 kDa的去糖基化CDA以及部分糖基化的分子质量为33 kDa的CDA,糖基化酶的品质得到了提升,比活力达到了77.27 U/mg,酶的得率达到110 mg/L,得率提升很大。
表 3为近年来一些CDA克隆表达的结果对比。
CDA的克隆表达能够大大提高不同微生物来源的CDA的产量,为以后的工业化生产奠定研究基础。
| 年份 | 研究人 | CDA来源 | 表达宿主 | 比活力(U/mg) | 表达形式/得率(mg/L) |
| 2012 | Kang等[38] | C.lindemuthianum UPS9 | P. pastoris | 77.27 | 可溶(110) |
| 2010 | Wang等[39] | A. nidulans | E. coli | 4.17 | 包涵体 |
| 2008 | Gauthier等[40] | R. circinans | P. pastoris | 965.13 | 可溶(0.62) |
| 2004 | Shrestha等[37] | C.lindemuthianum UPS9 | P. pastoris | 71.67 | 可溶(7.2) |
| 1999 | Tsigos等[34] | C.lindemuthianum ATCC56676 | E. coli | 4.22 | 包涵体 |
CDA在脱去甲壳素乙酰基方面具有重大的应用前景,众所周知,当前的脱乙酰基几乎全部采用热浓碱进行,这会产生大量的废弃碱液,造成环境污染和能源浪费,若CDA能够用于甲壳素脱乙酰制备壳聚糖则不存在上面的问题,还能生产出脱乙酰程度均匀、分子质量分布范围窄的高质量壳聚糖产品。例如,依据甲壳素脱乙酰酶的多点进攻可以定点脱去甲壳三糖、四糖等低聚甲壳素的乙酰基,从而得到低聚壳聚糖,而低聚壳聚糖能够得到某些高聚壳聚糖所没有的性质,如更好的抑菌性、水溶性及生物相容性等。
根据CDA在生物体内的功能和性质,还可将其用于人工生物合成壳聚糖,在一些接合菌中,几丁质合成酶同CDA 一起合成细胞壁中的壳聚糖,在这类菌的细胞壁中含有壳聚糖,并且有些真菌的壳聚糖含量及脱乙酰度均很高。例如,在M.rouxii细胞壁中几丁质和壳聚糖的比为 1∶3,A.coerulae中壳聚糖含量占细胞干重的8.4%,脱乙酰度高达95%,可以直接利用它来发酵生产壳聚糖[27]。现已有人工合成壳聚糖的报道,脱乙酰度也高达71%~74%[27]。 6 结论与展望
随着未来对高品质壳聚糖及壳聚寡糖需求的增加,CDA作为一种潜在的壳聚糖绿色生产关键产品,在应用上具有重大前景。这使得当前对CDA的研究成为了一个热点,关于CDA在不同菌种中发现的报道也日渐增多,但其大多数都是在真菌中发现的,由于真菌增殖较慢,大规模生产不实际,故而对其克隆表达研究至关重要。目前CDA的脱乙酰效率均不高,单纯的催化仅能脱去极少量乙酰基,其难点在于晶态甲壳素难以进行脱乙酰水解,故而研究如何处理以破坏掉甲壳素的晶体结构是以后的一个方向。这就需要科研工作者多在其催化机制及底物特异性、酶的构效关系上研究。目前,关于CDA结构的研究仍然非常少,且现有的结构研究已经表明不同来源CDA的结构及催化机制有一定的区别,这些研究对指导酶的克隆表达及未来酶的工业化生产和人工合成具有重大意义,故而可以预计,未来还会有更多关于CDA结构及机制方面的研究。目前,我国关于CDA的研究落后较多,大都只是在分离纯化方面的粗研究,对酶的催化机制及结构方面的研究几乎没有,这就需要我们以后的研究更加深入。相信随着研究的深入,CDA的规模化生产及将其用于生产高质量的壳聚糖将有着更加光明的前景。
| [1] | Kaur S,Dhillon G S. The versatile biopolymer chitosan: potential sources, evaluation of extraction methods and applications. Critical Reviews in Microbiology, 2014, 40(2): 155-175. |
| [2] | Araki Y,Ito E. A pathway of chitosan formation in Mucor rouxii: enzymatic deacetylation of chitin. Biochemical and Biophysical Research Communications,1974, 3: 669-675. |
| [3] | Kauss H, Jeblick W, Young D H. Chitin deacetylase from the plant pathogen colletotrichum- lindemuthianum. Plant Science Letters, 1983,28:231-236. |
| [4] | Gao X D, Katsumoto T, Onodera K. Purification and characterization of chitin deacetylase from Absidia coerulea. Journal of Biochemistry, 1995, 117(2): 257-263. |
| [5] | Carlos A, Oscar M N, Francisco S, et al.Purification of a heat stable chitin deacetylase from Aspergillus nidulans and its role in cell wall degradation. Current Microbiology,1995, 30: 49-54. |
| [6] | Pareek N, Vivekanand V, Saroj S, et al. Purification and characterization of chitin deacetylase from Penicillium oxalicum SAEM-51. Carbohydrate Polymers,2012,(2):1091-1097. |
| [7] | 万芳芳.高产甲壳素脱乙酰酶菌株的筛选及发酵研究.长沙:中南林业科技大学,生命科学与技术学院,2012,55. Wan F F. Screening and Fermentation of the Strains with High Yield Chitin Deacetylase. Changsha: Central South University of Forestry and Technology, The College of Life Science and Technology,2012,55. |
| [8] | Neith P, Stéphane T, Laurent D,et al. Activity of chitin deacetylase from Colletotrichum gloeosporioides on chitinous substrates. Carbohydrate Polymers,2013,96(1):227-232. |
| [9] | Yamada M, Kurano M, Inatomi S, et al. Isolation and characterization of a gene coding for chitin deacetylase specifically expressed during fruiting body development in the basidiomycete Flammulina velutipes and its expression in the yeast Pichia pastoris. FEMS Microbiology Letters, 2008, 289(2):130-137. |
| [10] | Baker L G, Specht C A, Donlin M J, et al. Chitosan, the deacetylated form of chitin, is necessary for cell wall integrity in Cryptococcus neoformans. Eukaryotic Cell, 2007, 6(5):855-867. |
| [11] | Srinivasan.Biotransformation of chitin to chitosan:USA,005739015A.1998-04-14. |
| [12] | 黄惠莉,叶存印,姚云艳. 枯草芽胞杆菌甲壳素脱乙酰酶的筛选及酶学性质. 微生物学通报,2004,31(5):33-37. Huang H L,Ye C Y, Yao Y Y,et al. Screening and properties of chitin deacetylase from Bacillus subtilis. Microbiology China, 2004,31(5):33-37. |
| [13] | 蔡俊,杜予民,杨建红,等.甲壳素脱乙酰酶产生菌的筛选及产酶条件.武汉大学学报(理学版), 2005,51(4):485-488. Cai J, Du Y M, Yang J H, et al. Selective strains and producing conditions of chitin deacetylase. Journal of Wuhan University (Natural Science),2005,51(4):485-488. |
| [14] | 蒋霞云,周培根,李燕,等. 几种霉菌产甲壳素脱乙酰酶活力比较及部分酶学性质.上海水产大学学报, 2006,15(2):211-215. Jiang X Y, Zhou P G, Li Y, et al. Comparisons of activities of chitin deacetylase from mold strains and study of its properties. Journal of Shanghai Fisheries University, 2006,15(2):211-215. |
| [15] | Sun Y Y, Zhang J Q, Wu S J. Statistical optimization for production of chitin deacetylase from Rhodococcus erythropolis HG05. Carbohydrate Polymers,2013,102(10):4. |
| [16] | 刘丽,赵祥颖,田延军,等. 甲壳素脱乙酰酶的研究及应用进展. 山东食品发酵,2009,4:23-25. Liu L,Zhao X Y,Tian Y J. Study and appilcation on chitin deacetylase. Shandong Food Ferment,2009,4:23-25. |
| [17] | Elmekawy A, Hegab H M, El-Baz A, et al. Kinetic properties and role of bacterial chitin deacetylase in the bioconversion of chitin to chitosan. Recent Patents on Biotechnology,2013,7(3): 234-241. |
| [18] | 李朝丽.甲壳素脱乙酰酶的活力优化研究.上海:东华大学,纺织学院,2009,5:53-54. Li C L. Study on Optimizing the Activity of Chitin Deaeetylase.Shanghai: Donghua University, Textile College,2009,5:53-54. |
| [19] | 张菁菁,董文宾,缑敬轩. 几丁质脱乙酰酶菌株的筛选鉴定及酶学性质. 食品工业科技,2012,33(6):244-249. Zhang J J,Dong W B,Gou J X. Screening identification and enzymatic characteristcs of chitin deacetylase strains. Science and Technology of Food Industry, 2012,33(6):244-249. |
| [20] | Zhao Y, Park R D, Muzzarelli R A. Chitin deacetylases: properties and applications. Marine Drugs,2010,8(1):24-46. |
| [21] | Ken T, Mayumi O K, Hayashi, K. Purification and characterization of extracellular chitin deacetylase from Colletotrichum lindemuthianum. Bioscience Biotechnology and Biochemistry,1996, 60:1598-1603. |
| [22] | Martinou A, Koutsioulis D, Bouriotis V. Expression, purification, and characterization of a cobalt-activated chitindeacetylase (Cda2p) from Saccharomyces cerevisiae. Protein Expression Purification,2002, 24(1):111-116. |
| [23] | Jeraj N, Kunic B, Lenasi H. Purification and molecular characterization of chitin deacetylase from Rhizopus nigricans. Enzyme and Microbial Technology,2006, 39(6):1294–1299. |
| [24] | Nidhi P, Vivekanand V, Saroj S. Purification and characterization of chitin deacetylase from Penicillium oxalicum SAEM-51. Carbohydrate Polymers, 2012,87(2):1091-1097. |
| [25] | Kafetzopoulos D, Martinou A, Bouriotis V. Bioconversion of chitin to chitosan: purification and characterization of chitin deacetylase from Mucor rouxii. Proceedings of the National Academy of Sciences, 1993, 90(7): 2564-2568. |
| [26] | Tsigos I, Bouriotis V. Purification and characterization of chitin deacetylase from Colletotrichum lindemuthianum. Journal of Biological Chemistry, 1995, 270(44): 26286-26291. |
| [27] | Cai C G, Yin H P. The Progress of Studies on Chitin Deacetylase Sources, Physicochemical Properties and Biological Engineering. In:Huo P. Proceedings of 2010 First International Conference on Cellular,Molecular Biology, Biophysics and Bioengineering(Volume 7). First International Conference on Cellular,Molecular Biology, Biophysics and Bioengineering, Qigihar,Hei longjiang,China,2010. China, Wuhan Institute of Technology, 2010:4. |
| [28] | Martinou A, Bouriotis V, Stokke B T, et al. Mode of action of chitin deacetylase from M. rouxii on partially N-acetylated chitosans. Carbohydr Res, 1998,311(2):71-78. |
| [29] | 彭益强,方柏山.甲壳素脱乙酰酶的研究概况和最新进展.中国医药工业杂志,2005, 36(11):79-85. Peng Y Q, Fang B S. Progress of chitin deacetylase reaction mechanism and Its gene expression. Chinese Journal of Pharmaceuticals, 2005, 36(11):79-85. |
| [30] | Win N N, Stevens W F. Shrim Pchitin as substrate for fungal chitin deacetylase. Applied Microbiology and Biotechnology, 2001, 57(3):334-341. |
| [31] | Blair D E, Hekmat O, Schuttelkopf A W. Structure and mechanism of chitin deacetylase from the fungal pathogen Colletotrichum lindemuthianum. Biochemistry,2006, 45(31):9416-9426. |
| [32] | Hoell I A, Gustav V K, Eijsink V G H. Structure and function of enzymes acting on chitin and chitosan. Biotechnology and Genetic Engineering Reviews,2010, 27: 331-366. |
| [33] | Tokuyasu K, Mitsutomi M, Yamaguchi I. Recognition of chitooligosaccharides and their N-acetyl groups by putative subsites of chitin deacetylase from a deuteromycete, Colletotrichum lindemuthianum. Biochemistry,2000, 39(30):8837-8843. |
| [34] | Ken T, Mayumi O K, Hayashi K, et al. Cloning and expression of chitin deacetylase from a deuteromycete,Colletotrichum lindemuthianum. Journal of Bioscience and Bioengineering,1999, 87(4):418-423. |
| [35] | Caufrier F, Martinou A, Dupont C. Carbohydrate esterase family 4 enzyme: substrate specificity.Carbohydrate Reseach, 2003, 338(7):687-692. |
| [36] | 蒋霞云,邹曙明,周培根.总状毛霉(Mucor racemosus)甲壳素脱乙酰酶全长cDNA的克隆及序列分析.农业生物技术学报. 2006,14(6):981-985. Jiang X Y, Zou S M, Zhou P G. Cloning and sequence analysis of complete chitin deacetylase(CDA) cDNA from Mucor racemosus. Journal of Agricultural Biotechnology,2006,14(6):981-985. |
| [37] | Shrestha B, Blondeau K, Stevens W F, et al. Expression of chitin deacetylase from Colletotrichum lindemuthianum in Pichia pastoris: Purification and characterization. Protein Expression and Purification,2004,38(2):196-204. |
| [38] | Kang L X, Chen X M, Zhai C. Synthesis and high expression of chitin deacetylase from Colletotrichum lindemuthianum in Pichia pastoris GS115. Journal of Microbiology and Biotechnology,2012, 22(9):1202-1207. |
| [39] | Wang Y, Song J Z, Yang Q. Cloning of a heat-stable chitin deacetylase gene from Aspergillus nidulans and its functional expression in Escherichia coli. Applied Biochemistry and Biotechnology,2010,162(3):843-854. |
| [40] | Gauthier C,Clerisse F,Dommes J, et al. Characterization and cloning of chitin deacetylases from Rhizopus circinans. Protein Expression and Purification,2008, 59(1):127-137. |
 2015, Vol. 35
2015, Vol. 35




