文章信息
- 张旭, 王晶晶, 刘建平
- ZHANG Xu, WANG Jing-jing, LIU Jian-ping
- 基于启动子和宿主改造的酿酒酵母表达系统优化研究
- The Optimization of Saccharomyces cerevisiae Expression System by Mutagenesis of Promoter and Host Strain
- 中国生物工程杂志, 2015, 35(1): 61-66
- China Biotechnology, 2015, 35(1): 61-66
- http://dx.doi.org/10.13523/j.cb.20150109
-
文章历史
- 收稿日期:2014-10-21
- 修回日期:2014-11-19
酿酒酵母(Saccharomyces cerevisiae)是一种低等单细胞真核生物,具有安全、生长繁殖快、培养简单、遗传操作便捷等特点,广泛应用于酿造、食品、医药等领域,一直是基础和应用研究的重要对象。酿酒酵母系统是最早发展起来用于表达异源蛋白的真核表达系统,第一个商品化的重组疫苗—乙肝疫苗即来自酿酒酵母,随后陆续有很多外源基因在该系统中成功表达[1, 2]。
影响外源基因在宿主内表达有多方面因素,包括启动子效率、宿主遗传特性、外源基因拷贝数及稳定性、培养条件等。近年来,利用基因工程方法和诱变育种来提高酿酒酵母表达蛋白能力、代谢产物等的研究进展较快。Miura S等人利用亚硝基胍诱变1 株 L-乳酸产生菌,突变株的乳酸产量超过90 g/L,达原始菌株产量的2倍[3]。张丽杰等以酿酒酵母 HY01 为出发菌,选用2 种低毒性化学诱变剂5-BU 和盐酸平阳霉素,在确定最佳配比的基础上,通过联用这2 种诱变剂进行复合诱变,获得了一定数量的正突变株,其中产能提高最高的1株发酵物中海藻糖含量达到16%,比出发菌株提高了10%[4]。
启动子(promoter)位于结构基因5'端上游区,是RNA聚合酶的结合区。启动子区的结构影响其与RNA聚合酶的亲和力,进而影响基因的转录表达效率。通过对启动子改造,筛选强启动子,是提高宿主重组蛋白表达水平的一种重要的方法[5]。Partow S等都通过筛选、构建强启动子实现了重组蛋白的高效表达[6, 7]。
pHR酿酒酵母表达系统是本实验室前期研究中基于酵母2μ质粒构建的一种新型酿酒酵母表达系统[8, 9]。与传统的以抗生素抗性基因作为筛选标记的酵母表达载体不同,除需表达的外源基因外,pHR表达载体所有元件序列均来自酿酒酵母自身,不携带抗药基因序列和任何大肠杆菌基因序列,以酿酒酵母Leu2基因作为营养筛选标记。外源基因表达质粒只能在酵母细胞内生存。外源基因通过同源重组方式克隆于表达载体上,通用性好、效率高。该系统以酿酒酵母Y16为宿主,表达载体为 pHR-PTEF-A,其启动子元件PTEF为酿酒酵母翻译延伸因子EF-1a基因的启动子。本研究通过诱变剂处理和易错PCR技术分别对宿主Y16和启动子PTEF进行突变改造,构建pHR-N酿酒酵母表达系统,提高其生产重组蛋白的能力。 1 材料与方法 1.1 菌株和质粒
大肠杆菌E.coli DH5α;酵母菌:S.cerevisiae S1502B (MATa ura3-52 leu2-3,112 trp1-289 his3-Δ),简称Y16。大肠杆菌-酿酒酵母穿梭质粒YEP353(ATCC number 37725)。酿酒酵母表达载体pHR-PTEF-A由本实验室构建。 1.2 主要试剂与仪器
KOD Plus Neo高保真DNA聚合酶,限制性内切酶、T4 DNA连接酶购于TaKaRa。质粒提取试剂盒和DNA凝胶回收试剂盒购于Axygen。甲基磺酸乙酯(Ethylmethyl sulfone,EMS)、X-Gal 和ONPG(o-Nitrophenyl-β-D-Galactopyranoside)购于Sigma。易错PCR试剂盒购于北京天恩泽生物技术公司。PCR扩增仪(Applied Biosystems);流式细胞仪(BD);分光光度仪(尤尼柯UV-2000)。 1.3 方 法 1.3.1 引物设计
所用引物由上海桑尼生物科技有限公司合成(表 1)。
| Primer application | Primer name | Primer sequence (5′-3′) |
| PCR PTEF | PTEF-F1 | ATGGATCCCATAGCTTCAAAATGTTTCTACTCC |
| PTEF-R1 | CGGAAGCTTTCATAAAACTTAGATTAGATTGCTATGCTTTC | |
| PCR HSA | HSA-F1 | AGATCCGATGCACACAAGAGTGAGGTTGC |
| HSA-R1 | ACGTCGACTCATTATAAGCCTAAGGCAGCTTGACT | |
| PCR GFP | GFP-F1 | CGTCGACAGTAAAGGAGAAGAACTTTTCACTG |
| GFP-R1 | CACTAGTTCATTATTTGTATAGTTCATCCATGCCA |
PCR获得的PTEF序列经酶切后克隆于YEP353载体。突变改造的PTEF、人血清白蛋白(human serum albumin,HSA)基因或绿色荧光蛋白(green fluorescent protein,GFP)基因分别经酶切后克隆于酿酒酵母表达载体pHR-PTEF-A。大肠杆菌转化采用42℃ 热激方法,酿酒酵母转化采用LiAC化学法[10]。分子生物学操作方法参考《分子克隆实验指南》(1996年第二版)和《酵母遗传学方法实验指南》(2000年第一版)。 1.3.3 易错PCR突变[11, 12]
按照易错PCR试剂盒说明书进行操作,反应总体系为60μl,PCR程序设定如下:

挑取少许YEPD固体培养基上的酿酒酵母Y16,接入新鲜YEPD液体培养中,30℃培养后,离心,无菌水洗涤2次,1ml PBS悬浮,加入40 μl EMS溶液,30℃温浴处理一定时间;离心收获菌体,5% 硫代硫酸钠溶液 1ml 洗涤2次,得到的酵母菌体用于转化筛选。 1.3.5 突变筛选方法1.3.5 .1 初筛
(1)启动子突变初筛[15]
YEP353载体携带LacZ报告基因,通过检测LacZ基因产物β-半乳糖苷酶(β-galactosidase,β-gal)的活性,能够反映LacZ前多克隆位点克隆的启动子的转录效率。经易错PCR突变后的启动子PTEF连接到质粒YEP353上,构建YEP353- PTEF-mutant质粒,连接产物转化Y16,涂布于选择性SD固体培养基上,30℃培养2~3天获得酵母转化子。随后转化子转接于含有X-Gal的固体培养基上,30℃继续培养,根据菌落蓝斑出现时间和颜色深浅(与对照酵母比较)筛选候选阳性克隆。
(2)诱变酵母菌株初筛
质粒YEP353-PTEF转化经EMS诱变处理的Y16菌体,涂布于选择性SD固体培养基上,30℃培养2~3天后获得转化子克隆,候选阳性克隆筛选方法同上所述。
对于初筛获得的候选阳性克隆进行复筛,确认其可靠性。采用的方法为ONPG法。重组酵母中表达的β-gal能够将ONPG解为半乳糖和黄色的邻-硝基苯酚(ONP)。将酵母克隆接入选择性SD液体培养基中30℃摇床培养24 h;按10%(V/V)接入1ml YEPD培养基中,培养48h,测OD600值;取100 μl菌液离心去上清,用1ml Z-Buffer洗一次;再用800μl Z-Buffer悬浮,置于-80℃,反复冻融3~4次;加入200μl ONPG反应一段时间,加入400μl 1mol/L NaCO3 终止反应;离心,取上清分光光度计测OD420。β-gal酶活性= (OD420×1000)/(OD600×V(ml)×T(min)) 2 结果 2.1 宿主酿酒酵母Y16诱变改造
选择YEPD中培养10h后的Y16酵母细胞,利用EMS进行诱变处理,测定EMS处理不同时间菌株的死亡率(结果略)。EMS最佳诱变条件为:PBS(pH=7.2)溶液中,EMS浓度40μl/ml,处理55min,Y16菌株死亡率为70%~80%。收集EMS诱变处理后存活的Y16酵母,继续培养待用。
质粒YEP353-PTEF转化诱变处理后的Y16酵母,获得的转化子分别测定β-gal酶活性,经过筛选鉴定,编号E14和E19的两个转化子β-gal酶活较未处理的Y16转化子(对照)酶活分别提高22.31%和26.53%(表 2)。
| 转化子 | β-gal酶活性 | 提高比率(%) |
| 对照 | 120.90±10.23 | 0.00 |
| A13 | 129.94±9.65 | 7.50% |
| A17 | 123.87±15.06 | 2.46% |
| E14 | 147.87±2.95 | 22.31% |
| E19 | 152.98±11.25 | 26.53% |
将E14和E19在非选择性YEPD培养基中连续传代培养,丢失酵母细胞中YEP353-PTEF质粒,经表型验证正确后,获得诱变菌株Y16-E14和Y16-E19。为确认菌株发生有利突变,将质粒YEP353-PTEF分别回转Y16-E14和Y16-E19,检测细胞β-gal酶活性。结果如表 3所示,Y16-E14和Y16-E19菌株产生β-gal酶活性分别比未改造的Y16菌株高73.53%和55.60%,由此获得了两株有利于重组β-gal酶表达的诱变酵母菌株Y16-E14和Y16-E19。
| 菌株 | β-gal酶活性提高比率(%) | |
| Y16-E14 | 334.34±11.90 | 73.53 |
| Y16-E19 | 299.80±17.03 | 55.60 |
| 对照(Y16) | 192.67±13.21 | 0.00 |
以启动子PTEF序列为模板,以PTEF-F1/PTEF-R1为引物进行易错PCR,PCR产物经BamHⅠ/HindⅢ酶切后,克隆于YEP353 载体,重组载体转化酵母Y16。经过初筛和复筛鉴定,发现编号E49-24、N36、N37三个转化子的β-gal酶活性较对照组分别提高了47.09%、46.55%、55.43%。三个转化子中YEP353-PTEF-mutant质粒抽提出来测序,发现三个质粒PTEF启动子区均在234位发生突变,即由T变为C(T234C),将突变启动子命名为PTEFv1。
为确认突变启动子PTEFv1的有效性和稳定性,将质粒YEP353-PTEFv1回转酵母Y16,检测重组酵母β-gal酶活,结果如表 4所示,装配突变启动子PTEFv1的重组酵母产生的β-gal酶活较对照重组酵母(启动子PTEF)提高61.47%,说明突变启动子PTEFv1转录效率明显强于野生型启动子PTEF。
| 重组酵母 | β-gal酶活性提高比例(%) | |
| Y16/PTEF-YEP353 | 127.52±29.58 | 0.00 |
| Y16/PTEFV1-YEP353 | 205.91±4.84 | 61.47 |
经过突变改造,我们获得了较PTEF启动子效率更高的PTEFv1启动子和有利于重组蛋白表达的诱变改造菌株Y16-E14、Y16-E19。随后将PTEFv1启动子和Y16-E14菌株应用于改造pHR酿酒酵母表达系统。将PTEFV1启动子替换酿酒酵母表达载体pHR-PTEF-A上的PTEF启动子,以Y16-E14作为宿主细胞,构建胞内表达GFP或分泌表达HSA的重组酵母,评价新构建的pHR-N表达系统的效能(表 5)。
| 组别 | Y16宿主 | Y16-E14宿主 |
| GFP组 | Y16/pHR-PTEF-GFP(Y1) | Y16-E14/pHR-PTEF-GFP(E1) |
| Y16/pHR-PTEFv1-GFP(Y2) | Y16-E14/pHR-PTEFv1-GFP(E2) | |
| HSA组 | Y16/pHR-PTEF-αF-HSA(Y3) | Y16-E14/pHR-PTEF-αF-HSA(E3) |
| Y16/pHR-PTEFv1-αF-HSA(Y4) | Y16-E14/pHR-PTEFv1-αF-HSA(E4) |
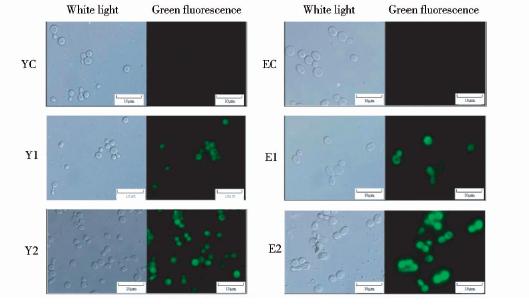
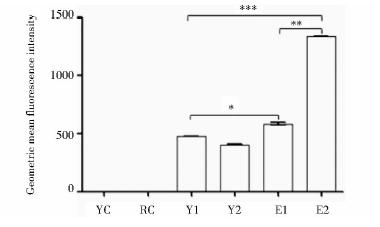
对照组分别为Y16/pHR-PTEF(YC)和Y16-E14/ pHR-PTEF(EC). αF,α-Factor信号肽分别利用荧光显微镜和流式细胞仪检测重组酵母表达GFP情况。荧光显微镜观察显示E2组酵母(Y16-E14/pHR-PTEFv1-GFP)荧光不仅强于E1组(Y16-E14/ pHR-PTEF-GFP)也明显强于Y1组(Y16/pHR-PTEF-GFP)或Y2组(Y16/pHR-PTEFv1-GFP),说明突变启动子PTEFv1和诱变改造后的宿主Y16-E14能够增强GFP蛋白表达(图 1)。流式细胞仪对重组酵母荧光强度进行定量检测分析,结果显示E2组酵母荧光强度显著高于其他各组(图 2),证实突变启动子PTEFv1和宿主Y16-E14对重组GFP产生的促进作用。
|
| 图 1 荧光显微镜检测重组酵母表达GFP情况 Fig. 1 Fluorescence microscope analysis of the recombinant yeasts expressing GFP |
|
| 图 2 流式细胞仪检测重组酵母平均荧光强度Fig. 2 Flow cytometry analysis of geometric mean fluorescence intensity of the recombinant yeastsCell number of each test sample is 5×104.*,P<0.05;**,P<0.01;***,P<0.001 |
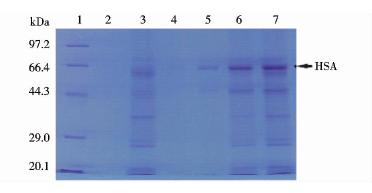
HSA组重组酵母培养2天后,取培养液上清进行SDS-PAGE分析,结果显示E4组的分泌的HSA量明显多于其他各组(图 3),说明E4组重组酵母(Y16-E14/pHR-PTEFv1-αF-HSA)分泌表达HSA能力最强,即改造的Y16-E14宿主和PTEFV1启动子有助于重组蛋白HSA的分泌表达。
|
| 图 3 SDS-PAGE鉴定各重组酵母分泌表达HSA情况Fig. 3 SDS-PAGE analysis of HSA secreted by the recombinant yeasts1:Marker;2: Corotrol YC;3: Corotrol EC;4:Y3;5:Y4;6:E3;7:E4 |
YEP353是一种大肠杆菌-酿酒酵母穿梭载体,将启动子克隆于该载体LacZ基因上游的多克隆位点区域,构建的重组载体转化酿酒酵母,通过重组酵母中LacZ基因表达产生的β-半乳糖苷酶(β-gal)活性测定可鉴别YEP353所克隆的启动子在酵母中的效率。本研究中,为了获得较已有的酿酒酵母强启动子PTEF效率更高的启动子,以PTEF序列为模板,易错PCR产生突变PTEF序列。突变启动子克隆于YEP353载体后转化酿酒酵母Y16菌株,获得重组酵母转化子文库。经过含X-Gal培养基平板初筛以及ONPG法复筛鉴定,N36等三个转化子产生的β-gal明显高于启动子未改造的对照酵母。测序后分析发现三个转化子重组YEP353质粒上克隆的突变PTEF序列完全一致,均仅有一个碱基突变(T234C)。筛选获得的三个突变启动子来自不同批次的易错PCR产物。实验中,虽然为了获得尽可能多的突变启动子,进行了多管多次易错PCR,但每一次PCR的退火温度、延伸时间等程序完全一致,在相同条件下,可能存在偏好突变位点的情况。T234C位点突变显著提高PTEF启动子的转录效率可能与该位点突变后影响启动子区结构,促进RNA聚合酶与启动子区结合有关,其具体机制有待实验确认。本研究证实通过易错PCR优化酵母启动子是一种行之有效的方法。
宿主也是表达系统需要考虑的重要因素,宿主细胞内生化环境(RNA转录和翻译速率、蛋白质折叠速率、稳定性等)对重组蛋白的产生具有重要影响[2, 18]。诱变、代谢工程等可以通过各种途径改变宿主细胞内环境,进而改善宿主生产重组蛋白的能力。国内外不少研究报道证实了化学诱变剂EMS改造宿主的有效性。为了提高酿酒酵母Y16生产重组蛋白(特别是分泌重组蛋白)的能力,我们采用EMS对Y16进行诱变处理。通过EMS对Y16酵母的处理,筛选获得了表达β-gal能力提高的二株突变酵母Y16-E14、Y16-E19。研究中发现与未诱变的Y16比较,Y16-E14酵母形态发生了明显变化,即Y16-E14酵母细胞个体更大。诱变改造的菌株生理特性是否发生变化、通过何种途径提高重组蛋白表达水平需要后续进一步研究探究。
将原有的pHR酿酒酵母表达载体启动子PTEF替换为突变启动子PTEFv1 、宿主酵母Y16替换为Y16-E14,获得预期性能有所改进的pHR-N酵母表达系统。为了解pHR-N酵母表达系统的普适性,我们分别以GFP、HSA为报告基因验证pHR-N系统胞内表达或分泌表达重组蛋白的能力。研究结果显示,相对启动子或宿主单一因素优化,PTEFv1和Y16-E14协同应用大大提高了重组蛋白胞内表达或分泌表达水平。
通过启动子和宿主两方面的改造,优化后的pHR-N表达系统表达外源基因的能力进一步提升,为拓展新型pHR酿酒酵母表达系统在生物医药领域的应用奠定了良好的基础。
| [1] | McAleer W J, Buynak E B, Maigetter R Z, et al. Human hepatitis B vaccine fromrecombinant yeast. Nature, 1984, 307:178-180. |
| [2] | Celik E, Calik P. Production of recombinant proteins by yeast cells. Biotechnology Advances, 2012, 30:1108-1118. |
| [3] | Miura S, Dwiarti L, Arimura T, et al. Enhanced production of L- lactic acid by ammonia-tolerant mutant strain Rhizopus sp. MK-96-1196. J Biosci Bioeng, 2004, 97(1):19-23. |
| [4] | 张丽杰,邸进申,全学军等.诱变选育高产海藻糖的酵母菌株. 生物技术,2005,15(1):31-33. Zhang L J, Di J S, Quan X J, et al. Breeding of high trehalose producing mutants of Saccharomyces cerevisiae. Biotechnology, 2005,15(1):31-33. |
| [5] | Alper H, Fischer C,Nevoigt E, et al. Tuning genetic control through promoter engineering. PNAS, 2005, 102 (36):12678-12683. |
| [6] | Partow S, Siewers V, Sara Bjørn S, et al. Characterization of different promoters for designinga new expression vector in Saccharomyces cerevisiae. Yeast, 2010; 27: 955-964. |
| [7] | Blount B A, Weenink T, Ellis T. Construction of synthetic regulatory networks in yeast. FEBS Letters, 2012, 586:2112-2121. |
| [8] | Yu J, Jian J X, Ji W M, et al. Glucose-free fructose production from Jerusalem artichoke using a recombinant inulinase-secreting Saccharomyces cerevisiae strain. Biotechnol Lett, 2011, 33:147-152. |
| [9] | 江佳稀,张宇飞,沈洪波等. 表达结核杆菌抗原的重组酿酒酵母免疫小鼠研究. 复旦学报(自然科学版), 2011,50(2):192-197. Jiang J X, Zhang Y F, Shen H B, et al. Induction of antigen-specific humoral immune response by subcutaneous vaccination with Saccharomyces cerevisiae expressing Mycobacterium tuberculosis antigen. Joural of Fudan University (Natural Science), 2011, 50(2):192-197. |
| [10] | Gietz R D, Schiestl R H. Frozen competent yeast cells that can be transformed with high efficiency using the LiAc/SS carrier DNA/PEG method. Nat Protoc, 2007, 2(1):1-4. |
| [11] | Cline J, Braman J C, Hogrefe H H. PCR fidelity of Pfu DNA polymerase and other thermostable DNA polymerases. Nucleic Acids Res, 1996, 24: 3546-3551. |
| [12] | Chaput J C, Woodbury N W, Stearns L A, et al. Creating protein biocatalysts as tools for future industrial applications. Expert Opin Biol Ther, 2008, 8: 1087-1098. |
| [13] | Burns P A, Allen F L, Glickman B W. DNA sequence analysis of mutagenicity and site specificity of ethyl methanesulfonate in Uvr+ and UvrB- strains of E.coli.Geneties, 1986,113:811- 819. |
| [14] | 程明,崔承彬,李长伟等. 化学诱变技术在微生物育种研究中的应用. 国际药学研究杂志. 2009, 36(6):412-417. Cheng M, Cui C B, Li C W, et al. Chemical mutation technique applied in microorganism breeding. Journal of International Pharmaceutical Research, 2009, 36(6):412-417. |
| [15] | Guarente L. Yeast promoters and lac-Z fusions designed to study expression of cloned genes in yeast. Methods Enzymol, 1983, 101:181-191. |
| [16] | Fashena S J, Serebriiskii IGand Golemis E A. The continued evolution of hybrid screening approaches in yeast: How to outwit different baits with different preys. Gene, 2000, 250: 1-14. |
| [17] | Menzel R. A microtiter plate-based system for the semiautomated growth and assay of bacterial cells for beta-galactosidase activity.Anal Biochem, 1989, 181:40-50. |
| [18] | Böer E, Steinborn G, Kunze G, et al. Yeast expression platforms. Appl Microbiol Biotechnol, 2007, 77:513-523. |
 2015, Vol. 35
2015, Vol. 35




