2. 广东省水环境与水产品安全工程技术研究中心,广东 广州 510225;
3. 广东省水禽健康养殖重点实验室,广东 广州 510225;
4. 广州市水产病害与水禽养殖重点实验室,广东 广州 510225
鱼类线粒体基因组(Mitochondrial DNA,mtDNA)与其他脊椎动物相似,为闭合环状的双链DNA分子,由重链(H链)和轻链(L链)组成,能进行自我复制、转录和翻译[1]。目前已报道的鱼类线粒体基因组大多编码37个基因(包括13个蛋白编码基因、22个tRNA基因、2个rRNA基因)以及一个非编码控制区(D-loop区)[2]。与核基因相比,线粒体基因组具有母系遗传、进化速度快、结构简单等特点,成为生物种群遗传、系统演化等方面研究的重要分子标记[3],广泛应用于鱼类物种起源、分子系统进化、遗传多样性和物种分子鉴定等领域的研究分析。
巨石斑鱼(Epinephelus tauvina)与斜带石斑鱼(Epinephelus coioides)隶属于硬骨鱼纲(Actinopterygii)鲈形目(Perciformes)鮨科(Serranidae)石斑鱼属(Epinephelus),主要分布于热带印度洋和太平洋近海,我国见于南海诸岛和台湾海域[4-6]。形态上,巨石斑鱼在个体发育过程中,体色斑纹与多种石斑鱼存在趋同现象,特别是与斜带石斑鱼,两者在体色、体形、斑纹等特征非常相似,表现为身体较修长,体表呈棕黑或棕黄色,身体与各鳍覆有许多橙红色或红棕色圆斑,尾鳍圆形等,利用形态学分类的方法不易将二者清晰区分。在养殖生产中,两者曾相互混淆,俗称“青斑”[7]。形态分类学上也有不少研究将斜带石斑鱼误认为是巨石斑鱼的情况。如Ben-Tuvia曾指出地中海中有关巨石斑鱼的洄游研究报道,其物种应该是斜带石斑鱼[8-9]。Sachithanandam等[10]分析了安达曼海巨石斑鱼及斜带石斑鱼群体样品,指出安达曼海及附近海域分布的巨石斑鱼其实是斜带石斑鱼的误鉴。Tan等[11]也指出中国南海及安达曼海鱼类分类资料中,巨石斑鱼与斜带石斑鱼存在误鉴情况。在分子系统进化研究上,也出现巨石斑鱼与斜带石斑鱼分类地位存在争议的情况。如在朱世华利用Cytb基因构建的石斑鱼属系统进化树上,巨石斑鱼与斜带石斑鱼的亲缘关系十分接近,两者聚为一支,序列差异率只有1%[12];周佳怡对中国沿海石斑鱼亚科鱼类DNA条形码分析也得到相似结果,两种鱼的遗传距离小于0.020[13]。而Craig等[14]综合线粒体基因与核基因对石斑鱼亚科鱼类分子系统进化研究,发现巨石斑鱼与斜带石斑鱼在进化树上的聚类位置相距较远,两者位于不同分支。Schoelinck等[15]、Ma等[16]在石斑鱼亚科分子系统学研究中也认为两者亲缘关系相对较远。在GenBank数据库中,也有部分上传的巨石斑鱼COⅠ基因序列与斜带石斑鱼序列相似性为99%以上的情况。至于两者亲缘关系是否十分接近,还是可能存在物种误鉴,需要对更多的基因序列信息进行比较分析才能进一步验证。目前有关巨石斑鱼线粒体基因组全长信息暂未公布,本研究通过测定巨石斑鱼与斜带石斑鱼线粒体基因组全长信息,从分子水平比较巨石斑鱼与斜带石斑鱼线粒体基因组的差异,结合形态特征数据分析,验证巨石斑鱼与斜带石斑鱼之间的物种有效性及亲缘关系,厘清物种鉴定争议问题。
1 材料与方法 1.1 实验材料本研究的巨石斑鱼与斜带石斑鱼样品主要采集于广东深圳水产码头。根据《中国鱼类系统检索》[5]、《台湾鱼类志》[6]、《Goupers of the World》[17]等资料对样品进行形态分类鉴定及详细数据测量,样品取肌肉于95%酒精中固定,用于基因组DNA提取。另外,从GenBank下载目前已测定上传的其他石斑鱼亚科鱼类线粒体全长序列,用于后续系统进化树的构建及分析。
1.2 试验方法 1.2.1 基因组DNA提取取约0.1 g的样品肌肉组织,利用海洋动物总基因组DNA提取试剂盒(天根生化),按照说明书操作进行提取,提取的基因组DNA用80 μL灭菌水溶解。采用1%琼脂糖凝胶进行电泳检测。同时每种DNA样品各取1 μL于核酸测定仪进行浓度测定,并选择OD260/OD280数值为1.8左右的DNA样品用于后续实验,确保提取的DNA浓度及纯度满足测序要求。
1.2.2 线粒体基因组序列扩增与测序参考目前已经公布的石斑鱼线粒体基因组序列进行比对分析,在保守序列区设计可扩增覆盖整个线粒体基因组的引物。引物信息见表 1。PCR反应总体积为50 μL,其中PCR MasterMix混合液(TAKARA)25 μL,灭菌水21 μL,上下游引物各1 μL,DNA模板2 μL。扩增条件为94 ℃预变性5 min;94 ℃变性30 s,50~58 ℃退火30 s,72 ℃延伸1~2 min,35~38个循环,最后72 ℃再延伸10 min。PCR产物用1%琼脂糖凝胶电泳检测,选取扩增成功的样品,使用PCR产物纯化试剂盒(天根生化)进行纯化回收,送天一辉远生物技术有限公司进行序列测定。
|
|
表 1 巨石斑鱼与斜带石斑鱼线粒体基因组扩增引物 Table 1 Primers used for complete mitogenome amplication of E. tauvina and E. coioides |
测序所得序列利用DNA Star软件进行比对、拼接与组装,辅以人工分析调整,拼装成完整全长序列。采用CGview软件绘制线粒体基因组环状结构图。结合GenBank下载的石斑鱼亚科鱼类线粒体全长序列,利用Clustal X、MEGA7.0、MegAlign等软件对蛋白质编码基因、tRNA和rRNA进行识别与注释,利用tRNAscan-SE Search Server软件对tRNA二级结构进行预测。利用MEGA 7.0软件分析巨石斑鱼与斜带石斑鱼线粒体基因组碱基组成与变异、遗传距离差异、密码子使用偏好性等信息。利用Model-Test 2.1.5进行最佳进化模型预测,使用RAxML 8.0软件基于最大似然法(Maximum likelihood,ML)构建进化树。
2 结果 2.1 巨石斑鱼与斜带石斑鱼的形态特征比较根据《中国鱼类系统检索》、《台湾鱼类志》、《Grou-pers of the world》等形态学分类资料中对巨石斑鱼及斜带石斑鱼的描述,结合作者对本研究采集的巨石斑鱼及斜带石斑鱼样品的观察比较,汇总出可区分巨石斑鱼及斜带石斑鱼的形态学特征数据(见表 2),这些数据用于后续两种石斑鱼形态与分子水平结合比较分析。
|
|
表 2 巨石斑鱼与斜带石斑鱼形态特征区分表 Table 2 Comparison analysis of morphological characteristics between E. tauvina and E. coioides |
巨石斑鱼和斜带石斑鱼线粒体基因(mtDNA)全长分别为16 787 bp(GenBank登录号:MW194890)和16 798 bp(GenBank登录号:MW752082),整体呈双链环状,由一条重链(H strand)与一条轻链(L strand)组成(见图 1)。与绝大多数脊椎动物线粒体基因组一样,两者mtDNA基因组包含37个基因(包括13个蛋白编码基因、22个tRNA基因和2个rRNA基因)和1个D-loop非编码控制区(见表 3)。其中L链上有9个基因,包括tRNA-Gln、tRNA-Ala、tRNA-Asn、tRNA-Cys、tRNA-Tyr、tRNA-Ser(TGA)、tRNA-Glu、tRNA-Pro和ND6,剩余的28个基因均位于H链上。两种石斑鱼大部分基因序列长度及间隔重复情况一致,少数基因存在差异(见表 3)。
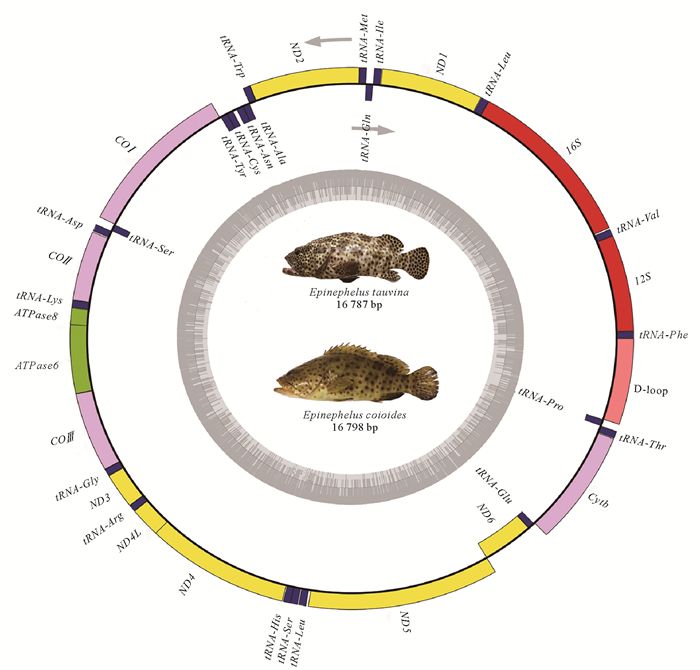
|
图 1 巨石斑鱼与斜带石斑鱼线粒体基因组结构图 Fig. 1 Mitogenome map of E. tauvina and E. coioides |
|
|
表 3 巨石斑鱼与斜带石斑鱼线粒体基因组结构组成 Table 3 Main structures of complete mitogenome in E. tauvina and E. coioides |
巨石斑鱼和斜带石斑鱼的线粒体基因组中均存在11处间隔区,间隔长度分别为75和78 bp,其中最大间隔出现在tRNA-Asn和tRNA-Cys这二者之间,分别为43和41 bp。此外,两者基因组中相邻基因还存在重叠现象(巨石斑鱼8处,斜带石斑鱼6处),即tRNA-Ile与tRNA-Gln(1 bp),ATPase8与ATPase6(10 bp),ATPase6与COⅢ(1 bp),ND4L与ND4(7 bp),ND5与ND6(4 bp),tRNA-Thr与tRNA-Pro(2 bp、1 bp),前者在16S rRNA与tRNA-Leu(TAA,3 bp),tRNA-Cys与tRNA-Tyr(1 bp)间也存在重复,其中最大重叠区域在ATPase8和ATPase6之间。其余基因对排列紧密,无间隔与重叠情况。
两种石斑鱼线粒体基因组的核苷酸组成如表 4所示,其中巨石斑鱼碱基含量为:A:4 853个(28.91%)、G:2 708个(16.13%)、C:4 846个(28.87%)、T:4 380个(26.09%),A+T=55.00%,C+G=45.00%。斜带石斑鱼mtDNA全长为16 798 bp,其碱基含量为:A:4 779个(28.45%)、G:2 773个(16.51%)、C:4 664个(27.76%)、T:4 582个(27.28%),A+T=55.73%,C+G=44.27%。两种石斑鱼的碱基组成A+T>C+G,并表现出明显的反G偏倚。
|
|
表 4 巨石斑鱼与斜带石斑鱼线粒体基因组的核苷酸组成 Table 4 Base composition of complete mitogenomes in E. tauvina and E. coioides |
巨石斑鱼与斜带石斑鱼线粒体基因组13个蛋白编码基因总长度均为11 428 bp,占线粒体全长的68.1%和68.0%。除了ND6基因以外,两种石斑鱼蛋白编码基因均位于H链上。起始密码子除了COⅠ基因(GTG)、ATPase6基因(巨石斑鱼为TTG,斜带石斑鱼为CTG)外,其他蛋白编码基因起始密码子均为ATG。终止密码子除ND5、ND6基因(巨石斑鱼为TAG,斜带石斑鱼为TAA),两种石斑鱼的其余基因均以TAA(ND1、COⅠ、ATPase8、ATPase6、ND4L)、不完全终止密码子TA(COⅢ)或T(ND2、COⅡ、ND3、ND4、Cytb)这3种鱼类mtDNA中常见终止密码子组成。两种石斑鱼相对同义密码子使用情况(RSCU)如图 2所示。除去起始和终止密码子,巨石斑鱼和斜带石斑鱼的密码子使用数分别为3 767和3 787个。其中,在密码子第三位中,巨石斑鱼C的使用频率最高(1 355个),斜带石斑鱼则是A的使用频率最高(1 257个),AT使用频率均高于GC。两种石斑鱼密码子使用分析情况如图 3所示,亮氨酸1(Leu1)的使用率最高(14.50%和14.23%),其次为异亮氨酸(Ile)(9.48%和9.37%),半胱氨酸(Cys)的使用率最低,分别为0.72%和0.77%。

|
( 每种氨基酸对应的左侧柱状图为巨石斑鱼,右侧柱状图为斜带石斑鱼。For each amino acid, the histogram on the left represent E. tauvina, the histogram on the right represent E. coioides. ) 图 2 巨石斑鱼和斜带石斑鱼线粒体基因组的同义密码子使用情况 Fig. 2 Relative synonymous codon usage (RSCU) of the mitogenome in E. tauvina and E. coioides |

|
( 每种氨基酸对应的左侧柱状图为巨石斑鱼,右侧柱状图为斜带石斑鱼。For each amino acid, the histogram on the left represent E. tauvina, the histogram on the right represent E. coioides. ) 图 3 巨石斑鱼和斜带石斑鱼线粒体基因组的氨基酸组成 Fig. 3 Amino acid composition of the mitogenome in E. tauvina and E. coioides |
巨石斑鱼与斜带石斑鱼rRNA基因由12S rRNA和16S rRNA组成,均位于tRNA-Phe和tRNA-Leu(TAA)基因之间,中间由tRNA-Val基因隔开。其中12S rRNA碱基长度分别为956和953 bp,16S rRNA长度分别为1 711和1 707 bp。与其他基因相比,两种石斑鱼的rRNA碱基组成并没有明显的反G偏倚现象,G含量(20.66%、20.98%)与T含量(21.67%、21.65%)相差不大,A含量相对较多。
巨石斑鱼与斜带石斑鱼22个tRNA基因大小范围在67~76 bp之间,总长度为1 566和1 558 bp。除tRNA-Val、tRNA-Cys、tRNA-Asp、tRNA-His、tRNA-Ser(GCT)、tRNA-Glu、tRNA-Thr外,其余tRNA基因长度在两种石斑鱼中均相同。其中位于L链的基因有8个,位于H链的基因有14个。利用tRNAscan-SE软件对两种石斑鱼22个tRNA二级结构进行预测,结果显示两者中均有21个tRNA基因能形成典型的三叶草形结构,包括7对碱基组成的受体臂(氨基酸臂),5对碱基组成的TψC臂,5对碱基组成的反密码臂及3~4对碱基组成的DHU臂,而两者的tRNA-Ser(GCT)基因的二级结构均缺少DHU臂。
2.5 控制区及WANCY区域与大部分硬骨鱼类相似,巨石斑鱼与斜带石斑鱼的线粒体基因组中也存在轻链复制起始区(Origin of light strand replication,OL),位于5个tRNA基因簇(WANCY区域)中的tRNA-Asn与tRNA-Cys之间,其中巨石斑鱼OL序列由43个碱基组成(5′-TCCCCCGCCTAACTCTTATTGAGTTTGAGTAGGC GGGGGAAGT-3′),而斜带石斑鱼由41个碱基组成(5′-TCCTCCGCCTACTATTTATGTTTAAATAGGCGGAGGAAAGC-3′),均能折叠成稳定的茎环二级结构,其保守结构为5′-GGCGG-3′。两种石斑鱼的控制区(D-loop区)均位于tRNA-Pro和tRNA-Phe基因间,长度分别为1 080和1 098 bp,不参与基因编码,而是调节mtDNA的复制和转录的片段。此序列显示出极大的AT偏好性,A+T含量为66.57%(巨石斑鱼)和69.03%(斜带石斑鱼),G+C含量仅为33.43%(巨石斑鱼)和30.97%(斜带石斑鱼)。
2.6 巨石斑鱼和斜带石斑鱼物种间线粒体基因组序列比较分析利用MEGA7.0软件对巨石斑鱼与斜带石斑鱼的序列进行比较分析,分析序列包括全基因组序列和常用于分子系统进化、亲缘关系研究的分子标记序列(13个蛋白编码基因、2个rRNA基因和D-loop区),结果如表 5所示,其中13个编码蛋白基因都统一去掉终止密码子。从表 5可知,巨石斑鱼与斜带石斑鱼线粒体全长分别为16 787和16 798 bp,两基因组之间相差11个碱基。基于Kimura-2-Parameter模型计算两线粒体基因组遗传距离为0.150,基因组存在较大差异。在13个蛋白质编码基因、2个rRNA基因及D-loop区这16个分子标记序列中碱基差异在24~322 bp之间,遗传距离差异为0.059~0.406。其中,两种石斑鱼的D-loop区差异最大,在约1 000 bp的序列长度中,存在322个碱基变异,遗传距离达0.406。12S rRNA和16S rRNA基因序列最为保守,两者碱基差异分别为55和96 bp,遗传距离分别为0.061和0.059。13个蛋白编码基因遗传距离在0.129~0.241之间,其中差异最大为ND6,遗传距离高达0.241,在519 bp序列中共有104 bp的碱基变异。COⅡ是差异最小的,遗传距离为0.129,在690 bp序列中含80 bp的碱基变异。在目前比较通用的物种鉴定标记COⅠ及Cytb基因,两基因在两种石斑鱼中碱基差异分别为188和177 bp,遗传距离分别为0.137和0.182,一般认为同一物种之间Cytb及COⅠ遗传距离不超过0.020,该结果显示巨石斑鱼与斜带石斑鱼已远超出种以上水平。
|
|
表 5 巨石斑鱼与斜带石斑鱼线粒体基因组16个基因序列比较分析 Table 5 Analysis 16 gene sequences between E. tauvina and E. coioides |
基于线粒体基因组12个蛋白编码基因序列,以鮨科鱼类吉氏低纹鮨(Hypoplectrus gemma)、燕赤鮨(Chelidoperca hirundinacea)作为外类群,利用最大似然法将巨石斑鱼、斜带石斑鱼与GenBank下载的其他31种石斑鱼亚科鱼类蛋白编码序列构建分子系统进化树(见图 4)。除去外类群,33种石斑鱼亚科鱼类共聚为5个分支。其中,石斑鱼属并未形成单系,与部分其他属的种类聚成两个平行分支,其中褐石斑鱼、云纹石斑鱼、纵带石斑鱼等10种石斑鱼属与光腭鲈属、驼背鲈属、下美鮨属、鸢鮨属的种类聚为一支(分支Ⅰ),位于进化树顶部,而另外10种石斑鱼聚为分支Ⅱ中;分支Ⅲ由烟鲈属与九棘鲈属组成;分支Ⅳ为侧牙鲈属,分支Ⅴ为鳃棘鲈属,进化地位最为原始,位于进化树基部。本研究的巨石斑鱼以100%支持率与蜂巢石斑鱼(Epinephelus merra)聚在一起,位于分支Ⅱ,而斜带石斑鱼以100%支持率与马拉巴石斑鱼(Epinephelus malabaricus)聚在一起,位于分支Ⅰ,两种石斑鱼的分类地位相对较远。

|
图 4 基于12个蛋白编码基因序列利用最大似然法构建的33种石斑鱼的分子系统进化树 Fig. 4 Molecular phylogenetic trees of 33 groupers constructed using maximum likelihood method based on 12 protein coding gene sequences |
巨石斑鱼与斜带石斑鱼的线粒体基因组结构呈双链闭合环状,全序列共编码37个基因(22个tRNA基因、13个编码蛋白基因和2个rRNA基因)和一个非编码的控制区序列。整个线粒体基因组基因排列顺序与大部分硬骨鱼类的结构一致,各基因长度也相差不大[18-20]。巨石斑鱼和斜带石斑鱼mtDNA的基因分布都呈非均一性,其中tRNA-Gln、tRNA-Ala、tRNA-Asn、tRNA-Cys、tRNA-Tyr、tRNA-Ser (TGA)、tRNA-Glu、tRNA-Pro 8个tRNA基因和ND6基因位于L链,其余基因均在H链,与连总强等[21]所指出的大多数鱼类L链和H链编码情况一致。位于H链上的线粒体基因更易发生水解和氧化,而13个编码蛋白基因仅ND6基因位于L链上,说明了其相对稳定性和特殊性[22]。
巨石斑鱼与斜带石斑鱼线粒体全基因组的碱基均表现出明显的反G偏倚现象,G碱基含量分别为16.13%和16.51%,与其他硬骨鱼类如牙鳕(Merlangius merlangus,16.2%)[23]、小黄鱼(Larimichthys polyactis,16%)[24]等线粒体基因中的G碱基含量相近,符合典型脊椎动物碱基组成特点[25-26]。两种石斑鱼的编码蛋白基因中绝大部分基因起始密码子为ATG,COⅠ基因起始密码子为GTG,与大部分硬骨鱼类线粒体基因组一致。而ATPase6基因则以TTG(巨石斑鱼)和CTG(斜带石斑鱼)作为起始密码子,此情况在Zhuang等[27]的研究中也有报道。终止密码子存在TAA、TAG及不完全密码子TA、T四种类型,与大多数硬骨鱼类相同[21],当中部分不完全终止密码子可在转录mRNA后由3′端的多聚腺苷酸化作用形成完整的TAA终止密码子[28-29]。部分鱼类出现过不同于以上4种类型的特殊终止密码子,如银龙鱼(Osteoglossum bicirrhosum)为AGG,蝴蝶鱼(Pantodon buchholzi)为AGA等[30],这些差异与突变有助于从分子水平鉴别不同物种,分析不同物种的分类关系及进化地位。
3.2 巨石斑鱼和斜带石斑鱼物种亲缘关系分析形态上,由于巨石斑鱼和斜带石斑鱼体色、体型、斑纹等特征异常相似,在形态分类、养殖生产上均存在两者混淆的情况。Ben-Tuvia[8]关于地中海巨石斑鱼以及Tan等[11]关于中国南海及安达曼海鱼类的分析记录中均存在巨石斑鱼与斜带石斑鱼的误鉴。在分子水平研究中,朱世华[12]利用Cytb基因、周佳怡[13]基于DNA条形码所构建的石斑鱼属系统进化树上,巨石斑鱼与斜带石斑鱼紧密聚在一起,亲缘关系相近。而Craig等[14]、schoelinck等[15]、Ma等[16]关于石斑鱼亚科分子系统进化研究中两种石斑鱼的分类位置相距较远,位于不同的分支,遗传差异较大。本研究根据采集的样品材料,结合《中国鱼类系统检索》[5]、《台湾鱼类志》[6]、《Groupers of the world》[17]等资料,比对分析了两种石斑鱼的形态学特征性状,总结出可有效区分两种石斑鱼的较典型差异特征,如巨石斑鱼最后四根背鳍基部有一大型黑斑或为一组小黑点,而斜带石斑鱼没有;巨石斑鱼不具有第一鳃弓侧骨片,斜带石斑鱼具有;巨石斑鱼幽门盲囊数为16~18个,斜带石斑鱼为50~60个。这些差异特征可作为区分巨石斑鱼和斜带石斑鱼的有效形态分类性状。
在线粒体基因组信息方面,巨石斑鱼与斜带石斑鱼的线粒体全长分别为16 787和16 798 bp,序列共存在11 bp长度差异,两者在部分基因的长度及序列间隔与重叠信息也存在区别(见表 3)。另外,筛选两种石斑鱼mtDNA中常用于分子系统进化研究的标记序列:13个蛋白编码基因、2个rRNA及D-loop序列比对分析,揭示了两者序列存在巨大差异。两种石斑鱼16个基因两两之间的遗传距离差异范围在0.059(16S rRNA)到0.406(D-loop)之间。Hebert[31]曾对11个门13 320个物种COⅠ序列进行研究,认为0.020(2%)的COⅠ遗传距离可以区分不同物种,种内序列差异极少超过0.020,大多数低于0.010(1%)。Avise与Walker[32]也确定了0.020(2%)的Cytb遗传距离可以区分不同物种。本研究中巨石斑鱼和斜带石斑鱼COⅠ及Cytb遗传距离差异达到了0.137和0.182,远大于0.020(2%)的临界值,其它14种基因在两种石斑鱼之间的遗传距离差异全部大于2%,包括序列进化相对保守的12S rRNA与16S rRNA,这揭示巨石斑鱼与斜带石斑鱼在分子水平上存在很大的遗传差异,远超出种以上水平。基于12个蛋白编码基因构建的分子系统进化树,巨石斑鱼以100%支持率与蜂巢石斑鱼聚在一起,位于第Ⅱ分支,而斜带石斑鱼与马拉巴石斑鱼聚在一起,位于第Ⅰ分支,分类地位相对较远。研究结果与Craig等[14]、Schoelinck等[15]、Ma等[16]基于多个标记构建的石斑鱼系统进化树结果一致,巨石斑鱼与斜带石斑鱼位于两个不同的独立分支。王颖汇[33]对中国近海分布的部分石斑鱼类分子系统研究中也表明,巨石斑鱼与斜带石斑鱼外部形态虽然相近,但两者在进化树上的位置相距甚远。显示两种石斑鱼虽然外部形态特征异常相似,但分子水平序列信息差异巨大,两者不能混淆为同一物种。但目前也有不少关于巨石斑鱼与斜带石斑鱼在分子水平十分接近的研究结果。Govindaraju等[34]基于RAPD标记技术对印度洋沿岸7种石斑鱼分类关系研究发现,斜带石斑鱼、巨石斑鱼和马拉巴石斑鱼聚为同一分支,遗传距离也十分接近。周佳怡[13]基于DNA条形码COⅠ基因石斑鱼分类研究,巨石斑鱼与斜带石斑鱼、马拉巴石斑鱼、青石斑鱼均交错聚集在同一分支,其认为这4种石斑鱼可能是近期辐射进化的结果。Sachithanandam等[10]基于DNA条形码研究分析了安达曼海巨石斑鱼和斜带石斑鱼的差异,结合NCBI上下载的巨石斑鱼COⅠ序列共同分析,发现安达曼海巨石斑鱼与斜带石斑鱼聚为同一个分支,遗传距离小于2%,而NCBI上下载采集于西太平洋的巨石斑鱼却位于其他分支,且与斜带石斑鱼的遗传距离大于10%。其认为安达曼海及附近海域分布的巨石斑鱼是斜带石斑鱼的误鉴,来自太平洋的巨石斑鱼是真正的巨石斑鱼,因此需要对安达曼海区的石斑鱼物种重新定义与归类。
目前,巨石斑鱼与斜带石斑鱼在形态与分子水平研究上均存在争议。本研究综合分析石斑鱼的相关分类资料,揭示两种石斑鱼在形态上存在一定的区别特征。综合线粒体基因组序列遗传分歧度分析,两者在基因序列水平存在巨大差异,已超出物种间的遗传变异。研究结果表明,无论从外部形态上,还是从分子水平上进行判断,两种石斑鱼是存在较大差异的独立物种。至于目前部分研究认为巨石斑鱼与斜带石斑鱼亲缘关系十分接近,其原因是物种的误鉴,还是物种近期的辐射进化,还是不同海区巨石斑鱼群体存在多样性差异,还需要进一步研究落实。建议在全球存在巨石斑鱼分布的不同海区采集巨石斑鱼群体,针对不同地理种群进行分子标记分析,厘清不同巨石斑鱼群体的遗传多样性差异,进而最终确定巨石斑鱼的种质情况。
| [1] |
郭新红, 刘少军, 刘巧, 等. 鱼类线粒体DNA研究新进展[J]. 遗传学报, 2004, 31(9): 983-1000. Guo X H, Liu S J, Liu Q, et al. New progresses on mitochondrial DNA in fish[J]. Acta Genetica Sinica, 2004, 31(9): 983-1000. (  0) 0) |
| [2] |
Pereira S L. Mitochondrial genome organization and vertebrate phylogenetics[J]. Genetics and Molecular Biology, 2000, 23(4): 745-752. DOI:10.1590/S1415-47572000000400008 (  0) 0) |
| [3] |
杨杨, 宋小晶, 唐文乔, 等. 克氏光唇鱼线粒体基因组测定及光唇鱼属的系统发育分析[J]. 动物学杂志, 2018, 53(2): 207-219. Yang Y, Song X J, Tang W Q, et al. Complete mitochondrial genome of Acrossocheilus kreyenbergii, with phylogenetic analysis of genus Acrossocheilus[J]. Chinese Journal of Zoology, 2018, 53(2): 207-219. (  0) 0) |
| [4] |
陈大刚, 张美昭. 中国海洋鱼类[M]. 青岛: 中国海洋大学出版社, 2015: 974, 980. Chen D G, Zhang M Z. Marine Fishes of China[M]. Qingdao: China Ocean University Press, 2015: 974. (  0) 0) |
| [5] |
成庆泰, 郑葆珊. 中国鱼类系统检索[M]. 北京: 科学出版社, 1987: 287-293. Cheng Q T, Zheng B S. Systematic Synopsis of Chinese Fishes[M]. Beijing: Science Press, 1987: 287-293. (  0) 0) |
| [6] |
沈世杰. 台湾鱼类志[M]. 台北: 台湾大学动物学系, 1993: 360-363. Shen S J. Fishes of Taiwan[M]. Taipei: Department of Zoology, Taiwan University, 1993: 360-363. (  0) 0) |
| [7] |
方琼珊, 郑乐云, 林琪, 等. 三种石斑鱼的中文定名问题[J]. 福建水产, 2009(3): 17-21. Fang Q S, Zheng L Y, Lin Q, et al. The denomination of the Chinese names of three species of grouper called "Qingban" as usual name[J]. Journal of Fujian Fisheries, 2009(3): 17-21. DOI:10.3969/j.issn.1006-5601.2009.03.004 (  0) 0) |
| [8] |
Ben-Tuvia A, A Lourie. A Red Sea grouper Epinephelus tauvina caught on the Mediterranean coast of Israel[J]. Israel J Zool, 1969, 18: 245-247. (  0) 0) |
| [9] |
Froese R, Pauly D. Fish Base[DB/OL]. (2018-02-01)[2020-10-08]. http://www.fishbase.org.
(  0) 0) |
| [10] |
Sachithanandam V, Mohan P M, Nagarajan M, et al. Identification and reappraisal of Epinephelus coioides and Epinephelus tauvina through a DNA barcoding approach from the Andaman waters[J]. Indian Journal of Geo-Marine Sciences, 2015, 44(1): 1778-1786. (  0) 0) |
| [11] |
Tan S M, Lim P Y, Tetsushi S K, et al. A Colour Guide to the Fishes of the South China Sea and the Andaman Sea[M]. Singapore: Southeast Asian Fisheries Development Center, 1982: 45.
(  0) 0) |
| [12] |
朱世华, 杨迎春, 郑文娟, 等. 从细胞色素b部分序列探讨石斑鱼属的分子系统发育关系[J]. 水生生物学报, 2006, 30(4): 432-438. Zhu S H, Yang Y C, Zheng W J, et al. Molecular phylogenetic relationship of Epinephelus based on sequences of mtDNA Cytb[J]. Acta Hydrobiologica Sinica, 2006, 30(4): 432-438. DOI:10.3321/j.issn:1000-3207.2006.04.011 (  0) 0) |
| [13] |
周佳怡. 中国沿海石斑鱼亚科的DNA条形码研究[D]. 广州: 暨南大学, 2011: 28-44. Zhou J Y. DNA Barcoding of Epinephelinae in Coastal Waters of China[D]. Guangzhou: Jinan University, 2011: 28-44. (  0) 0) |
| [14] |
Craig M T, Hastings P A. A molecular phylogeny of the groupers of the subfamily Epinephelinae (Serranidae) with a revised classification of the Epinephelini[J]. Ichthyological Research, 2007, 54(1): 1-17. DOI:10.1007/s10228-006-0367-x (  0) 0) |
| [15] |
Schoelinck C, Hinsinger D D, Dettaï A, et al. A Phylogenetic re-analysis of groupers with applications for ciguatera fish poisoning[J]. PLoS One, 2014, 9(8): 1-15. (  0) 0) |
| [16] |
Ma K Y, Craig M T, Chaot J H, et al. The historical biogeography of groupers: Clade diversification patterns and processes[J]. Molecular Phylogenetics and Evolution, 2016, 100: 21-30. DOI:10.1016/j.ympev.2016.02.012 (  0) 0) |
| [17] |
Heemstra P C, Randall J E. Groupers of the World[M]. Rome: FAO Species Catalogue, 1993.
(  0) 0) |
| [18] |
史宝, 柳学周, 刘永山, 等. 黄条鰤线粒体全基因组测序及结构特征分析[J]. 中国水产科学, 2019, 26(3): 405-415. Shi B, Liu X Z, Liu Y S, et al. Complete sequence and gene organization of mitochondrial genome of Seriola aureovittata[J]. Journal of Fishery Sciences of China, 2019, 26(3): 405-415. (  0) 0) |
| [19] |
葛倩芸, 蔡原, 王建福, 等. 鲤和鲫线粒体(mtDNA)全基因组分析[J]. 基因组学与应用生物学, 2020, 39(1): 37-43. Ge Q Y, Cai Y, Wang J F, et al. Complete genome analysis of mtDNA in Carp and Crucian[J]. Genomics and Applied Biology, 2020, 39(1): 37-43. (  0) 0) |
| [20] |
Miya M, Takeshima H, Endo H, et al. Major patterns of higher teleostean phylogenies: A new perspective based on 100 complete mitochondrial DNA sequences[J]. Molecular Phylogenetics and Evolution, 2003, 26(1): 121-138. DOI:10.1016/S1055-7903(02)00332-9 (  0) 0) |
| [21] |
连总强, 滚双宝, 李力, 等. 基于第二代测序技术兰州鲇线粒体基因组全序列测定与分析[J]. 水生生物学报, 2017, 41(2): 334-345. Lian Z Q, Gun S B, Li L, et al. Sequencing and Analysis of the complete mitochondrial genome of Siluruslanzhouensis based on next generation sequencing technologies[J]. Acta Hydrobiologica Sinica, 2017, 41(2): 334-345. (  0) 0) |
| [22] |
Brown W M, Prager E M, Wang A, et al. Mitochondrial DNA sequences of primates: Tempo and mode of evolution[J]. Journal of Molecular Evolution, 1982, 18(4): 225-239. DOI:10.1007/BF01734101 (  0) 0) |
| [23] |
毛明光, 顾杰, 刘瑞婷, 等. 太平洋鳕线粒体全基因组测序及结构特征分析[J]. 水生生物学报, 2019, 43(1): 17-26. Mao M G, Gu J, Liu R T, et al. Analysis of complete mitochondrial genome sequences of Gadus macrocephalus[J]. Acta Hydrobiologica Sinica, 2019, 43(1): 17-26. (  0) 0) |
| [24] |
柳淑芳, 吴仁协, 朱玲, 等. 小黄鱼(Larimichthys polyactis)线粒体基因组结构与特征[J]. 海洋与湖沼, 2010, 41(6): 885-894. Liu S F, Wu R X, Zhu L, et al. Complete sequence and gene organization of mitochondrial DNA of the small yellow croaker Larimichthys polyactis[J]. Oceanologia et Limnologia Sinica, 2010, 41(6): 885-894. (  0) 0) |
| [25] |
张方, 米志勇. 动物线粒体DNA的分子生物学研究进展[J]. 生物工程进展, 1998, 18(3): 25-31. Zhang F, Mi Z Y. Advance in molecular biology of animal mitochondrial DNA[J]. Progress in Biotechnology, 1998, 18(3): 25-31. (  0) 0) |
| [26] |
陈涛. 麦穗鱼(Pseudorasbora parva)线粒体基因组序列测定及分析[D]. 西安: 陕西师范大学, 2010: 30-31. Chen T. Mitochondrial Genome Sequence and Analysia of Pseudorasbora parva[D]. Xi'an: Shaanxi Normal University, 2010: 30-31. (  0) 0) |
| [27] |
Zhuang X, Qu M, Zhang X, et al. A comprehensive description and evolutionary analysis of 22 grouper (Perciformes, Epinephelidae) mitochondrial genomes with emphasis on two novel genome organizations[J]. PLoS One, 2013, 8(8): 1-14. (  0) 0) |
| [28] |
张燕萍, 郑红梅, 邵芳, 等. 沙塘鳢线粒体基因组全序列的测定和分析[J]. 常熟理工学院学报(自然科学), 2016, 30(4): 97-104. Zhang Y P, Zheng H M, Shao F, et al. Sequencing and analysis of the complete mitochondrial genome of Odontobutis[J]. Journal of Changshu Institute Technology(Natural Sciences), 2016, 30(4): 97-104. (  0) 0) |
| [29] |
段晓克. 壮体长春鳊线粒体基因组全长测定及团头鲂密码子偏好模型分析[D]. 武汉: 华中农业大学, 2015: 20. Duan X K. Studies on Complete Mitochondrial Genome of Parabramis pekinensis strenosoma and Codon Usage Patterns of Megalobrama amblycephala[D]. Wuhan: Huazhong Agricultural University, 2015: 20. (  0) 0) |
| [30] |
Inoue J G, Miya M, Tsukamoto K, et al. A mitogenomic perspective on the basal teleostean phylogeny: Resolving higher-level relationships with longer DNA sequences[J]. Molecular Phylogenetics and Evolution, 2001, 20(2): 275-285. (  0) 0) |
| [31] |
Hebert P D N, Ratnasingham S, Ward J R. Barcoding animal life: Cytochrome c oxidase subunit 1 divergences among closely related species[J]. Proceedinngs of the Royal Society Biological Sciences, 2003, 270: 96-99. (  0) 0) |
| [32] |
Avise J C, Walker D. Species realities and numbers in sexual vertebrates: perspective from an asexually transmitted genome[J]. Proceedings of the National Academy of Sciences of the United States of America, 1999, 96(3): 992-995. (  0) 0) |
| [33] |
王颖汇. 中国近海部分石斑鱼类分子系统学的研究[D]. 厦门: 厦门大学, 2007: 10-11, 26-56. Wang Y H. Molecular Phylogenetic Relationships of Part China Seas Groupers[D]. Xiamen: Xiamen University, 2007: 10-11, 26-56. (  0) 0) |
| [34] |
Govindaraju G S, Jayasankar P. Taxonomic relationship among seven species of groupers (genus Epinephelus; family Serranidae)as revealed by RAPD fingerprinting[J]. Marine Biotechnology, 2004, 6: 229-237. (  0) 0) |
2. Guangdong Provincial Water Environment and Aquatic Products Security Engineering Technology Research Center, Guangzhou 510225, China;
3. Guangdong Province Key Laboratory of Waterfowl Healthy Breeding, Guangzhou 510225, China;
4. Guangzhou Key Laboratory of Aquatic Animal Diseases and Waterfowl Breeding, Guangzhou 510225, China
 2022, Vol. 52
2022, Vol. 52


