牡蛎是世界性广布种类,是我国乃至世界最重要的海产经济贝类之一。2016年中国牡蛎养殖产量达483万t,居世界首位[1]。长牡蛎(Crassostrea gigas)又称太平洋牡蛎,是我国北方沿海最主要的养殖种类。在北方海域长牡蛎的繁殖期在每年的5~8月,此时体内大部分营养用于繁殖后代,体内糖原大量消耗,软体部重量急剧减少,口感差,不宜供应市场。因此,牡蛎养殖业亟需引进优良牡蛎新品种来供应夏季市场。
岩牡蛎(Crassostrea nippona),又称夏牡蛎,属珍珠贝目(Pterioida)牡蛎科(Ostreidae),主要分布在日本、韩国等东亚地区,具有抗病能力强、适应范围广、生长速度快、产量高等特点。岩牡蛎产卵期在8~9月,且有多批次放精产卵的特点,夏季体内糖原含量仍保持在较高水平,味道较好,弥补了长牡蛎夏季产卵不能上市的空缺,深受市场欢迎[2]。
为了丰富我国牡蛎的养殖种类,我们从日本引进岩牡蛎进行人工育苗和良种选育。在引种繁育中发现,岩牡蛎与长牡蛎成体区别明显,但稚贝不易区分。由于牡蛎的外壳可塑性较强和分布区域的重叠性,仅仅依据外部形态学特征鉴定牡蛎种类存在很强的主观因素,这给牡蛎新品种的选育与养殖带来极大困难。因此,迫切需要一种快速有效的鉴别岩牡蛎与长牡蛎的技术方法。
聚合酶链式反应-限制性片段长度多态性分析(PCR-RFLP)技术是一种常用的快速简便分析物种组成的方法[3],与PCR产物测序的序列对比鉴定方法相比,其操作简单、快捷,节约成本。本研究基于岩牡蛎与长牡蛎16S rDNA与COI基因片段开发了一种快速高效的PCR-RFLP鉴定技术,首次从分子生物学水平上鉴别区分这2种重要的经济贝类。
1 材料和方法 1.1 实验材料实验所用的岩牡蛎与长牡蛎取自山东荣成养殖基地。每种牡蛎各取壳高2~3 cm的稚贝18个个体,解剖取闭壳肌,于-80 ℃超低温冰箱保存,用于提取DNA。
1.2 PCR扩增与测序采用常规“酚—氯仿”抽提法提取样品基因组DNA,用1.0%琼脂糖凝胶电泳检测所提取的DNA质量,并用超微量分光光度计(NanoDrop 2000)测定DNA浓度,最终稀释至50 ng/μL。参照Palumbi等[4]设计的引物(16Sar:5′-CGCCTGTTTATCAAAAACAT-3′,16Sbr:5′-CCGGTCTGAACTCAGATCACGT-3′),以基因组DNA为模板,扩增岩牡蛎和长牡蛎的16S rDNA片段。根据岩牡蛎(GenBank登录号:HM015198)与长牡蛎(GenBank登录号:AB033687)线粒体COI的相对保守区域,利用Premier 5.0软件设计出一对引物序列(COIY-F:5′-CTATTAGAAATATGCGGTCTG-3′,COIY-R:5′-AACAAAGTAAGTATCGTGAAG-3′),用于扩增2种牡蛎的COI基因片段。PCR反应体系为10 μL:50 ng DNA,0.25 U Taq DNA聚合酶,1 μL 10× PCR buffer,0.8 μL dNTPs,1 μmol/L上下引物,5.15 μL灭菌超纯水。PCR反应程序为:94 ℃预变性3 min;94 ℃变性30 s,56 ℃(16S rDNA)或54 ℃(COI)退火45 s,72 ℃延伸1 min,共32个循环;72 ℃延伸5 min,4 ℃保存。取2 μL PCR产物,经1.5%的琼脂糖凝胶电泳成像检测。随机挑选部分个体的PCR产物送至青岛华大基因生物有限公司进行测序。
1.3 PCR-RFLP分析测序得到的序列,运用软件Clustal W进行序列比对。使用软件BioEdit进行酶切位点的分析,最后选取同一种内切酶Alu Ⅰ对COI与16S rDNA基因片段进行酶切。酶切反应体系为10 μL,包括5 μL的PCR产物,0.5 μL的限制性内切酶Alu Ⅰ(5 U;Takara),1 μL的10 × buffer酶切缓冲液,3.5 μL的灭菌超纯水。酶切反应体系在37 ℃反应过夜,酶切产物用2.0%琼脂糖凝胶电泳成像检测。
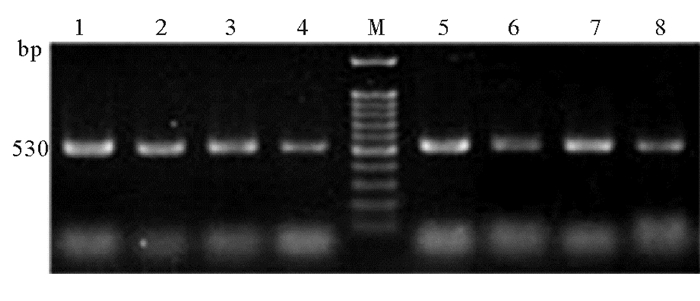
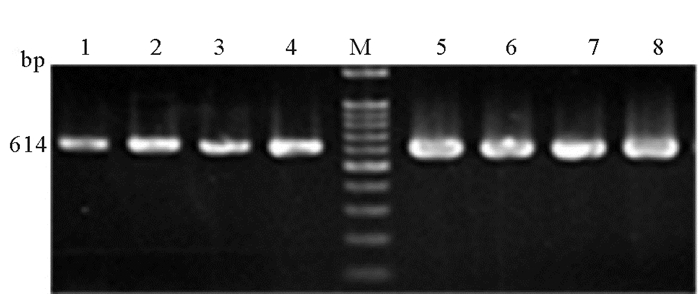
2 结果与分析 2.1 PCR扩增结果PCR扩增结果如图 1和图 2所示,岩牡蛎和长牡蛎16S rDNA与COI均扩增出一条单一片段,未发现其片段长度多态性。16S rDNA测序得到的片段大小为530 bp(见图 1),COI测序得到的片段大小为614 bp(见图 2)。
|
(1~4:长牡蛎;5~8:岩牡蛎;M:100 bp DNA marker。1~4:C. gigas;5~8:C. nippona;M:100 bp DNA marker.) 图 1 长牡蛎和岩牡蛎16S rDNA片段PCR产物电泳图 Fig. 1 Electrophoretic profile of PCR products of 16S rDNA of C. gigas and C. nippona |
|
(1~4:长牡蛎;5~8:岩牡蛎;M:100 bp DNA marker。1~4:C. gigas;5~8:C. nippona;M. 100 bp DNA marker.) 图 2 长牡蛎和岩牡蛎COI片段PCR产物电泳图 Fig. 2 Electrophoretic profile of PCR products of COI of C. gigas and C. nippona |
序列比对及酶切位点分析后发现,在长牡蛎16S rDNA扩增产物中距多核苷酸序列5′端的第457和458位碱基之间的磷酸二酯键为限制性内切酶Alu Ⅰ酶切位点(AGCT),而岩牡蛎16S rDNA扩增产物有2个Alu Ⅰ酶切位点,分别在370和371碱基之间和406和407碱基之间(见图 3)。在长牡蛎COI扩增产物中距多核苷酸序列5′端的第102和103位碱基间的磷酸二酯键与第576和577位碱基间的磷酸二酯键为内切酶Alu Ⅰ酶切位点,岩牡蛎COI扩增产物只有1个Alu Ⅰ酶切位点,在第378和379碱基之间(见图 4)。

|
(下划线标出的为Alu Ⅰ酶切位点。Sites of Alu Ⅰ(AGCT) are underlined with single lines.) 图 3 长牡蛎和岩牡蛎16S rDNA序列比对结果及酶切位点 Fig. 3 Sequence alignment and restriction-site polymorphisms in 16S rDNA sequences of C. gigas and C. nippona |

|
(下划线标出的为Alu Ⅰ酶切位点。Sites of Alu Ⅰ(AGCT) are underlined with single lines.) 图 4 长牡蛎和岩牡蛎COI序列比对结果及酶切位点 Fig. 4 Sequence alignment and restriction-site polymorphisms in COI sequences of C. gigas and C. nippona |
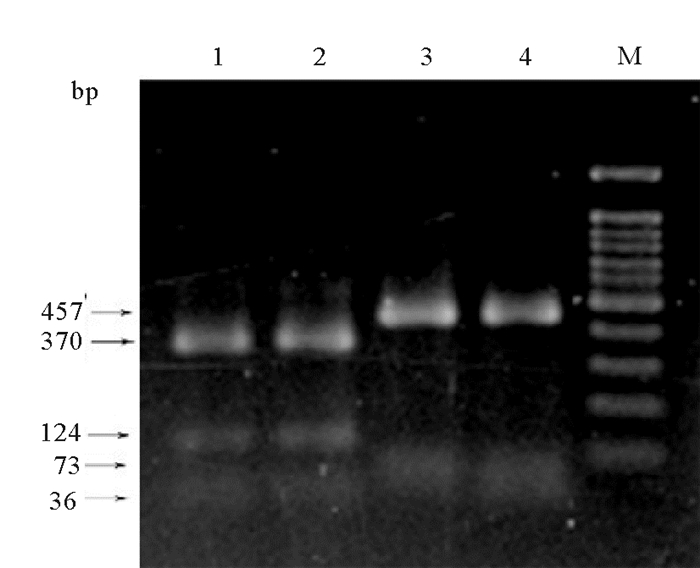
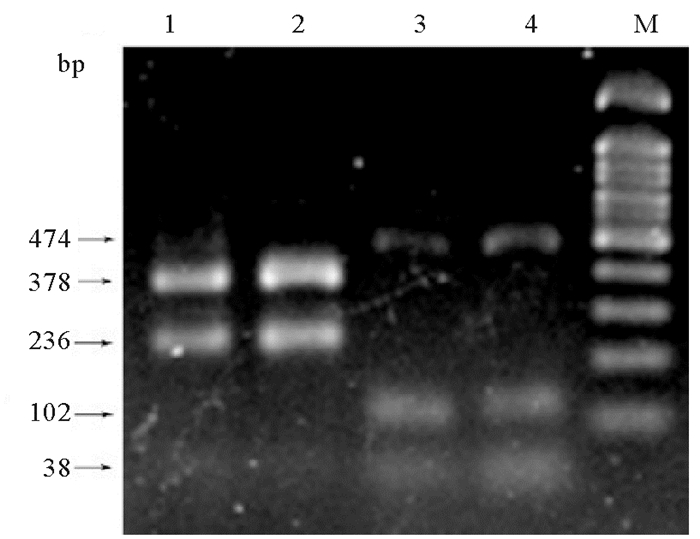
限制性内切酶Alu Ⅰ对岩牡蛎和长牡蛎的PCR扩增产物的酶切结果如图 5和6所示,与预期酶切分析结果一致。岩牡蛎16S rDNA片段酶切后得到36、124和370 bp 3个片段,而长牡蛎16S rDNA扩增片段经Alu Ⅰ酶切后得到73和457 2个片段(见图 5)。COI基因片段经Alu Ⅰ酶切后,岩牡蛎产生236和378 2个片段,长牡蛎产生38、102和474 bp 3个片段(见图 6)。虽然有些酶切片段因为太小在琼脂糖凝胶成像中显示模糊,但这并不影响将这2种牡蛎直观地区分开来。
|
(1~2:岩牡蛎;3~4:长牡蛎;M:100 bp DNA marker。1~2:C. nippona;3~4:C. gigas;M:100 bp DNA marker.) 图 5 长牡蛎和岩牡蛎16S rDNA酶切结果 Fig. 5 RFLP patterns of 16S rDNA of C. gigas and C. nippona after digestion with Alu Ⅰ |
|
(1~2:岩牡蛎;3~4:长牡蛎;M:100 bp DNA marker。1~2: C. nippona;3~4: C. gigas;M:100 bp DNA marker.) 图 6 长牡蛎和岩牡蛎COI酶切结果 Fig. 6 RFLP patterns of COI of C. gigas and C. nippona after digestion with Alu Ⅰ |
岩牡蛎是正在开展全人工繁育的牡蛎养殖新品种,具有较高的经济价值。在引种繁育阶段,牡蛎物种的鉴定十分重要。牡蛎品种出现混杂,会直接影响人工育苗效果。由于牡蛎外部形态特征(如贝壳形状)常随栖息环境的不同而发生很大的改变,依据传统形态分类的方法对其进行物种鉴定存在很大困难。随着科学技术的发展,分子生物学技术的广泛应用解决了此类难题。在以前的研究中,多种分子生物学技术已经应用到牡蛎的物种鉴别中,包括测序[5],变性梯度凝胶电泳(GDDE)[6],特异性多重PCR[7],高分辨率熔解曲线(HRM)[8]等技术。上述方法通常成本较高,或操作繁琐,或设备昂贵,对操作人员技术要求较高。
PCR-RFLP技术是在PCR和DNA序列分析基础上发展产生的RFLP技术,因方法简单、快捷、成本较低,已广泛应用于水产经济物种的分析鉴定。例如,Sumathi等[9]利用PCR-RFLP技术成功鉴定了5种石斑鱼;庄怡等[10]利用PCR-RFLP技术成功鉴定鲫鱼6个不同品系;文菁等[11]利用开发的PCR-RFLP方法成功鉴定了16种经济海参;Pie等[12]利用PCR-RFLP技术快速鉴定了巴西海岸3种重要的养殖牡蛎。
线粒体DNA作为真核细胞的核外遗传因子,结构简单,易于分离,进化速度比核DNA快,已广泛应用于水产动物的种类鉴别中[13-14]。线粒体DNA中,16S rDNA与COI基因片段保守性相对较高,引物可以种间通用,是种类鉴别中常用的基因片段。本研究选用Palumbi等[4]开发的一对通用引物在2种牡蛎中成功扩增出一条530 bp的16S rDNA基因片段,同时设计筛选了一对特异性引物,在岩牡蛎与长牡蛎中成功扩增出一条长614 bp的线粒体COI片段,PCR产物被限制性内切酶Alu Ⅰ酶切后,经琼脂糖凝胶电泳成像显示出大小不同的酶切片段,成功将岩牡蛎与长牡蛎鉴别出来。随机挑选了岩牡蛎与长牡蛎各8个个体进行了测序,测序结果与酶切实验结果一致,未发现酶切位点突变个体,验证了本研究PCR-RFLP鉴定方法的可行性与可靠性。
本研究基于线粒体DNA PCR-RFLP技术开发出的岩牡蛎与长牡蛎鉴定方法操作简单,仅仅需要PCR扩增目的基因片段,经特异性内切酶酶切、电泳后,即可有效鉴别岩牡蛎与长牡蛎。鉴定结果准确可靠,有利于节约成本,提高牡蛎样品的鉴定效率,同时为岩牡蛎养殖新品种的开发提供了技术手段。
4 结语本研究基于岩牡蛎与长牡蛎16S rDNA与COI基因片段开发了一种快速高效的PCR-RFLP鉴定方法。利用通用引物与本研究开发引物扩增2种牡蛎的线粒体16S rDNA与COI基因片段,应用同一种限制性核酸内切酶Alu Ⅰ对PCR产物进行酶切,并对酶切产物进行琼脂糖凝胶电泳。根据酶切图谱可有效鉴别岩牡蛎与长牡蛎。这种简单、可靠、经济的PCR-RFLP鉴定方法,能够为岩牡蛎养殖新品种的开发提供技术支撑。
| [1] |
农业部渔业渔政管理局. 中国渔业统计年鉴2016[M]. 北京: 中国农业出版社, 2016. Fisheries and Fishery Administration, the Ministry of Agriculture. China Fishery Statistical Yearbook 2016[M]. Beijing: China Agriculture Press, 2016. (  0) 0) |
| [2] |
李文姬. 岩牡蛎的生物学及其养殖[J]. 水产科学, 2007, 26(12): 689-690. Li W J. Biology and cultivation of oyster Crassostrea nippona[J]. Fisheries Science, 2007, 26(12): 689-690. DOI:10.3969/j.issn.1003-1111.2007.12.013 (  0) 0) |
| [3] |
Karl S A, Bowen B W, Avise J C. Global population genetic structure and male-mediated gene flow in the green turtle (Chelonia mydas): RFLP analyses of anonymous nuclear loci[J]. Genetics, 1992, 131(1): 163-173.
(  0) 0) |
| [4] |
Palumbi S R, Martin A, Romano S, et al. The Simple Fool's Guide to PCR[M]. Honolulu: University of Hawaii Press, 1991.
(  0) 0) |
| [5] |
Reece K S, Cordes J F, Stubbs J B, et al. Molecular phylogenies help resolve taxonomic confusion with Asian Crassostrea oyster species[J]. Marine Biology, 2008, 153(4): 709-721. DOI:10.1007/s00227-007-0846-2
(  0) 0) |
| [6] |
Yu H, Li Q. Denaturing gradient gel electrophoresis as a tool for the identification of commercially important Crassostrea oysters in China[J]. Fisheries Science, 2008, 74(6): 1354-1356. DOI:10.1111/fis.2008.74.issue-6
(  0) 0) |
| [7] |
Wang H, Guo X. Identification of Crassostrea ariakensis and related oysters by multiplex species-specific PCR[J]. Journal of Shellfish Research, 2008, 27(3): 481-487. DOI:10.2983/0730-8000(2008)27[481:IOCAAR]2.0.CO;2
(  0) 0) |
| [8] |
Jin Y L, Li Q, Kong L F, et al. High-resolution melting (HRM) analysis: A highly sensitive alternative for the identification of commercially important Crassostrea oysters[J]. Journal of Molluscan Studies, 2014, 81(1): 167-170.
(  0) 0) |
| [9] |
Sumathi G, Jeyasekaran G, Shakila R J, et al. Molecular identification of grouper species using PCR-RFLP technique[J]. Food Control, 2015, 51: 300-306. DOI:10.1016/j.foodcont.2014.11.026
(  0) 0) |
| [10] |
庄怡, 刘必谦, 唐杰. 利用PCR-RFLP技术对鲫鱼6个不同品系的鉴定[J]. 生物技术通报, 2012(11): 144-149. Zhuang Y, Liu B Q, Tang J. Identifying six strains of Carassius auratus by PCR-RFLP on mtDNA ND3/ND4L/ND4 gene[J]. Biotechnology Bulletin, 2012(11): 144-149. (  0) 0) |
| [11] |
文菁, 胡超群, 张吕平, 等. 16种商品海参16S rRNA的PCR-RFLP鉴定方法[J]. 中国水产科学, 2011, 18(2): 451-457. Wen J, Hu C Q, Zhang L P, et al. PCR-RFLP identification of 16 commercial sea cucumber species on the basis of 16S rRNA gene[J]. Journal of Fishery Sciences of China, 2011, 18(2): 451-457. (  0) 0) |
| [12] |
Pie M R, Ribeiro R O, Boeger W A, et al. A simple PCR-RFLP method for the discrimination of native and introduced oyster species (Crassostrea brasiliana, C. rhizophorae and C. gigas; Bivalvia: Ostreidae) cultured in Southern Brazil[J]. Aquaculture Research, 2006, 37(15): 1598-1600. DOI:10.1111/are.2006.37.issue-15
(  0) 0) |
| [13] |
Bakke I, Johansen S D. Molecular phylogenetics of gadidae and related gadiformes based on mitochondrial DNA sequences[J]. Marine Biotechnology, 2005, 7(1): 61-69. DOI:10.1007/s10126-004-3131-0
(  0) 0) |
| [14] |
Klinbunga S, Khamnamtong N, Tassanakajon A, et al. Molecular genetic identification tools for three commercially cultured oysters (Crassostrea belcheri, Crassostrea iredalei, and Saccostrea cucullata) in Thailand[J]. Marine Biotechnology, 2003, 5(1): 27-36. DOI:10.1007/s10126-002-0047-4
(  0) 0) |
 2018, Vol. 48
2018, Vol. 48


