2. 中国海洋大学海洋生物多样性与进化研究所, 山东 青岛 266003;
3. 青岛海洋科学与技术国家实验室, 山东 青岛 266100
细胞核是细胞中最大的细胞器,在不同的细胞类型、细胞周期的不同阶段[1-2]、迁移状态[3]和分化状态[4]下,细胞核的位置是不同的。细胞核位置对细胞骨架组织、信号传导[5-8]和转录控制[9-10]具有重要的意义。研究发现,调控细胞核运动的细胞质机械力系统主要通过与核膜的连接作用于细胞核[4, 11-14],在细胞迁移和分化等过程中维持细胞核的位置或位移。LINC复合体就是介导细胞质机械力连接到核膜上的一个重要连接装置。LINC复合体包括KASH结构域蛋白、SUN结构域蛋白及其在核内、核外的分子连接网络[13]。KASH蛋白定位于外核膜上,因其C端跨膜区的氨基酸序列与人的Syne-1和Syne-2蛋白的C端氨基酸序列、果蝇的Klarsicht蛋白和秀丽隐杆线虫的ANC-1蛋白同源而得名[15-16],这段序列被称为Klarsicht/ANC-l/Syne Homology (KASH)结构域。LINC复合物的特异性由KASH蛋白的N端决定,KASH蛋白的大小和结合细胞骨架种类的能力各不相同。SUN蛋白定位于内核膜上,因其C端氨基酸的序列与酵母Sad-1蛋白和线虫UNC-84蛋白的存在一定的相似性而得名,这段序列被称为SUN结构域[17]。KASH蛋白和SUN蛋白通过特定的结构域结合。有研究报道,这2类蛋白具有连接细胞核骨架与细胞质骨架的功能,并能够将胞质细胞骨架的机械力传导至核骨架上,从而引起核形变及位置的变化等[4, 13, 18-21]。基于模式生物的研究结果表明,LINC复合体结构破坏会导致多种细胞功能异常,如细胞迁移和卵发育异常、受精卵分裂失败等,KASH蛋白和SUN蛋白功能缺陷或丧失也会导致一系列的人类疾病,如Emery-Dreifuss肌营养不良症、心肌病等[13, 22-23]。
海鞘(Ciona robusta)属于脊索动物门、尾索动物亚门,是无脊椎动物向脊椎动物进化的中间类群。海鞘的脊索在幼虫期为两端闭合的直管,而在幼体向成体变态发育过程中退化消失。海鞘脊索发育的最初步骤是将原肠胚前端的单层上皮样细胞转变为圆柱形杆状细胞,第一阶段是汇聚延伸,包括脊索细胞的间插运动和一定程度的脊索伸长,而第二阶段的伸长,是脊索在缢缩环的作用下变窄并沿前后轴延长和增加体积[24-25]。值得注意的是,在胚胎发育的延伸阶段,脊索细胞的细胞核定位在细胞的后侧[26],且这一现象仅存在于延伸时期。这一主动的核位移受到PCP (Planar cell polarity)极性信号的调控,且对于海鞘胚胎发育或器官形成过程有重要作用,但脊索细胞平衡细胞核位置的机制以及LINC复合体是否参与这个过程尚不清楚。目前,LINC复合体中的关键元件KASH和SUN蛋白已经在一些后生动物、真菌和植物中被发现和阐释[4, 15, 27],查阅相关数据得知,在海鞘体内存在KASH和SUN蛋白。但是其在海鞘不同发育时期的表达模式目前不清楚。
1 材料与方法 1.1 实验动物采集与样品保存本研究选取的实验动物为玻璃海鞘采集于山东省荣成市寻山公司养殖区,采集后,清洗表面淤泥及杂质,然后循环海水中进行暂养养殖条件:水温为(18±1) ℃,盐度为33±2,持续通气,选择状态良好的动物进行后续实验。
1.2 实验动物卵的受精与胚胎收集取3~5只性腺发育成熟成体海鞘,分别取卵和精子,精子和卵混合受精5~10 min后清洗受精卵。放置于装满海水的培养皿中培养。收集正常发育至不同阶段的胚胎备用。
1.3 KASH和SUN基因的克隆从ANISEED网站中获取玻璃海鞘的转录组数据[28-29],用已知物种的KASH和SUN的核苷酸序列和氨基酸序列与转录组数据中通过序列比对,最终共获得2条KASH序列和2条SUN序列。使用美国国立生物技术信息中心数据库(NCBI)的Open Reading Frame Finder工具初步预测这4条序列的开放阅读框序列,据此结果设计引物并由公司合成(上海生物工程公司)(见表 1)。然后以玻璃海鞘的cDNA为模板,PCR扩增出以上序列,经过切胶回收后将基因连接到blunt3载体上,经转化后送上海生物工程公司测序。
|
|
表 1 本研究所用引物序列 Table 1 Primer sequences used in this study |
使用NCBI网站的Conserved Domains Database工具和SMART网站预测蛋白质的结构域;使用MEGA 10.0软件构建系统进化树,使用GENEDOC软件和DNAMAN软件进行序列比对并将比对结果可视化。
1.5 荧光定量PCR使用Primer5软件根据序列设计扩增片段长为80~150 bp的引物(见表 1),送至上海生物工程公司合成,使用南京诺唯赞公司的模板示踪型染料法定量PCR检测试剂盒ChamQTM SYBR Color qPCR Master Mix(货号Q411-02)根据操作流程进行预实验,确定条带单一且CQ值正常,并进行实验。使用Excel软件计算Cr_KASH1、Cr_KASH2、Cr_SUN1和Cr_SUN2基因的相对表达量,使用Prism 9.0软件计算Cr_KASH1、Cr_KASH2、Cr_SUN1和Cr_SUN2不同发育时期表达量之间的显著性差异。
1.6 原位杂交使用Primer5软件根据序列设计扩增片段长为400 bp的引物(见表 1),送至上海生物工程公司合成。原位杂交参考Christiaen等[30]的实验方法,杂交温度为53~56 ℃。
1.7 电穿孔和细胞系转染根据先前报道的海鞘胚胎电穿孔实验的方法进行电穿孔实验[31]。使用Lipofectamine 3000试剂转染NIH/3T3细胞系质粒(供应商:Thermo Fisher Scientific)。
1.8 抑制剂处理转染后的细胞,在37 ℃、5% CO2的条件下培养至细胞覆盖24孔细胞培养板孔板底部面积的80%,然后添加200 nmol/L红海海绵素B(Latrunculin B,Sigma, L5288)或1.6 μmol/L诺考达唑(Nocodazole, Sigma, M1404)于培养液中孵育30 min。PBS清洗后,用4%的多聚甲醛固定,用phalloidin及DAPI染色后封片并进行荧光观察。
2 结果分析 2.1 海鞘KASH和SUN基因序列分析通过序列比对,在海鞘基因组里找到2段与KASH蛋白有同源性的序列,Cr_KASH1 (Transcript ID : KH2012:KH.C3.502.v1.A.SL3-1)和Cr_KASH2(Transcript ID: KH2012:KH.C3.564.v2.A.SL2-1)。其中Cr_KASH1与Homo sapiens SYNE1的同源性为53.08%,Cr_KASH2与Homo sapiens SYNE2的同源性为31.49%,两段与SUN蛋白同源性较高的序列是Cr_SUN1(Transcript ID: KH2012:KH.L18.41.v1.A.SL1-1)和Cr_SUN2(Transcript ID: KH2012:KH.L40.14.v1.B.SL1-1)。其中Cr_SUN1与Danio rerio SUN1的同源性为51.56%,Cr_KASH2与Drosophila virilis SUN2的同源性为17.05%。NCBI网站的ORF Finder工具预测分析显示,Cr_KASH1的开放阅读框(ORF)长4 407 bp,所编码蛋白包含3个与细胞骨架结合的SPEC结构域和1个与SUN蛋白结合的KASH结构域,以及1个跨越核膜的螺旋结构域;Cr_KASH2的ORF长1 926 bp,所编码蛋白包括3个与细胞骨架结合的SPEC结构域和1个螺旋卷曲;Cr_SUN1的ORF由2 562 bp构成,所编码蛋白包含1个SUN结构域;Cr_SUN2的ORF长为1 590 bp,所编码蛋白缺失SUN结构域。
2.2 海鞘KASH和SUN同源比对和进化分析将海鞘KASH蛋白和SUN蛋白与NCBI中的其他物种的KASH蛋白和SUN蛋白的氨基酸序列进行比对,发现他们在KASH结构域和SUN结构域上同源(见图 1)。利用MEGA 10.0软件的Neighbor-Joining分析法对氨基酸序列进行系统发育进化分析、KASH的系统发育进化树显示,Cr_KASH1同哺乳动物的Syne-1和Syne-2蛋白的亲缘关系最接近,Cr_KASH2同果蝇的MSP-300(Muscle-specific protein 300 kDa)亲缘关系最近(见图 2A)。MSP-300是果蝇内发现的一种特有KASH蛋白,同果蝇的肌肉组织细胞核的定位有关[32]。根据SUN的系统发育进化树可以看出,Cr_SUN1和Cr_SUN2均在同一分支,同哺乳动物亲缘关系较远,Cr_SUN2同线虫体内的UNC-83亲缘关系最近(见图 2B)。用于同源比对和进化分析的序列来源总结于表 2。

|
(A: 玻璃海鞘Cr_KASH1和Cr_KASH2与其他模式生物KASH蛋白的KASH结构域,红色星号标记玻璃海鞘中的2种KASH蛋白。B: 玻璃海鞘Cr_SUN1和Cr_SUN2与其他模式生物SUN蛋白的SUN结构域,红色星号标记玻璃海鞘中的2种SUN蛋白。C: 编码Cr_KASH和Cr_SUN蛋白的基因结构分析。A: The KASH domains of Cr_KASH1 and Cr_KASH2 in Ciona robusta and KASH proteins in other model species. Two KASH proteins in Ciona robusta are marked by red asterisks. B: The SUN domains of Cr_SUN1 and Cr_SUN2 in Ciona robusta and SUN proteins in other model species. Two SUN proteins in Ciona robusta are marked by red asterisks. C: Structural analysis of genes encoding Cr_KASH and Cr_SUN. ) 图 1 海鞘KASH和SUN的序列比对 Fig. 1 Sequence alignment of Ciona robusta KASH and SUN |

|
(A.邻接法构建的KASH蛋白的系统进化树。红色圆点指示海鞘KASH蛋白在进化树中的位置。B. 邻接法构建的SUN蛋白的系统进化树。红色三角指示海鞘SUN蛋白在进化树中的位置。构建进化树所用的氨基酸序列与图 3一致。A. Phylogenetic tree of KASH protein, red dots indicate the position of KASH protein in the Ciona robusta. B. Phylogenetic tree of SUN protein, red triangles indicate the position of SUN protein in the Ciona robusta. The amino acid sequence used to construct the phylogenetic tree was consistent with Figure 3. ) 图 2 KASH蛋白和SUN蛋白的系统进化树 Fig. 2 Phylogenetic tree of KASH and SUN proteins |
|
|
表 2 用于同源比对和进化分析的序列 Table 2 Sequences for homology alignment and evolutionary analysis |
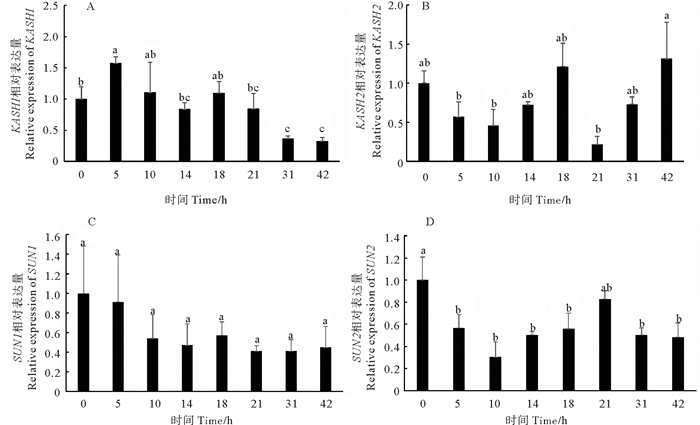
荧光定量结果显示,Cr_KASH1基因从受精卵到胚胎尾部退缩完成期间均有表达,其中从受精卵时期到110细胞期期间表达量较高,而在受精后10 h时表达量下降,在受精后10~14 h表达量维持在较低水平。而在受精后14~18 h期间Cr_KASH1基因的表达量开始出现了回升,在受精后18 h后相对表达量又持续下降(见图 3A)。Cr_KASH2基因从受精卵到胚胎尾部退缩完成期间均有表达,在110细胞期表达量较高,随后在受精后10 h表达量下降,受精后10~14 h表达量有回升,而在受精后14~18 h表达量显著升高,受精后18~21 h表达量显著下降,受精后31~42 h时期表达量显著上升(见图 3B)。
|
(A: Cr_KASH1基因在不同发育时期的表达水平。B: Cr_KASH2基因在不同发育时期的表达水平。C: Cr_SUN1基因在不同发育时期的表达水平。D: Cr_SUN2基因在不同发育时期的表达水平。同一基因不同字母表示不同时期胚胎中的表达量存在显著性差异(P<0.05)。A: The relative expression level of Cr_KASH1 at different developmental stages. B: The relative expression level of Cr_KASH2 at different developmental stages. C: The relative expression level of Cr_SUN1 at different developmental stages. D: The relative expression level of Cr_SUN2 at different developmental stages. For each gene, different letters indicate significant differences(P < 0.05)among the embryo at the different stages. ) 图 3 玻璃海鞘KASH和SUN基因在不同发育时期的表达水平 Fig. 3 The relative expression level of KASH and SUN at different development stage of Ciona robusta |
海鞘Cr_SUN1基因从受精卵到胚胎尾部退缩完成期间均有表达,各时期间的表达量无显著差异(见图 3C)。Cr_SUN2基因从受精卵到胚胎尾部退缩完成期间均有表达,从受精卵时期到110细胞期表达量较高,随后在10 h时期表达量下降,10~14 h期表达量保持在较低水平,而在受精后14~21 h期间表达量有回升,但受精后21 h相对表达量下降(见图 3D)。
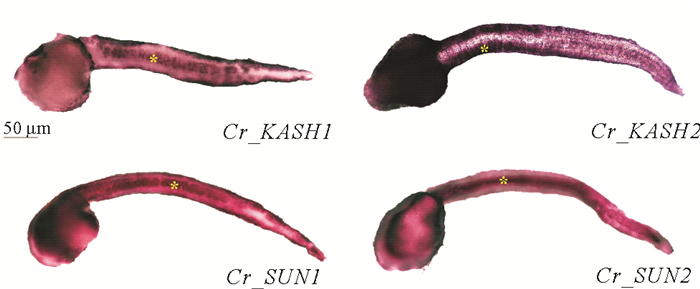
2.4 Cr_KASH、Cr_SUN在胚胎发育早期的表达模式为了确定Cr_KASH和Cr_SUN在海鞘胚胎组织中的表达模式,本文作者对上述4个基因在胚胎尾芽期进行了原位杂交分析。染色结果显示:Cr_KASH1在表皮和脊索组织位置高表达; Cr_KASH2在头部和表皮组织中高表达,脊索处未检测到明显信号; Cr_SUN1和Cr_SUN2均在脊索和表皮组织高表达(见图 4)。
|
(图中黄色星号指示原位杂交探针信号位置,Cr_KASH1-脊索,Cr_KASH2-肌肉,Cr_SUN1-脊索,Cr_SUN2-脊索。The yellow asterisk indicates the signal location of the in situ hybridization probe, Cr_KASH1-notochord, Cr_KASH2-muscle, Cr_SUN1-notochord, and Cr_SUN2-notochord. ) 图 4 KASH和SUN基因在玻璃海鞘胚胎中的表达模式 Fig. 4 Expression patterns of KASH and SUN genes in the embryos of Ciona robusta |
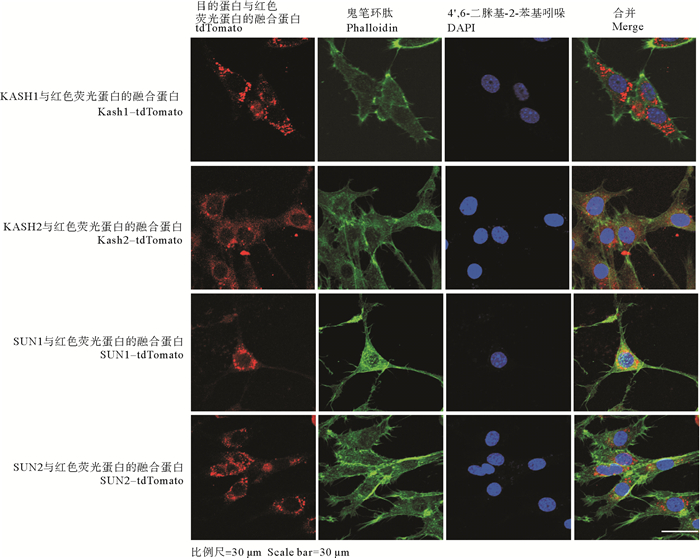
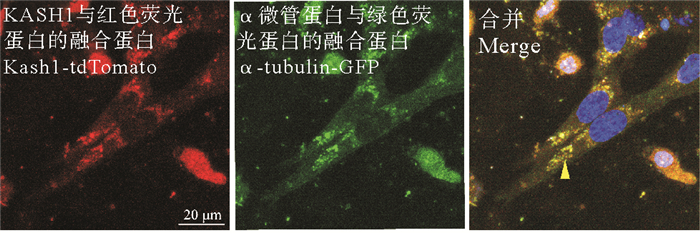
以往有研究报道,NIH/3T3细胞在迁移过程中细胞核居于细胞运动方向的后侧[3, 33],这一定位模式与海鞘脊索细胞的细胞核向后运动的现象相似。为了观察Cr_KASH和Cr_SUN在细胞中的位置,本文作者在NIH/3T3细胞系中表达Cr_KASH和Cr_SUN,并观察它们的亚细胞定位。结果显示,Cr_KASH1在NIH/3T3细胞中定位在细胞核周围,主要集中在其两极(见图 5)。进而与微管分布进行对比发现Cr_KASH1与α-tubulin可以共定位(见图 6)。与Cr_KASH1不同,Cr_KASH2除定位在核周之外,在胞质中也有分布,用Phalloidin染色后观察到,Cr_KASH2与F-actin有共定位。Cr_SUN1、Cr_SUN2都围绕核周分布(见图 5)。
|
图 5 KASH蛋白和SUN蛋白在NIH/3T3细胞中的亚细胞定位 Fig. 5 Sub-cellular localization of KASH and SUN proteins in the NIH/3T3 cell lines |
|
(图中黄色箭头指示Cr_KASH1和α-tubulin共同定位的位置。比例尺=20 μm。The yellow arrow indicates the location of both Cr_KASH1 and α-tubulin. Scale bar=20 μm. ) 图 6 Cr_KASH1和α-tubulin在NIH/3T3细胞中的定位 Fig. 6 Localization of KASH1 protein and α-tubulin in the NIH/3T3 cell lines |
由于Cr_KASH1与α-tubulin以及Cr_KASH2与F-actin有共定位的现象,本文作者进一步破坏细胞微丝系统或微管系统后,观察这2个蛋白的定位变化。诺考达唑处理的细胞中,α-tubulin的分布也发生紊乱,证实药物发挥了作用,且Cr_KASH1蛋白大部分在细胞质中呈弥散状分布,仅有少量在核膜上分布。加入红海海绵素B后,部分Cr_KASH2蛋白围绕核周,其余在细胞质中呈弥散状分布(见图 7)。
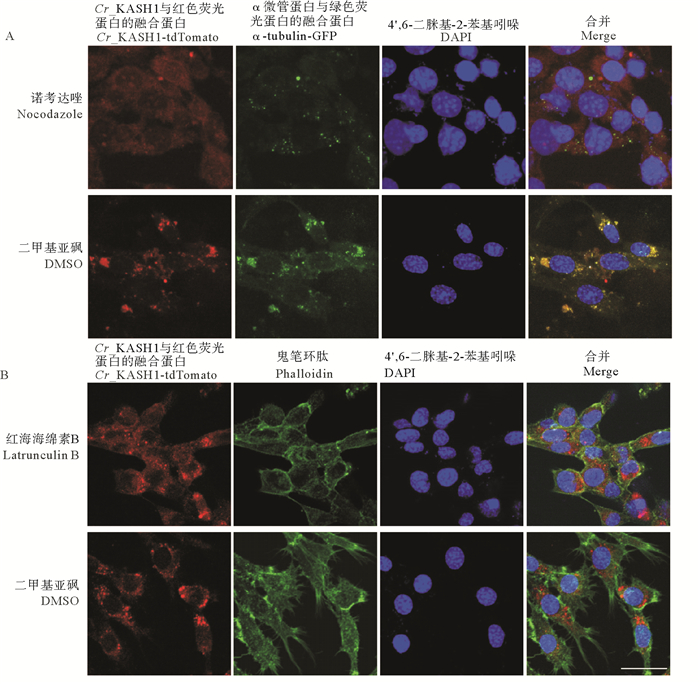
|
(A: 诺考达唑处理后,Cr_KASH1蛋白(红色)和α-tubulin(绿色)的亚细胞定位,以及对照组(加等量DMSO),Cr_KASH1蛋白(红色)和α-tubulin(绿色)的亚细胞定位。B: 红海海绵素处理后,Cr_KASH2蛋白(红色)和phalloidin(绿色)的亚细胞定位,以及对照组(加等量DMSO),Cr_KASH2蛋白(红色)和phalloidin(绿色)的亚细胞定位。比例尺=30 μm。A: Subcellular localization of Cr_KASH1 (red) and α-tubulin (green) after treatment with Nocodazole, as well as the subcellular localization of Cr_KASH1 (red) and α-tubulin(green) in the control group (with equal amounts of DMSO). B: Subcellular localization of Cr_KASH1 (red) and phalloidin (green) after treatment with Latrunculin B, as well as the subcellular localization of Cr_KASH1 (red) and phalloidin (green) in the control group (with equal amounts of DMSO). Scale bar =30 μm. ) 图 7 药物处理后KASH蛋白在NIH/3T3细胞中的亚细胞定位 Fig. 7 Localization of KASH1 protein and α-tubulin in the NIH/3T3 cell lines after drug treatment |
本文通过PCR扩增获得Cr_KASH1、Cr_KASH2、Cr_SUN1和Cr_SUN2的ORF序列,并对其DNA序列及蛋白序列进行了结构分析,Cr_KASH1、Cr_KASH2、Cr_SUN1均有保守的结构域,并在序列相似性和进化树分析中发现与其他物种中的含KASH或SUN结构域的蛋白有同源性,Cr_SUN2虽然没有保守的SUN2结构域,但是与含SUN结构域的蛋白同源。分析Cr_KASH1、Cr_KASH2、Cr_SUN1和Cr_SUN2的表达模式发现,Cr_KASH1、Cr_SUN1、Cr_SUN2均特异性的定位在了脊索组织中,而Cr_KASH2则在肌肉中表达较强。
在其他物种的研究中,KASH蛋白也呈现组织特异性表达的现象,并且这些特异性表达KASH蛋白的细胞或组织往往都具有特殊的细胞功能或细胞核定位模式。在哺乳动物中,共鉴定出5种KASH蛋白:Syne-1、Syne-2、Nesprin-3、Nesprin-4和KASH5蛋白。其中,Syne-1和Syne-2定位在核膜上,Syne-1在骨骼肌中发挥作用[34],Syne-2在胚胎期小鼠大脑皮层神经细胞发育过程中有重要作用[35],Nesprin-3广泛表达在多个组织中,尤其在骨骼肌和心肌细胞中有特异的高表达。Nesprin-4仅在耳蜗的高度分泌细胞和毛细胞处有表达[36-37]。在线虫中,目前有3种KASH蛋白被鉴定出来,UNC-83、ANC-1和ZYG-12蛋白。其中,UNC-83定位在核膜周围,仅局限于迁移的细胞中有表达[38]。ANC-1蛋白分子量较大,根据结构域分析显示,它的C端有1个KASH结构域可以与细胞骨架相结合,N端有1个SUN结构域可以连接到核膜上[11, 39-40]。ZYG-12主要在受精卵时期发挥作用,参与雌、雄原核的结合[41]。在果蝇中,目前已知的KASH蛋白有2种,分别是Klarsicht和MSP-300,果蝇的Klarsicht主要参与的是果蝇眼原基的发育,当其命运决定形态发生沟下的细胞分化为感光细胞后,细胞核从底层迁移到表层,这一步骤是眼原基发育所必需的[1, 42-43]。MSP300的研究发现,它定位在肌肉和内胚层,以及肌肉之间的连接处,参与果蝇肌肉纤维网络的组织[32]。在海鞘中,本文作者检索到了2种KASH蛋白,它们的定位也有所不同,Cr_KASH1在脊索组织表达量较高,而Cr_KASH2则在肌肉有特异表达,因此二者或许在海鞘胚胎发育过程中有不同的贡献,导致不同组织的细胞核行为特殊。
SUN蛋白是1个保守的定位在内核膜上的蛋白,能够与定位在外核膜上的KASH蛋白形成1个跨膜的分子桥梁,因此关于SUN蛋白,在先前的报道中它往往与某一种或几种KASH蛋白具有相似的表达模式和定位模式。在哺乳动物中,目前发现了4种SUN蛋白,其中SUN1和SUN2是最早被鉴定出的SUN蛋白,它们在所有的组织中均有表达,是KASH锚定在核膜上所必需的[44-48]。果蝇中检索到2个含有SUN结构域的蛋白,其中一种SUN蛋白是Klaroid,与果蝇的KASH蛋白Klarsicht的表达在同样的组织中,一起参与果蝇眼原基的发育,突变后都有相同的表型出现,也证实了二者有密不可分的关系[49]。在线虫中,目前被报道了2种SUN蛋白,也都与KASH蛋白有联系,SUN蛋白Matefin已经证实与KASH蛋白ZYG-12有相同的组织特异性,UNC-84与ANC-1和UNC-83联系而将其锚定在核膜上。这些模式物种的研究均证实了,通常SUN蛋白与KASH蛋白表达在同一类型组织中,并形成复合体来连接细胞骨架与核骨架。本研究发现,在海鞘中的2种SUN蛋白,它们特异性的定位在了脊索组织中,具有与KASH蛋白Cr_KASH1非常相似的表达模式和亚细胞定位,因此它们也可能联系在一起,共同发挥生物学功能。
本文作者在培养的NIH/3T3细胞系中转染海鞘Cr_KASH1、Cr_KASH2、Cr_SUN1和Cr_SUN2的表达载体,观察到这些蛋白均围绕细胞核周围定位,值得注意的是在胞质中,海鞘Cr_KASH1与α-tubulin、Cr_KASH2与肌动蛋白均有共定位。而在分别用药物破坏微管和微丝系统后,Cr_KASH1和Cr_KASH2在胞质中的定位也受到破坏,表明二者与微管系统均有相互联系。哺乳动物的Syne-1和Syne-2可以结合肌动蛋白, 它们的亚型也与微管系统的马达蛋白、驱动蛋白kinesin-1和动力蛋白dynein相互作用,但是否直接结合尚不清楚[4, 35, 50]。在小鼠和斑马鱼中敲除编码Syne-1或Syne-2的基因导致新皮层和视网膜中内核膜上存在缺陷,免疫共沉淀实验结果也证实了小鼠的Syne-1与驱动蛋白kinesin-1和动力蛋白dynein有相互作用[4, 51]。由此可知,Syne-1和Syne-2均具有结合2种细胞骨架的能力。本文作者通过在NIH/3T3细胞系中将Cr_KASH1与α-tubulin共转发现,二者存在共定位,即证实了海鞘的Cr_KASH1与微管系统存在相互作用,但结合模式尚未可知。同时Cr_KASH1的qPCR和原位杂交结果显示了,Cr_KASH1在海鞘胚胎的尾芽延伸阶段的脊索组织中有特异性的表达。而根据以往报道可知,海鞘胚胎的脊索在尾芽延伸时期会发生迁移、排列以及极性建立的一系列过程,而在延伸过程中,细胞核往往会迁移至细胞的后侧,并被固定[24, 52]。Cr_KASH1的亚细胞定位显示,它定位在核周,并且与微管有共定位,Cr_KASH1蛋白作为连接细胞骨架与细胞核骨架重要的一个连接蛋白,在这期间Cr_KASH1基因表达量增加,即表明在这个过程中KASH蛋白发挥了重要作用。Cr_SUN1在胚胎发育的整个时期均有表达,但其mRNA在脊索特异性高表达。Cr_SUN2的表达模式与亚细胞定位与之十分相似,并且二者在胚胎尾芽延伸时期的脊索组织中均存在高表达,它们也可能是脊索特殊细胞核定位模式的参与者。
| [1] |
Patterson K, Molofsky A B, Robinson C, et al. The functions of Klarsicht and nuclear lamin in developmentally regulated nuclear migrations of photoreceptor cells in the Drosophila eye[J]. Molecular Biology of the Cell, 2004, 15(2): 600-610. DOI:10.1091/mbc.e03-06-0374 (  0) 0) |
| [2] |
Tomlinson A, Ready D F. Sevenless: A cell-specific homeotic mutation of the drosophila eye[J]. Science, 1986, 231(4736): 400-402. DOI:10.1126/science.231.4736.400 (  0) 0) |
| [3] |
Gomes E R, Jani S, Gundersen G G. Nuclear movement regulated by Cdc42, MRCK, myosin, and actin flow establishes MTOC polarization in migrating cells[J]. Cell, 2005, 121(3): 451-463. DOI:10.1016/j.cell.2005.02.022 (  0) 0) |
| [4] |
Gundersen G G, Worman H J. Nuclear Positioning[J]. Cell, 2013, 152(6): 1376-1389. DOI:10.1016/j.cell.2013.02.031 (  0) 0) |
| [5] |
Muchir A, Wu W, Worman H J. Reduced expression of A-type lamins and emerin activates extracellular signal-regulated kinase in cultured cells[J]. Biochimica et Biophysica Acta, 2009, 1792(1): 75-81. DOI:10.1016/j.bbadis.2008.10.012 (  0) 0) |
| [6] |
Hale C M, Shrestha A L, Khatau S B, et al. Dysfunctional connections between the nucleus and the actin and microtubule networks in laminopathic models[J]. Biophysical Journal, 2008, 95(11): 5462-5475. DOI:10.1529/biophysj.108.139428 (  0) 0) |
| [7] |
Muchir A, Pavlidis P, Decostre V, et al. Activation of MAPK pathways links LMNA mutations to cardiomyopathy in emery-dreifuss muscular dystrophy[J]. Journal of Clinical Investigtion, 2007, 117(5): 1282-1293. DOI:10.1172/JCI29042 (  0) 0) |
| [8] |
Folker E S, Östlund C, Luxton G W G, et al. Lamin A variants that cause striated muscle disease are defective in anchoring transmembrane actin-associated nuclear lines for nuclear movement[J]. Proceedings of the National Academy of Sciences, 2011, 108(1): 131-136. DOI:10.1073/pnas.1000824108 (  0) 0) |
| [9] |
Goetz S C, Anderson K V. The primary cilium: A signalling centre during vertebrate development[J]. Nature Review Genetics, 2010, 11(5): 331-344. DOI:10.1038/nrg2774 (  0) 0) |
| [10] |
Del Bene F, Wehman A M, Link B A, et al. Regulation of neurogenesis by interkinetic nuclear migration through an apical-basal notch gradient[J]. Cell, 2008, 134(6): 1055-1065. DOI:10.1016/j.cell.2008.07.017 (  0) 0) |
| [11] |
Gurusaran M, Davies O R. A molecular mechanism for LINC complex branching by structurally diverse SUN-KASH 6: 6 assemblies[J]. Elife, 2021, 10: e60175. DOI:10.7554/eLife.60175 (  0) 0) |
| [12] |
Carley E, Stewart R M, Zieman A, et al. The LINC complex transmits integrin-dependent tension to the nuclear lamina and represses epidermal differentiation[J]. Elife, 2021, 10: e58541. DOI:10.7554/eLife.58541 (  0) 0) |
| [13] |
Stroud M J, Banerjee I, Lowe J, et al. LINC complex proteins in cardiac structure, function, and disease[J]. Circulation Research, 2014, 114(3): 538. DOI:10.1161/CIRCRESAHA.114.301236 (  0) 0) |
| [14] |
Horn H F. LINC complex proteins in development and disease[J]. Current Topics in Developmental Biology, 2014, 109(109): 287. (  0) 0) |
| [15] |
Razafsky D, Hodzic D. Bringing KASH under the SUN: The many faces of nucleo-cytoskeletal connections[J]. Journal of Cell Biology, 2009, 186(4): 461-472. DOI:10.1083/jcb.200906068 (  0) 0) |
| [16] |
Fridolfsson H N, Starr D A. Kinesin-1 and dynein at the nuclear envelope mediate the bidirectional migrations of nuclei[J]. Journal of Cell Biology, 2010, 191(1): 115-128. DOI:10.1083/jcb.201004118 (  0) 0) |
| [17] |
Bouzid T, Kim E, Riehl B D, et al. The LINC complex, mechanotransduction, and mesenchymal stem cell function and fate[J]. Journal of Biological Engineering, 2019, 13(1): 68. DOI:10.1186/s13036-019-0197-9 (  0) 0) |
| [18] |
Hao H, Starr D A. SUN/KASH interactions facilitate force transmission across the nuclear envelope[J]. Nucleus (Austin, Tex), 2019, 10(1): 73-80. (  0) 0) |
| [19] |
D'Angelo M A, Hetzer M W. The role of the nuclear envelope in cellular organization[J]. Cellular and Molecular Life Sciences, 2006, 63(3): 316-332. DOI:10.1007/s00018-005-5361-3 (  0) 0) |
| [20] |
Yin L M, Schnoor M, Jun C D. Structural characteristics, binding partners and related diseases of the calponin homology (CH) domain[J]. Frontiers in Cell and Developmental Biology, 2020, 8: 342. DOI:10.3389/fcell.2020.00342 (  0) 0) |
| [21] |
Dejardin T, Carollo P S, Sipieter F, et al. Nesprins are mechanotransducers that discriminate epithelial-mesenchymal transition programs[J]. Journal of Cell Biology, 2020, 219(10): e201908036. DOI:10.1083/jcb.201908036 (  0) 0) |
| [22] |
Starr D A, Han M. ANChors away: An actin based mechanism of nuclear positioning[J]. Journal of Cell Science, 2003, 116(2): 211-216. DOI:10.1242/jcs.00248 (  0) 0) |
| [23] |
Adames N. R. Microtubule interactions with the cell cortex causing nuclear movements in Saccharomyces cerevisiae[J]. Journal of Cell Biology, 2000, 149(4): 863-874. DOI:10.1083/jcb.149.4.863 (  0) 0) |
| [24] |
Dong B, Horie T, Denker E, et al. Tube formation by complex cellular processes in Ciona intestinalis notochord[J]. Developmental Biology, 2009, 330(2): 237-249. DOI:10.1016/j.ydbio.2009.03.015 (  0) 0) |
| [25] |
Jiang D, Smith W C. Ascidian notochord morphogenesis[J]. Developmental Dynamics, 2007, 236(7): 1748-1757. DOI:10.1002/dvdy.21184 (  0) 0) |
| [26] |
Jiang D, Munro E M, Smith W C. Ascidian prickle regulates both mediolateral and anterior-posterior cell polarity of notochord cells[J]. Current Biology, 2005, 15(1): 79-85. DOI:10.1016/j.cub.2004.12.041 (  0) 0) |
| [27] |
Zhou X, Graumann K, Evans D E, et al. Novel plant SUN-KASH bridges are involved in RanGAP anchoring and nuclear shape determination[J]. Journal of Cell Biology, 2012, 196(2): 203-211. DOI:10.1083/jcb.201108098 (  0) 0) |
| [28] |
Dardaillon J, Dauga D, Simion P, et al. ANISEED 2019: 4D exploration of genetic data for an extended range of tunicates[J]. Nucleic Acids Research, 2020, 48(1): 668-675. (  0) 0) |
| [29] |
Brozovic M, Dantec C, Dardaillon J, et al. ANISEED 2017: Extending the integrated ascidian database to the exploration and evolutionary comparison of genome-scale datasets[J]. Nucleic Acids Research, 2018, 46(1): 718-725. (  0) 0) |
| [30] |
Christiaen L, Wagner E, Shi W, et al. Whole-mount in situ hybridization on sea squirt (Ciona intestinalis) embryos[J]. Cold Spring Harbor Perspectives in Biology, 2009, 2009(12): PDB. PROT5348. (  0) 0) |
| [31] |
Bhattachan P, Rae J, Yu H, et al. Ascidian caveolin induces membrane curvature and protects tissue integrity and morphology during embryogenesis[J]. Faseb Journal, 2020, 34(1): 1345-1361. DOI:10.1096/fj.201901281R (  0) 0) |
| [32] |
Rosenberghasson Y, Renertpasca M, Volk T. A Drosophila dystrophin-related protein, MSP-300, is required for embryonic muscle morphogenesis[J]. Mechanisms of Development, 1996, 60(1): 83-94. DOI:10.1016/S0925-4773(96)00602-8 (  0) 0) |
| [33] |
Starr D A. A nuclear-envelope bridge positions nuclei and moves chromosomes[J]. Journal of Cell Science, 2009, 122(5): 577-586. DOI:10.1242/jcs.037622 (  0) 0) |
| [34] |
Grady R M, Starr D A, Ackerman G L, et al. Syne proteins anchor muscle nuclei at the neuromuscular junction[J]. Proceedings of the National Academy of Sciences of the United States of America, 2005, 102(12): 4359-4364. DOI:10.1073/pnas.0500711102 (  0) 0) |
| [35] |
Zhang X, Lei K, Yuan X, et al. SUN1/2 and Syne/Nesprin-1/2 complexes connect centrosome to the nucleus during neurogenesis and neuronal migration in mice[J]. Neuron, 2009, 64(2): 173-187. DOI:10.1016/j.neuron.2009.08.018 (  0) 0) |
| [36] |
Horn H F, Brownstein Z, Lenz D R, et al. The LINC complex is essential for hearing[J]. Journal of Clinical Investigtion, 2013, 123(2): 740-750. (  0) 0) |
| [37] |
Roux K J, Crisp M L, Liu Q, et al. Nesprin 4 is an outer nuclear membrane protein that can induce kinesin-mediated cell polarization[J]. Proceedings of the National Academy of Sciences, 2009, 106(7): 2194-2199. DOI:10.1073/pnas.0808602106 (  0) 0) |
| [38] |
Starr D A, Hermann G J, Malone C J, et al. unc-83 encodes a novel component of the nuclear envelope and is essential for proper nuclear migration[J]. Development, 2001, 128(24): 5039-5050. DOI:10.1242/dev.128.24.5039 (  0) 0) |
| [39] |
Starr D A, Han M. Role of ANC-1 in tethering nuclei to the actin cytoskeleton[J]. Science, 2002, 298(5592): 406-409. DOI:10.1126/science.1075119 (  0) 0) |
| [40] |
Horvitz H R, Sulston J E. Isolation and genetic characterization of cell-lineage mutants of the nematode Caenorhabditis elegans[J]. Genetics, 1980, 96(2): 435-454. DOI:10.1093/genetics/96.2.435 (  0) 0) |
| [41] |
Malone C J, Misner L, Bot N L, et al. The C. elegans hook protein, ZYG-12, mediates the essential attachment between the centrosome and nucleus[J]. Cell, 2004, 115(7): 825-836. (  0) 0) |
| [42] |
Mosley-Bishop K L, Qinghong L, Patterson K, et al. Molecular analysis of the klarsicht gene and its role in nuclear migration within differentiating cells of the Drosophila eye[J]. Current Biology, 1999, 9(21): 1211-1220. DOI:10.1016/S0960-9822(99)80501-6 (  0) 0) |
| [43] |
Fischer J A. Drosophila Klarsicht has distinct subcellular localization domains for nuclear envelope and microtubule localization in the eye[J]. Genetics, 2004, 168(3): 1385-1393. DOI:10.1534/genetics.104.028662 (  0) 0) |
| [44] |
Crisp M. Coupling of the nucleus and cytoplasm: Role of the LINC complex[J]. Journal of Cell Biology, 2006, 172(1): 41-53. DOI:10.1083/jcb.200509124 (  0) 0) |
| [45] |
Haque F, Lloyd D J, Smallwood D T, et al. SUN1 interacts with nuclear lamin A and cytoplasmic nesprins to provide a physical connection between the nuclear lamina and the cytoskeleton[J]. Molecular Biology of the Cell, 2006, 26(10): 3738-3751. DOI:10.1128/MCB.26.10.3738-3751.2006 (  0) 0) |
| [46] |
Padmakumar V C, Libotte T, Lu W, et al. The inner nuclear membrane protein Sun1 mediates the anchorage of Nesprin-2 to the nuclear envelope[J]. Journal of Cell Science, 2005, 118(15): 3419-3430. DOI:10.1242/jcs.02471 (  0) 0) |
| [47] |
Hodzic D M, Yeater D B, Bengtsson L, et al. Sun2 is a novel mammalian inner nuclear membrane protein[J]. Journal of Biological Chemistry, 2004, 279(24): 25805-25812. DOI:10.1074/jbc.M313157200 (  0) 0) |
| [48] |
Malone C J, Fixsen W D, Horvitz H R, et al. UNC-84 localizes to the nuclear envelope and is required for nuclear migration and anchoring during C. elegans development[J]. Development, 1999, 126(14): 3171-3181. DOI:10.1242/dev.126.14.3171 (  0) 0) |
| [49] |
Kracklauer M P, Banks S M, Xie X, et al. Drosophila klaroid encodes a SUN domain protein required for Klarsicht localization to the nuclear envelope and nuclear migration in the eye[J]. Fly (Austin), 2007, 1(2): 75-85. DOI:10.4161/fly.4254 (  0) 0) |
| [50] |
Yu J, Lei K, Zhou M, et al. KASH protein Syne-2/Nesprin-2 and SUN proteins SUN1/2 mediate nuclear migration during mammalian retinal development[J]. Human Molecular Genetics, 2011, 20(6): 1061-1073. DOI:10.1093/hmg/ddq549 (  0) 0) |
| [51] |
Tsujikawa M, Omori Y, Biyanwila J, et al. Mechanism of positioning the cell nucleus in vertebrate photoreceptors[J]. Proceedings of the National Academy of Sciences of the United States of America, 2007, 104(37): 14819-14824. DOI:10.1073/pnas.0700178104 (  0) 0) |
| [52] |
Peng H, Qiao R, Dong B. Polarity establishment and maintenance in ascidian notochord[J]. Frontiers in Cell and Developmental Biology, 2020, 8: 597446. DOI:10.3389/fcell.2020.597446 (  0) 0) |
2. Institute of Evolution & marine biodiversity, Ocean University of China, Qingdao 266003, China;
3. Laboratory for Marine Biology and Biotechnology, Qingdao National Laboratory for Marine Science and Technology, Qingdao 266100, China
 2023, Vol. 53
2023, Vol. 53


