2. 海洋生物学与生物技术功能实验室,青岛海洋科学与技术国家实验室,山东 青岛 266237;
3. 海洋渔业科学与食物产出过程功能实验室,青岛海洋科学与技术国家实验室,山东 青岛 266237
棘皮动物在生物进化史上占有独特的地位,它们是无脊椎动物中进化地位最高的门类,同时也是后口动物中最为古老的种类[1]。仿刺参作为棘皮动物的代表性物种,具有极其重要的研究价值。在水产养殖业中,其因营养价值和药用价值高,已经成为了中国、日本、韩国和俄罗斯等地最具价值的水产经济物种之一[2];在生物学方面,其夏眠和组织再生的生物学特性已经逐渐成为研究热点[3],仿刺参也逐渐成为无脊椎动物中重要的研究物种。鉴于仿刺参在经济上以及生物学研究上越来越重要的研究价值,在基因组和转录组水平上对其进行生物学深度分析,已经成为仿刺参研究的重要任务。近年来,由于RNA-seq技术的广泛应用,仿刺参在转录组水平上的研究也有了较大的进展,然而DNA甲基化作为重要的表观遗传调控机制,在仿刺参中的研究仍然比较匮乏,赵业等曾用同裂酶酶切检测过仿刺参非夏眠状态下组织的DNA甲基化状况[4],但是组织中各基因的DNA甲基化的详细信息并不完善。
DNA甲基化是DNA分子层面表观修饰的重要方式。在真核生物中,DNA甲基化参与了多种生物体内的生理生化过程,具有重要的生物学功能,包括维持胚胎的正常发育[5-6];使X染色体失活[7],失活的X染色体上大部分基因的CpG是甲基化的,而在活化的染色体上是非甲基化的[8];维持染色体稳定性:DNA甲基化被证明与转座子活性相关,在哺乳动物中转座子相关的DNA序列均呈现出高度甲基化的状态,同时保持转录沉默状态[9]。随着DNA甲基化成为表观遗传学的研究热点,其相关生物学特性也被广泛研究。随着测序技术的不断发展,全基因DNA甲基化的检测方法也不断推陈出新,MethylRAD-Seq技术结合了甲基化修饰依赖性内切酶和简化基因组限制性酶切分型技术的特点,其优点在于可以在不知道基因组背景信息的前提下大规模检测全基因组范围内的甲基化位点,而且该技术建库流程简单、技术重复性好、假阳性率低[6]。
相关研究表明,无脊椎动物基因组甲基化水平通常低于脊椎动物[10],而且甲基化标记更趋向于出现在基因功能区而不是基因间区[11],也暗示了无脊椎动物甲基化规律与脊椎动物有所差异。基于生物体各组织的甲基化状态有所差异,本论文选择了3种仿刺参研究常用且重要的组织(肠道、呼吸树和性腺常出现于仿刺参研究中,而且其萎缩退化和再生特性是仿刺参夏眠、再生等研究的理想材料)进行后续研究,通过建立仿刺参3种组织的甲基化文库,利用本实验室已经拼接完成的部分仿刺参基因组数据和表达谱数据(未发表,表达谱数据与本研究中的甲基化数据来源于同一批但非同一个体的组织材料)初步探究仿刺参组织甲基化状况及其特异性,为丰富仿刺参甲基化研究内容和无脊椎动物的甲基化发生规律提供可用信息。
1 材料与方法 1.1 实验材料本研究选用的仿刺参来源于辽宁省大连市獐子岛黑石礁海区(124°47′E, 39°3′N),组织为成体仿刺参正常状态下的肠道、呼吸树和性腺三种组织。材料取出后立即用PBS冲洗去除材料表面海水以及其他杂质,然后用RNAwait浸泡保存于1.5 mL EP管中,带回实验室后用吸管将RNAwait吸取干净,置于-80℃冰箱长期保存,用于后续DNA提取和甲基化文库的构建。
1.2 Methy RAD-Seq测序文库的构建首先,采用传统的酚氯仿抽提法提取仿刺参基因组DNA,其中裂解液更换为CTAB(Tris-HCl 1 mol/L,pH=8.0;EDTA 0.5 mol/L),DNA的质量用1%的琼脂糖凝胶电泳检测;其次,参照Wang等[12]MethylRAD文库构建方法,应用MspJ Ⅰ内切酶进行组织甲基化文库构建(见图 1)[6],构建过程如下:
(1) MspJ Ⅰ酶切基因组DNA:
MspJ Ⅰ内切酶:MspJ Ⅰ是一种识别甲基化位点的核酸内切酶,它有特定的识别位点,并在该识别位点两侧进行酶切,得到一个36 bp的酶切片段。MspJ Ⅰ识别位点见图 1。
首先将事先提取的高质量基因组DNA进行浓度稀释,将浓度控制在100 ng/μL,然后将DNA样品轻弹混匀,配置如下酶切体系:
|
|
酶切条件:37 ℃酶切4 h。
(2) 酶切标签连接接头:
以第一步酶切产物为底物,每个DNA样品配置如下反应体系:
|
|
连接条件:4 ℃连接16 h。
(3) 一轮PCR富集MSPJ Ⅰ酶切标签:
|
|
反应条件:98 ℃变性5 s,60 ℃退火20 s,72 ℃延伸10 s,进行14~18个循环,最后72 ℃延伸5 min。
(4) 二轮PCR引入Barcode序列:
|
|
反应条件:98 ℃变性5 s,60 ℃退火20 s,72 ℃延伸10 s,进行5~7个循环,最后72 ℃延伸5 min。
(5) PCR产物纯化回收
使用QIAGEN PCRPurification Kit回收纯化PCR产物。
最后用Qubit测定纯化后样品浓度,按照每个文库DNA总量相等的原则,将文库等量混样,使用Illumina Hiseq2000平台进行Single end 50 bp测序。
试剂:
MspJ Ⅰ(New EngLandBioLabs,产品型号R0661L)
T4 DNA Ligase(New EngLandBioLabs,产品型号M0202L)
Phusion(New EngLandBioLabs,产品型号M0535L)
QIAGEN PCR Purification Kit(Qiagen,产品型号28106.0)
Qubit 2.0 fluorometer(Invitrogen,产品型号Q32866)
1.3 测序数据处理及分析 1.3.1 测序数据处理由于仿刺参基因组未完全拼接完成,大部分scaffold长度较短,所以本研究从已拼接的基因组中筛选出最长的1 000条scaffold作为参照基因组进行后续分析。
数据质量控制:测序数据为单端50 bp的reads,为保证数据质量,首先对测序得到的fastq格式的文件处理,进行数据质量过滤。质量过滤的条件为:(1)首先截取reads的前30 bp作为有效序列;(2)过滤掉含有总长度10%数量的N(不确定的碱基)的测序reads;(3)过滤掉含有总长度20%数量的低质量(质量值低于10)碱基的reads;(4)过滤掉含有超过10个连续碱基的reads;(5)统计测序得到的总的reads量、每个文库的数据量,以及经过质量控制后的数据量,以检测文库的质量。
甲基化标签在仿刺参基因组上的分布:首先从仿刺参参照基因组中提取含有CG二核苷酸、长度为30 bp和含有CHG三核苷酸、长度为29 bp的序列,使用soap软件[14-15],将高质量标签与基因组提取的标签进行唯一比对,统计每个位点的甲基化标签深度,以reads深度代表位点的甲基化水平。分别统计各个样本在不同位点处的甲基化水平,若样本的某个标签在检测深度大于5,则认为此样本的该位点发生了甲基化。
计算甲基化频率可以反映基因组的总体甲基化水平,甲基化频率是指平均出现一个甲基化位点的碱基数目,即相应区域长度/该区域内甲基化标签数目,计算公式为相应区域总长度÷该区域内甲基化标签数量。
1.3.2 不同组织基因甲基化水平差异性分析参照RPKM计算方式,将基因甲基化水平计算公式定义为:total methylated tags/(mapped reads(millions)×gene lenth(kb))。计算并统计出各组织中基因的甲基化水平。
在各组织基因甲基化水平已知的基础上,利用R软件包中的“edgeR”函数分别进行两种组织之间基因甲基化水平的差异分析,然后将3组差异分析中的差异基因(p < 0.05为筛选阈值)汇总进行Gene Ontology(GO)功能富集分析,从而更好地了解各组织之间甲基化水平差异基因所涉及的相关生物学过程。
1.3.3 基因甲基化水平与表达水平之间的相关性分析基因甲基化位点发生位置不同,对基因表达调控的作用也有所区别[16]。在脊椎动物中,通常启动子区域发生甲基化伴随着基因转录沉默或转录抑制[16-17],与此相比,无脊椎动物中DNA甲基化规律与调控机制不同。为了更好的了解仿刺参组织中甲基化位点在启动子与基因功能区对基因表达调控之间的相关性,本研究通过整合已有的甲基化文库数据与仿刺参表达谱数据,对各部分基因进行了分析。首先,在获得参照基因组基因相关的甲基化标签位置信息的基础上,将基因分为以下3部分:只在启动子区域有甲基化标签分布的基因、只在基因功能区有甲基化标签分布的基因以及在启动子区域和基因功能区均有甲基化标签分布的基因;其次,分别绘制各组织所有基因以及各部分基因甲基化水平与表达量之间的相关性散点图,进而使用R软件包中的“cor.test”函数进行相关性检验(method=“pearson”)分析基因甲基化水平与表达水平之间的相关性。
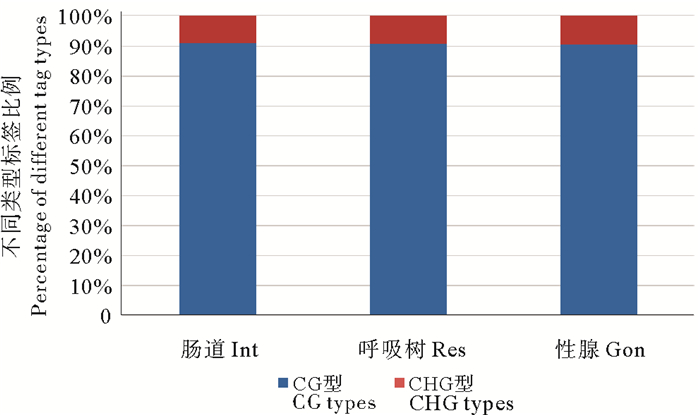
2 实验结果 2.1 仿刺参组织的MethylRAD文库数据统计及甲基化位点鉴定对已经拼接完成的仿刺参最长的前1 000条scaffold中的MspJ Ⅰ酶酶切位点通过计算机进行电子酶切分析,数据显示总共有4 234 227个酶切位点,CG和CHG型各占1 699 838和2 534 389个,当酶切位点中H=C时,甲基化标签为CCG型,为方便统计分析,本研究后续分析将此类标签归为CG型。测序结果显示,3个样本总共获得77 505 862条reads,其中高质量reads有77 432 392条,占总reads的99.9%以上,前1 000条scaffold上总唯一比对reads数为6 598 460条,具体信息见表 1。对3种组织样本的酶切标签进行甲基化位点进行鉴定分析,其中CG型甲基化位点比例明显高于CHG型甲基化位点,且不同组织中各甲基化位点类型所占比例表现出高度一致性。肠道、呼吸树和性腺3种组织检测到的甲基化位点数分别为114 417、128 251和136 423,其中检测深度大于5的位点数分别为45 202、52 288和56 786,各组织的甲基化位点类型见表 2。图 2所示的是3种组织中甲基化位点分型比例,从图中可以清晰的看出CG型甲基化位点占甲基化位点的主要部分,约91%(其中CCG类型约为8.6%),而CHG型只占9%左右,2种类型的标签比例与电子酶切结果相反。
|
|
表 1 MethylRAD文库测序原始数据 Table 1 The primary data of MethylRAD libraries |
|
|
表 2 甲基化位点数目及类型统计 Table 2 Number and type statistics of methylation sites |
|
图 2 CG和CHG型甲基化位点比例 Fig. 2 Percentage of methylation sites of CG and CHG |
利用仿刺参参照基因组,将MethylRAD文库测序中检测到的MspJ Ⅰ甲基化标签定位到基因区及非基因区。甲基化标签在基因的启动子区(promoter)(转录起始位点上游2k的范围),UTR区,CDS区,内含子区(intron)以及在非基因区的基因间区(intergenic)均有分布。甲基化标签分布结果(见表 3)显示:肠道、呼吸树和性腺3种组织的甲基化标签均有较大比例分布于基因间区,位点数目分别为20 176(44.64%)、23 409(44.77%)和26 076(45.92%);CDS区域略低于内含子区,位点数目为10 135(22.42%)、11 515(22.02%)和11 573(20.38%),内含子区域位点数目分别为12 494(27.64%)、14 646(28.01%)和16 059(28.28%);启动子区域位点数目分别为2 022(4.47%)、2 298(4.39)和2 572(4.53%);占比最小的区域为UTR区,为375(0.83%)、420(0.80%)和506(0.89%)。对2种不同类型的甲基化标签分别进行统计。从表 3中可以看出3种组织的2种甲基化位点分布状况具有很高的一致性,不同类型的甲基化位点在不同区域的分布比例略有差别。CG型甲基化位点在基因间区的分布比例明显高于其它区域,CDS区域所占比例低于内含子区域;而CHG型甲基化位点在基因间区的分布比例相对较低,在基因功能区与CG型位点分布不同,CHG型在CDS区的占比要高于内含子区域。
|
|
表 3 甲基化标签在不同区域的分布 Table 3 Distribution of methylation tags in different region |
为了深入了解仿刺参甲基化位点的分布特征,我们通过统计基因间区和基因功能区各区域的长度,分别计算得到各组织甲基化标签在不同区域的标签频率。统计结果(见表 4)显示3种组织在各区域的标签密度趋势一致性较高。CDS区域标签密度明显大于其它区域,分别为1 447碱基、1 273碱基和1267碱基;标签密度最小的区域为基因间区和5’UTR区域,分别是11 748碱基、10 144碱基、9 103碱基和7 722碱基、6 499碱基、5 416碱基,低于已报道的其他无脊椎动物[18]。
|
|
表 4 甲基化标签在不同区域的频率 Table 4 Frequency of methylation tags in different region |
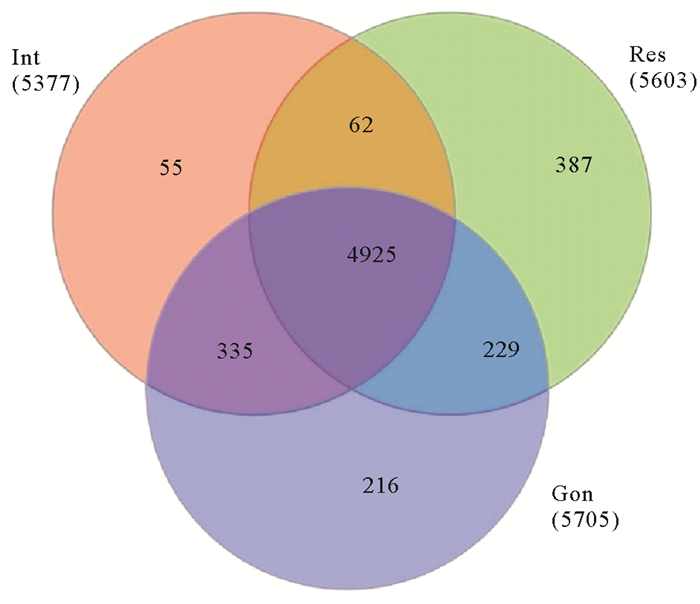
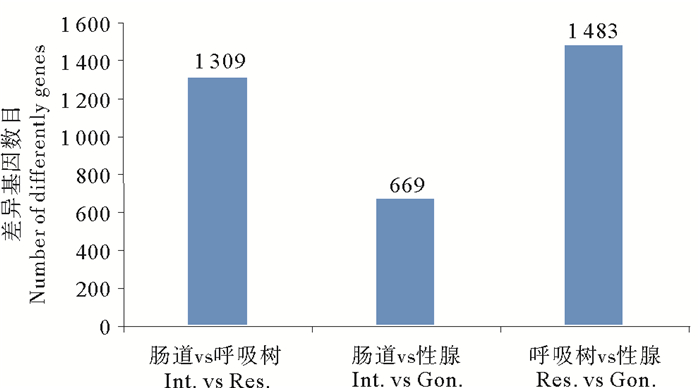
3种组织之间在甲基化位点及分布特征上存在较高的一致性,但是各组织也存在其本身的特异性。3种组织甲基化基因韦恩图(见图 3)显示:各组织均有其本身的特异甲基化基因,其中呼吸树组织特异甲基化基因最多,有387个,肠道组织特异甲基化最少,有55个,性腺组织有216个。利用R软件包中“edgeR”函数对3个组织的甲基化基因进行差异分析,筛选(p < 0.05为阈值)出各组织之间的差异基因(见图 4),结果显示呼吸树组织和性腺组织之间甲基化水平差异基因最多,为1 483个,肠道组织与性腺组织之间甲基化水平差异基因最少,有669个。对3种组织甲基化水平差异基因进行功能富集分析,分析结果(见表 5)显示组织间甲基化水平差异基因涉及到多种重要的生物学过程,包括生物体繁殖、胁迫应对机制、大分子合成及物质代谢、能量代谢等重要生命活动相关生物过程。
|
图 3 三种组织甲基化基因分析 Fig. 3 Analysis of methylated gene in three tissues |
|
图 4 三种组织甲基化基因差异分析结果 Fig. 4 Analysis of differentially methylated genes in three tissues |
|
|
表 5 三种组织甲基化水平差异基因GO富集分析 Table 5 GO enrichment of differently methylated genes in three tissues |
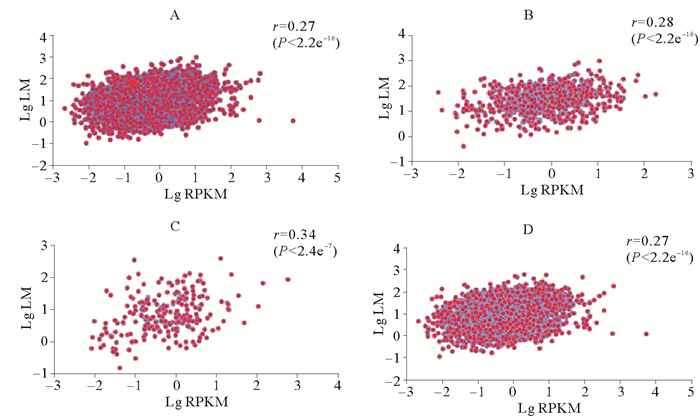
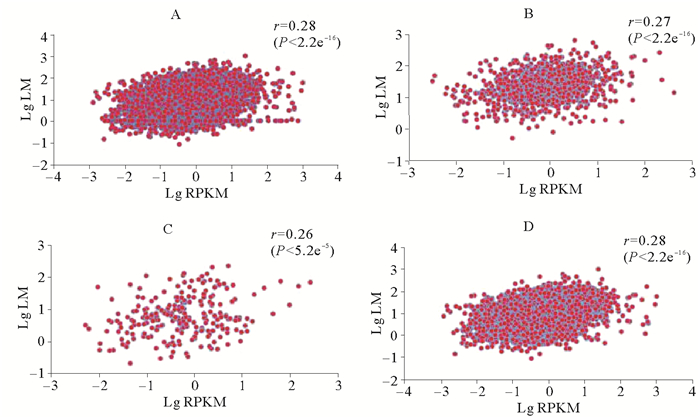
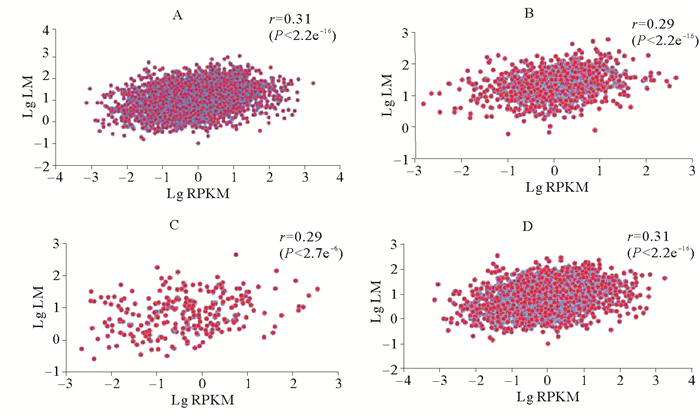
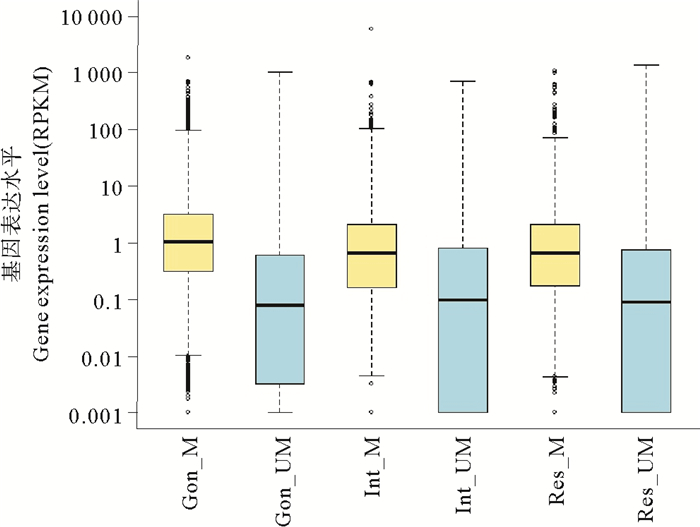
图 5~7所示是3种组织中有甲基化修饰基因的甲基化水平与表达水平的相关性。脊椎动物甲基化位点主要分布基因间区,启动子区域发生DNA甲基化对基因表达主要起抑制作用[18]。为了验证仿刺参组织中DNA甲基化是否符合这一规律,我们分别调查了仿刺参3个组织3类甲基化基因(如1.3所述分类)及全部甲基化基因的甲基化水平与基因表达水平的相关性。从图中可以看出,与脊椎动物不同,仿刺参3种组织所有甲基化基因的甲基化水平与表达水平均呈现出弱的正相关性,相关系数均为0.30左右。为了进一步验证这种正相关性,我们绘制了3种组织甲基化基因与非甲基化基因的基因表达水平分布图(见图 8),结果显示各组织甲基化基因(黄色箱体所示)表达水平明显高于非甲基化基因(蓝色箱体所示)。
|
A.所有甲基化基因甲基化水平与表达水平相关性;B.在启动子和基因功能区均有甲基化标签分布的基因甲基化水平与表达水平相关性;C.只在启动子区域有甲基化标签分布的基因甲基化水平与表达水平相关性;D.只在基因功能区有甲基化标签分布的基因甲基化水平与表达水平相关性;LM代表基因的甲基化水平;RPKM代表基因的表达水平;r代表相关系数。 A. The association between level of methylation and expression level about all the methylated genes; B. The association between level of methylation and expression level about the gene that methylation tags located in promoter and gene body region; C.The association between level of methylation and expression level about the gene that methylation tags only located in promoter region; D. The association between level of methylation and expression level about the gene that methylation tags only located in gene body respectively in the tissue of intestine; LM represent genes' level of methylation; RPKM represent the expression level of gene; r represent correlation coefficient. 图 5 肠道组织各部分基因甲基化水平与表达量相关性 Fig. 5 The association between genes' level of methylation and expression level in intestine |
|
A.所有甲基化基因甲基化水平与表达水平相关性;B.在启动子和基因功能区均有甲基化标签分布的基因甲基化水平与表达水平相关性;C.只在启动子区域有甲基化标签分布的基因甲基化水平与表达水平相关性;D.只在基因功能区有甲基化标签分布的基因甲基化水平与表达水平相关性;LM代表基因的甲基化水平;RPKM代表基因的表达水平;r代表相关系数。 A. The association between level of methylation and expression level about all the methylated genes; B. The association between level of methylation and expression level about the gene that methylation tags located in promoter and gene body region; C.The association between level of methylation and expression level about the gene that methylation tags only located in promoter region; D. The association between level of methylation and expression level about the gene that methylation tags only located in gene body respectively in the tissue of intestine; LM represent genes' level of methylation; RPKM represent the expression level of gene; r represent correlation coefficient. 图 6 呼吸树组织各部分基因甲基化水平与表达水平相关性 Fig. 6 The association between genes' level of methylation and expression level in respiratory tree |
|
A.所有甲基化基因甲基化水平与表达水平相关性;B.在启动子和基因功能区均有甲基化标签分布的基因甲基化水平与表达水平相关性;C.只在启动子区域有甲基化标签分布的基因甲基化水平与表达水平相关性;D.只在基因功能区有甲基化标签分布的基因甲基化水平与表达水平相关性;LM代表基因的甲基化水平;RPKM代表基因的表达水平;r代表相关系数。 A. The association between level of methylation and expression level about all the methylated genes; B. The association between level of methylation and expression level about the gene that methylation tags located in promoter and gene body region; C.The association between level of methylation and expression level about the gene that methylation tags only located in promoter region; D. The association between level of methylation and expression level about the gene that methylation tags only located in gene body respectively in the tissue of intestine; LM represent genes' level of methylation; RPKM represent the expression level of gene; r represent correlation coefficient. 图 7 性腺组织各部分基因甲基化水平与表达量相关性 Fig. 7 The association between genes' level of methylation and expression level in gonad |
|
M:甲基化基因,UM:非甲基化基因。 M and UM represent methylated and unmethylated genes in tissues. 图 8 甲基化基因和非甲基化基因的表达水平 Fig. 8 Expression level of methylated and unmethylated genes |
通过对仿刺参甲基化位点鉴定及分布特征的分析显示:仿刺参3种组织的甲基化位点主要分布于基因功能区,与之前文献报道的其他无脊椎动物如海胆(Strongylocentrotus purpuratus)[19]、玻璃海鞘(Cionaintestinalis)[20]、牡蛎(Crassostreagigas)[21]等基因组甲基化位点分布状况类似。甲基化位点类型分析显示CG型甲基化位点明显多于CHG型甲基化位点,而在电子酶切产生的数据中CHG型位点多于CG型,可能的原因在于实际情况中符合酶切位点的碱基组成可能未发生甲基化,同时表明CG型碱基组成更容易发生甲基化。对基因功能区甲基化位点特征分析显示,甲基化位点密度要远小于已报道的无脊椎动物[18],主要的原因可能是:(1)为防止假阳性位点而提高筛查标准,导致遗漏部分真实甲基化位点;(2)参照基因组并未拼接完全,影响标签比对结果。
仿刺参组织DNA甲基化总体分布特征(标签在功能区的分布比例)差别较小,而甲基化水平与基因表达水平呈弱的正相关性。对各组织之间DNA甲基化图谱的比较分析发现仿刺参各组织之间甲基化差异程度较小,甲基化位点分布以及类型一致性较高。不同组织中甲基化位点的总体分布规律及基因甲基化水平与表达水平相关性呈现出较高的一致性,这可能跟特定物种的甲基化分布规律有关。同时,各组织的甲基化也存在各自的特异性。首先各组织均有其特有的一部分甲基化基因,其次各组织之间相同基因的甲基化水平有所差异,仿刺参3种组织甲基化差异的基因涉及多种生物学过程,主要包括繁殖、压力应激、大分子合成等,对应于不同组织的不同功能。
对3种组织甲基化基因的甲基化水平与表达水平的相关性分析发现,甲基化标签的分布区域基本不影响基因甲基化水平与基因表达量之间的相关性,而且各部分基因甲基化水平与表达量均呈现出弱的正相关趋势;而图 8则显示各组织中发生甲基化的基因表达水平普遍高于未发生甲基化的基因,且3种组织结果一致。以上结果暗示了仿刺参基因组中DNA甲基化可能起到促进基因表达的作用,之前牡蛎的研究结果也呈现出了类似的规律[22]。这种促进表达的可能的机制一是不同于脊椎动物甲基化主要分布在promoter区,而已研究的无脊椎动物(如仿刺参、牡蛎等)的甲基化主要发生在基因区,暗示了它们甲基化的功能可能存在一定的差异,二是甲基化会抑制某些转座子的转录从而促进其宿主基因的表达[23],另外,某些组蛋白修饰(如H3K36me3)能够召集甲基化酶[24],从而促进基因的表达。但是考虑到DNA甲基化与基因表达相互关系的复杂性及相关证据目前还较少,因此对于无脊椎动物中DNA甲基化与基因表达相互关系的规律探究还需要更深入的研究和更多的证据。
本文通过分析仿刺参3种组织的DNA甲基化文库数据,得到了组织的甲基化位点类型、分布以及概况等详细信息,分析了其甲基化特性,并通过整合甲基化文库数据与已有的表达谱数据发现仿刺参基因组DNA甲基化与基因表达之间呈弱的正相关性,为无脊椎动物基因组甲基化研究提供可用信息。
| [1] |
廖玉麟. 中国动物志, 棘皮动物门, 海参纲[M]. 北京: 科学出版社, 1997, 334-335. Liao Y. FAUNA SINICA, Echinodermata, Holothuroidea[M]. BeiJing: Science Press, 1997, 334-335. (  0) 0) |
| [2] |
袁秀堂, 杨红生, 陈慕雁, 等. 刺参夏眠的研究进展[J]. 海洋科学, 2007, 31(8): 88-90. Yuan X, Yang H, Chen M, et al. Research advances in aestivation of sea cucumber Apostichopus japonicus(Selenka)[J]. Marine Sciences, 2007, 31(8): 88-90. (  0) 0) |
| [3] |
Carnevali M D C, Burighel P. Regeneration in Echinoderms and Ascidians[M]. eLS: John Wiley and Sons, Ltd, 2010.
(  0) 0) |
| [4] |
Zhao Y, Chen M, Storey K B, et al. DNA methylation levels analysis in four tissues of sea cucumber Apostichopus japonicus based on fluorescence-labeled methylation-sensitive amplified polymorphism (F-MSAP) during aestivation[J]. Comparative Biochemistry and Physiology Part B Biochemistry and Molecular Biology, 2014, 181: 26-32.
(  0) 0) |
| [5] |
Feng S, Jacobsen S E, Reik W. Epigenetic reprogramming in plant and animal development[J]. Science, 2010, 330(6004): 622-627. DOI:10.1126/science.1190614
(  0) 0) |
| [6] |
吕佳. 高效低成本全基因组DNA甲基化检测技术(MethylRAD-Seq)的建立及其在海洋贝类中的应用[D]. 青岛: 中国海洋大学, 2013. Lv J. Establishment of an Efficient and Cost-effective Method for Genome-wide DNA Methylation Profiling (MethylRAD-Seq) and Its Application in Marine Bivalves[D]. QingDao: Ocean University of China, 2013. (  0) 0) |
| [7] |
Mohandas T, Sparkes R S, Shapiro L J. Reactivation of an inactive human X chromosome: Evidence for X inactivation by DNA methylation[J]. Science, 1981, 211(4480): 393-396. DOI:10.1126/science.6164095
(  0) 0) |
| [8] |
Lee JT. Homozygous Tsix mutant mice reveal a sex-ratio distortion and revert to random X-inactivation[J]. Nature Genetics, 2002, 32(1): 195-200. DOI:10.1038/ng939
(  0) 0) |
| [9] |
Martienssen R A, Colot V. DNA methylation and epigenetic inheritance in plants and filamentous fungi[J]. Science, 2001, 293(5532): 1070-1074. DOI:10.1126/science.293.5532.1070
(  0) 0) |
| [10] |
Tweedie S, Charlton J, Clark V, et al. Methylation of genomes and genes at the invertebrate-vertebrate boundary[J]. Molecular & Cellular Biology, 1997, 17(3): 1469-1475.
(  0) 0) |
| [11] |
Head J A. Patterns of DNA methylation in animals: An ecotoxicological perspective[J]. Integrative and Comparative Biology, 2014, 54(1): 77-86. DOI:10.1093/icb/icu025
(  0) 0) |
| [12] |
Wang S, Lv J, Zhang L, et al. Methyl RAD: A simple and scalable method for genome-wide DNA methylation profiling using methylation-dependent restriction enzymes[J]. Open Biology, 2015, 5(11): 150130. DOI:10.1098/rsob.150130
(  0) 0) |
| [13] |
Cohen-Karni D, Xu D, Apone L, et al. The MspJI family of modification-dependent restriction endonucleases for epigenetic studies[J]. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(27): 11040-11045. DOI:10.1073/pnas.1018448108
(  0) 0) |
| [14] |
Du H, Bao Z, Hou R, et al. Transcriptome sequencing and characterization for the sea cucumber Apostichopus japonicus (Selenka, 1867)[J]. Plos One, 2012, 7(3): e33311. DOI:10.1371/journal.pone.0033311
(  0) 0) |
| [15] |
Sun L, Chen M, Yang H, et al. Large scale gene expression profiling during intestine and body wall regeneration in the sea cucumber Apostichopus japonicus[J]. Comparative Biochemistry and Physiology D-genomics and Proteomics, 2011, 6(2): 195-205. DOI:10.1016/j.cbd.2011.03.002
(  0) 0) |
| [16] |
王瑞娴, 徐建红. 基因组DNA甲基化及组蛋白甲基化[J]. 遗传, 2014, 36(3): 191-199. Wang R, Xu J. Genomic DNA methylation and histone methylation[J]. Hereditas, 2014, 36(3): 191-199. (  0) 0) |
| [17] |
Schübeler D. Function and information content of DNA methylation[J]. Nature, 2015, 517(7534): 321-326. DOI:10.1038/nature14192
(  0) 0) |
| [18] |
Sarda S, Zeng J, Hunt B G, et al. The Evolution of invertebrate gene body methylation[J]. Molecular Biology and Evolution, 2012, 29(8): 1907-1916. DOI:10.1093/molbev/mss062
(  0) 0) |
| [19] |
Bird A P, Taggart M H, Smith B A. Methylated and unmethylated DNA compartments in the sea urchin genome[J]. Cell, 1979, 17(4): 889-901. DOI:10.1016/0092-8674(79)90329-5
(  0) 0) |
| [20] |
Suzuki M M, Kerr A R, De S D, et al. CpG methylation is targeted to transcription units in an invertebrate genome[J]. Genome Research, 2007, 17(5): 625-631. DOI:10.1101/gr.6163007
(  0) 0) |
| [21] |
Gavery M R, Roberts S B. DNA methylation patterns provide insight into epigenetic regulation in the Pacific oyster (Crassostrea gigas)[J]. BMC Genomics, 2010, 11(1): 483. DOI:10.1186/1471-2164-11-483
(  0) 0) |
| [22] |
Olson C E, Roberts S B. Genome-wide profiling of DNA methylation and gene expression in Crassostrea gigas male gametes[J]. Frontiers in Physiology, 2014, 5: 224.
(  0) 0) |
| [23] |
Yoder J A, Walsh C P, Bestor T H. Cytosine methylation and the ecology of intragenomic parasites[J]. Trends Genet, 1997, 13: 335-340. DOI:10.1016/S0168-9525(97)01181-5
(  0) 0) |
| [24] |
Hahn M A, Wu X, Li A X, et al. Relationship between gene body DNA methylation and intragenic H3K9me3 and H3K36me3 chromatin marks[J]. PlosOne, 2011, 6: e18844. DOI:10.1371/journal.pone.0018844
(  0) 0) |
2. Laboratory for Marine Biology and Biotechnology, Qingdao National Laboratory for Marine Science and Technology, Qingdao 266237, China;
3. Laboratory for Marine Fisheries Science and Food Production Processes, Qingdao National Laboratory for Marine Science and Technology, Qingdao 266237, China
 2018, Vol. 48
2018, Vol. 48



