2. 新疆维吾尔自治区水产科学研究所,新疆 乌鲁木齐 830000;
3. 重庆市水产技术推广总站,重庆 400020;
4. 乌鲁木齐市百汇鱼生渔业科技有限公司,新疆 乌鲁木齐 830026;
5. 浙江海洋大学水产学院,浙江 舟山 316022
鱼菜共生是将无土栽培技术和水产养殖技术有机结合起来,同时生产动物和植物产品的一种生态系统[1]。在鱼菜共生系统中,水生动物排泄废物,水中的微生物将这些废物转化为营养物质,植物可以利用这些废物和营养物质,并将其清除从而改善水质,从而形成互惠互利的良性循环[2]。池塘生态系统中除动植物外,还存在大量的细菌和真菌等微生物。真菌种类繁多,在自然界分布广泛,其中淡水真菌的种类超过600种,淡水真菌由其陆地祖先进化而来,逐渐演化出一套适应水生环境扩散繁殖的机制。真菌是鱼菜共生系统中一类非常重要的微生物,它们一方面参与有机质分解,为水生植物和鱼类提供营养物质[3],另一方面许多病原真菌还会影响动植物生长和繁殖[4-5]。因此,水生真菌在维持鱼菜共生系统平衡、促进物质循环和能量流动过程中发挥着十分重要的作用。
然而真菌数量庞大、形态多样且群落结构复杂,其生长及生理生化特征也会因环境不同而发生变化[6-7]。仅依靠形态学、生理学和生态学特征为依据进行真菌的分类,难以满足准确鉴定的要求。近年来,随着分子生物技术的发展,以核酸和蛋白质等分子生物学性状来判别和区分不同分类阶元和进化类群真菌的研究,逐渐弥补了传统分类的不足。特别是以Illumina Hiseq高通量测序平台为代表的新一代测序技术的产生,其通量大、时间短、精确度高和信息量丰富等优点,成为了鉴别研究真菌群落结构组成的新方向和热点领域[8-9]。核糖体基因内转录间隔区(Internal transcribed spacer,ITS)所包含的2个区间序列ITS1和ITS2是目前广泛用于真菌分类研究的分子标记,二者分别位于真核生物核糖体rDNA序列的18S rDNA和5.8S rDNA以及5.8S rDNA和28S rDNA之间。它们作为非编码区序列具有较高的保守性,承受的选择压力较小、相对变化较大,且易于被通用引物扩增,能够为分类鉴定和系统学分析提供详尽的遗传学信息[10]。
本研究基于Illumina Hiseq 2500高通量测序平台,利用双末端测序的方法,构建小片段文库对新疆水生野生动物救护中心和乌鲁木齐市米东区长山子镇水源地养殖基地鱼菜共生池塘水样及水培蔬菜根系ITS序列进行双末端测序,通过对真菌群落结构和多样性的对比研究,探究养殖池塘水域真菌微生物的时空组成变化及对环境因子的响应机制,以期为调控改善水产养殖水质,构建健康绿色养殖系统奠定理论基础。
1 材料与方法 1.1 试验地点概况及样本采集试验样品于2017年9月采自位于新疆昌吉回族自治州五家渠市滨湖乡的新疆水生野生动物救护中心(87°34′E,44°15′N)和新疆乌鲁木齐市米东区长山子镇水源地养殖基地(87°59′E,44°08′N),并在两处各选取1口普通池塘水样作空白对照,样品信息及缩写见表 1。试验池塘和对照池塘面积均为4 333 m2、平均水深1.80 m,安装型号和功率相同的投饵机和增氧机,设有独立的进排水系统,且水质符合GB11607渔业水质标准。当年5月中旬投放规格80~100克/尾的大规格罗非鱼鱼种,放养量为1.5尾/m2。鱼菜共生池塘水面架设直径为75 mm的PVC排水管制作的浮床(360 cm×120 cm),浮床由上下两层聚乙烯网片包裹,其中上层为疏网,网眼直径3 cm;下层为密网,网眼直径0.8 cm,网片垂直间距约3 cm。水培空心菜为当年菜地育成,剪取长度15 cm茎秆于6月中旬移植至浮床,扦插株距10 cm,根部露出下层网片约4 cm。每处浮床面积均为415 m2,约占池塘养殖面积的10.4%。
|
|
表 1 样品来源信息及缩写 Table 1 The origin and abbreviation of the investigated samples |
在4口池塘中随机选取8个取样点,距离水面约15 cm深处采集10 mL等体积水样,混匀后装入灭菌离心管。空心菜根系悬浮于PBS缓冲液(0.15 mol/L NaCl,0.1 mol/L Na2EDTA,pH=8.0)中,充分摇匀后使用孔径0.22 μm(Millipore)无菌滤膜过滤,将滤膜剪碎置于灭菌冷冻管中。上述所有样品均置于-80 ℃保存。
1.2 基因组DNA提取、PCR扩增和产物回收使用CTAB法[11]分别提取以下6组样品基因组DNA:新疆水生野生动物救护中心鱼菜共生池塘水样(JH.KX)、乌鲁木齐市米东区长山子镇水源地养殖基地鱼菜共生池塘水样(MD.KX)、新疆水生野生动物救护中心对照池塘水样(JH.C)、乌鲁木齐市米东区长山子镇水源地养殖基地对照池塘水样(MD.C)、新疆水生野生动物救护中心鱼菜共生池塘空心菜根系水样(JH.R)、乌鲁木齐市米东区长山子镇水源地养殖基地鱼菜共生池塘空心菜根系水样(MD.R),每组样品设置3个生物学重复。经1%琼脂糖凝胶电泳检测DNA提取质量,并使用NanoDrop® ND-1000紫外分光光度计检测DNA浓度后,取适量的DNA样品于离心管中,使用无菌水稀释至1 ng/μL备用。根据测序区域的选择,使用带Barcode的特异引物,选择美国New England Biolabs公司生产的Phusion High-Fidelity PCR Master Mix with GC Buffer和高效高保真酶进行ITS1的宏基因组PCR扩增。上下游通用引物序列分别为:ITS5-1737F(5‘-GGAAGTAAAAGTCGTAACAAGG-3’)和ITS2-2043R(5‘-GCTGCGTTCTTCATCGATGC-3’)。50 μL PCR反应体系包括5×Phusion Master Mix 10 μL,10 μmol/L的上下游引物各2.5 μL,10 mmol/L的dNTPs 1 μL,Phusion DNA聚合酶0.5 μL,基因组DNA 10 μL,加无核酸酶纯水至终体积50 μL。PCR反应程序为:98 ℃预变性1 min;98 ℃变性10 s,50 ℃退火30 s,72 ℃延伸45 s,共30个循环;最后72 ℃延伸8 min,4 ℃保存。PCR产物经2%浓度的琼脂糖凝胶电泳检测,使用胶回收试剂盒(QIAquick PCR Purification Kit 250T)对PCR目的条带进行纯化回收。
1.3 文库构建和高通量测序使用TruSeq® DNA PCR-Free Sample Preparation Kit建库试剂盒进行文库构建,构建好的文库分别采用Qubit和Q-PCR定量,合格后送至陕西博瑞德生物科技有限公司,根据所扩增的ITS1区域特点进行高通量测序。
1.4 生物信息学分析截去Barcode和引物序列后,使用FLASH软件[12]对测序得到的原始Tags数据(Raw tags)进行拼接[13],使用Qiime 1.7.0软件[14]对Raw tags进行截断,并将得到的数据集进一步过滤。通过UCHIME软件[15]与Unite数据库(https://unite.ut.ee/)进行比对检测,最终去除其中的嵌合体及其短序列[16],得到有效Tags(Effective tags)数据。
使用Uparse 7.0.1软件[17]在97%的一致性水平下,将Effective tags进行聚类成为分类操作单元(Operational taxonomic units, OTUs),并尽可能多的对其代表序列进行物种注释。使用Qiime软件中的Blast方法[18]与Unite数据库进行物种注释分析[19],并分别统计各样本在不同分类水平的群落组成。使用MUSCLE 3.8.31软件[20]进行快速多序列比对,基于全部OTUs代表序列构建系统发生关系。
使用Qiime软件计算Alpha多样性指数(Observed species、Shannon指数、Simpson指数、Chao1指数、ACE指数、Goods coverage等),使用R 2.15.3软件绘制稀释曲线并进行多样性指数组间差异分析。运用Qiime软件计算Unifrac距离、构建非加权配对算术平均(Unweighted pair group method using arithmetic average, UPGMA)聚类树。分别使用R软件中的WGCNA、Stats和ggplot2软件包绘制主坐标分析图(Principal coordinates analysis, PCoA)。
2 结果与分析 2.1 真菌群落OTU注释及结构组成本研究基于Illumina HiSeq 2500高通量测序平台,对鱼菜共生池塘和普通池塘水体和水培蔬菜根系真菌进行测定和分析,共鉴定出真菌6门、25纲、72目、145科、139属和167种。获得长度在17~430 bp之间的Raw reads 544 095条,过滤得到平均长度为256 bp的有效序列78 348条,6份样品的有效序列数量分别为73 462条(JH.R)、78 901条(JH.KX)、64 664条(JH.C)、89 221条(MD.KX)、94 176条(MD.R)和69 664条(MD.C)。用于构建OTU分类信息单元且获得注释信息的平均Tags数为75 840条,有效数据量达到96.80%。在相似度大于97%的水平上检测到OTU平均数为541(见表 2)。上述数据可以看出,该方法适用于两种不同养殖模式池塘水样及水培植物根系真菌种群组成及变异分析。
|
|
表 2 样品的OTUs聚类注释和Alpha多样度指数统计表 Table 2 OTUs clustering, annotation and Alpha diversity indices of different samples |
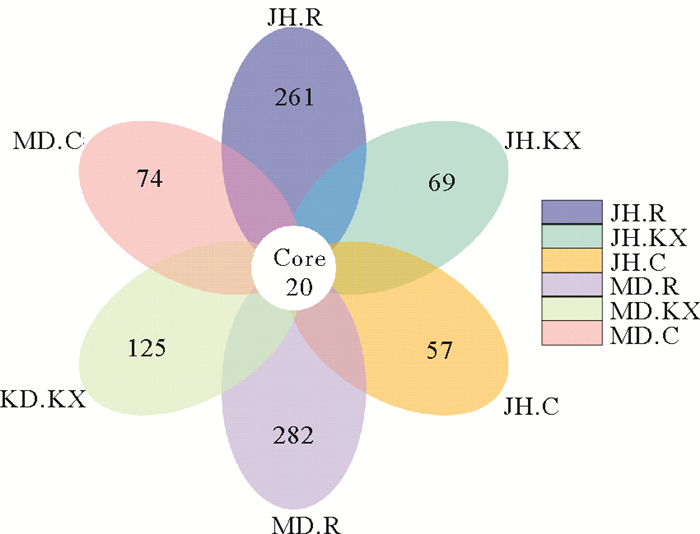
基于OTU的花瓣图可用于表示多个样品的OTU共有和独有物种情况,在对所有样品进行均一化处理之后绘制的花瓣图可以看出(见图 1),MD.R样品中特有OUT数量最高(282),其次为JH.R(261),JH.C特有OUT数量最低,仅有57个。此外,从两两样品真菌群落组成的相似程度来看,6个样本共有的OUT数量不高,仅为20个。
|
图 1 样品OTU分布的花瓣图 Fig. 1 Flower plot of OTU distribution in different samples |
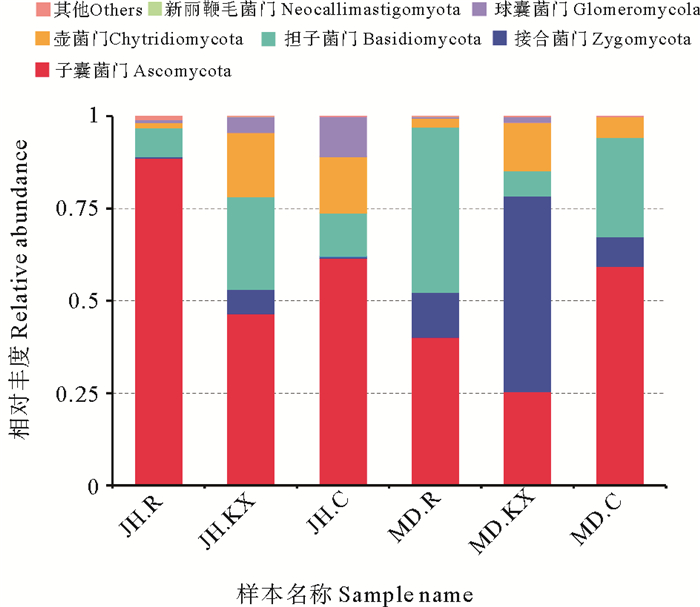
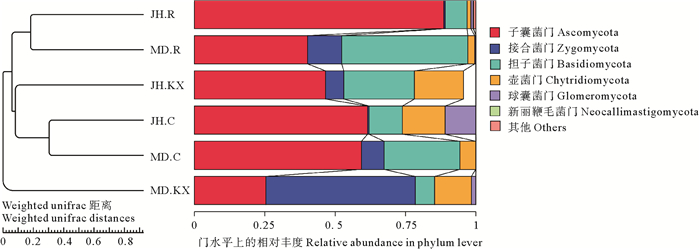
根据物种注释结果,选取每个样品在门的水平上最大丰度排名前6的物种,生成物种相对丰度柱形累加图(见图 2),分别为新丽鞭毛菌门(Neocallimastigomycota)、球囊菌门(Glomeromycota)、壶菌门(Chytridiomycota)、担子菌门(Basidiomycota)、接合菌门(Zygomycota)和子囊菌门(Ascomycota)。从真菌分布相对丰度变化的整体趋势来看,新疆水生野生动物救护中心3个样品(JH.R、JH.KX和JH.C)中检测到的优势菌均为子囊菌门(Ascomycota);而乌鲁木齐市米东区长山子镇水源地养殖基地仅对照池塘水样(MD.C)优势菌均为子囊菌门(Ascomycota),其他2个样品(MD.R和MD.KX)占优势地位的真菌群落分别为担子菌门(Basidiomycota)和接合菌门(Zygomycota)。此外,两处试验地点鱼菜共生池塘水样(JH.KX和MD.KX)中子囊菌门(Ascomycota)相对丰度均低于对应的对照组水样(JH.C和MD.C),而接合菌门(Zygomycota)和壶菌门(Chytridiomycota)相对丰度则高于对照组。从空心菜根系真菌微生物群落组成来看,新疆水生野生动物救护中心样品(JH.R)相对含量最多的子囊菌门(Ascomycota)丰度高达88.58%,明显高于其他样品;而乌鲁木齐市米东区长山子镇水源地养殖基地样品(MD.R)中,子囊菌门(Ascomycota)和担子菌门(Basidiomycota)相对丰度大致相当,分别为40.16%和44.83%。
|
( Others代表图中所示之外的其他所有门的相对丰度之和。Others represent the sum of relative abundances of all other phylums except those in the figure. ) 图 2 门水平上的物种相对丰度柱形图 Fig. 2 Relative abundance histogram of species on phylum level |
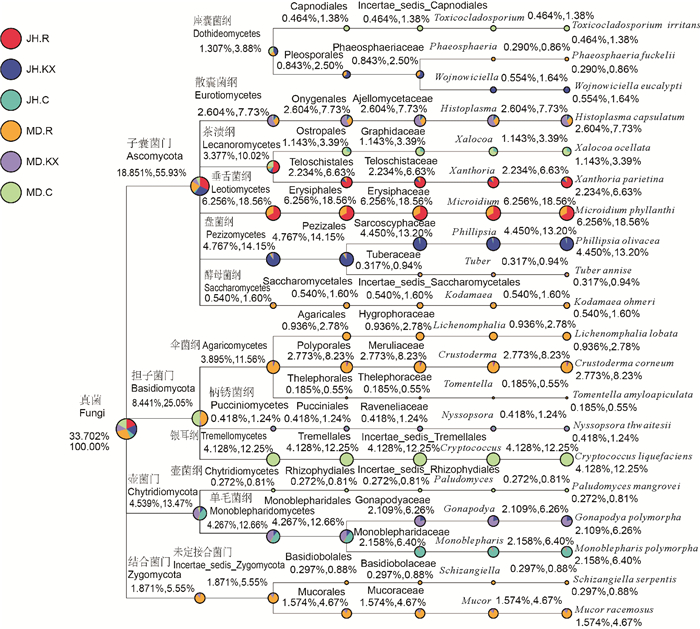
对每个样品或每个分组的物种分类结果,筛选特别关注的物种(默认选择最大相对丰度前20的种)进行物种分类统计[21]。分组的物种分类树如图 3所示,图 3中扇形的大小表示分组在该分类上相对丰度的比例大小,下方数字为相对丰度百分率,前者表示占所有分类物种的百分率,后者表示占所选取物种的百分率。从图 3中可以看出,样品中相对丰度较高的4个门分别为子囊菌门(Ascomycota)、担子菌门(Basidiomycota)、壶菌门(Chytridiomycota)和结合菌门(Zygomycota)。其中MD.R所涉及的真菌门类较多,特别是在担子菌门(Basidiomycota)和接合菌门(Zygomycota)中相对丰度比较较高。隶属于子囊菌门(Ascomycota)、锤舌菌纲(Leotiomycetes)、白粉菌目(Erysiphales)的白粉菌科(Erysiphaceae)菌群是根系样品(MD.R和JH.R)特有的。从目的角度来看,仅银耳目(Tremellales)和煤炱目(Capnodiales)为MD.C的独有菌群。
|
图 3 各分组中特定物种分类树 Fig. 3 Specific taxonomy tree in each group |
群落物种多样性的Alpha多样性又被称为生境内的多样性,主要关注局域均匀生境下的物种数目,可用于分析样品内的微生物群落多样性,以反映样品内微生物群落的丰富度和多样性。其中Chao1指数和ACE指数主要用于描述群落丰富度,而Shannon指数和Simpson指数则多用于评估群落多样性[22]。使用Qiime软件对各样本进行均一化处理(临界值Cutoff=61 392),并对97%一致性阈值时的Alpha多样度指数进行统计(见表 2)。Goods coverage代表样本文库的覆盖率,可表明高通量测序结果能否反映样本中微生物的真实情况,其值越大表明样本中序列被测出的概率越高[23]。从表 2中可得知,6个样品的测序覆盖度均在0.998以上,表明样本中真菌覆盖率高、已基本覆盖到所有物种,且测序深度满足样品中真菌多样性分析的需要,可用于下一步数据分析。乌鲁木齐市米东区长山子镇水源地养殖基地鱼菜共生池塘空心菜根系(MD.R)真菌的香农多样性指数、辛普森多样性指数、Chao1多样性指数和ACE多样性指数均最高,而来自同一采样点对照池塘(MD.C)中真菌多样性组成均最低。此外,Observed species代表样品中OTUs的直观数量统计情况,MD.R数值最高,也表明该样品物种丰富度最高。
2.4 样品间差异比较分析Beta多样性也被称为生境间的多样性,代表了根据环境梯度不同,生境群落之间物种组成的相异性或物种依据环境梯度的更替速率。作为生物多样性的重要组成部分,Beta多样性是对不同样品的微生物群落构成差异的比较分析,主要关注时空环境格局相关的物种组成变化[24-25]。Unifrac距离是利用微生物序列间的进化信息计算而来,能够用于衡量两个样品间的相异度,值越小表示这样品在物种多样性方面存在的差异越小[26]。根据OTUs信息计算得出的Weighted unifrac距离,不同样品真菌间变化范围为1.493~1.970,其中相异系数最大值出现在MD.KX和JH.R之间,表明二者真菌组成差异最大。
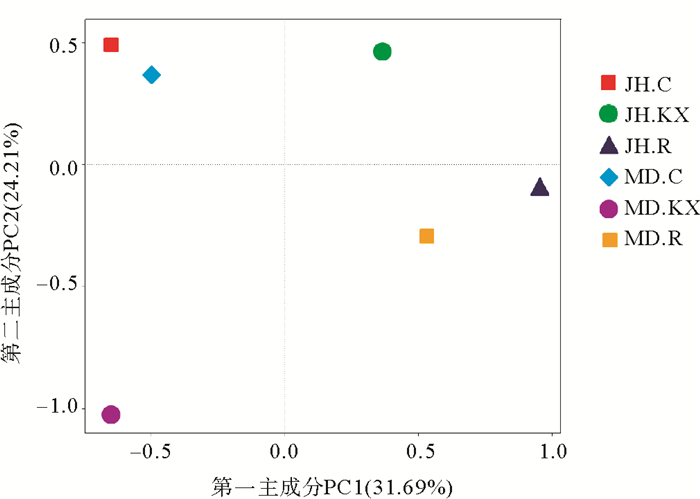
基于Weighted unifrac距离进行多变量统计学PCoA分析,选取贡献率最大的主坐标组合进行作图(见图 4),群落结构相似度最高的样品(JH.C和MD.C)聚集在一起,表明二者真菌群落结构高度相似,而MD.KX在散点图上与其他样品均远远分开,其菌落组成与其他样品存在较大差异。
|
图 4 基于Weighted unifrac距离的PCoA分析 Fig. 4 Principal coordinates analysis(PCoA)based on weighted unifrac distances |
以Weighted Unifrac距离矩阵做UPGMA聚类分析,并将聚类结果与各样品在门水平上的物种相对丰度进行整合,构建聚类关系树如图 5所示,空心菜根系(JH.R和MD.R)、空白对照池塘水样(JH.C和MD.C)真菌群落结构组成相似,两两彼此间首先聚类,而MD.KX与其他样品间距离最远、相似度最低,与PCoA分析结果相似。
|
图 5 基于Weighted unifrac距离构建的UPGMA聚类树 Fig. 5 UPGMA phylogenetic tree constructed based on Weighted unifrac distances |
植物共生菌是与宿主长期共同生活的一类特殊微生物,这些微生物与寄主协同进化,在植物生长发育、抗逆性方面起着重要作用。共生真菌通常与宿主是互惠的,植物的光合产物可以为真菌提供碳源,真菌可以为植物提供营养,帮助植物吸收矿物质,提高宿主吸收营养的能力,产生有生物活性的化合物如抗生素、生物碱等可以提高植物抗性,在植物的适应环境中起到重要作用[27]。植物根系共生菌被发现存在于95%的植物种类上[28-29]。共生真菌的种类丰富,先后从陆生和水生栖息地被分离出来的植物共生菌包括子囊菌(Ascomycetes)[30]、担子菌(Basidiomycete)、半知菌(Adelomycete)和一些卵菌(Oomycetes)[31-32]。有关水生植物共生菌的研究已有报道,如Fisher等观察到河岸植物的根部由于缺少基质而导致水生真菌富集[33]。在鱼菜共生环境下,水生真菌与水生蔬菜相互作用,能够促进蔬菜更好生长。本研究中,从各样品的OTUs聚类和注释情况来看,乌鲁木齐市米东区长山子镇水源地养殖基地(MD)池塘中真菌数量要高于相应新疆水生野生动物救护中心(JH)池塘。较多种类和数量的真菌一方面作为植物的共生菌,另一方面作为水体中的分解者,产生的多种代谢产物促进了植物的生长,这也从一方面解释了蔬菜种植效果乌鲁木齐市米东区长山子镇水源地养殖基地(MD)要优于新疆水生野生动物救护中心(JH)的原因。
从样品分布花瓣图可以看出,两处池塘真菌显示出同一趋势,即蔬菜根系中真菌数量最多、其次为鱼菜共生池塘水样、最少的为普通池塘空白对照水样。植物根系共生真菌能够形成庞大的菌丝网络结构,扩大宿主植物根系吸收范围、提高植物对营养物质的利用率[34]。根系共生真菌还对植物吸收磷等养分具有重要促进作用[35],本试验中,鱼菜共生池塘平均总氮(1.67 mg/L)、总磷(0.26 mg/L)、硝酸盐氮(0.004 mg/L)、氨氮(0.519 mg/L)含量均低于对照池塘(1.74、1.08、0.06和0.539 mg/L),其中总磷和硝酸盐氮下降最明显。乌鲁木齐市米东区长山子镇水源地养殖基地(MD)鱼菜共生池塘真菌数量高于疆水生野生动物救护中心(JH),从空心菜的生长情况来看,整个养殖期间前者共收获空心菜6 250 kg,而后者因空心菜长势不佳,未形成采摘规模。植物长势呈现出与真菌数量一致的现象,也表明植物的生长反过来能促进真菌的繁殖,植物与真菌是一种互利互惠的共生关系。UPGMA聚类树显示两处根系的真菌聚在一起,表明根系特殊的环境催生了某些特有菌类,这些菌类适应了与根系的共生环境,从而形成了根系共有菌群。
两地点根系中共有的真菌为子囊菌门(Ascomycota)中的锤舌菌纲(Leotiomycetes)和石黄衣属(Xanthoria)。有报道称锤舌菌纲为湿地土壤的主要类群[36],该类群菌类能够适应水生生活环境。在一些植物根系土壤、植物组织共生菌也发现锤舌菌纲的存在[37-38],表明这些真菌与植物生长有着密切的关系。而石黄衣是一种是以真菌为主导的,真菌与藻类共生的地衣类群,其中的一些种类能够适应寒冷气候条件,如高寒山地常见的丽石黄衣(Xanthoria elegans)。石黄衣富含多种次生代谢产物[39],通过对根系作用能够调节植物的生长。除共有真菌外,乌鲁木齐市米东区长山子镇水源地养殖基地(MD)空心菜根系中独有的真菌种类较多,包括伞菌纲(Agaricomycetes)、接合菌门(Zygomycota)和酵母菌纲(Saccharomycetes)。酵母菌是人们所熟悉的、也是也较早应用于人类生活实践的一类真菌。酵母菌细胞含有丰富的蛋白质、矿物质和维生素,能够作为饲养动物的替代性饲料,不仅有利于提高鱼类成活率、促进生长,还能够促进鱼类免疫器官的发育、增强鱼类对弧菌病毒的免疫能力。4个月的养殖周期内,该养殖基地鱼菜共生池塘罗非鱼成活率高达98.8%,亩产734 kg,平均体重744 g,高于对照池塘(成活率96.5%,亩产726 kg,平均体重735 g)。此外,酵母菌能够在厌氧和需氧的条件下生存,通过将水体中的有机质转化为自身繁殖的养分,极大的降低了水体中有害物质的含量,有效防止了水体富营养化,优化和改善了养殖水体质量[40]。鱼菜共生池塘中真菌、植物、动物形成了一系列复杂的互利共生关系网,通过优化配置鱼类和蔬菜种类、数量、密度,能够形成一套高效、环保的水产养殖生态循环系统。
4 结语本研究基于Illumina HiSeq 2500高通量测序平台,对鱼菜共生这种新型农业生态复合种养体系中养殖水体和水培蔬菜根系的真菌宏基因组ITS基因序列进行了测定,并探讨了其真菌群落多样性及分布格局。研究结果显示,不同地点、不同样品类型间真菌群落组成和丰度均具有一定差异,新疆水生野生动物救护中心和乌鲁木齐市米东区长山子镇水源地养殖基地鱼菜共生池塘空心菜根系的真菌群落多样性最高且群落结构高度相似。乌鲁木齐市米东区长山子镇水源地养殖基地的鱼菜共生池塘的蔬菜长势明显好于新疆水生野生动物救护中心,并且蔬菜长势与真菌数量呈正相关,推测植物根系与根系共生菌协同作用促进了植物生长。另一方面,鱼菜共生池塘的真菌种类和数量均多于普通池塘,这表明植物的生长可能有利于真菌繁殖。
| [1] |
丁永良, 张明华, 张建华, 等. 鱼菜共生系统的研究[J]. 中国水产科学, 1997, 4(5): 70-75. Ding Y L, Zhang M H, Zhang J H, et al. Researches on fish and vegetable co-existing system[J]. Journal of Fishery Sciences of China, 1997, 4(5): 70-75. DOI:10.3321/j.issn:1005-8737.1997.05.013 (  0) 0) |
| [2] |
Love D C, Fry J P, Laura G, et al. An international survey of aquaponics practitioners[J]. PLoS ONE, 2014, 9(7): 102662. DOI:10.1371/journal.pone.0102662
(  0) 0) |
| [3] |
Kaushik N K, Hynes H B N. The fate of dead leaves that fall into streams[J]. Archiv Fur Hydrobiologie, 1971, 68(4): 465-515.
(  0) 0) |
| [4] |
Scott W W. Research on fungi associated with diseases of fish and fish eggs[J]. North American Journal of Aquaculture, 1961, 23(1): 33-33.
(  0) 0) |
| [5] |
Biedunkiewicz A, Ozimek T. Qualitative and quantitative changes of potentially pathogenic fungi in a hydrophyte wastewater treatment plant[J]. Polish Journal of Environmental Studies, 2009, 18(2): 161-166.
(  0) 0) |
| [6] |
戴芳澜. 真菌的形态和分类[M]. 北京: 科学出版社, 1987. Dai F L. Morphology and Taxonomy of Fungi[M]. Beijing: Science Press, 1987. (  0) 0) |
| [7] |
Kirk P M, Cannon P F, Minter D W, et al. Dictionary of the Fungi (10th Edition)[M]. Clayton: Csiro Publishing, 2008.
(  0) 0) |
| [8] |
Degnan P H, Ochman H. Illumina-based analysis of microbial community diversity[J]. The ISME Journal, 2012, 6(1): 183-194. DOI:10.1038/ismej.2011.74
(  0) 0) |
| [9] |
Caporaso J G, Lauber C L, Walters W A, et al. Ultra-high-throughput microbial community analysis on the Illumina HiSeq and MiSeq platforms[J]. ISME Journal: Multidisciplinary Journal of Microbial Ecology, 2012, 6(8): 1621-1624.
(  0) 0) |
| [10] |
Schocha C L, Seifertb K A, Huhndorf S, et al. Nuclear ribosomal internal transcribed spacer (ITS) region as a universal DNA barcode marker for Fungi[J]. PNAS, 2012, 109(16): 6241-6246. DOI:10.1073/pnas.1117018109
(  0) 0) |
| [11] |
Murray M G, Thompson W F. Rapid isolation of high molecular weight plant DNA[J]. Nucleotide Acids Research, 1980, 8(19): 4321-4325. DOI:10.1093/nar/8.19.4321
(  0) 0) |
| [12] |
Mago T, Salzberg S L. FLASH: Fast length adjustment of short reads to improve genome assemblies[J]. Bioinformatics, 2011, 27(21): 2957-2963. DOI:10.1093/bioinformatics/btr507
(  0) 0) |
| [13] |
Bokulich N A, Subramanian S, Faith J J, et al. Quality-filtering vastly improves diversity estimates from Illumina amplicon sequencing[J]. Nature Methods, 2012, 10(1): 57-59.
(  0) 0) |
| [14] |
Caporaso J G, Kuczynski J, Stombaugh J, et al. QⅡME allows analysis of high-throughput community sequencing data[J]. Nature Methods, 2010, 7(5): 335-336. DOI:10.1038/nmeth.f.303
(  0) 0) |
| [15] |
Edgar R C, Haas B J, Clemente J C, et al. UCHIME improves sensitivity and speed of chimera detection[J]. Bioinformatics, 2011, 27(16): 2194-2200. DOI:10.1093/bioinformatics/btr381
(  0) 0) |
| [16] |
Haas B J, Gevers D, Earl A M, et al. Chimeric 16S rRNA sequence formation and detection in Sanger and 454-pyrosequenced PCR amplicons[J]. Genome Research, 2011, 21(3): 494-504. DOI:10.1101/gr.112730.110
(  0) 0) |
| [17] |
Edgar R C. UPARSE: Highly accurate OTU sequences from microbial amplicon reads[J]. Nature Methods, 2013, 10(10): 996-998. DOI:10.1038/nmeth.2604
(  0) 0) |
| [18] |
Altschul S F, Gish W, Miller W, et al. Basic local alignment search tool[J]. Journal of Molecular Biology, 1990, 215(3): 403-410. DOI:10.1016/S0022-2836(05)80360-2
(  0) 0) |
| [19] |
Urmas K, Nilsson R H, Abarenkov K, et al. Towards a unified paradigm for sequence-based identification of fungi[J]. Molecular Ecology, 2013, 22(21): 5271-5277. DOI:10.1111/mec.12481
(  0) 0) |
| [20] |
Edgar R C. MUSCLE: Multiple sequence alignment with high accuracy and high throughput[J]. Nucleic Acids Research, 2004, 32(5): 1792-1797. DOI:10.1093/nar/gkh340
(  0) 0) |
| [21] |
Desantis T Z, Hugenholtz P, Keller K, et al. NAST: A multiple sequence alignment server for comparative analysis of 16S rRNA genes[J]. Nucleic Acids Research, 2006, 34: 394-399.
(  0) 0) |
| [22] |
Li B, Zhang X, Guo F, et al. Characterization of tetracycline resistant bacterial community in saline activated sludge using batch stress incubation with high-throughput sequencing analysis[J]. Water Research, 2013, 47(13): 4207-4216. DOI:10.1016/j.watres.2013.04.021
(  0) 0) |
| [23] |
Kraková L, Šoltys K, Budiš J, et al. Investigation of bacterial and archaeal communities: Novel protocols using modern sequencing by Illumina miseq and traditional DEGG-cloning[J]. Extremophiles, 2016, 20(5): 795-808. DOI:10.1007/s00792-016-0855-5
(  0) 0) |
| [24] |
Whittaker R H. Evolution and measurement of species diversity[J]. Taxon, 1972, 21(2/3): 213-251.
(  0) 0) |
| [25] |
斯幸峰, 赵郁豪, 陈传武, 等. Beta多样性分解:方法、应用与展望[J]. 生物多样性, 2017, 25(5): 464-480. Si X F, Zhao Y H, Chen C W, et al. Beta-diversity partitioning: Methods, applications and perspectives[J]. Biodiversity Science, 2017, 25(5): 464-480. (  0) 0) |
| [26] |
Lozupone C, Lladser M E, Knights D, et al. UniFrac: An effective distance metric for microbial community comparison[J]. ISME Journal, 2010, 5(2): 169-172.
(  0) 0) |
| [27] |
于小娟, 胡玉金, 刘润进. 真菌与植物共生机制研究进展[J]. 微生物学杂志, 2017, 37(1): 98-104. Yu X J, Hu Y J, Liu R J. Advances in mechanism study of symbiosis between fungi and plants[J]. Journal of Microbiology, 2017, 37(1): 98-104. DOI:10.3969/j.issn.1005-7021.2017.01.016 (  0) 0) |
| [28] |
冯邦, 杨祝良. 外生菌根共生:共生真菌多样性及菌根形成的分子机制[J]. 中国科学:生命科学, 2019, 49(4): 436-444. Feng B, Yang Z L. Ectomycorrhizal symbioses: Diversity of mycobionts and molecular mechanisms that entail the development of ectomycorrhizae[J]. Scientia Sinica Vitae, 2019, 49(4): 436-444. (  0) 0) |
| [29] |
Mugendi D N. Principles and applications of soil micro-biology[J]. Agroforestry Systems, 2001, 51(3): 253-255.
(  0) 0) |
| [30] |
Carroll G C. Fungal endophytes in stems and leaves, from latent pathogen to mutualistic symbiont[J]. Ecology, 1988, 69(1): 2-9. DOI:10.2307/1943154
(  0) 0) |
| [31] |
Petrini O. Taxonomy of endophytic fungi of aerial plant tissues[M]//Fokkema N J, Van den Henvel J. Microbiology of the Phyllosphere. Cambridge: Cambridge University Press, 1986: 175-187.
(  0) 0) |
| [32] |
Sinclair J B, Cerkauskas R F. Latent infection vs endophytic colonization by fungi[M]//Redlin S S, Carris L M. Endophytic Fungi in Grasses and Woody Plants.[s.l.]: APS Saint Paul, 1996: 121-132.
(  0) 0) |
| [33] |
Fisher P J, Petrini O, Webster J. Aquatic hyphomycetes and other fungi living aquatic and terrestrial roots of Alnus glutinosa[J]. Mycological Research, 1991, 95(5): 543-547. DOI:10.1016/S0953-7562(09)80066-X
(  0) 0) |
| [34] |
刘春卯, 贺学礼, 徐浩博, 等. 蒙古沙冬青AM真菌物种多样性研究[J]. 生态环境学报, 2013, 22(7): 1148-1152. Liu C M, He X L, Xu H B, et al. Species diversity of arbuscular mycorrhizal fungi in the rhizosphere of Ammopiptanthus monglicus[J]. Ecology and Environmental Science, 2013, 22(7): 1148-1152. DOI:10.3969/j.issn.1674-5906.2013.07.010 (  0) 0) |
| [35] |
王思佳, 赖文珍, 谢贤安, 等. 桉树根系共生真菌的分离与鉴定[J]. 西北植物学报, 2018, 38(8): 1553-1561. Wang S J, Lai W Z, Xie X A, et al. Isolation and identification of symbiotic fungi in Eucalyptus roots[J]. Acta Botanica Boreali-Occidentalia Sinica, 2018, 38(8): 1553-1561. (  0) 0) |
| [36] |
陈会海, 许修宏, 李洪涛. 应用PCR-DGGE分析三江湿地土壤真菌群落结构的多样性[J]. 环境科学研究, 2012, 25(11): 1272-1278. Chen H H, Xu X H, Li H T. Diversity analysis of soil fungal community structure in Sanjiang wetland by PCR-DGGE[J]. Research of Environmental Sciences, 2012, 25(11): 1272-1278. (  0) 0) |
| [37] |
徐雪雪, 王东, 秦舒浩, 等. 沟垄覆膜连作马铃薯根际土壤真菌多样性分析[J]. 水土保持学报, 2015, 29(6): 301-306. Xu X X, Wang D, Qin S H, et al. Diversity analysis of fungal communities in potato continuous cropping soil under different patterns of ridge-furrow film mulching[J]. Journal of Soil and Water Conservation, 2015, 29(6): 301-306. (  0) 0) |
| [38] |
韦艳梅, 周雅琴, 李力, 等. 白及内生真菌多样性研究[J]. 广西植物, 2016, 36(7): 832-836. Wei Y M, Zhou Y Q, Li L, et al. Diversity of endophytic fungi associated with Bletilla striata[J]. Guihaia, 2016, 36(7): 832-836. (  0) 0) |
| [39] |
王海英, 李红梅, 石楠, 等. 五种石黄衣中次生代谢产物的高压液相色谱分析[J]. 菌物系统, 2003, 22(4): 536-541. Wang H Y, Li H M, Shi N, et al. The HPLC analysis of the lichen substances in five species of Xanthoria (Ascomycota)[J]. Mycosystema, 2003, 22(4): 536-541. DOI:10.3969/j.issn.1672-6472.2003.04.008 (  0) 0) |
| [40] |
何杨. 酵母菌在水产养殖中的应用[J]. 饲料博览, 2018, 317(9): 24-28. He Y. Application of yeast in aquaculture[J]. Animal Nutrition, 2018, 317(9): 24-28. DOI:10.3969/j.issn.1001-0084.2018.09.007 (  0) 0) |
2. Xinjiang Fisheries Research Institute, Urumqi 830000, China;
3. Chongqing Fisheries Technology Extension Station, Chongqing 400020, China;
4. Baihui Yusheng Fishery Science and Technology Co., Ltd., Urumqi 830026, China;
5. Fisheries College, Zhejiang Ocean University, Zhoushan 316022, China
 2021, Vol. 51
2021, Vol. 51


