2. 青岛市海洋减灾中心,山东 青岛 266003
小黄鱼(Larimichthys polyactis)是鲈形目(Perciformes)石首鱼科(Sciaendae)黄鱼属(Larimichthys)的一种经济鱼类,主要栖息在河口附近及沿岸海域[1]。小黄鱼是暖温性底层洄游鱼类,主要摄食甲壳类、幼鱼以及其他种类的浮游生物[2]。小黄鱼是西北太平洋所特有的重要经济鱼类,在中国渤海、黄海和东海海域均有分布。曾经我国小黄鱼的资源量非常丰富,在我国的捕捞渔业中一直占有重要地位,是我国著名的“四大海产”之一[2]。然而随着捕捞压力的加大,小黄鱼不仅产量降低且有低龄化趋势[3]。虽然从二十世纪九十年代开始,国家出台了一系列保护措施,但目前我国小黄鱼渔业资源状况仍不容乐观[4]。
为了研究小黄鱼的种质资源状况,制定更合理的保护策略,很多学者从资源量、洄游、形态学、繁殖生物学等不同方面展开研究[4-7],利用分子标记手段对小黄鱼群体遗传学的研究也有多篇报道[8-10]。海洋鱼类遗传多样性和遗传结构研究是解析物种分布规律与资源变动情况的有效手段[11],目前鱼类群体遗传学的研究报道多集中于分析不同地理群体空间尺度上的遗传多样性差异。然而也有研究发现,同一海域不同时间采集的样品也会存在遗传学差异[12],这使得群体遗传学研究变得更加复杂。若群体存在时间尺度上的遗传分化,空间上的遗传多样性和遗传结构也会由于采样时间的不同而产生一定的差异[12-13]。此外,群体时间尺度上的遗传差异也能在一定程度上反映物种资源量的波动情况。目前小黄鱼群体遗传学研究均集中在不同地理群体间[4-7],尚未见其同一地点不同时间尺度上的群体遗传学的研究报道。
由于操作简便和结果准确,线粒体DNA标记和微卫星DNA标记已成为群体遗传学研究中较常用的分子标记[14-17]。本研究利用线粒体DNA控制区序列和微卫星标记技术对青岛近海小黄鱼不同季节的采集样品进行了群体遗传学研究,旨在探讨小黄鱼遗传多样性的变化,从而分析小黄鱼分布规律与资源变动情况,为小黄鱼资源的合理保护提供基础资料。
1 材料和方法 1.1 样品采集本研究于2019—2020年在青岛近海通过当地渔民小型船近岸采集得到了3、5、8和12月共计4个月份的青岛近海小黄鱼样品,同时从GenBank上下载了2005年的序列数据(KF412731~KF412751)[9],共计117尾样品的序列进行分析。对新采集的样品进行形态鉴定,然后取其肌肉组织并加入95%乙醇,样品和肌肉组织均放入-20 ℃冰箱进行保存(见表 1)。
|
|
表 1 小黄鱼样品采集信息与遗传多样性指数 Table 1 Sample information and genetic statistics of L.polyactis populations |
使用传统的苯酚-氯仿法提取小黄鱼基因组DNA[17-18]。依据吴仁协等[19]扩增的小黄鱼线粒体DNA全序列(GenBank: NC 013754),利用Primer Premier 5软件设计小黄鱼线粒体DNA控制区引物,然后进行PCR扩增[20]。引物序列为F2-CGGACGTC-GGGGGTTAAAT和R2-ATGGGGAGCAACCACA-AGAA。参考Song等[21]的研究来配置PCR反应体系,体系总体积为25 μL。PCR反应程序:94 ℃预变性5 min; 94 ℃变性45 s,53 ℃退火45 s,72 ℃延伸45 s,37个循环; 72 ℃延伸10 min。PCR产物经过电泳检测后,将扩增效果较好的产物送到北京擎科生物有限公司青岛分公司进行双向测序。
从Liu等[22]筛选的17对微卫星引物中合成12对(Lpo103、Lpo104、Lpo105、Lpo106、Lpo109、Lpo110、Lpo111、Lpo112、Lpo113、Lpo114、Lpo115、Lpo116)用于本研究中样品的扩增。12对引物的正向引物在合成时添加荧光接头,共3种荧光接头,分别为HEX、FAM和TAMARA,利用3种接头的不同颜色来识别数据。PCR反应体系和反应条件参考Liu等[22]的研究来配置。使用琼脂糖对扩增产物进行检测,合格的产物送至北京擎科生物有限公司青岛分公司进行基因分型。
1.3 数据分析使用DNASTAR(DNASTAR,Inc.,Medison,WI)软件包中的Seqman根据测序峰图校正和比对序列,使用MEGA 7.0软件构建小黄鱼单倍型系统发育树[23],经过1 000次bootstrap抽样对系统发育树的可靠性进行评估。使用Arlequin 3.5软件获得单倍型多样度、核苷酸多样度、遗传分化等指标[24]。使用Lamarc 2.0软件估算参数θ(θ=Ne·μ,其中Ne为有效种群大小,μ为突变速率)[25],根据θ计算各季节小黄鱼样本有效种群Ne的大小[26]。根据小黄鱼控制区的突变速率为(3%~12%)/百万年[17],使用Lamarc 2.0软件计算各群体间的基因流M(M=m/μ)和生长率参数g。
使用Genemarker v.1.91软件读取每个个体的各位点的等位基因长度[27]; 使用Fstat v.2.9软件计算各个位点的遗传多样性指标,包括等位基因丰度、等位基因数、观测杂合度和期望杂合度[28]; 使用Excel Microsatellite Toolkit(MS-tools)计算各位点的多态信息含量(PIC)[29]; 使用Genepop v.4.0软件来检测哈代温伯格平衡和连锁不平衡[30]; 使用Structure v.2.3软件确定小黄鱼样本的分配模式[31-32]; 使用Fstat v.2.9软件计算小黄鱼样本的遗传分化指数(Fst)[28]; 使用Population v.1.2软件计算遗传距离[33]; 使用Genetix软件进行三维因子主成分分析[34]。
2 结果 2.1 基于线粒体DNA控制区的遗传学分析经过与Xiao等[9]扩增的小黄鱼序列进行比对后获得447 bp的同源序列,该序列用于后续分析。结果显示:序列的平均A、T、C、G含量分别为27.46%、35.75%、14.43%、22.36%,A+T的含量高于C+G,展现了碱基组成的偏倚。多态位点数量、单倍型多样度和核苷酸多样度见表 1,本研究中青岛近海小黄鱼存在着较高的遗传多样性,且单倍型多样度和核苷酸多样度均略高于2005年的数据。
本研究中在117条序列中共检测出63个多态位点,这些多态位点共定义了81个单倍型。2019年8月采集的样本有最多的单倍型,为24个,2005年4月样本的单倍型数最少,为16个(见表 2)。
|
|
表 2 各群体小黄鱼单倍型统计 Table 2 Statistical table of haplotypes in each population of L. polyactis |
以大黄鱼为外群(GenBank: EU339149)构建的单倍型NJ系统树显示,81个单倍型杂乱分布,没有显著的遗传结构(见图 1);最小跨度树也显示出同样的结果(见图 2)。

|
图 1 以大黄鱼为外群(GenBank: EU339149)构建的单倍型NJ系统树 Fig. 1 The phylogenetic analyzes for L. polyactis investigated using Neighbor-joining tree (NJ) with Larimichthys crocea as the outgroup (GenBank: EU339149) |
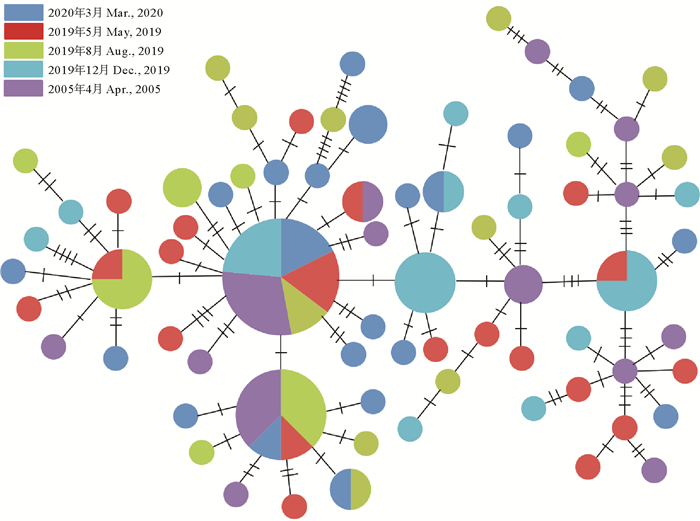
|
图 2 基于小黄鱼线粒体DNA控制区序列构建的单倍型网络图 Fig. 2 Haplotype network diagram based on mitochondrial DNA control region sequence of L. polyactis |
小黄鱼不同季节样本间的遗传分化指数Fst值整体较小,仅部分季节样本间存在微弱的遗传分化(见图 3)。为进一步探究青岛近海小黄鱼不同季节样本的遗传差异,对所有的小黄鱼样本进行了3种组合的AMOVA分析,分别是一个基因池(全部样本)、一个基因池(近期小黄鱼样本)、两个基因池(近期小黄鱼样本、2005年小黄鱼样本)。AMOVA分析结果显示,3种分组模式下,小黄鱼样本内部的遗传变异均大于95%,不同季节小黄鱼样本间无显著遗传差异。
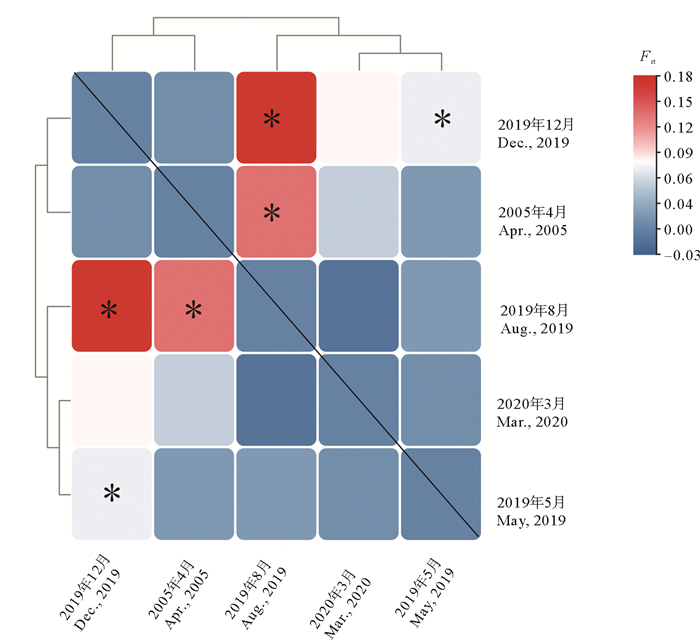
|
图 3 小黄鱼群体间的Fst值 Fig. 3 The pairwise Fst values among L. polyactis populations |
本研究中青岛近海小黄鱼有效种群数量高于2005年的数据,且生长指数也均呈现较大的正值。种群基因流分析结果显示本研究中不同季节样本间的基因流较高,而2005年的样本与近期各月份的样本基因流是较低的(见表 3)。
|
|
表 3 小黄鱼群体的有效种群、生长指数及基因流 Table 3 Effective population, growth parameter and gene flow of L. polyactis |
基于12个位点对2019—2020年小黄鱼样本的遗传多样性分析,结果见表 4。不同季节小黄鱼样本共获得了185个等位基因,等位基因丰富度的范围是11.752~21.695。所有位点的多态信息含量的范围是0.841~0.941,平均多态信息含量为0.891,所有样本均表现出较高的遗传多样性水平(见表 4)。哈代温伯格平衡检测结果表明,经Bonferroni校正后,不同季节小黄鱼样本在2个位点上偏离平衡状态(P>0.05)。本研究中小黄鱼的平均观测杂合度、平均期望杂合度和平均多态信息含量均高于2009年[35](见表 5),这一结果与基于线粒体DNA控制区的分析结果基本一致。
|
|
表 4 基于12个微卫星位点的小黄鱼群体的遗传多样性参数 Table 4 Summary statistics for the variability 12 polymorphic microsatellite loci in L. polyactis populations |
|
|
表 5 近两年小黄鱼样本的遗传多样性与历史数据的对比 Table 5 Comparison of population genetic diversity in recent two years with historical data |
小黄鱼不同季节样本的遗传分化指数Fst值范围为-0.024(2019年5月和2019年8月相比较)~0.152(2019年5月和2020年3月相比较)(见表 6),遗传距离(δμ)2范围为0.279(2019年5月和2019年8月相比较)~0.414(2019年5月和2020年3月相比较)(见表 6)。利用主成分分析获得的三个变异因子的贡献率分别为50.29%、29.25%和20.64%。结果表明,不同季节小黄鱼样本均有较大部分重叠,不能被有效区分(见图 4)。
|
|
表 6 小黄鱼样本的Fst值(对角线下)与遗传距离(δμ)2(对角线上) Table 6 Pairwise Fst (above diagonal) and (δμ)2 (below diagonal) among populations of L. polyactis |
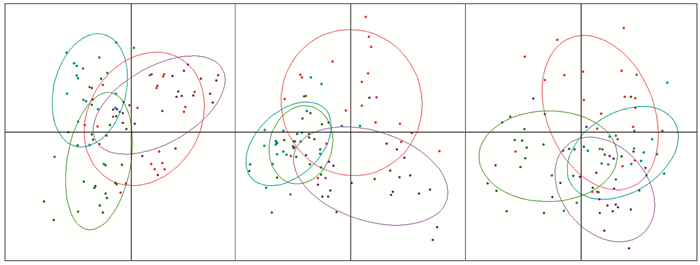
|
(紫色:2019年5月,红色:2019年8月,绿色:2019年12月; 蓝色:2020年3月。Purple: May, 2019, Red: Aug., 2019, Green: Dec., 2019, Blue: Mar., 2020. ) 图 4 小黄鱼样本的主成分分析 Fig. 4 Principal component analysis of L. polyactis populations |
基于混合模型对不同季节小黄鱼样本进行了STRUCTURE分析,结果表明,K=3为组群分配的最优值,当K=3时,各季节样本的分配比例相对一致(见图 5)。
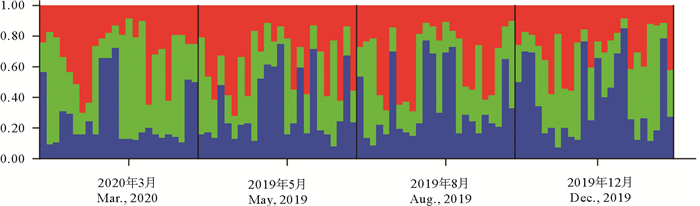
|
图 5 不同季节小黄鱼的STRUCTURE条形图(K=3) Fig. 5 STRUCTURE bar plots (K=3) from 12 microsatellite loci of L. polyactis populations |
物种的遗传多样性是生物进化的原始材料,在物种进化过程中起到了重要作用。利用线粒体DNA标记和微卫星标记的多态性计算不同群体的遗传学参数,继而通过统计学手段检测群体的遗传结构、遗传多样性和群体历史动态是非常有效的,并且已经被广泛的应用到海洋鱼类的群体遗传学分析中[16, 36-37]。群体时间尺度上的遗传变异的原因可能是存在一定地理隔离的物种在不同时期出现在同一区域内,或者是该物种在同一时期出现在同一区域但彼此没有混合。鱼类的洄游和鱼卵、仔稚鱼随洋流漂浮的特性为这一现象的产生提供了条件。研究物种时间尺度上的遗传多样性和遗传结构能更有效、更全面地鉴别种群,估算种群的遗传多样性变化,对渔业资源的管理和保护有着重要意义。
本研究中线粒体DNA控制区序列分析的结果表明青岛近海小黄鱼群体呈现较高的单倍型多样性和较低的核苷酸多样性,这表明小黄鱼群体内的遗传多样性较高。这种遗传模式也存在于其他海洋鱼类之中[38-39]。物种在遗传漂变的作用下会损失部分频率较低的单倍型,而短时间内群体规模的快速增长可能会造成随机遗传漂变减小,从而部分稀有单倍型被保存下来,造成了小黄鱼群体有较高的遗传多样性[40]。微卫星标记的研究结果也支持了这一结论。
与Xiao等[9]和Liu等[35]的数据相比较,本研究中线粒体DNA控制区序列和微卫星标记的分析结果均表明青岛近海小黄鱼的遗传多样性指数略有升高。且基于线粒体DNA标记估算的近期小黄鱼的有效种群也高于2005年的数据,这可能与小黄鱼的适应能力较强以及国家相关禁渔法规的出台有关[41]。
遗传分化指数Fst是估测群体内遗传相似性的有效手段[42]。本研究中两种标记的分析结果均表明不同季节小黄鱼样本间存在较低的遗传分化,同时主成分分析和STRUCTURE分析结果也表明小黄鱼的遗传变异主要来自于群体内部。Xiao等[9]和Liu等[35]等基于线粒体DNA和微卫星标记的研究结果显示我国近海小黄鱼各群体间有较高的基因流,因此尽管小黄鱼存在大范围洄游现象,但青岛近海各季节小黄鱼样本没有显著的遗传分化[5, 43]。
小黄鱼与大黄鱼同属于“四大海产”,我国近海的大黄鱼种群在经历了二十世纪八十年代的急剧衰退后,资源量始终难以恢复,而小黄鱼的资源量却有逐渐上升的趋势[44]。尽管遗传学研究的结果表明小黄鱼现存的种群保持着较高的遗传多样性和支持其长期续存的进化潜力,但是目前小黄鱼的小型化和低龄化状况不容乐观[45]。有研究表明小黄鱼目前的生长方式属于负异速生长类型,即体长的增长快于体质量的增加,也证实了小黄鱼群体有小型化和提早性成熟现象[46]。因此,应制定更合理的保护政策,同时加强对渔民的宣传教育,提高小黄鱼的最小可捕规格,切实有效实现小黄鱼渔业资源的可持续发展。
4 结语本研究结果显示青岛近海小黄鱼不同季节样本间没有显著的遗传分化,表明我国近海不同地理群体的小黄鱼可能存在较高的基因流。青岛近海小黄鱼群体具有较高的遗传多样性暗示其资源量有恢复的趋势。但小黄鱼个体的小型化与早熟化现象也急需我们采取更合理的保护措施。
| [1] |
朱元鼎, 罗云林, 伍汉霖, 等. 中国石首鱼类分类系统的研究和新属新种的叙述[M]. 上海: 上海科学技术出版社, 1963: 50-51. Zhu Y D, Luo Y L, Wu H L, et al. Classification System of Sciaenidae in China and Description of New Genera and Species[M]. Shanghai: Shanghai Science and Technology Press, 1963: 50-51. (  0) 0) |
| [2] |
黄昊, 程起群. 小黄鱼生物学研究进展[J]. 现代渔业信息, 2010, 25(9): 9-12. Huang H, Cheng Q Q. Recent progress and prospect of small yellow croaker (Pseudosciaena polyactis Bleeker)[J]. Modern Fisheries Information, 2010, 25(9): 9-12. DOI:10.3969/j.issn.1004-8340.2010.09.003 (  0) 0) |
| [3] |
林龙山, 程家骅. 东海区小黄鱼渔业生物学现状的分析[J]. 中国海洋大学学报(自然科学版), 2004, 34(4): 565-570. Lin L S, Cheng J Y. An analysis of the current situation of fishery biology of small yellow croaker in the East China Sea[J]. Periodical of Ocean University of China, 2004, 34(4): 565-570. (  0) 0) |
| [4] |
韩青鹏, 单秀娟, 万荣, 等. 基于地统计二阶广义线性混合模型的黄海冬季小黄鱼时空分布和资源量指数估算[J]. 水产学报, 2019, 43(7): 1603-1614. Han Q P, Shan X J, Wan R, et al. Spatiotemporal distribution and the estimated abundance indices of Larimichthys polyactis in winter in the Yellow Sea based on geostatistical delta-generalized linear mixed models[J]. Journal of Fisheries of China, 2019, 43(7): 1603-1614. (  0) 0) |
| [5] |
徐兆礼, 陈佳杰. 小黄鱼洄游路线分析[J]. 中国水产科学, 2009, 16(6): 931-940. Xu Z L, Chen J J. Analysis on migratory routine of Larimichthy polyactis[J]. Journal of Fishery Sciences of China, 2009, 16(6): 931-940. DOI:10.3321/j.issn:1005-8737.2009.06.014 (  0) 0) |
| [6] |
韩真, 肖永双, 高天翔. 中国近海9个小黄鱼群体的形态学比较研究[J]. 南方水产科学, 2012, 8(3): 25-33. Han Z, Xiao Y S, Gao T X. Comparison of morphological characteristics of 9 Larimichthys polyactis populations in China[J]. South China Fisheries Science, 2012, 8(3): 25-33. DOI:10.3969/j.issn.2095-0780.2012.03.004 (  0) 0) |
| [7] |
郑学斌, 杜晨, 王景倩, 等. 小黄鱼(Larimichthys polyactis)精子的生理特性及超低温冷冻保存研究[J]. 海洋与湖沼, 2020, 51(1): 193-205. Zheng X B, Du C, Wang J Q, et al. Physiological characteristics and cryopreservation effect of Larimichthys polyactis sperm[J]. Oceanologia et Limnologia Sinica, 2020, 51(1): 193-205. (  0) 0) |
| [8] |
Meng Z N, Zhuang Z M, Jin X S, et al. Genetic diversity in small yellow croaker (Pseudosciaena polyactis) by RAPD analysis[J]. Chinese Biodiversity, 2003, 11(3): 197-203. (  0) 0) |
| [9] |
Xiao Y S, Song N, Li J, et al. Significant population genetic structure detected in the small yellow croaker Larimichthys polyactis inferred from mitochondrial control region[J]. Mitochondrial DNA, 2013, 26(3): 409. (  0) 0) |
| [10] |
Li Y, Han Z Q, Song N, et al. New evidence to genetic analysis of small yellow croaker (Larimichthys polyactis) with continuous distribution in China[J]. Biochemical Systematics & Ecology, 2013, 50: 331-338. (  0) 0) |
| [11] |
Maria R R, Romana E, Minoru I, et al. Genetic diversity in farmed Asian Nile and red hybrid tilapia stocks evaluated from microsatellite and mitochondrial DNA analysis[J]. Aquaculture, 2004, 236(1): 131-150. (  0) 0) |
| [12] |
Heyden S V D, Lipinski M R, Matthee C A. Remarkably low mtDNA control region diversity in an abundant demersal fish[J]. Molecular Phylogenetics & Evolution, 2010, 55(3): 1183-1188. (  0) 0) |
| [13] |
Pujolar J M, Maes G E, Volckaert F A M. Genetic patchiness among recruits in the European eel (Anguilla anguilla)[J]. Marine Ecology Progress Series, 2006, 307(1): 209-217. (  0) 0) |
| [14] |
Crandall K A, Posada D, Vasco D. Effective population sizes: Missing measures and missing concepts[J]. Animal Conservation, 1999, 2(4): 317-319. DOI:10.1111/j.1469-1795.1999.tb00078.x (  0) 0) |
| [15] |
Wang L, Liu S, Zhuang Z M, et al. Population genetic studies revealed local adaptation in a high gene-flow marine fish, the small yellow croaker (Larimichthys polyactis)[J]. PLoS One, 2013, 8(12): e83493. DOI:10.1371/journal.pone.0083493 (  0) 0) |
| [16] |
Shan B B, Liu Y, Song N, et al. Genetic diversity and population structure of black sea bream (Acanthopagrus schlegelii) based on mitochondrial control region sequences: The genetic effect of stock enhancement[J]. Regional Studies in Marine Science, 2020, 35: 101188. DOI:10.1016/j.rsma.2020.101188 (  0) 0) |
| [17] |
Xiao Y S, Zhang Y M, Gao T X, et al. Genetic diversity in the mtDNA control region and population structure in the small yellow croaker Larimichthys polyactis[J]. Environmental Biology of Fishes, 2009, 85(4): 303-314. DOI:10.1007/s10641-009-9497-0 (  0) 0) |
| [18] |
Sambrook J, Fritsch E F, Maniatis T. Molecular Cloning: A Laboratory Manual[M]. 2nd eds. New York: Cold Spring Harbor Laboratory Press, 1989.
(  0) 0) |
| [19] |
吴仁协. 小黄鱼线粒体基因组全序列和群体遗传学研究[D]. 厦门: 厦门大学, 2009. Wu R X. Studies on Complete Mitochondrial Genome and Population Genetics of Larimichthys polyactis (Bleeker 1877)[D]. Xiamen: Xiamen University, 2009. (  0) 0) |
| [20] |
Lalitha S. Primer premier 5[J]. Biotech Software & Internet Report, 2000, 1(6): 270-272. (  0) 0) |
| [21] |
Song N, Zhang X M, Sun X F, et al. Population genetic structure and larval dispersal potential of spotted tail goby Synechogobius ommaturus of the Northwest Pacific[J]. Journal of Fish Biology, 2010, 77: 388-402. DOI:10.1111/j.1095-8649.2010.02694.x (  0) 0) |
| [22] |
Liu B J, Gao T X, Liu J X. Development of 17 novel polymorphic microsatellites in the small yellow croaker Larimichthys polyactis[J]. Conservation Genetics Resources, 2014, 6(2): 397-399. DOI:10.1007/s12686-013-0102-7 (  0) 0) |
| [23] |
Kumar S, Stecher G, Tamura K. MEGA7: Molecular evolutionary genetics analysis version 7.0 for bigger datasets[J]. Molecular Biology and Evolution, 2016, 33(7): 1870-1874. DOI:10.1093/molbev/msw054 (  0) 0) |
| [24] |
Excoffier L, Lischer H E L. Arlequin suite ver 3.5: A new series of programs to perform population genetics analyses under Linux and Windows[J]. Molecular Ecology Resources, 2010, 10(3): 564-567. DOI:10.1111/j.1755-0998.2010.02847.x (  0) 0) |
| [25] |
Felsenstein J, Kuhner M K, Yamato J, et al. Likelihoods on coalescents: A Monte Carlo sampling approach to inferring parameters from population samples of molecular data[J]. Lecture Notes-Monograph Series, 1999, 33: 163-185. (  0) 0) |
| [26] |
Kuhner M K, Smith L P. Comparing likelihood and Bayesian coalescent estimation of population parameters[J]. Genetics, 2006, 175(1): 155-165. (  0) 0) |
| [27] |
Hulce D, Li X, Snyderleiby T, et al. GeneMarker genotyping software: Tools to increase the statistical power of DNA fragment analysis[J]. Journal of Biomolecular Techniques, 2011, 22(Suppl): 35. (  0) 0) |
| [28] |
Goudet J. FSTAT (version 1.2): A computer program to calculate F-statistics[J]. Journal of Heredity, 1995, 86: 485-486. DOI:10.1093/oxfordjournals.jhered.a111627 (  0) 0) |
| [29] |
Park S D E. Trypanotolerance in West African Cattle and the Population Genetic Effects of Selection[D]. Dublin: University of Dublin, 2001.
(  0) 0) |
| [30] |
Rousset F. GENEPOP: A complete re-implementation of the GENEPOP software for Windows and Linux[J]. Molecular Ecology Notes, 2008, 8(1): 103-106. DOI:10.1111/j.1471-8286.2007.01931.x (  0) 0) |
| [31] |
Pritchard J K, Tephens M S, Onnelly P D. Inference of population structure using multilocus genotype data[J]. Genetics, 2000, 155: 945-959. DOI:10.1093/genetics/155.2.945 (  0) 0) |
| [32] |
Evanno G, Regnaut S, Goudet J. Detecting the number of clusters of individuals using the software STRUCTURE: A simulation study[J]. Molecular Ecology, 2005, 14: 2611-2620. DOI:10.1111/j.1365-294X.2005.02553.x (  0) 0) |
| [33] |
Raymond M, Rousset F. GENEPOP (version 1.2): Population genetics software for exact tests and ecumenicism[J]. Journal of Heredity, 1995, 86(3): 248-249. DOI:10.1093/oxfordjournals.jhered.a111573 (  0) 0) |
| [34] |
Belkhir K. Genetix 4.05, Logiciel Sous Windows TM Pour la Genetique des Populations 4.05. Laboratoire Genome, Populations, Interactions, CNRS UMR 5000[D]. Montpellier, France: University de Montpellier Ⅱ, 2004.
(  0) 0) |
| [35] |
Liu B J, Zhang B, Dong X D, et al. Population structure and adaptive divergence in a high gene flow marine fish: The small yellow croaker (Larimichthys polyactis)[J]. PLoS One, 2016, 11(4): e0154020. DOI:10.1371/journal.pone.0154020 (  0) 0) |
| [36] |
Tajima F. Evolutionary relationship of DNA sequences in finite populations[J]. Genetics, 1983, 105(2): 437-460. DOI:10.1093/genetics/105.2.437 (  0) 0) |
| [37] |
Song N, Ma G Q, Zhang X M, et al. Genetic structure and historical demography of Collichthys lucidus inferred from mtDNA sequence analysis[J]. Environmental Biology of Fishes, 2014, 97(1): 69-77. DOI:10.1007/s10641-013-0124-8 (  0) 0) |
| [38] |
Ishikawa S, Aoyama J, Tsukamoto K, et al. Population structure of the Japanese eel Anguilla japonica as examined by mitochondrial DNA sequencing[J]. Fisheries Science, 2001, 67: 246-253. DOI:10.1046/j.1444-2906.2001.00227.x (  0) 0) |
| [39] |
Zheng W J, Lai Y H, You X Y, et al. Genetic diversity of Pseudosciaena polyactis in Zhoushan based on mitochondrial DNA D-loop region sequences[J]. Zoological Research, 2012, 33(3): 329-336. (  0) 0) |
| [40] |
Rogers A, Harpending H. Population growth makes waves in the distribution of pairwise genetic difference[J]. Molecular Biology and Evolution, 1992, 9: 552-569. (  0) 0) |
| [41] |
刘炳舰. 小黄鱼和日本鳗鲡群体遗传结构及本地适应性研究[D]. 北京: 中国科学院大学, 2017. Liu B J. Genetic Structure and Native Adaptability of Larimichthys polyactis and Japanese Eel Populations[D]. Beijing: University of Chinese Academy of Sciences, 2017. (  0) 0) |
| [42] |
Meirmans P G, Hedrick P W. Assessing population structure: Fst and related measures[J]. Molecular Ecology Resources, 2015, 11: 5-18. (  0) 0) |
| [43] |
殷名称. 鱼类早期生活史研究与其进展[J]. 水产学报, 1991, 15(4): 348-358. Yin M C. Research on the early life history of fishes and its progress[J]. Journal of Fisheries of China, 1991, 15(4): 348-358. (  0) 0) |
| [44] |
林龙山, 姜亚洲, 刘尊雷, 等. 黄海南部和东海小黄鱼资源分布差异性研究[J]. 中国海洋大学学报(自然科学版), 2010, 40(3): 1-6. Lin L S, Jiang Y Z, Liu Z L, et al. Analysis of the distribution difference of small yellow croaker between the southern Yellow Sea and the East China Sea[J]. Periodical of Ocean University of China, 2010, 40(3): 1-6. (  0) 0) |
| [45] |
Lin L S, Li Z L, Jin Y Z. Current status of small yellow croaker resources in the southern Yellow Sea and the East China Sea[J]. Chinese Journal of Oceanology and Limnology, 2011, 29(3): 547-555. DOI:10.1007/s00343-011-0182-8 (  0) 0) |
| [46] |
李忠炉, 金显仕, 单秀娟, 等. 小黄鱼体长-体质量关系和肥满度的年际变化[J]. 中国水产科学, 2011, 18(3): 602-610. Li Z L, Jin X S, Shan X J, et al. Inter-annual changes on body weight-length relationship and relative fatness of small yellow croaker (Larimichthys polyactis)[J]. Journal of Fishery Sciences of China, 2011, 18(3): 602-610. (  0) 0) |
2. Qingdao Marine Hazard Mitigation Service, Qingdao 266003, China
 2022, Vol. 52
2022, Vol. 52


