2. 青岛海洋科学与技术试点国家实验室 海洋渔业科学与食物产出过程功能实验室, 山东 青岛 266237;
3. 日照山海天旅游度假区渔政监督管理站, 山东 日照 276800
在气候变化、过度捕捞、海洋环境污染等多重压力的作用下,鱼类种群出现了小型化、低龄化的特征,种类组成则发生了暖水种增加的变化,海洋生态系统正面临着严峻的挑战[1-2]。海洋鱼类栖息地的有效监测对于开展海洋生态系统和海洋生物多样性的保护、管理和研究至关重要[3]。生物多样性监测可以有效评估群落结构和功能的时空变化[4],进而分析环境和气候等因素对生物多样性产生的影响。大范围和长时期的连续监测能够揭示该地区物种的变化以及不同物种独特的生活史现象,为海洋生态系统的保护和管理政策的制定提供重要依据。
传统的海洋生物多样性监测多采用拖网调查的方式进行[5]。拖网调查提供了对鱼类丰度和生物量的估计,同时能获取被采集样品的年龄结构、性别比例、性腺成熟度和摄食情况等基础生物学信息。虽然渔业拖网调查在生物多样性调查和评估当中具有突出贡献,但其采样方式和要求都存在一定的局限性[6-7]。渔业拖网调查费用昂贵,且要求调查人员具备专业的分类学鉴定能力[8-9]。在拖网过程中,受限于拖网类型, 只能捕获特定水层的生物,特定的网口设计也会导致遗漏小型或稀有物种。此外,拖网会对海洋环境造成破坏,破坏海底原有地形的植被,危及海洋生物的生存和繁殖[10],从而对海洋生物造成潜在伤害。在时间和空间尺度上都有着一定的缺陷。
近年来对入侵物种和稀有物种监测的研究表明,环境DNA metabarcoding是一种低成本、灵敏性高且无入侵性的调查方法[11-12]。随着高通量测序技术的发展和普及,环境DNA metabarcoding(eDNA metabarcoding)被广泛地应用于海洋鱼类多样性调查和渔业管理,能够对传统渔业拖网调查提供良好的补充[13-14]。Closek等利用MitoFish-U通用引物调查了加州中部洋流中两年间的鱼类群落结构变化,且与拖网调查进行了比较[15]。Aglieri等的研究表明,环境DNA metabarcoding能够有效地捕获沿海鱼类群落的分类多样性和功能多样性[16]。舒璐等通过环境DNA metabarcoding技术调查了洱海的鱼类多样性,结果表明环境DNA metabarcoding可用于快速检测洱海鱼类的多样性和空间分布[17]。环境DNA metabarcoding的应用扩大了生物多样性的研究范围,越来越多的研究通过这种方法进行物种鉴定和生物多样性评估[18]。与水性环境DNA(Aqueous eDNA)相比,沉积物环境DNA(Sediment eDNA)多被用于讨论过去更长时间尺度的生物信息。有研究表明,部分游离的环境DNA片段无法长时间悬浮在水中而向下积聚在沉积物上[19]。Turner等的研究发现沉积物环境DNA的可检测周期比水性环境DNA的可检测周期更长[11]。文献[20]比较了沉积物环境DNA和水性环境DNA检测的物种数量差异,结果表明沉积物环境DNA能检测到更多的物种。Siegenthaler等的研究表明,沉积物环境DNA和水性环境DNA检测到的物种仅有部分重叠[21]。因此,在生物多样性监测中加入沉积物环境DNA采样能够提高生物多样性检测的准确性和丰富度。
对于较少站位和层位的环境DNA采样,其检测物种分布和多样性的准确性取决于DNA在水中的扩散程度,有研究通过海洋动力模型模拟了环境DNA在水平方向的传输[22]。但在垂直分层作用的海洋环境中,环境DNA的分布规律尚不清楚。Jeunen等的研究表明,存在表底盐度差异的峡湾地区水体分层限制了淡水生物环境DNA信号的垂向扩散[23]。黄海是典型的陆架海域,水深相对较浅,具有较强的海洋水动力过程,同时在夏季冷水团的作用下出现了垂直温度分层[24]。在浅海温度分层水体环境下环境DNA metabarcoding技术反映海洋生物多样性和空间分布的效果尚待评估。
本研究根据2020年夏季南黄海西部13个站位的环境DNA采样和底拖网调查数据,基于Alpha多样性指数和Beta多样性分析,比较了环境DNA metabarcoding和底拖网两种方法检测鱼类多样性的能力,探讨了环境DNA metabarcoding技术监测海洋生物的多样性和空间分布的效果。此外,还比较了水性环境DNA和沉积物环境DNA检测鱼类多样性的能力,为环境DNA metabarcoding相关研究提供数据支持。
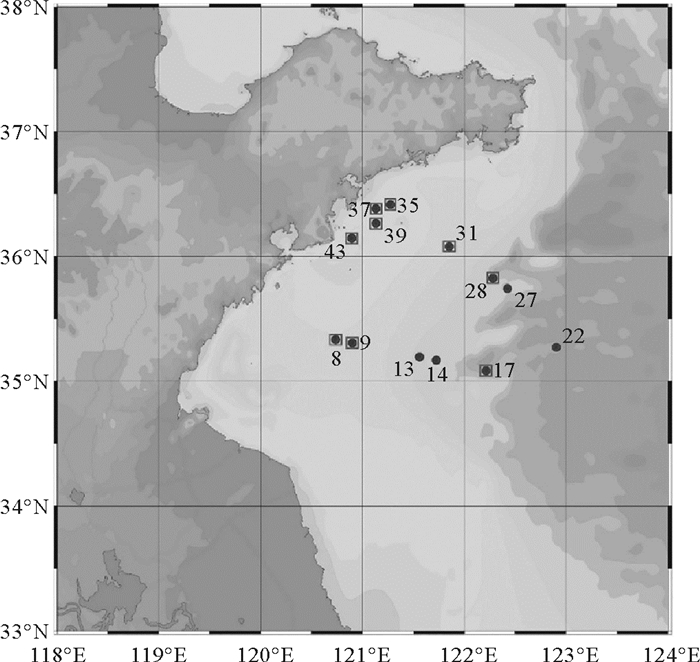
1 材料与方法 1.1 环境DNA样品采集本次采样于2020年7月27日至8月2日进行,选取南黄海西部13个采样点进行环境DNA采样和底拖网,各采样站位见图 1。其中,沉积物样品仅在9个站位被成功采集。
|
(图中实心圆标注的站位为采水和拖网站位,方形标注的站位为成功采集到沉积物的站位。Locations marked with solid circles are stations for collecting water and trawling, and locations marked with squares are stations for successfully collecting sediments. ) 图 1 环境DNA采样和拖网调查站位图 Fig. 1 Stations of environmental DNA sampling and trawl survey |
环境样品的采集和保存根据Zhang等人的方法进行[25]。每个采样点使用采水器采集表层水和底层水各5 L, 分别保存至已消毒的一次性塑料桶中,并按照水和保存液10∶1的比例加入500 mL的DNA保存缓冲液(Tiandz Inc.)以防止DNA降解。在每次采水之前,使用纯净水对采水器进行充分地冲洗。海底表层沉积物通过箱式采泥器进行采集,使用一次性手套收集1 g沉积物样品并装入5 mL的纯净冻存管中,并加入DNA组织保存液(Tiandz Inc.)以防止DNA降解。为了避免交叉污染,使用一次性样品密封袋包裹每个采样的容器。所有样品放入冷冻室进行避光冷冻保存直至上岸进行下一步操作。
在采集完水样和沉积物样品后,使用温盐深仪(CTD)收集了每个站位水体剖面的深度(Depth,D)和温度(Temperature,T)参数。
1.2 环境DNA样品鉴定 1.2.1 DNA提取在将样品带回实验室后,立即使用真空抽滤设备将水样过滤到滤膜(47 mm,0.2 μm)上。具体过程如下:每份水样由3个滤器进行抽滤,每个滤器抽滤1 L,抽滤结束后收集3个滤器上的滤膜并加入DNA组织保存缓冲液(Tiandz Inc.),在-80 ℃下冷冻保存。在每份样品抽滤完成后对过滤漏斗和过滤器进行充分的高温洗涤和消毒。为了检测是否存在外源性污染,在样品抽滤过程中设置4个阴性对照。所有样品抽滤结束后,对滤膜和沉积物样品进行DNA提取。通过CTAB法提取滤膜上的DNA,每份样品单独提取。使用MagPure Soil DNA LQ Kit试剂盒从每个冻存管中提取沉积物中的DNA。提取的DNA快速置于-20 ℃下冷冻保存。
1.2.2 12S rRNA基因扩增子测序使用MiFish-U引物[26](F:5′-GTCTAACTCGCGCCGC-3′,R:5′-CATAGGGTATCTAATCCATCTGG-3′)来扩增样品中的鱼类DNA。通过两次PCR程序生成扩增子。第一次PCR使用两个通用引物对进行,总反应体积为12.0 μL,包括6.0 μL的2×KAPA HiFi Hot Start Ready Mix,正反引物各3.6 pmol,1.0 μL模板DNA和水。第一次PCR扩增程序为:95 ℃预变性3 min;35个循环包括:98 ℃变性20 s,65 ℃退火15 s,72 ℃延伸15 s;最后72 ℃延伸5 min。将第一次PCR扩增后的产物用纯水稀释至原浓度的1/10,并作为模板用于接下来的PCR。第二次PCR是将MiSeq适配体序列和8 bp索引序列添加到两个扩增子末端,总反应体积是12.0 μL,包括6.0 μL的2×KAPA HiFi Hot Start Ready Mix,3.6 pmol的正向引物和反向引物,1.0 μL的模板DNA和水。第二次PCR扩增程序为:95 ℃预变性3 min;12个循环包括:98 ℃变性20 s,72 ℃延伸30 s;最后72 ℃延伸5 min。在单独的量化步骤之后,扩增子被汇集成等量,并使用Illlumina MiSeq平台和MiSeq Reagent Kit v2(Illumina, San Diego, CA, USA)进行双端2×150 bp测序。
1.2.3 序列处理和鱼类鉴定使用FastQC程序处理测序数据。与条形码完全匹配的原始测序读数被分配到各自的样本,并被确定为有效的序列。使用FLASH组装成对的读数。在进行嵌合体检测后,剩余的高质量序列被聚类到序列相似性为97%的操作分类单元(Operational taxonomic unit, OTU)。使用默认参数从每个OTU中选择一个代表序列,通过代表序列集与MitoFish数据库[27]对比进行物种注释。
1.3 拖网样品采集和鉴定拖网数据来自2020年7月—8月的南黄海西部海域进行的底拖网渔业资源调查。在每个站位采集环境DNA样品和环境数据后,开始进行底拖网采样。采用220 kW单拖底拖网渔船作为调查船,布设调查站位13个。拖网调查均在白天进行,每站拖网时间1 h,平均拖速3 kn。调查使用的底拖网网口宽约15 m、高约7.53 m,囊网网目为13 mm。每次拖网先挑选出大型和稀有鱼种后,根据渔获物数量按照1/4或1/8的比例进行留存,且保证渔获物中所有鱼种均被留存。调查采样按照《海洋调查规范》GB.12763.6—2007进行,渔获物样品冰鲜保存,带回实验室进行种类鉴定和生物学测定。鱼种的鉴定参照陈大刚等编著的《中国海洋鱼类》[29]进行, 获取数据主要包括各种渔获生物的丰度和生物量,采用随机取样法收集各主要种类样品,包括目标鱼种、主要经济鱼种和渔获优势种。
1.4 弱化样品污染环境DNA metabarcoding的敏感性导致了在采样和实验过程中容易出现污染问题。由于采用渔船进行采样的条件限制,在采集完水样后无稳定环境开展抽滤作业,且现场无超低温冷冻保存设备,可能导致样品之间存在污染。为弱化污染的影响在数据分析时根据Yamamoto等[30]的研究方法对数据进行了筛选:
(1) 去除了空白对照组中出现的外源性鱼种。
(2) 样品中总读数小于50的OTU被认为不存在,因此手动将这些OTU进行去除。
1.5 统计分析根据筛选后的OTU计算了每个环境DNA样品的Alpha多样性指数,其中包括:Richness、Shannon、Simpson、Pielou、Chao1和Invsimpson。Alpha多样性指数用于反映水样和沉积物中的鱼类多样性差异。
使用R软件对拖网的生物样品进行了基于物种组成的聚类分析,用于显示采样站位间的鱼类组成差异。为探讨水样和沉积物中的鱼类组成差异,本研究进行了基于Bray-curtis距离的非度量多维标度(Non-metric Multidimensional Scaling, NMDS)分析。使用SPSS 25软件进行Wilcoxon检验,用于环境DNA metabarcoding和底拖网获取的鱼类种数是否存在显著差异。通过基于Bray-curtis距离的主坐标分析(Principal Coordinates Analysis,PCoA)来比较环境DNA metabarcoding和底拖网两种方法的物种组成差异,并通过Anosim分析确定这种差异是否显著。
2 结果与分析 2.1 环境DNA metabarcoding鉴定的鱼类组成环境DNA metabarcoding在水样和沉积物样品中总共检测出45种海洋鱼类(见表 1)。其中,小黄鱼(Larimichthys polyactis,27.20%)、方氏云鳚(Pholis fangi,21.16%)、黄鮟鱇(Lophius litulon,18.67%)、日本鳀(Engraulis japonicus,8.59%)、小眼绿鳍鱼(Chelidonichthys spinosus,4.06%)占有相对较高的读数(Reads)。
|
|
表 1 环境DNA metabarcoding和拖网获得的鱼类组成 Table 1 Fish species detected by eDNA metabarcoding and trawl |
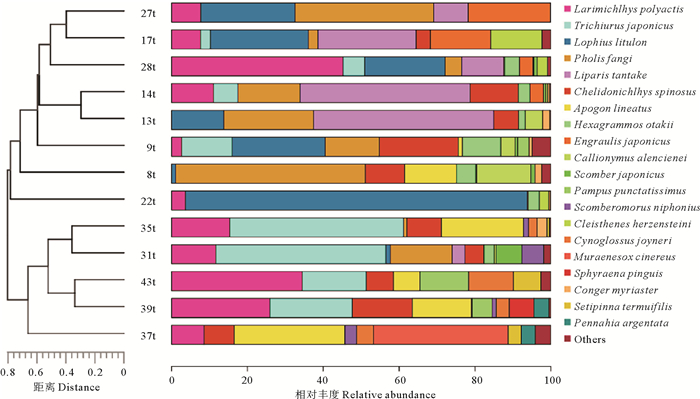
在13次底拖网调查中一共确定了32种鱼类。对底拖网数据进行基于物种组成的聚类分析,结果显示分成两支。沿岸站位(31、35、37、39、43)分成一支,离岸站位分成一支(见图 2)。在所有的拖网站位中,小黄鱼(L. polyactis,16.14%)、日本带鱼(Trichiurus japonicus,16.13%)、黄鮟鱇(Lophius litulon,12.95%)、方氏云鳚(P. fangi,12.80%)、细纹狮子鱼(Liparis tanakae,12.45%)占有较高的资源密度。
|
(“27t”表示27号站位底拖网采样,下同。The "27t" means bottom trawl sampling at station 27, other similar. ) 图 2 拖网捕获的鱼类组成 Fig. 2 Fish species caught by trawl |
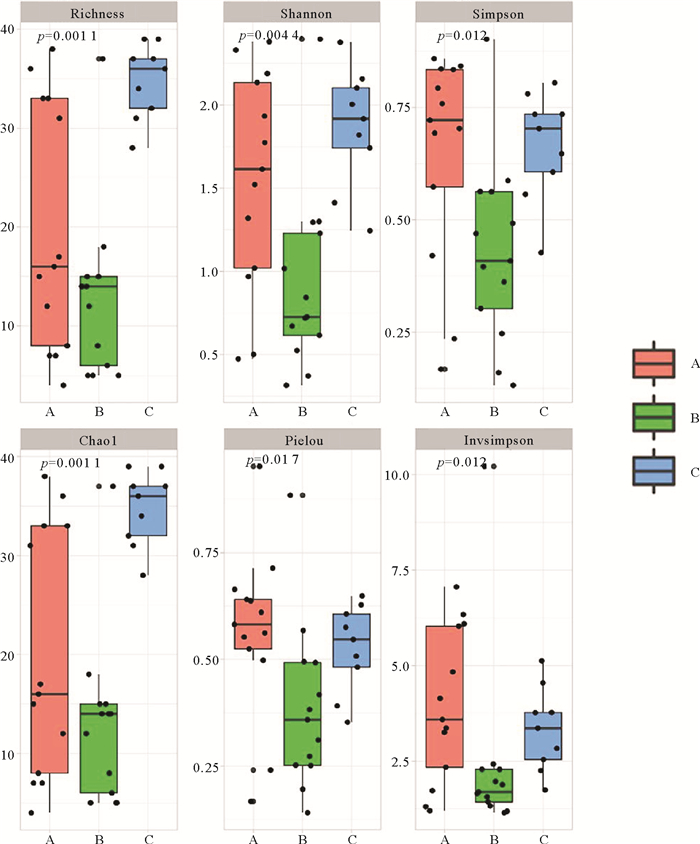
在物种组成上,表层水中检测出36种鱼类,底层水中检测出32种鱼类,沉积物中检测到36种鱼类,水样和沉积物中同时检测到的重叠鱼类有30种。Alpha多样性指数显示,沉积物样品的Richness指数、Simpson指数和Chao1指数均高于水样(见图 3)。
|
(“A”组表示表层水样,“B”组表示底层水样,“C”组表示沉积物。Group "A" represents surface water sample, group "B" represents bottom water sample, and group "C" represents sediment. ) 图 3 水性和沉积物环境DNA的Alpha多样性指数 Fig. 3 Alpha diversity index of eDNA in water and sediment |
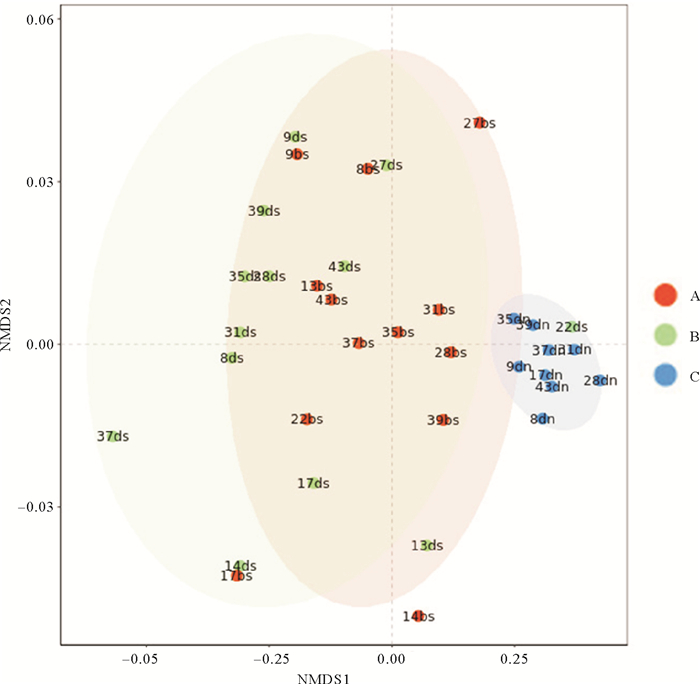
基于Bray-curtis距离的NMDS分析评估了水样和沉积物中的鱼类组成差异(见图 4)。结果显示,水样和沉积物分开聚集,沉积物样品之间的相似性更高,这说明两者之间检测到的鱼类组成存在一定差异。
|
(“A”组表示表层水样,“B”组表示底层水样,“C”组表示沉积物。Group "A" represents surface water sample, group "B" represents bottom water sample, and group "C" represents sediment. ) 图 4 基于Bray-curtis距离的鱼类组成NMDS分析 Fig. 4 NMDS analysis of fish composition based on Bray curtis distance |
环境DNA metabarcoding和底拖网调查相结合共鉴定出54种鱼类,其中环境DNA metabarcoding共检测出45种海洋鱼类。通过底拖网调查,在13个站位中共捕获32种鱼类。与底拖网捕捞的结果相比,环境DNA metabarcoding检测到的鱼种数量更多(p<0.05)。两种调查方法检测到的重叠物种有23种(见表 1)。
拖网数据的PCoA结果显示在空间上形成三个聚集,分别是沿岸站位(35、37、39、43)、中部站位(8、9、13、14、31)和深水站位(17、22、27、28)(p<0.05)。环境DNA metabarcoding的PCoA结果未出现空间上的聚集(见图 5)。
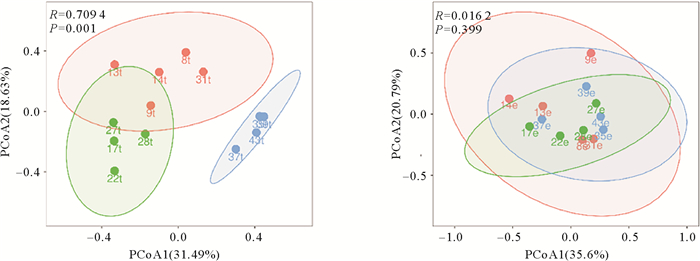
|
(“t”表示底拖网采样,“e”表示环境DNA metabarcoding采样。The "t" indicates bottom trawl sampling and "e" indicates eDNA metabarcoding sampling. ) 图 5 基于Bray-curtis距离的主坐标分析(PCoA) Fig. 5 Principal Cbuyo-ordinates Analysis (PCoA) based on Bray-curtis distance |
在本研究中,环境DNA metabarcoding从35个样本中共扩增出了1 562种鱼类序列OTU,这些OTU在注释和筛选之后共确定为45种鱼类,而底拖网调查在13个站位中共捕获了32种鱼类。环境DNA metabarcoding和底拖网调查相结合共鉴定出54种鱼类。与底拖网捕捞结果相比,环境DNA metabarcoding检测到的鱼种数量更多(p<0.05)。两种调查方法检测到的重叠物种有23种。根据底拖网数据的PCoA分析显示,沿岸站位、中部站位和深水站位出现了分开聚集,在环境DNA metabarcoding的结果中并未显示出类似的差异。造成这种情况的原因可能是环境DNA采样能够捕获整个水域深度的鱼类信息,而底拖网捕获的鱼类信息多数为底层鱼种,底层的环境差异导致了鱼类组成的差异,所以在结果中出现了不同区域的聚集现象。
3.2 鱼类空间分布在环境DNA metabarcoding中的表达水样和沉积物中检测到的鱼类组成存在部分重叠,这与Siegenthaker等[21]的研究结果相似。从Alpha多样性上来看,沉积物显示出了更高的多样性指数。有研究表明从鱼体中释放出来的游离DNA会随着沉降而富集在海底的沉积物中,从而极大地减缓降解速度[31-32]。因此,在进行环境DNA调查时,水样和沉积物同时采样能够对获取的鱼类信息进行互相补充,使调查结果能更真实地表征当地的生物多样性。
在表层和底层水样的多样性对比中,表层水的Alpha多样性指数高于底层水。在物种组成上,表层和底层检测到的鱼种存在部分重叠,这可能反映了游离的环境DNA在垂直方向上存在混合。有研究表明,鱼类在释放出环境DNA片段后,大多数环境DNA颗粒太大(大于1 μm),无法保持悬浮状态,因此会被重力向下输送[31, 33-34]。但伴随着潮流混合、人为因素以及浮游生物和部分鱼类垂直迁移的习性,这些底部的环境DNA会发生向上输送的再悬浮[35-36]。在环境DNA me-tabarcoding调查中,底层水样出现了生活在中上层的鱼类,比如日本鳀。而像黄鮟鱇这种底栖鱼类同样出现在表层水样的结果中。在夏季,由于黄海冷水团的影响,远岸部分站位底层温度较低(见图 6),与表层相比出现较大的温差。这种环境DNA在垂直方向上混合的现象表明,温度分层不会影响游离的环境DNA在陆架海域垂直方向上的传输。这与Jeunen等[23]的研究结果相似,由于垂直方向的水体混合,在海洋层内的表、底采样深度上仍然能够检测到相似数量的底栖物种和游泳物种。因此,在有水体分层的海洋环境中,需要建立更合理的采样策略以提高结果的准确性。
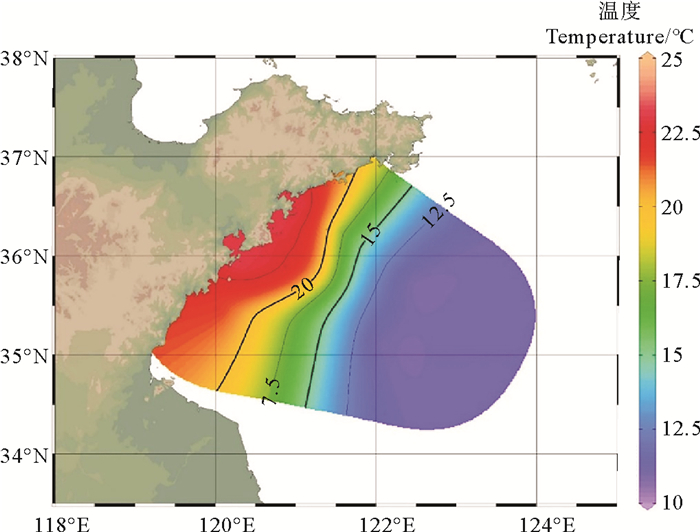
|
图 6 采样区域的底层温度 Fig. 6 Sea bottom temperature of the sampling area |
由于参考序列数据库不完整以及线粒体DNA序列本身的限制,对于某些鱼种和分类群,传统拖网调查可能比环境DNA metabarcoding的表现更好[37]。例如在底拖网采样的结果中,有部分鱼种未被环境DNA metabarcoding发现。在本研究中,部分OTU仅被注释到科级以上的分类水平,这使得环境DNA结果难以与拖网结果进行匹配,为了更直观地对比两种调查方法的结果,这部分OTU没有加入到与拖网调查的比较中。此外,某些海水硬骨鱼类线粒体DNA序列并不保守,具有高度的同源性,对于这些鱼种仅仅通过环境DNA metabarcoding难以区分,例如鲽形目、平鲉属、东方鲀属、黑棘鲷和真鲷等[26, 38-39]。未来的研究急需解决这些问题,尽可能避免环境DNA metabarcoding在资源调查方面产生的局限。
3.4 渔业资源调查的有效补充以目前的研究进展来看,环境DNA metabarcoding技术为传统的渔业资源评估模式提供了一种新思路。环境DNA metabarcoding以其独特的技术优势,在入侵物种和稀有物种监测、生物多样性调查和生物量评估预测等研究中发挥了巨大作用。此外,环境DNA metabarcoding对种群的生活史过程更敏感,能够分析鱼类营养结构、预测鱼类生活史时期、评估鱼类迁移模式等。有研究表明,鱼类在某个生活史阶段或某个生命活动中会释放出异常数量的环境DNA,根据这一特性可以预估种群的生活史时期[40]。Takeuchi等利用特异性引物通过环境DNA metabarcoding识别了北太平洋西部日本鳗鱼(Anguilla japonica)的产卵场[41]。Tabassum等根据来自罗斯海地区7个栖息地的粪便DNA metabarcoding和定量PCR(qPCR)分析了阿德利企鹅(Pygoscelis adeliae)的摄食[42];Tsuji等通过环境DNA分析以一种非侵入性的方式量化了产卵活动[40]。这些研究对于预测海洋生态系统功能以及对渔业资源的管理和保护都具有重要意义。
出于渔业管理目的,种群评估主要依赖于在大空间尺度上整合多种拖网的捕捞量数据,这种调查需要耗费大量财力、物力和时间[43]。相比于传统的拖网调查,环境DNA metabarcoding分析提供了使用标准化方法短时、高效地调查生物多样性的机会,从而减少对专业分类学鉴定人员的依赖[25, 44];本研究显示环境DNA技术比底拖网调查能更准确、更全面地表征生物多样,通过传统底拖网调查无法采集到的鱼类,也能在环境DNA数据中得以体现。在传统拖网调查的基础上,环境DNA metabarcoding的加入有助于海洋生态系统的监测,增强对不同空间尺度上海洋生物分布和多样性的调查能力。沉积物样品中的鱼类DNA信息是不可忽视的,在运用环境DNA metabarcoding调查的过程中,加入对沉积物样品的采集和分析能够更准确地反映研究区域的物种组成和多样性。此外,不同季节连续的环境DNA调查不仅能够监测生物多样性和分布等方面的内容,同时还能够建立基于环境DNA技术的渔业资源评估模式,获取种群的季度变化和年际变化信息,为种群的定量评估提供重要依据。
4 结语生物多样性的有效监测对于海洋生态系统的管理和保护具有重要意义。环境DNA metabarcoding作为一种无入侵性且灵敏度高的调查方法能够有效弥补传统渔业资源调查的不足和弊端。在本研究中,通过采集水样、沉积物环境DNA和底拖网生物样品,比较了环境DNA metabarcoding和底拖网这两种调查方法监测鱼类生物多样性的能力,评估了环境DNA metabarcoding获取鱼类生物多样性和空间分布的效果。研究结果表明,环境DNA metabarcoding比拖网调查能捕获更多的鱼类组成信息,但两种方法检测到的鱼类组成存在部分重叠。因此,环境DNA metabarcoding可以作为传统拖网调查的一种补充工具,为渔业管理和保护提供依据。
致谢: 感谢曹畅博士在样品采集过程中提供的帮助;感谢王业辉博士、宋娜老师对文章内容提出的宝贵意见。
| [1] |
Jackson J B C, Kirby M X, Berger W H, et al. Historical overfishing and the recent collapse of coastal ecosystems[J]. Sciense, 2001, 293(5530): 629-637. DOI:10.1126/science.1059199 (  0) 0) |
| [2] |
Somero G N, Beers J M, Chan F, et al. What changes in the carbonate system, oxygen, and temperature portend for the Northeastern Pacific Ocean: A physiological perspective[J]. BioScience, 2016, 66(1): 14-26. DOI:10.1093/biosci/biv162 (  0) 0) |
| [3] |
Lubchenco J, Gropud-colvert K. Making waves: The science and politics of ocean protection[J]. Science, 2015, 350(6259): 382-383. DOI:10.1126/science.aad5443 (  0) 0) |
| [4] |
熊满堂, 王普泽, 叶少文, 等. 丹江口水库浮游植物群落时空特征及鱼产力评估[J]. 中国水产科学, 2021, 28(6): 715-727. Xiong M T, Wang P Z, Ye S W, et al. Spatio-temporal characteristics of the phytoplankton community and assessment of fish productivity in the Danjiangkou Reservoir, the water source for the South-to-North Water Diversion Project, China[J]. Journal of Fishery Sciences of China, 2021, 28(6): 715-727. (  0) 0) |
| [5] |
Sakuma K M, Field J C, Mantua N J, et al. Anomalous epipelagic micronekton assemblage patterns in the neritic waters of the California current in spring 2015 during a period of extreme ocean conditions[J]. California Cooperative Oceanic Fisheries Investigations Reports, 2016, 57: 164-183. (  0) 0) |
| [6] |
Stoeckle B C, Kuehn R, Geist J. Environmental DNA as a monitoring tool for the endangered freshwater pearl mussel (Margaritifera margaritifera L.): A substitute for classical monitoring approaches?[J]. Aquatic Conservation: Marine and Freshwater Ecosystems, 2016, 26(6): 1120-1129. DOI:10.1002/aqc.2611 (  0) 0) |
| [7] |
秦传新, 左涛, 于刚, 等. 环境DNA在水生生态系统生物量评估中的研究进展[J]. 南方水产科学, 2020, 16(5): 123-128. Qin C X, Zuo T, Yu G, et al. Advances in research of environmental DNA (eDNA) in biomassassessment of aquatic ecosystems[J]. South China Fisheries Science, 2020, 16(5): 123-128. (  0) 0) |
| [8] |
单秀娟, 李苗, 王伟继. 环境DNA(eDNA)技术在水生生态系统中的应用研究进展[J]. 渔业科学进展, 2018, 39(3): 23-29. Shan X J, Li M, Wang W J. Application of environmental DNA technology in aquatic ecosystem[J]. Progress in Fishery Sciences, 2018, 39(3): 23-29. DOI:10.19663/j.issn2095-9869.20171025001 (  0) 0) |
| [9] |
高天翔, 陈治, 王晓艳. 近海鱼类多样性调查新方法——环境DNA分析技术[J]. 浙江海洋大学学报(自然科学版), 2018, 37(1): 1-7. Gao T X, Chen Z, Wang X Y. Environmental DNA, a new method for fish diversity investigation in the coastal waters[J]. Jounral of Zhejiang Ocean University(Natural Science), 2018, 37(1): 1-7. DOI:10.3969/j.issn.1008-830X.2018.01.001 (  0) 0) |
| [10] |
姜维, 赵虎, 邓捷, 等. 环境DNA分析技术——一种水生生物调查新方法[J]. 水生态学杂志, 2016, 37(5): 1-7. Jiang W, Zhao H, Deng J, et al. Detection of aquatic species using environmental DNA[J]. Jounral of Hydroecology, 2016, 37(5): 1-7. DOI:10.15928/j.1674-3075.2016.05.001 (  0) 0) |
| [11] |
Turner C R, Miller D J, Coyne K J, et al. Improved methods for capture, extraction, and quantitative assay of environmental DNA from Asian bigheaded carp(Hypophthalmichthys spp.)[J]. PLoS One, 2014, 9(12): e114329. DOI:10.1371/journal.pone.0114329 (  0) 0) |
| [12] |
Davy C M, Kidd A G, Wilson C C. Development and validation of environmental DNA (eDNA) markers for detection of freshwater turtles[J]. PLoS One, 2015, 10(7): e0130965. DOI:10.1371/journal.pone.0130965 (  0) 0) |
| [13] |
Ficetola G F, Manenti R, Taberlet P. Environmental DNA and metabarcoding for the study of amphibians and reptiles: Species distribution, the microbiome, and much more[J]. Amphibia-Reptilia, 2019, 40(2): 129-148. DOI:10.1163/15685381-20191194 (  0) 0) |
| [14] |
Itakura H, Wakiya R, Yamamoto S, et al. Environmental DNA analysis reveals the spatial distribution, abundance, and biomass of Japanese eels at the river-basin scale[J]. Aquatic Conservation: Marine and Freshwater Ecosystems, 2019, 29(3): 361-373. DOI:10.1002/aqc.3058 (  0) 0) |
| [15] |
Closek C J, Santora J A, Starks H A, et al. Marine vertebrate biodiversity and distribution within the central california current using environmental DNA (eDNA) metabarcoding and ecosystem surveys[J]. Frontiers in Marine Science, 2019, 6: 732. DOI:10.3389/fmars.2019.00732 (  0) 0) |
| [16] |
Aglieri G, Baillie C, Mariani S, et al. Environmental DNA effectively captures functional diversity of coastal fish communities[J]. Molecular Ecology, 2021, 30(13): 3127-3139. DOI:10.1111/mec.15661 (  0) 0) |
| [17] |
舒璐, 林佳艳, 徐源, 等. 基于环境DNA宏条形码的洱海鱼类多样性研究[J]. 水生生物学报, 2020, 44(5): 1080-1086. Shu L, Lin J Y, Xu Y, et al. Investigating the fish diversity in erhai lake based on environmental DNA metabarcoding[J]. Acta Hydrobiologica Sinica, 2020, 44(5): 1080-1086. (  0) 0) |
| [18] |
陈炼, 吴琳, 刘燕, 等. 环境DNA metabarcoding及其在生态学研究中的应用[J]. 生态学报, 2016, 36(15): 4573-4582. Chen L, Wu L, Liu Y, et al. Application of environmental DNA metabarcoding in ecology[J]. Acta Ecologica Sinica, 2016, 36(15): 4573-4582. (  0) 0) |
| [19] |
Maggi F. The settling velocity of mineral, biomineral, and biological particles and aggregates in water[J]. Journal of Geophysical Research: Oceans, 2013, 118(4): 2118-2132. DOI:10.1002/jgrc.20086 (  0) 0) |
| [20] |
Shaw J L A, Clarke L J, Wedderburn S D, et al. Comparison of environmental DNA metabarcoding and conventional fish survey methods in a river system[J]. Biological Conservation, 2016, 197: 131-138. DOI:10.1016/j.biocon.2016.03.010 (  0) 0) |
| [21] |
Siegenthaler A, Wangensteen O S, Soto A Z, et al. Metabarcoding of shrimp stomach content: Harnessing a natural sampler for fish biodiversity monitoring[J]. Molecular Ecology Resources, 2019, 19(1): 206-220. DOI:10.1111/1755-0998.12956 (  0) 0) |
| [22] |
Andruszkiewicz E A, Koseff J R, Fringer O B, et al. Modeling environmental DNA transport in the coastal ocean using Lagrangian particle tracking[J]. Frontiers in Marine Science, 2019, 6: 477. DOI:10.3389/fmars.2019.00477 (  0) 0) |
| [23] |
Jeunen G J, Lamare M D, Knapp M, et al. Water stratification in the marine biome restricts vertical environmental DNA (eDNA) signal dispersal[J]. Environmental DNA, 2019, 2(1): 99-111. (  0) 0) |
| [24] |
Li J C, Jiang F, Wu R, et al. Tidally induced temporal variations in growth of young-of-the-year pacific cod in the Yellow Sea[J]. Journal of Geophysical Research: Oceans, 2021, 126(6): 1-16. (  0) 0) |
| [25] |
Zhang H, Yoshizawa S, Iwasaki W, et al. Seasonal fish assemblage structure using environmental DNA in the Yangtze Estuary and its adjacent waters[J]. Frontiers in Marine Science, 2019, 6: 10. DOI:10.3389/fmars.2019.00010 (  0) 0) |
| [26] |
Miya M, Sato Y, Fukunaga T, et al. MiFish, a set of universal PCR primers for metabarcoding environmental DNA from fishes: Detection of more than 230 subtropical marine species[J]. Royal Society Open Science, 2015, 2(7): 150088. DOI:10.1098/rsos.150088 (  0) 0) |
| [27] |
Iwasaki W, Fukunaga T, Isagozawa R, et al. MitoFish and MitoAnnotator: A mitochondrial genome database of fish with an accurate and automatic annotation pipeline[J]. Molecular Biology and Evolution, 2013, 30(11): 2531-2540. DOI:10.1093/molbev/mst141 (  0) 0) |
| [28] |
自然资源部第三海洋研究所. 海洋调查规范第6部分: 海洋生物调查: GB/T 12763.6—2007[S]. 北京: 中国标准出版社, 2007: 172. Third Institute of Oceanography, Ministry of Natural Resources. Specifications for Oceanographic Survey Part 6: Marine Biological Survey: GB/T 12763.6—2007[S]. Beijing: Standards Press of China, 2007: 172. (  0) 0) |
| [29] |
陈大刚, 张美昭. 中国海洋鱼类[M]. 青岛: 中国海洋大学出版社, 2015. Chen D G, Zhang M Z. Chinese Marine Fish[M]. Qingdao: China Ocean University Press, 2015. (  0) 0) |
| [30] |
Yamamoto S, Masuda R, Sato Y, et al. Environmental DNA metabarcoding reveals local fish communities in a species-rich coastal sea[J]. Scientific Reports, 2017, 7: 40368. DOI:10.1038/srep40368 (  0) 0) |
| [31] |
Pilliod D S, Goldberg C S, Arkle R S, et al. Factors influencing detection of eDNA from a stream-dwelling amphibian[J]. Molecular Ecology Resources, 2014, 14(1): 109-116. DOI:10.1111/1755-0998.12159 (  0) 0) |
| [32] |
Sakata M K, Yamamoto S, Gotoh R O, et al. Sedimentary eDNA provides different information on timescale and fish species composition compared with aqueous eDNA[J]. Environmental DNA, 2020, 2(4): 505-518. DOI:10.1002/edn3.75 (  0) 0) |
| [33] |
Hansen B K, Bekkevold D, Clausen L W, et al. The sceptical optimist: Challenges and perspectives for the application of environmental DNA in marine fisheries[J]. Fish and Fisheries, 2018, 19(5): 751-768. DOI:10.1111/faf.12286 (  0) 0) |
| [34] |
Barnes M A, Turner C R, Jerde C L, et al. Environmental conditions influence eDNA persistence in aquatic systems[J]. Environmental Science & Technology, 2014, 48(3): 1819-1827. (  0) 0) |
| [35] |
Holliland P B, Ahlbeck I, Westlund E, et al. Ontogenetic and seasonal changes in diel vertical migration amplitude of the calanoid copepods Eurytemora affinis and Acartia spp. in a coastal area of the northern Baltic proper[J]. Journal of Plankton Research, 2012, 34(4): 298-307. DOI:10.1093/plankt/fbs001 (  0) 0) |
| [36] |
Hafker N S, Meyer B, Last K S, et al. Circadian clock involvement in zooplankton diel vertical migration[J]. Current Biology, 2017, 27(14): 2194-2201. DOI:10.1016/j.cub.2017.06.025 (  0) 0) |
| [37] |
Liu S L, Yang C T, Zhou C R, et al. Filling reference gaps via assembling DNA barcodes using high-throughput sequencing-moving toward barcoding the world[J]. Gigascience, 2017, 6(12): 1-8. (  0) 0) |
| [38] |
Wang Y H, Duan J N, Shi H L, et al. Species identification of small fish in Xixuan Island coastal waters of Zhoushan using DNA barcoding[J]. Journal of Applied Ichthyology, 2020, 36(1): 75-84. DOI:10.1111/jai.13995 (  0) 0) |
| [39] |
Zhu F, Zhang Z, Chen S, et al. The complete mitochondrial genome of the hybrid of Pagrus major (♀)×Acanthopagrus schlegelii (♂)[J]. Mitochondrial DNA Part A, 2016, 27(4): 2980-2981. DOI:10.3109/19401736.2015.1060471 (  0) 0) |
| [40] |
Tsuji S, Shibata N. Identifying spawning activity in aquatic species based on environmental DNA spikes[J]. Environmental DNA, 2021, 3(1): 190-199. (  0) 0) |
| [41] |
Takeuchi A, Watanabe S, Yamamoto S, et al. First use of oceanic environmental DNA to study the spawning ecology of the Japanese eel Anguilla japonica[J]. Marine Ecology Progress Series, 2019, 609: 187-196. (  0) 0) |
| [42] |
Tabassum N, Lee J H, Lee S R, et al. Molecular diet analysis of Adelie penguins (Pygoscelis adeliae) in the Ross Sea using fecal DNA[J]. Biology-Basel, 2022, 11(2): 182. (  0) 0) |
| [43] |
马鸿娟, Kathryn S, 马利民, 等. 环境DNA及其在水生生态系统保护中的应用[J]. 生态学杂志, 2016, 35(2): 516-523. Ma H J, Kathryn S, Ma L M, et al. Environmental DNA and its application in protecting aquatic ecosystems[J]. Chinese Journal of Ecology, 2016, 35(2): 516-523. (  0) 0) |
| [44] |
Cowart D A, Matabos M, Brandt M I, et al. Exploring environmental DNA (eDNA) to assess biodiversity of hard substratum faunal communities on the lucky strike vent field (mid-atlantic ridge) and investigate recolonization dynamics after an induced disturbance[J]. Frontiers in Marine Science, 2020, 6(783): 21. (  0) 0) |
2. Laboratory for Marine Fisheries Science and Food Production Processes, Pilot National Laboratory for Marine Science and Technology(Qingdao), Qingdao 266237, China;
3. Rizhao Shan Hai Tian Tourist Resort Fishery Supervision and Management Station, Rizhao 276800, China
 2023, Vol. 53
2023, Vol. 53


