2. 青岛海洋科学与技术试点国家实验室,海洋生物学与生物技术功能实验室,山东 青岛 266237;
3. 青岛海洋科学与技术试点国家实验室,海洋渔业科学与食物产出过程功能实验室,山东 青岛 266237;
4. 中国海洋大学三亚海洋研究院,热带海洋生物种质资源开发与种业工程实验室,海南 三亚 572000
随着现代分子生物学技术的快速发展,分子育种技术已成为遗传改良的重要手段[1]。全基因组选择育种[2]利用全基因组范围的遗传标记计算基因组育种值,在分子水平对优良品质进行选育,大大加快了育种进程,已成为动植物育种领域的一大研究热点。近年来,作为动植物中开展全基因组选择育种工作的重要技术支撑,高通量基因分型技术呈迅猛发展势头。随着二代测序(Next generation sequencing, NGS)平台的建立及测序成本的不断降低,全基因组重测序技术已成为动植物群体遗传学及育种研究的有效方法,但处理大量样品且物种基因组较大时费用仍然是非常昂贵的[3]。简化基因组测序技术[4-7]通过限制性内切酶酶切基因组DNA,降低基因组复杂度,实现基因组内低成本大规模单核苷酸多态性(Single nucleotide polymorphisms, SNPs)标记的分型[8]。但此类技术仅限于酶切标签上位点的检测,使其更适用于全基因组范围的非靶向SNPs标记开发和应用。然而基因相关位点,如位于候选基因或候选基因附近的SNP位点,可能与表型性状变异有更大关联,在全基因组选择育种中具有更为重要的价值。随着越来越多物种基因组信息的获取及丰富的SNP标记资源的积累,对靶向位点分型可以更好地实现全基因组选择育种的研究需求。
基因芯片技术是伴随着“人类基因组计划”发展而来的技术,其最早应用于DNA的测序。该技术基于核酸杂交的基本原理,通过检测芯片中固定的已知探针堆与待测样品的杂交谱重组出样品序列[9]。基因芯片技术广泛应用于农畜牧业的遗传育种研究中,如群体进化分析[10]、优良性状基因的挖掘[11]、育种值的估算[12]等。目前在牛、猪、鸡、山羊、绵羊等家畜以及水稻、玉米、马铃薯、小麦等植物中,相应的基因芯片都已被大量开发,这极大推动了相关育种工作。但对非模式生物如扇贝等水产生物而言,芯片开发的研究相对较少,主要由于其缺乏商业化芯片,芯片的定制和生产费用比较昂贵[13]。其次芯片技术位点固定[13],位点选择灵活性不高,无法满足个性化要求。最后芯片技术所需的昂贵的专用仪器也是限制其推广应用的一大原因[14]。液相分子杂交技术HD-Marker(High-throughput, sequencing-based GoldenGate approach)可在单个管内同时实现1万多个靶位点的基因分型[15],近期已提高其通量至86 000。该技术通过杂交-延伸-连接等反应获取靶位点的碱基信息,后续通过PCR扩增将捕获后的完整序列富集,然后进行高通量测序。HD-Marker将液相杂交技术的位点选择灵活性和高通量测序技术通量高、成本低的优势有效结合,无需固化探针,可自由选择覆盖基因组几百至几万个标记位点进行靶向分型,为非模式生物的低成本靶位点基因分型提供了一种兼容不同通量级别的理想技术手段[15]。但育种工作者仍然面临着短期内需快速完成大批量样本基因分型工作的挑战。自动化是解决大批量样本基因分型过程中手工操作耗时耗力问题的有效途径。Hamilton、Eppendorf、Tecan、Beckman等几家主要的工作站生产厂家不仅在核酸提取、药物筛选等应用上提供全自动化的解决方案,还可以搭建自动化文库构建平台,且它们的自动化移液工作站在法医学、临床以及NGS服务平台等实验室获得了广泛的应用[16-18]。
本研究旨在建立标准化的、自动化的、高效高通量的HD-Marker文库构建流程,以满足短时快速对大规模样本并行基因分型的需求。本研究依托自动化移液系统(Freedom EVO 100-8),根据HD-Marker技术的流程按照实验需求配置了工作台的桌面,测定和调节了重要移液参数并且编写了自动化实验程序,并以虾夷扇贝(Patinopecten yessoensis)100个位点的探针池为原料,从扩增效果均匀性、实验成功率、文库捕获率及分型准确性等方面验证了自动化文库构建流程的可靠性。
1 材料与方法 1.1 实验材料自动化移液系统Freedom EVO 100-8;真空抽滤模块,包括真空抽滤器主机、真空抽滤模块底座、真空抽滤96孔板适配器; 4位加样针载架(可回放至深孔板结构的吸头盒中); 4位微板载架。主要耗材包括96孔PCR板(生工生物工程股份有限公司,货号F600960-0010)、1.5 mL EP管(青岛赛尚科贸有限公司,货号LXGHY-15)、100 mL Through试剂槽(Tecan,货号10613049)、12通道试剂槽(生工生物工程股份有限公司,货号F619714-0001)。
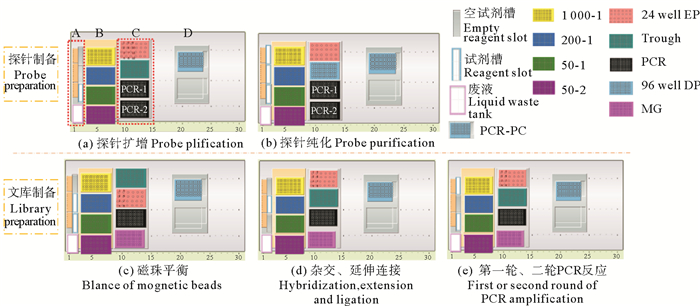
1.2 实验方法 1.2.1 自动化文库构建工作台的桌面配置桌面配置主要涉及原料槽、枪头以及样品PCR管等的合理摆放。应遵循的基本原则为:(1)使步骤之间板位的放置协同性尽可能高,无需对桌面布局产生较大的更改; (2)使加样机械臂的移液操作基本遵循最少路径,提高移液的效率。图 1所示根据HD-Marker实验流程确立的几个主要实验步骤的桌面布局。桌面布局主要由四大部分组成,从左到右依次为:第一部分(A)为100 mL试剂槽组,共有3个位置,由上而下标记为A1~A3,用于存放用量较大的原料; 第二部分(B)为加样针载架,共有4个位置,由上而下标记为B1~B4,用于存放大中小三种规格的枪头盒; 第三部分(C)为试剂配制区域,共有4个位置,由上而下标记为C1~C4,用于放置EP管原料、母液配置试剂槽以及96孔PCR管等; 第四部分(D)为用于PCR产物纯化的组件,主要配备真空抽滤96孔板适配器。在自动化流程中,主要针对第一部分(A)和第三部分(C)的桌面进行更改。
|
(A~D:分别代表桌面布局的第一至四部分; 1 000/200-1:1 000/200 μL的加样针载架; 50-1和50-2:50 μL的加样针载架1和2;24 well EP:24孔离心管架; Trough:母液试剂配置槽; PCR:96孔PCR板; 96 well DP:96深孔板; MG:磁力架; Filter:PCR产物纯化的组件) 图 1 主要实验步骤的桌面布局 Fig. 1 Layout of the desktop for main steps |
(1) 自动化液体处理系统吸放液针头的标准空间定位。为了使8通道加样机械臂能够准确的探测到液面的位置或者PCR管、EP管的底部,必须对使用的耗材进行空间定位,确定位置参数X、Y、Z。需要进行定位的耗材主要包括96孔PCR板(规格:200 μL)、12通道试剂槽、100 mL试剂槽、磁力架上放置的96孔PCR板(规格: 100 mL)。主要步骤如下,加载枪头后将机械臂移至96孔PCR板的1号管上方,使用微调功能调节针头至96孔板A排1列空位的正中间,将针头下探到管底上方1~2 mm的高度获得合适的Z轴坐标后,单击“ < ”使当前调节的值覆盖原来的Z轴坐标值,与X、Y轴的调节以及其他耗材的调节也遵循以上的方式。
(2) 移液参数的调节。为了使移液操作精确,液体处理的配置设置也需要反复进行优化和调节。其中主要调节的部分包括:吸液速度和喷液速度、延迟时间及浸入液面的高度等。
(3) 容器吸液安全余量的确定。通常为了保证所吸取的液体足量,吸液会设置在Zmax位置-2 mm左右的距离,用自动化液体处理系统吸取定量液体后,测定管内剩余的2 mm安全距离内剩余的体积即为安全余量,不同的耗材于2 mm距离处的安全余量分别进行测定。
(4) 移液准确度和精度的测定。采用去离子水作为移液液体,并用天平称量的方式对自动化液体处理系统的移液精准度进行测定。环境温度、水温均为20 ℃,电子天平内相对湿度60%~90%。测定从试剂槽中吸取定量体积到96孔板之间的移液精准度。利用自动化液体处理系统进行5、10、100、1 000 μL 4个体积量的移液操作,并称重,重复15次。
1.2.3 自动化探针制备方法的建立(1) 探针扩增流程的建立。通过一次流程,实现192管PCR母液配置及分液操作,10 min内完成操作。整个实验过程中涉及到的原料管板位放置及体系如下(×8代表 8通道的枪头,×12代表循环12次)。
① PCR反应所需要的H2O放置在100 mL试剂槽(图 1标记为试剂槽); Oligo模板、引物Fwd-Primer(10 μmol/L)、引物Rev-Primer(10 μmol/L)分别放置于24微孔板EP的1~3位,OneTaqmix(NEB)放置5~8位(见图 1)。
② 在12通道母液槽(Trough)的第一列和第二列中进行母液的配置,用平行8根枪头从100 mL试剂槽位置吸取H2O体积5 760 μL(720 μL×8),移液到第一列,循环两次,第二次喷液设置在第二列,用平行3根枪头分别从24 well EP的1、2、3位置分别吸取Oligo模板体积80 μL、引物Fwd- Primer体积480 μL、引物Rev-Primer体积480 μL,循环2次,步骤同上; 用平行4根枪头从24 well EP的5~8位置吸取体积OneTaqmix体积5 760 μL(900 μL×4),循环两次,步骤同上。
③ 配置好母液后进行分液操作,每次调取8根枪头从母液槽的第一列吸取母液体积480 μL(60 μL×8),分别加到96孔PCR板(图 1(a)标记为PCR-1)的第一列每孔,循环12次按列分配母液。母液槽第二列中的母液分配至第二张96孔PCR板(图 1(a)标记为PCR-2)。
④ 取出两个96孔PCR板,共计192管,贴上PCR膜后随即放入PCR仪中,完成扩增反应。
(2) 探针纯化流程的建立。待PCR反应结束后,对192管PCR产物进行纯化操作,采用的是真空抽滤法同时进行96个样品的纯化。整个实验过程中涉及到的原料管板位放置及体系如下(×8代表 8通道的枪头,×12代表循环12次)。
① 纯化操作所需要的PM缓冲液、PE缓冲液、EB洗脱液分别放置在100 mL试剂槽位置(见图 1(b)),用于样品与PM混合的收集版放置于4位微板载架的site2(图 1(b)标记为96 well DP),两份96孔PCR产物分别放置在4位微板载架的site3和site4(见图 1(b)标记为PCR-1及PCR-2)。真空抽滤96孔板适配器放置在真空抽滤模块底座的site1(图 1(b)标记为PCR-PC)。
② 产物与PM缓冲液的混匀在96 well EP中进行,首先用平行8根枪头将PCR-1中所有液体转移至96孔深孔板,PCR-1及PCR-2的每两列转移至96 well EP的同一列。
③ 用平行8根枪头转移PM缓冲液5 760 μL(360 μL×8×12)至96 well EP进行混匀操作,然后平行转移至PCRPC中,开启压力设置,施加压强至350 mmPa,计时6 min; 降压至0。再进行一次压强为600 mmPa的加压,计时3 min结束后释放掉压力,纯化柱中的液体抽滤到废液桶中。
④ 转移PE的步骤与步骤(3)类似,需要修改PE缓冲液体积为86 400 μL(900 μL×8×12),PE加到收集管以后,开启压力,压力值及时间设置同上,重复2次。
⑤ 最后一遍PE抽滤后,静置5 min,使滤膜充分干燥,此时需要手动将收集管下方放置回收管。
⑥ 转EB的步骤与步骤(3)类似,需要修改EB缓冲液的体积为5 280 μL(55 μL×8×12)。EB加到收集管静置10 min,使DNA充分的溶解以后,开启压力,压力值及时间设置同上,重复2次。
1.2.4 自动化文库制备方法的建立(1) 磁珠平衡。通过一次流程,实现96管平衡磁珠的准备,磁珠的洗涤和平衡等操作在10 min内即可完成。整个实验过程中涉及到的原料管、96孔板的放置及体系如下(×8代表 8通道的枪头,×12代表循环12次)。
① 平衡磁珠的缓冲液(0.5×SSC buffer)放置在100 mL试剂槽(图 1(c)标记为试剂槽),链霉素亲和磁珠放置在24孔EP的1~3位。96孔PCR板分别放置在4位微板载架的site3(图 1(c)标记为PCR),磁力架放置在4位微板载架的site4(图 1(c)标记为MG)。
② 调取8根枪头吸取磁珠混悬液,吸液操作之前进行吹洗操作,使磁珠呈均匀悬浮状态,保证每一次的吸取都等量,吸取磁珠体积为20 μL×8,喷液在96孔PCR板,循环12次,完成96孔的磁珠分配。
③ 磁珠分配后,利用Roma抓扳手将96孔PCR板移至磁力架上,静置5 min,使磁液分离,分离后移除上清。此处需要设置移液两次,使上清液充分移除。
④ Roma抓扳手将磁力架上的96孔PCR板移回至4位微板载架的site3位置,进行二轮磁珠清洗,后续操作步骤同上,平衡好的磁珠最终回位到site3,继续进行下一轮反应。
(2) 杂交反应。杂交反应的体系主要在平衡好的磁珠及杂交液中进行,整个实验过程中涉及到的原料管、96孔板的放置及体系如下(×8代表 8通道的枪头,×12代表循环12次)。
① 杂交液放置在100 mL试剂槽site2 (图 1(d)标记为试剂槽),制备好探针放置于4位微板载架的site2位(标记为Trough),上一步平衡好的磁珠放置在site3板位(图 1(d)标记为PCR),样品DNA保存于96孔PCR板中,放置在4位微板载架的site4位(图 1(d)标记为MG)。
② 调取8根枪头吸取杂交液体积为30 μL×8×12(×8代表 8通道的枪头,×12代表循环12次),移至site3板位96孔PCR板中。
③ 调取8根枪头吸取探针体积为10 μL×8×12,移至site3板位96孔PCR板中。
④ 调取8根枪头吸取样品DNA体积为10 μL×8×12,移至site3板位96孔PCR板中,混匀。
⑤ 将96孔板进行封膜置于PCR仪中杂交过夜。
⑥ 延伸反应。杂交反应完成后,涉及到一些上清的移除和冲洗操作与平衡磁珠的过程类似,配置延伸连接反应的母液可以参照探针扩增,其中涉及到需要更改的体积变量(×8代表 8通道的枪头,×12代表循环12次):连接缓冲液体积为2 212.8 μL(23.05 μL×8×12)、连接酶体积为120 μL(1.25 μL×8×12)、dNTP体积为24 μL(0.25 μL×8×12)。
(4) 一轮、二轮PCR。对延伸连接后的模板进行两轮PCR,可以参照探针扩增的程序编译模板,其中需要对反应体系中所涉及到的移液体积及分液体积进行修改,主要的体积变量为引物primer1体积为38.4 μL(0.4 μL×8×12)、引物primer2体积为38.4 μL(0.4 μL×8×12)、dNTP体积为115.2 μL(1.2 μL×8×12)、Phusion酶体积为38.4 μL(0.4 μL×8×12)、H2O体积为38.4 μL(0.4 μL×8×12)。完成母液分配后,将96孔板进行封膜置于PCR仪中进行扩增。富集完成的产物参照探针纯化流程进行纯化,后随机选取8个文库根据Qubit定量结果将样品等量混合后使用Illumina Hiseq X-ten平台(PE150)进行高通量测序。
2 结果与分析 2.1 HD-Marker文库自动化构建流程的搭建 2.1.1 吸放液针标准空间定位校准及移液参数的调节通过细致定位和微调,样品容器的水平位置及Z轴相关参数都已建立(见表 1),确保了枪头能够在Ztravel的参数范围内平稳无障碍的移动。对Z参数的设置除了包括Ztravel(Z移动),还包括Ztravel和Zstart的调节。其中ZDispense定位校准的作用是保证喷液时液体能够准确的打入目标孔位,但又不会使液体溅入到相邻孔位,通常在该位置管边缘向下3~5 mm处。另外Zstart的位置是配合页面探测功能使用的,即从该位置启用页面探针功能,吸液时根据页面的高度同步下降,有效的避免枪头外壁挂载液体造成浪费,此类吸液模式适用于内切酶、聚合酶等黏稠并且成本较高的试剂。其他的吸液操作多采用Zmax位置吸液,这样能够有效的保证吸取的液体足量,并且减少由于探测模式产生的误报情况。
|
|
表 1 自动化液体处理系统针头位置定位校准结果 Table 1 Needle position correction result of automated liquid handling system |
每次移液所涉及到的吸液和喷液参数基本都有其特定的标准,该参数的设置与移液体积、液体质地、吸液喷液的位置等都息息相关。总体原则是:(1)基本1 s内完成吸液,对于体积较小的试剂,一般不宜太快; 经测试对于10 μL(最小吸液体积)体积来说,吸液速度一般不宜超过40 μL/s; (2)吸入液体后需要有一定的等待延迟时间,通常为300~1 000 ms,如果吸取黏稠的液体需要适当的延长等待时间,保证吸取的液体足量; (3)喷液速度的设置通常在400~600 μL/s,平稳有效的喷液速度的调试非常重要,如果喷液速度过慢,产生的压力不足以释放掉枪头内所有液体会造成移液的不精准,但如果喷液速度过快容易产生液体的喷溅造成污染; (4)不同液体质地其吸液和喷液需要根据具体情况采取合适的吸液高度。
2.1.2 各容器液体吸液的安全余量测定尽管Tecan自动化液体处理系统在吸液时可以选择页面探测模式或者吸液页面跟随的模式,但实际操作中面对如酶、SDS等黏稠或者容易起泡的液体时并不灵敏,容易产生报错。因此在吸取液体时,多数情况会选择在最低下探高度(Zmax)的位置进行吸液,而此时若液体不足会影响反应体系的准确性。为保证吸取液体足量准确,对不同液体装载容器进行安全余量的测定是至关重要的。经测试,1.5 mL EP管,最低安全余量25 μL; 12孔母液槽中,单槽最低安全余量为300 μL; 母液试剂配置槽100 mL,最低安全余量3 mL; 96深孔板等用于液体全部转移时,无需测量吸液安全余量。
2.1.3 移液精准度测定为了评价移液操作的可靠性,本文作者对5、10、100和1 000 μL的移液体积进行了准确度和精度的检测。其中移液体积为1 000 μL时,各加样针组间相对误差范围在0.1%~0.9%之间,变异系数平均为0.86%;移液体积为100 μL时,各加样针组间相对误差范围在2.0%~5.6%之间,变异系数平均为4.19%;移液体积为10 μL时,各加样针组间相对误差范围在2.2%~4.7%之间,变异系数平均为5.43%;移液体积为5 μL时,各加样针组间相对误差范围在1.2%~6.2%之间,变异系数平均为8.09%。结果显示,Freedom EVO 100-8自动化液体处理系统对于10 μL以上的移液体积的处理,其准确度及精密度都较为理想。但在5 μL体积下,其整体准确度及精密度稍显不足。因此在后续进行实验步骤设置时,会充分考虑到精准移液的体积限制,尽可能使移液体积均控制在10 μL以上。表 2所示的是自动化液体处理系统的8个加样针的移液相对误差和变异系数。
|
|
表 2 自动化液体处理系统移液准确度和精密度 Table 2 Accuracy and precision of fluid transfer using automated liquid handling system |
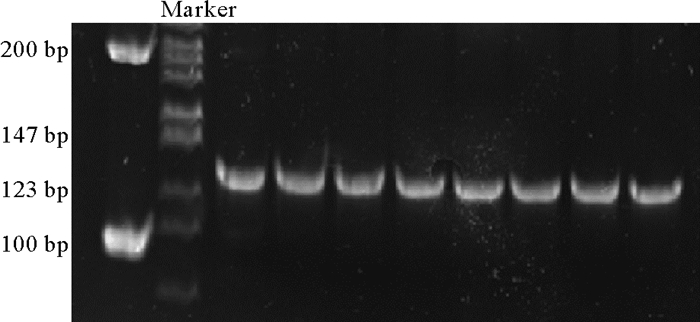
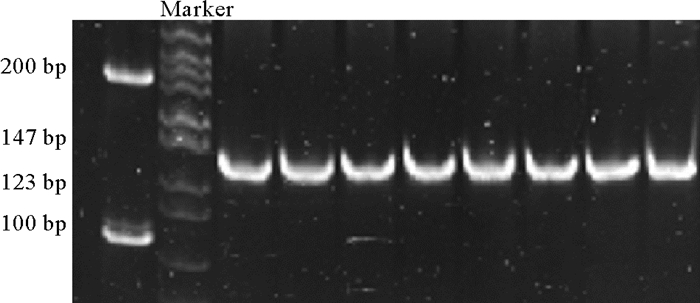
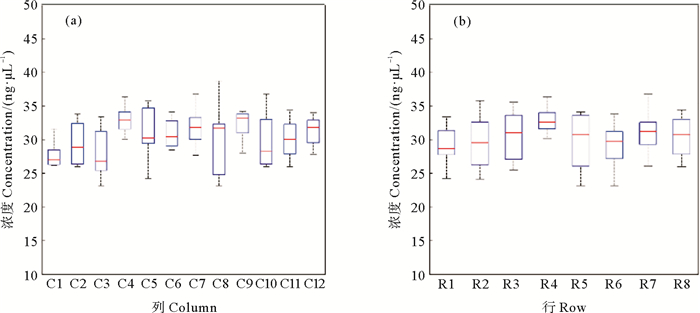
利用自动移液工作站同步进行96管探针的扩增,将母液分配至单管,且液面的高度均匀一致,并在10 min内完成了所有样品的PCR体系制备。待PCR反应结束后,利用工作站真空抽滤法同时进行了96管PCR产物的纯化。电泳图检测结果显示所有管内均没有非特异性扩增(见图 2),并且纯化后的产物具有均匀一致的亮度(见图 3);浓度检测结果显示,所有管内的浓度范围在23.1~38.7 ng/μL之间。96管PCR纯化产物按照列或行分析时,单因素方差分析结果表明行或列间无显著差异,浓度分布基本均匀一致(见图 4),表明自动化PCR扩增以及纯化的均匀性。
|
图 2 探针扩增检测电泳图 Fig. 2 Polyacrylamide gel electrophoresis results of PCR product of oligo pools |
|
图 3 纯化产物PAGE检测图 Fig. 3 Polyacrylamide gel electrophoresis results of purified product |
|
((a)12列之间的浓度分布箱型图; (b) 8行之间的浓度分布箱型图。(a) Boxplot of mean product concentration per column; (b) Boxplot of product concentration per row. ) 图 4 探针纯化后列之间和行之间的浓度箱型图 Fig. 4 Box diagrams between columns and rows after purification |
利用调试好的自动化分型流程,制备了32个虾夷扇贝(P. yessoensis)DNA样品的HD-Marker文库。其中成功建库的样品29个,剩下的3个样品的文库无法满足高通量测序的要求,自动化的建库成功率为90.63%。最终获得的29个文库浓度分布在8.9~10.6 ng/μL之间,且文库浓度均匀,质量符合测序要求。
随机选取8个成功建库的样品进行PE150测序,从特异性、捕获率及分型准确率等几个指标评价自动化制备HD-Marker文库测序结果的可靠性。结果显示,在所有样品中特异性均在80.0%以上,位点捕获率均在97%以上,分型位点比例能达到100%。与标准WGS文库数据相比,位点分型准确性也在97%~100%之间(见表 3)。
|
|
表 3 自动化分型文库的测序结果 Table 3 Sequencing result of automated genotyping library |
与人工构建文库的测序结果相比较,自动化文库与人工构建的文库在位点测序深度(覆盖某位点的reads数目)上具有较高的一致性,8个自动化文库的Pearson系数[19]均能达到0.96(见图 5)。综上所述,利用自动化移液工作站制备的HD-Marker基因分型文库能够获得较为稳定可靠的测序结果。
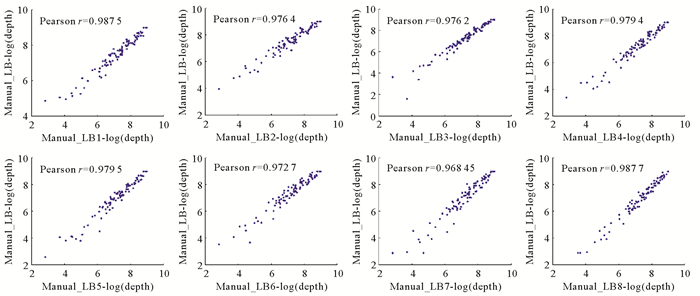
|
(Manual_LB-log(depth):人工制备文库测序深度的log值; LB1-LB8:8个自动化制备的文库。Manual_LB-log(depth): Log value of sequencing depth of manual library; LB1-LB8:8 automated libraries. ) 图 5 自动化制备的文库与人工建库相比测序深度的一致性 Fig. 5 The correlation of sequencing depth between automated library and manual library |
当样品数量较大(>96个样品)时基于自动化工作站的自动化文库制备所需时间仅为手动的1/7(见表 4),尽管分步之间的衔接仍需人工干预,但对人工需求相比全人工手动建库大大降低。其次,机器预编译的程序原则上不会存在漏加、错加和少加等情况,构建的文库具有较高的均匀性,特别适用于大批量样品的基因分型文库的构建。但相对于人工手动建库,自动化文库制备所需成本较高,约为手动建库的1.1倍,但总体而言,自动化文库的构建能够有效的减少人力,并且获得均一稳定的建库结果,在短时间内快速实现大量样品的高通量测序文库构建。
|
|
表 4 自动化制备文库与手动建库的比较 Table 4 The comparison between automated and manual construction of HD-Marker library |
短时高效获取大量样品的SNP分型信息是实现动植物全基因组选择育种的关键要素。除模式生物、人及畜牧动物等可依靠基因芯片快速获取分型信息[9-12],大量生物尤其非模式生物主要通过二代测序平台的检测分析获取分型结果。但其首要的步骤就是构建高质量的测序文库,而测序文库的构建涉及大量手工操作,尤其处理大规模样品是一项十分耗时耗力的工作。本研究首次建立了基于自动化移液工作站的自动化高通量HD-Marker基因分型文库构建流程,并对其进行了评估。
本研究建立的自动化文库构建流程一次反应可以同时运行96个样品的文库构建,整体运行时间仅为人工操作的1/7。为解决大量样品纯化效率低的问题,本研究建立了基于96孔真空抽滤法的PCR产物纯化方式,加样和纯化步骤全程可自动化完成,改变以往单个离心柱回收进行纯化的方式,能够大幅度提高探针制备的效率。通过电泳图和浓度纯度的结果,判断出不同管之间产物的得率无显著差异,说明依托自动化移液工作站建立的真空抽滤法适用于HD-Marker的探针制备环节。另外,文库制备的全部流程反应中均包含链霉素磁珠,因此磁珠的平衡、磁珠与液体的混匀以及磁液分离等操作都是影响实验的关键步骤。因此,本文作者对涉及到磁珠操作时的吸喷液速度及次数进行了优化调整,既能保证磁珠充分悬浮于液体中,又能得到较好的磁液分离效果。
本研究还比较了使用自动化移液工作站构建基因分型文库与人工手动建库的优缺点,发现自动化移液工作站构建基因分型文库有诸多优点:(1)准确:自动化工作站按照设定好的程序自动进行实验,不会出现漏加或者错加等实验错误。(2)稳定:重复进行多次实验,结果均一性程度高。(3)高效:自动化工作站设定好程序,可以无人值守工作,提高实验室工作效率。(4)实验多元化:全自动化移液工作站通常都整合丰富多样的实验设备,如振荡器Te-Shake,磁珠提取模块Te-MagS,真空提取模块Te-Vac等,在药物和抗体筛选[20-21]、核酸蛋白提取[22]、免疫组化[23]、酶免实验[24]等方向应用广泛。但自动化移液工作站的使用也存在一定的不足,针对性的建议如下:(1)原装进口枪头的成本较高,后续可在某些可循环利用的实验环节将枪头丢弃操作优化为放回枪头盒中,或者通过一些不与样品DNA直接接触的方式进一步降低耗材使用的成本; (2)机械吸液无法像人工充分吸取试剂槽底部的试剂,因此在原料的损耗上相比人工要多,但可以通过试剂的回收再利用降低试剂的损耗。(3)本研究中使用的Tecan Freedom EVO自动化液体处理系统配备1 mL注射器,但从本研究结果中可看出在低于10 μL体积的定量中,准确性及精密度不够理想,因此流程建立时需尽可能规避这部分误差。
总体来说,基于TECAN工作站建立的自动化HD-Marker文库构建流程,可依托预编译的控制程序实现标准化自动化地HD-Marker基因分型文库构建,并且能够获得均匀稳定的建库质量,保证整体测序的质量。可以有效地代替人工操作,避免和减少人为操作导致的实验误差,并提高批量样品并行处理的效率,在动植物的遗传育种研究中具有重要的应用价值。
| [1] |
Ribaut J M, de Vicente M C, Delannay X. Molecular breeding in developing countries: Challenges and perspectives[J]. Current Opinion in Plant Biology, 2010, 13(2): 213-218. DOI:10.1016/j.pbi.2009.12.011 (  0) 0) |
| [2] |
Meuwissen T H, Hayes B J, Goddard M E. Prediction of total genetic value using genome-wide dense marker maps[J]. Genetics, 2001, 157(4): 1819-1829. DOI:10.1093/genetics/157.4.1819 (  0) 0) |
| [3] |
Davey J W, Hohenlohe P A, Etter P D, et al. Genome-wide genetic marker discovery and genotyping using next-generation sequencing[J]. Nature Reviews Genetics, 2011, 12(7): 499-510. DOI:10.1038/nrg3012 (  0) 0) |
| [4] |
Baird N A, Etter P D, Atwood T S, et al. Rapid SNP discovery and genetic mapping using sequenced RAD markers[J]. PLoS One, 2008, 3(10): e3376. DOI:10.1371/journal.pone.0003376 (  0) 0) |
| [5] |
Elshire R J, Glaubitz J C, Sun Q, et al. A robust, simple genotyping-by-sequencing (GBS) approach for high diversity species[J]. PloS One, 2011, 6(5): e19379. DOI:10.1371/journal.pone.0019379 (  0) 0) |
| [6] |
Wang S, Meyer E, McKay J K, et al. 2b-RAD: A simple and flexible method for genome-wide genotyping[J]. Nature Methods, 2012, 9(8): 808-810. DOI:10.1038/nmeth.2023 (  0) 0) |
| [7] |
Wang S, Liu P, Lv J, et al. Serial sequencing of isolength RAD tags for cost-efficient genome-wide profiling of genetic and epigenetic variations[J]. Nature Protocols, 2016, 11(11): 2189-2200. DOI:10.1038/nprot.2016.133 (  0) 0) |
| [8] |
Andrews K R, Good J M, Miller M R, et al. Harnessing the power of RADseq for ecological and evolutionary genomics[J]. Nature Reviews Genetics, 2016, 17(2): 81-92. DOI:10.1038/nrg.2015.28 (  0) 0) |
| [9] |
Pease A C, Solas D, Sullivan E J, et al. Light-generated oligonucleotide arrays for rapid DNA sequence analysis[J]. Proceedings of the National Academy of Sciences, 1994, 91(11): 5022-5026. DOI:10.1073/pnas.91.11.5022 (  0) 0) |
| [10] |
Caas-álvarez J J, González-Rodríguez A, Munilla S, et al. Genetic diversity and divergence among Spanish beef cattle breeds assessed by a bovine high-density SNP chip[J]. Journal of Animal Science, 2015, 93(11): 5164-5174. DOI:10.2527/jas.2015-9271 (  0) 0) |
| [11] |
Zhu B, Li Q, Liu R, et al. Genome-wide association study of H/L traits in chicken[J]. Animals (Basel), 2019, 9(5): 260. (  0) 0) |
| [12] |
Junqueira V S, Cardoso F F, Oliveira M M, et al. Use of molecular markers to improve relationship information in the genetic evaluation of beef cattle tick resistance under pedigree-based models[J]. Journal of Animal Breeding and Genetics, 2017, 134(1): 14-26. DOI:10.1111/jbg.12239 (  0) 0) |
| [13] |
Albrechtsen A, Nielsen F C, Nielsen R. Ascertainment biases in SNP chips affect measures of population divergence[J]. Molecular Biology and Evolution, 2010, 27(11): 2534-2547. DOI:10.1093/molbev/msq148 (  0) 0) |
| [14] |
Jiang Z, Wang H, Michal J J, et al. Genome wide sampling sequencing for snp genotyping: Methods, challenges and future development[J]. International Journal of Biological Sciences, 2016, 12(1): 100-108. DOI:10.7150/ijbs.13498 (  0) 0) |
| [15] |
Lv J, Jiao W, Guo H, et al. HD-Marker: A highly multiplexed and flexible approach for targeted genotyping of more than 10 000 genes in a single-tube assay[J]. Genome Research, 2018, 28(12): 1919-1930. DOI:10.1101/gr.235820.118 (  0) 0) |
| [16] |
Stangegaard M, Frøslev T G, Frank-Hansen R, et al. Automated extraction of DNA from blood and PCR setup using a Tecan Freedom EVO liquid handler for forensic genetic STR typing of reference samples[J]. Journal of Laboratory Automation, 2011, 16(2): 134-140. DOI:10.1016/j.jala.2010.11.003 (  0) 0) |
| [17] |
匡金枝, 聂同钢, 杨智, 等. 法医物证DNA自动化检验技术体系的研究[J]. 中国法医学杂志, 2009, 24(3): 164-167. Kuang J Z, Nie T G, Yang Z, et al. A study of automation workstation for the DNA extraction of forensic biological evidence[J]. Chinese Journal of Forensic Medicine, 2009, 24(3): 164-167. DOI:10.3969/j.issn.1001-5728.2009.03.007 (  0) 0) |
| [18] |
Vogeser M, Spöhrer U. Automated processing of whole blood samples for the determination of immunosuppressants by liquid chromatography tandem-mass spectrometry[J]. Clinical Chemistry and Laboratory Medicine, 2006, 44(9): 1126-1130. (  0) 0) |
| [19] |
Sedgwick P. Pearson's correlation coefficient[J]. British Medical Journal, 2012, 345: e4483. DOI:10.1136/bmj.e4483 (  0) 0) |
| [20] |
Ye Q Z, Nan F J, Hu L Y, et al. High-throughput screening for human collagenase1 inhibitors[J]. Acta Pharmacologyica Sinica, 2000, 21(11): 1021-1026. (  0) 0) |
| [21] |
Wierling P S, Bogumil R, Knieps-Grünhagen E, et al. High-throughput screening of packed-bed chromatography coupled with SELDI-TOF MS analysis: Monoclonal antibodies versus host cell protein[J]. Biotechnology and Bioengineering, 2007, 98(2): 440-450. DOI:10.1002/bit.21399 (  0) 0) |
| [22] |
严江伟, 王顺霞, 任贺, 等. 应用自动化工作站提取常见生物样本DNA[J]. 中国法医学杂志, 2007, 2(22): 73-75. Yan J W, Wang S X, Ren H, et al. Study on the application of the automation workstation in DNA extraction of biological samples[J]. Chinese Journal of Forensic Medicine, 2007, 2(22): 73-75. (  0) 0) |
| [23] |
Rumballe B, Georgas K, Little M H. High-throughput paraffin section in situ hybridization and dual immunohistochemistry on mouse tissues[J]. Cold Spring Harbor Protocols, 2008(8): pdb.prot5030. (  0) 0) |
| [24] |
张鹏, 魏巍, 李玉燕, 等. 使用全自动酶免工作站进行酶联免疫吸附试验的效果评估[J]. 家禽科学, 2020(7): 10-14. Zhang P, Wei W, Li Y Y, et al. Effect evaluation of enzyme-linked immunosorbent assay with automatic ELISA workstation[J]. Poultry Science, 2020(7): 10-14. DOI:10.3969/j.issn.1673-1085.2020.07.003 (  0) 0) |
2. Laboratory for Marine Biology and Biotechnology, Pilot Qingdao National Laboratory for Marine Science and Technology(Qingdao), Qingdao 266237, China;
3. Laboratory for Marine Fisheries Science and Food Production Processes, Pilot Qingdao National Laboratory for Marine Science and Technology(Qingdao), Qingdao 266237, China;
4. Laboratory of Tropical Marine Germplasm Resources and Breeding Engineering, Sanya Oceanographic Institution, Ocean University of China, Sanya 572000, China
 2022, Vol. 52
2022, Vol. 52


