2. 中国水产科学院黄海水产研究所,山东 青岛 266071;
3. 山东出入境检验检疫局,山东 青岛 266002
志贺氏菌属(Shigella)是一类革兰氏阴性杆菌,是人类细菌性痢疾最为常见的致病菌,又称痢疾杆菌。人主要通过食用被志贺氏菌污染的食品而感染发病,以溃疡、大肠化脓性炎症以及全身中毒症状为主要临床特征。据统计,每年全球的人类痢疾感染病例有1.647亿,其中有1.632亿病例发生在发展中国家,并导致110万人死亡,志贺氏菌病的危害远远超过现有的想象[1]。
根据志贺氏菌表面抗原结构的不同,可以将志贺氏菌属分为4类,包括痢疾志贺氏菌(S. dysenteriae)、鲍氏志贺氏菌(S. boydii)、福氏志贺氏菌(S. flexneri)、宋内氏志贺氏菌(S. sonnei)4个菌种。志贺氏菌的菌型分布因地域不同而有所差异,了解菌群分布与不同菌型的变迁,对于预防痢疾和制备菌苗具有重大意义。此外,在公共卫生领域和院内感染评价常进行特定地域食品致病菌检测、医院感染、局部流行病学调查及临床诊断,也需要准确而快速简便的分型及检测技术。传统的分型技术主要基于病原菌的表型如生理、生化特征等[2],如血清型分型、耐药性分型、噬菌体分型等,但存在分辨率低、不可重复等诸多局限性。随着分子生物学的快速发展,基于病原菌基因特征的分子分型方法以其重复性好、特异性强、敏感性高等特点逐步取代传统表型分型方法,成为病原菌分型的趋势。比较成熟的分子分型技术有脉冲场凝胶电泳(PFGE)、多位点序列分析(MLST)、扩增片段长度多态性分析(AFLP)、多位点可变数量串联重复序列(VNTR)、限制性片段长度多态性分析(RFLP)、基因芯片、随机扩增多态性分析(RAPD)、质粒图谱分析等[3]。然而,上述方法操作较繁琐,程序比较复杂。针对4种志贺氏菌的快速分型目前尚未有报道,还是停留在生化和血清学鉴定的水平上,因此建立其快速、简便的分型方法具有重要的现实意义。
微生物菌体在中红外光谱(4 000~400 cm-1波数范围)处有特定吸收且具有很强的指纹特征。微生物干涉后的红外光谱经傅立叶变换处理便可得到傅立叶变换红外光谱。FT-IR技术读取的是微生物菌体的所有组成成分化学键的振动情况,提供整个微生物菌体组成成分的光谱定量信息[4],结合化学计量学运算方法,实现对微生物的分类、鉴定[5-9]。前期,我们应用FT-IR技术对微生物的分类鉴定进行了大量研究,探讨了FT-IR技术用于微生物分类鉴定的影响因素[10],对大肠杆菌、沙门氏菌、李斯特氏菌等分别进行了研究[11-13],证明FT-IR技术具有强大的微生物分类鉴定能力,而且分辨率和灵敏度非常高。但是,上述研究仅就FT-IR技术在不同分类层次上的的分类鉴定能力进行了研究,尚未就某个致病菌领域进行全面系统的研究,因此还不能真正应用。基于此,本文选择志贺氏菌属所有4种志贺氏菌,首先读取4种志贺氏菌的傅里叶变换红外光谱,构建其标准图谱数据库,然后应用PCA和HCA两种聚类方法,尝试建立4种志贺氏菌的快速分型方法,这将会为志贺氏菌的分型与检测提供了一种有效的工具。
1 材料与方法 1.1 材料与试剂 1.1.1 菌种S. dysenteriae CICC 23829、S. flexneri CICC 21678、S. boydii CICC 21680、S. sonnei CICC 21679、S. enteritidis CICC 21482购自中国工业微生物菌种保藏管理中心(CICC),出血性大肠埃希氏菌O157:H7 ATCC 43895(Escherichia. Coli O157: H7, E. coli O157: H7)购自美国模式培养物集存库(ATCC)。接种菌种到营养肉汤中进行活化,37 ℃培养18~24 h,备用。
1.1.2 试剂超纯水,电阻18.2 mΩ;0.9%生理盐水;无水乙醇,分析纯级别;志贺氏菌增菌肉汤-新生霉素,麦康凯(MAC)琼脂,木糖赖氨酸脱氧胆酸盐(XLD)琼脂,缓冲蛋白胨水(BPW),四硫磺酸钠煌绿(TTB)增菌液,亚硒酸盐胱氨酸(SC)增菌液,亚硫酸铋(BS)琼脂,改良EC肉汤,改良山梨醇麦康凯(CT-SMAC)琼脂,营养肉汤,购自北京陆桥生物科技有限公司,按说明书配置。改良科玛嘉O157显色琼脂购自法国科玛嘉公司。
1.2 设备与材料ZnSe窗片,直径25 mm,厚度2 mm,透过波长是7 800~440 cm-1,透过率大于68%;CR22G Ⅲ离心机,日本日立公司;核酸蛋白分析仪,Bio-rad公司;(36±1)℃恒温恒湿培养箱,3M公司;VERTEX70型傅立叶变换红外光谱仪,布鲁克公司;45 ℃干燥箱,3M公司。
1.3 方法 1.3.1 样品制备接种4种志贺氏菌S. dysenteriae,S. flexneri,S. boydii和S. sonnei活化肉汤培养物于盛有10 mL营养肉汤的带塞大试管中,(36 ±1) ℃培养18~24 h,核酸蛋白分析仪测定菌液浓度。
分别吸取1 mL各菌的肉汤培养物于1.5 mL离心管中,5 000g离心5 min,弃上清,用1 mL无菌生理盐水悬浮洗涤沉淀,再用1 mL超纯水洗涤2次。最后用50 μL超纯水悬浮。准确吸取10 μL菌悬液于ZnSe窗片中心位置,涂抹均匀,水平置于45 ℃干燥箱中进行烘干。
1.3.2 光谱采集应用傅立叶变换红外光谱仪对制备好的样品进行光谱采集,采集模式采用透过率模式(采用气氛补偿功能去除环境中的CO2和水蒸气的吸收光谱干扰)。实验参数:光谱分辨率4 cm-1,波段范围4 000~600 cm-1[14-15],64次光谱累计求平均[16]。
1.3.3 光谱处理及数据分析每个样品至少做6次试验,每次做3个重复,取平均,取6个有效数据。
光谱预处理:应用OPUS 6.5软件,对采集的4种菌的4 000~600 cm-1波数范围光谱依次进行透过率-吸光度转化、基线校正(消除基线漂移),矢量归一化处理。然后将归一化后的光谱转化为DPT数据点格式,进而转化为Excel数据格式,得到4种志贺氏菌标准菌株的红外光谱数据库。
选择各菌1 800~900 cm-1波数范围光谱数据分别导入Statistica 6.0和Matlab 6.5软件,分别进行分级聚类分析(Hierarchical cluster analysis,HCA)和主成分分析(Principal component analysis,PCA),得到4种志贺氏菌FT-IR光谱的树状分级聚类分布图和主成分聚类分布图。
1.4 方法验证用活化好的S. sonnei CICC 21679、S. enteritidis CICC 21482和E. coli O157: H7 ATCC 43895菌悬液污染同一份新鲜猪肉样品。分别根据《食品安全国家标准食品微生物学检验志贺氏菌检验》(GB 4789.5-2012)、《食品安全国家标准食品微生物学检验沙门氏菌检验》(GB 4789.4-2010)和《食品卫生微生物学检验大肠埃希氏菌O157:H7/NM检验》(GB/T 4789.36-2008)进行检验,挑取MAC和XLD琼脂上的可疑志贺氏菌、BS和XLD琼脂上的可以沙门氏菌以及CT-SMAC和改良科玛嘉O157显色琼脂上的可疑E. coli O157: H7分别接种营养肉汤中,细菌培养及样品制备方法同1.3.1。
根据1.3.2方法采集上述可疑菌的光谱数据,并按1.3.3对其进行预处理,将处理好的数据与建立的4种志贺氏菌的标准光谱数据库合并,利用Statistica 6.0软件进行HCA,得到分级聚类图。
2 结果 2.1 平均光谱及光谱数据库的建立4种志贺氏菌的光谱经基线校正、适量归一化预处理后,对每种菌的6个重复数据取平均,得到各种菌的平均光谱(见图 1)。光谱间的一些差异可以用肉眼进行观察区分,特别是在1 000~1 600 cm-1波数范围内的光谱差异较明显,推测这应该是区分4种志贺氏菌的主要光谱区域。但是对其进行准确分类需借助计算机及化学计量学方法。此外,每个波数或每段波数范围的光谱与细菌成分或结构具有特异的对应关系,因此,细菌间的差异可以通过光谱分析直接具体到其生物大分子组成或其结构上[12]。
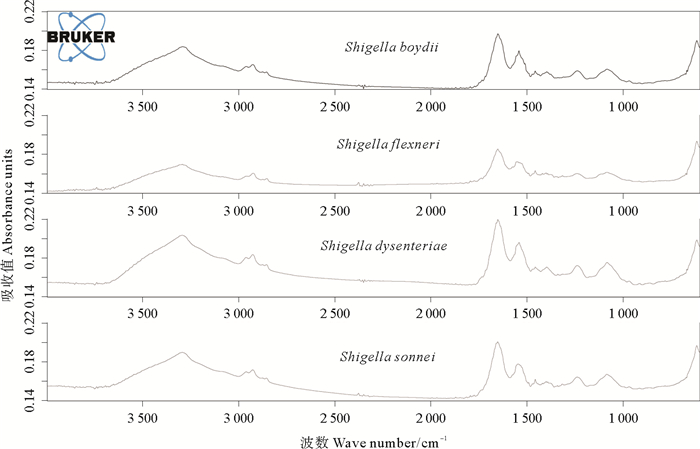
|
图 1 4种志贺氏菌的平均光谱图 Fig. 1 Average FT-IR spectra of four species of Shigella |
对志贺氏菌属内4种菌的FT-IR光谱进行一系列预处理后,得到归一化后的光谱,即建立了志贺氏菌属内S. dysenteriae,S. flexneri,S. boydii和S. sonnei 4种菌的标准的FT-IR光谱数据库。该数据库提供了一个标准的比对和参考,利用这个数据库,我们可以对未知可疑目标菌进行快速分型。方法为:首先严格按照样品制备条件和光谱采集条件获得可疑目标菌的光谱,对光谱按照同样的方法进行预处理后,与建立的数据库合并,然后对合并后的光谱数据重新进行PCA或HCA聚类分析,根据可疑目标菌与数据库中4种Shigella光谱数据的聚类情况便可对可疑菌作出分型的初步判断。
2.2 数据分析 2.2.1 主成分分析(PCA)PCA分析属于一种非监督型统计方法,通过线性变换从多个变量中筛选出几个较少的综合变量,筛选出的这几个综合变量应对于PCA分析具有较大贡献,且能反应原始变量的绝大多数信息。主成分(Pricipale component, PC)即是通过线性变换从多个变量中筛选出的能反应原始变量信息、对PCA分析具有主要贡献的综合变量。本研究选取PC1、PC2和PC3等3个PCs作为聚类依据,得到主成分聚类三维分布图(如图 2)。由图 2可以看出,PCA分析可以将志贺氏菌属内S. dysenteriae,S. flexneri,S. boydii和S. Sonnei 4种菌进行准确聚类,也说明4种Shigella光谱之间具有差异性。对于可疑目标菌,将其光谱与构建的4种Shigella标准菌株的光谱数据库合并,对合并后的数据进行PCA分析,根据聚类分布图中可疑目标菌光谱数据在标准菌株光谱数据库中的的聚类情况,即可对可疑目标菌的分型情况作出初步判断。

|
图 2 4种志贺氏菌光谱的PCA聚类分布图 Fig. 2 Results of PCA cluster |
3个主成分PC1、PC2和PC3权重见图 3。由图 3可知,在1 800~900 cm-1波数范围内,3个PCs均具有较大的权重值,其中PC1权重值最大,PC2和PC3次之,反映出3个PCs对PCA聚类分析的贡献大小。3个PCs对于聚类的总贡献率为99.81%(分别为99.3%、0.36%和0.15%),说明这3个PCs能反应出原始变量的绝大多数信息,采用这3个PCs建立的PCA聚类分析方法具有可行性。
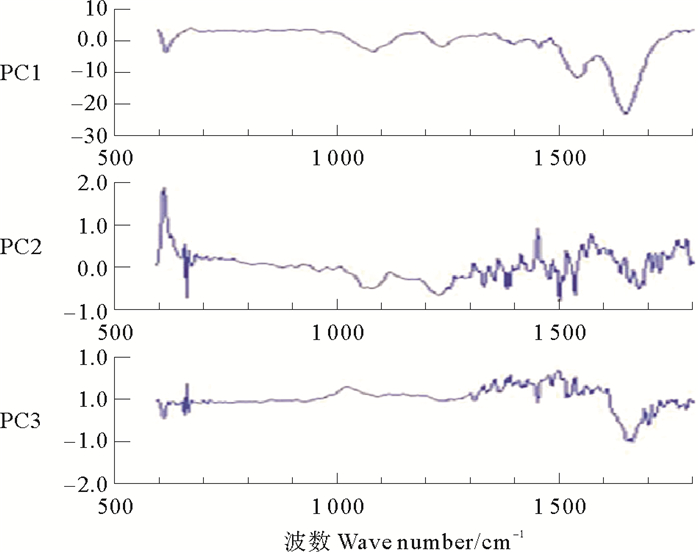
|
图 3 4种志贺氏菌光谱的主成分权重图 Fig. 3 Loadings plot of PCs obtained from PCA analysis of FT-IR spectra of four species of Shigella |
对4种Shigella的光谱数据进行HCA分析,利用皮尔森积矩相关系数(Ward′s method,Pearson r),用光谱间的距离作为判定依据,得到树状聚类分布图,见图 4。结果证明HCA分析可以将4种Shigella进行成功聚类。同PCA,对于可疑目标菌,将其光谱与构建的4种Shigella标准菌株的光谱数据库合并,对合并后的数据进行HCA分析,通过观察树状聚类分布图中可疑目标菌光谱与标准菌株的聚类情况,即可对可疑目标菌的分类鉴定情况进行判断。
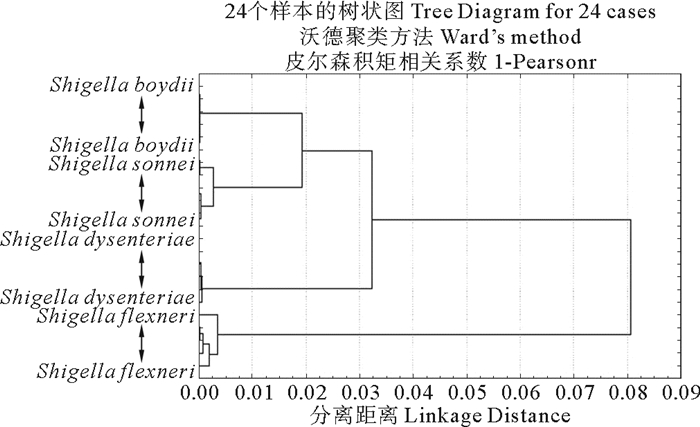
|
图 4 4种志贺氏菌光谱的HCA聚类分布图 Fig. 4 Results of HCA cluster |
将污染样品的可疑菌的光谱数据与建立的4种志贺氏菌的标准光谱数据库合并,将合并数据进行HCA聚类分析,得到聚类分布图,见图 5。由图 5可知,分离的可疑志贺氏菌被聚类到了S. sonnei聚类群中,分离的可疑S. enteritidis和E. coli未被聚类到4种志贺氏菌的任一种聚类群中,而是各自被单独聚为一类。而且就光谱间的距离而言,可疑志贺氏菌与S. sonnei聚类群距离最近,而可疑S. enteritidis和E. coli与4种志贺氏菌的聚类群距离都较远。以上结果说明建立的4种志贺氏菌标准光谱数据库和HCA方法可以将志贺氏菌进行准确分型,而且对于非志贺氏菌也可以对其准确排除,避免了假阳性结果的出现。
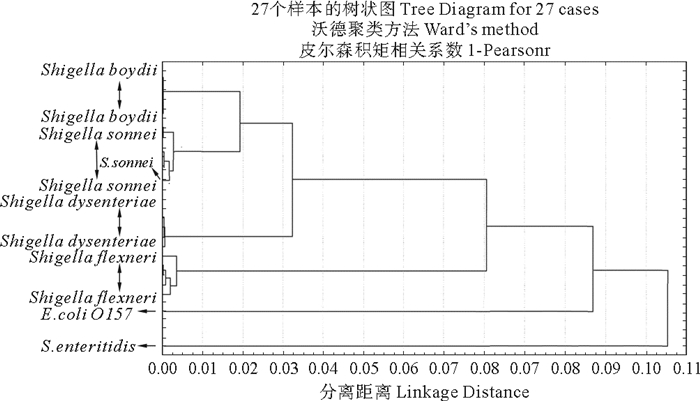
|
图 5 方法验证HCA聚类分析结果 Fig. 5 HCA results of test |
本研究首先通过对志贺氏菌属内4种菌的FT-IR光谱进行处理,得到了S. dysenteriae, S. flexneri, S. boydii和S. Sonnei 4种菌的FT-IR光谱数据库,用以未知可疑目标菌的快速分型。这对于志贺氏菌的检测及分型非常有意义。我们建立了志贺氏菌属所有4种Shigella的光谱数据库,为其分型检测奠定了基础。细菌的中红外光谱具有非常典型的指纹特征,光谱数据库的建立相当于为可疑目标菌提供了一个比对和参考,因此可以大大简便细菌的分类鉴定过程。在微生物分类鉴定领域,数据库的概念都是基于其生化代谢特征,基于其光谱建立数据库的相关报道较少,因此本研究提出的FT-IR光谱数据库的概念会为细菌的分型及检测提供一个新的思路和研究方向。但是,由于细菌是活的生命体,其组成成分随生命状态而异,这样反应的光谱特征可能就会有差异,因此本文建立的光谱数据库及采集的光谱必须是以统一的样品制备条件为前提。对此,前期我们已通过实验验证了细菌浓度、培养条件及加热灭火等条件对FT-IR技术用于细菌分类鉴定均具有明显的影响[10]。
本研究应用PCA或HCA分析方法,建立了4种志贺氏菌的快速分型方法。实验结果说明2种分析方法均能实现对4种Shigella的快速分类。其中PCA分析方法可在确保光谱差异性的前提下将数据维数减少至二维或三维图,但这可能会导致原本很好聚类的数据点看似发生“交叉或重叠”,体现在二维或三维图中就不能成功分类。在多种细菌数据分析时容易发生上述情况。但是本研究志贺氏菌属一共只有4种菌,因此不会发生这种情况。HCA以光谱之间的距离作为分类依据,分类结果通过平面树状图直观的体现,不会发生重叠或交叉问题。在实际微生物分类鉴定中,2种方法可以结合运用。据文献报道[16],微生物一定波数范围光谱特征与其细菌结构组成具有特定对应关系,在1 800~900 cm-1波数范围光谱有3个“指纹区域”具有特定吸收,即酰胺区、混合区(包括脂肪酸、磷酸化合物和蛋白质)和多糖区。从图 1便可看出在这个区域4种菌光谱之间具有差异性,另外,结合我们的分析结果,本研究选择1 800~900 cm-1范围光谱进行分析。
为验证所建立数据库和分型方法的准确性,本研究对从污染样品分离的可疑菌进行了HCA聚类分析。结果充分说明我们应用FT-IR技术建立的方法可以准确实现4种志贺氏菌的准确快速分型。与传统表型方法及分子生物学分型方法相比,该方法大大简化了操作过程,从采集光谱到数据分析1小时内便可完成。这为志贺氏菌临床诊断、流行病学调查、食物中毒的预警和溯源、食品中志贺氏菌检测及医院感染监控,志贺氏菌的分型与检测提供了一种有效的工具,也为其他致病菌的分型研究提供了一种新的思路。但是,作为一种分型技术,本研究建立的分型技术仅仅是对志贺氏菌属“四大类”志贺氏菌进行了“粗分”。而每一种志贺氏菌又有若干血清型,对不同的血清型进行“细分”,或许这将是FT-IR技术用于分型研究未来的工作。
| [1] |
任慧婧, 刘芸, 王美会, 等. 志贺氏菌检测方法的研究进展[J]. 河南农业, 2015, 7: 49-50. Ren H J, Liu Y, Wang M H, et al. The advancement on detection methods for Shigella[J]. Henan Agriculture, 2015, 7: 49-50. (  0) 0) |
| [2] |
管峰, 杨季芳. 食源性致病菌溯源分型技术研究进展[J]. 浙江万里学院学报, 2012, 4: 96-100. Guan F, Yang J F. Study on typing of foodborne pathogens tracking[J]. Journal of Zhejing Wanli University, 2012, 4: 96-100. (  0) 0) |
| [3] |
王庆忠, 娄峥, 宣瑛. 病原菌分子分型方法研究进展[J]. 检验医学, 2009, 24(5): 397-400. Wang Q Z, Lou Z, Xuan Y. Stydy on molecular typing methods for pathogens[J]. Laboratory Medicine, 2009, 24(5): 397-400. (  0) 0) |
| [4] |
Naumann D. Some ultrastructural information on intact, living bacterial cells and related cell-wall fragments as given by FTIR[J]. Infrared Physics and Technology, 1984(24): 233-238.
(  0) 0) |
| [5] |
Naumanan D, Helm D, Labischinski H. Microbiological characterization by FT-IR spectroscopy[J]. Nature, 1991(351): 6321-6381.
(  0) 0) |
| [6] |
Helm D, Labischinski H, Schallehn G, et al. Classification and identification of bacteria by fourier transform infrared spectroscopy[J]. Journal of Genaral and Applied Microbiology, 1991a(137): 69-79.
(  0) 0) |
| [7] |
Helm D., Labischinski H., Naumann D. Elaboration of a procedure for identification of bacteria using fourier transform IR spectral libraries: A stepwise correlation approach[J]. Jouranl of Microbiological Methods, 1991b(14): 127-142.
(  0) 0) |
| [8] |
Jaurequiberry M, Madoz L V, Giuliodori M J, et al. Identification of Escherichia coli and Trueperella pyogenes isolated from the uterus of dairy cows using routine bacteriological testing and fourier transform infrared spectroscopy[J]. Acta Veterinaria Scandinavica, 2016, 58(1): 81. DOI:10.1186/s13028-016-0262-z
(  0) 0) |
| [9] |
Johler S, Stephan R, Althaus D, et al. High-resolution subtyping of Staphylococcus aureus strains by means of Fourier-transform infrared spectroscopy[J]. Systematic and Applied Microbiology, 2016, 39(3): 189-194. DOI:10.1016/j.syapm.2016.03.003
(  0) 0) |
| [10] |
杨丽君, 王静, 李兆杰, 等. 傅立叶变换红外光谱技术用于细菌检测的影响因素研究[J]. 食品科学, 2013, 34(8): 190-194. Yang L J, Wang J, Li Z J, et al. Several factors for the detection of bacteria by FT-IR spectroscopy[J]. Food Science, 2013, 34(8): 190-194. DOI:10.7506/spkx1002-6630-201308040 (  0) 0) |
| [11] |
王静, 杨丽君, 李兆杰, 等. 傅立叶变换红外光谱技术对两株大肠杆菌的鉴别[J]. 微生物学通报, 2012, 39(2): 1844-1850. Wang J, Yang L J, Li Z J, et al. Differentiation of two strains of Escherichia coli by FT-IR spectroscopy[J]. Microbiology China, 2012, 39(2): 1844-1850. (  0) 0) |
| [12] |
杨丽君, 李兆杰, 宋晓华, 等. 傅立叶变换红外光谱技术对5种沙门氏菌的快速分类鉴定[J]. 微生物学通报, 2013, 40(2): 373-379. Yang L J, Li Z J, Song X H, et al. Rapid differentiation and identification of five species of Salmonella by FT-IR spectroscopy[J]. Microbiology China, 2013, 40(2): 373-379. (  0) 0) |
| [13] |
杨丽君, 李兆杰, 王静, 等. 傅立叶变换红外光谱技术对3种李斯特氏菌的快速分类鉴定[J]. 食品与生物技术学报, 2013, 32(2): 169-173. Yang L J, Li Z J, Wang J, et al. Rapid differentiation and identification of three species of Listeria by FT-IR spectroscopy[J]. Journal of Food Science and Biotechnology, 2013, 32(2): 169-173. (  0) 0) |
| [14] |
Lin M, Al-Holy M, Chang SS, et al. Rapid discrimination of Alicyclobacillus strains in apple juice by fourier transform infrared spectroscopy[J]. International Journal of Food Microbiology, 2005(105): 369-376.
(  0) 0) |
| [15] |
Rodriguez-Saona LE, Khambaty FM, Fry FS, et al. Rapid detection and identification of bacterial strains by fourier transform nearinfrared spectroscopy[J]. Journal of Agricultural and Food Chemistry, 2001(49): 574-579.
(  0) 0) |
| [16] |
Dziuba B, Babuchowski A, Nalecz D, et al. Identification of lactic acid bacterica using FTIR spectroscopy and cluster analysis[J]. International Dairy Journal, 2007(17): 183-189.
(  0) 0) |
| [17] |
Al-Qadiri HM, Lin M, Cavinato AG, et al. Fourier transform infrared spectroscopy, detection and identification of Escherichia coli O157:H7 and Alicyclobacillus strains in apple juice[J]. International Journal of Food Microbiology, 2006(111): 73-80.
(  0) 0) |
2. Yellow Sea Fisheries Institute, Chinese Academy of Fishery Sciences, Qingdao 266071, China;
3. Shandong Entry-exit Inspection and Quarantine Bureau, Qingdao 266002, China
 2018, Vol. 48
2018, Vol. 48

