2. 中国水产科学研究院东海水产研究所,上海 200090;
3. 江苏省海洋水产研究所,江苏 南通 226007;
4. 浙江海洋大学水产学院,浙江 舟山 316022
黑鲷(Acanthopagrus schlegelii)隶属鲈形目(Percoiformes)鲷科(Sparidae)棘鲷属(Acanthopagrus),亦称黑棘鲷[1],俗称黑加吉、海鲋等,广泛分布于日本、朝鲜及中国沿海地区[2]。黑鲷环境适应性强,生长迅速,且不作长距离洄游,是中国重要的增养殖种类[2]。由于过度捕捞、生态环境变化等原因,黑鲷自然群体资源量下降[3],其增殖放流活动逐步得到应用与推广。江苏省海洋水产研究所于1986年开展黑鲷人工增殖放流活动,截至1992年,共放流三次黑鲷幼鱼,数量达54.1万尾[4-5]。2018—2020年,在山东省海阳市的增殖放流工作中,共放流黑鲷658.28万尾[6]。我国每年的全国“放鱼日”会放流各种水生生物苗种,其中就包括大量的黑鲷放流[7-8]。
海洋生物增殖放流活动所产生的遗传学影响一直以来备受关注。Hamasaki等[9]对鹿儿岛湾(Kagoshima Bay)湾内海域、湾外海域以及其他海域的真鲷(Pagrus major)群体进行遗传学分析,结果显示湾内野生群体的遗传多样性低于湾外野生群体,推测是长期的增殖放流活动产生的影响。Gonzalez等[10]利用微卫星DNA标记对日本广岛湾(Hiroshima Bay)放流的黑鲷幼鱼以及回捕群体进行了遗传多样性监测,结果发现野生群体和孵化场鱼苗之间有很高的遗传相似性。Shan等[11]利用线粒体DNA控制区序列对珠江口流域的亲鱼、放流苗种、回捕群体等进行遗传多样性分析,结果发现与野生种群相比,亲鱼和放流苗种的遗传多样性指数较低,珠江口流域黑鲷群体的遗传多样性也低于其他海区。育苗场在繁育过程中使用亲鱼数量较少的局限性以及亲鱼或放流个体来自其他地区,都可能对野生群体造成遗传学影响。因此,放流群体和海捕群体的遗传多样性监测工作对了解种质资源现状以及研究增殖放流活动对野生群体产生的遗传学影响有重大意义。
线粒体DNA具有遵循母系遗传且不易发生基因重组的特点,线粒体DNA控制区序列与其他编码区相比,突变比率较高,目前已被广泛应用于海洋鱼类的群体遗传多样性研究中[12-14]。例如,赵祥等[15]利用线粒体DNA控制区对黄姑鱼(Nibea albiflora)的养殖群体与野生群体进行比较研究,发现黄姑鱼养殖群体遗传多样性显著偏低。Iguchi等[16]利用线粒体DNA控制区对日本香鱼(Plecoglossus altivelis)野生群体和放流群体进行研究,结果显示放流群体的遗传多样性低于野生群体,为保护野生群体的遗传多样性,作者建议放流群体的遗传多样性应尽可能与野生群体保持一致。
目前我国黑鲷放流工作正在大规模开展,但尚未见到监测舟山、莱州和南通地区的黑鲷放流群体对野生群体影响的研究。因此,本研究基于线粒体DNA控制区序列对采自舟山、莱州和南通的黑鲷亲鱼、放流苗种及海捕群体进行遗传多样性分析,旨在了解我国近海黑鲷的种质资源现状,评估增殖放流活动可能会对野生群体产生的遗传学影响,为后续的增殖放流活动提供参考。
1 材料与方法 1.1 样品采集本研究所用舟山亲鱼群体样品采自册子岛育苗场,亲鱼均为舟山近海捕捞,莱州和南通放流群体分别来自莱州顺昌水产有限公司(亲本来自莱州近海)、江苏省海洋水产研究所如东养殖基地(亲本来自如东近海),舟山育苗场鱼苗来自册子岛育苗场,舟山放流场鱼苗来自册子岛育苗场以及西轩岛基地。样品采集时间均与放流时间相近。海捕群体分别来自莱州近海、南通近海和舟山近海。放流群体放流地点与海捕群体采集地点相近,实验样品共764尾,具体样品采集信息见表 1。所有样品经形态学鉴定、测量后剪取背部肌肉置于冻存管中,加入无水乙醇固定并于-20 ℃中保存备用。
|
|
表 1 本研究中所用的黑鲷样品信息 Table 1 The sampling information of Acanthopagrus schlegelii in this study |
将黑鲷的肌肉组织经蛋白酶k消化后采用标准酚-氯仿方法提取样品DNA[17],根据黑鲷线粒体DNA全基因组序列(NCBI登录号: NC_018553.1),使用Primer Premier 6软件[18]设计引物序列并进行线粒体DNA控制区高变区序列的扩增。正反引物序列分别为:hdd-F: 5’-ACCCTTACTATCAACTCCCAAA-3’;hdd-R: 5’-ATCGCCACCATTAACTTATGC-3’,PCR体系共25 μL,1.1×T3 Super PCR Mix 22 μL,正、反向引物及模板DNA各1 μL。PCR反应条件参照Shan等[11],退火温度改为58 ℃。PCR扩增产物经1.5%的琼脂糖凝胶电泳检测后送至北京擎科生物科技有限公司青岛分公司进行双向测序。
1.3 数据分析测序所得序列均利用DNA Star(DNA Star, Inc. Madison, USA)软件包进行比对、人工校正,利用Arlequin软件[19]对黑鲷样品进行遗传多样性参数分析,如单倍型数目、多态位点数目、单倍型多样度、核苷酸多样度等。同时利用Arlequin软件计算两两群体间的遗传分化指数FST并进行AMOVA分析。
2 结果对本研究中764尾黑鲷样品序列进行比对、处理,获得403 bp的控制区序列用于遗传学分析。这些序列的A、T、C、G碱基的平均含量为39.39%、30.46%、19.90%和10.25%。A+T含量(69.85%)显著高于C+G含量(30.15%),显示出明显的碱基组成偏倚。8个群体共检测到54个多态位点,定义了69个单倍型,亲鱼群体、放流群体和海捕群体的单倍型分别为7个、46个和49个,其中亲鱼群体与放流群体、海捕群体的共享单倍型分别为5个和6个,放流群体与海捕群体的共享单倍型为27个(见表 2)。在本研究中,海捕群体的单倍型多样度和核苷酸多样度分别为0.850~0.951、0.008~0.009,其中南通海捕群体遗传多样性最高。亲鱼群体的单倍型多样度和核苷酸多样度分别为0.849和0.011,放流群体的单倍型多样度和核苷酸多样度分别为0.786~0.935和0.005~0.009,其中舟山放流场的放流群体遗传多样最高(见表 3)。整体上黑鲷亲鱼群体、放流群体以及海捕群体的遗传多样性均较高,海捕群体的遗传多样性高于放流群体的遗传多样性。
|
|
表 2 黑鲷各群体单倍型数目统计表 Table 2 Statistics of haplotypes in each population of Acanthopagrus schlegelii |
|
|
表 3 黑鲷群体遗传多样性指数 Table 3 Genetic diversity index of Acanthopagrus schlegelii populations |
基于黑鲷线粒体DNA控制区序列构建的单倍型网络图表明黑鲷群体不存在明显的谱系结构(见图 1)。群体间遗传分化指数FST值显示(见图 2),黑鲷亲鱼群体与放流群体、放流群体间以及放流群体与海捕群体间均产生中等程度的遗传分化。根据黑鲷群体来源,将黑鲷群体分为几个基因池进行AMOVA分析(见表 4),将8个群体作为一个基因池进行分析,结果显示群体内变异比例为93.76%,将黑鲷群体分为2个基因池(亲鱼群体和海捕群体为一个基因池,放流群体为一个基因池),结果显示群体内变异百分比为94.45%,按来源将黑鲷群体分为3个基因池(亲鱼群体、海捕群体和放流群体各为一个基因池),群体内变异百分比为94.39%,按地理区域将黑鲷群体分为3个基因池,群体内变异百分比为94.09%。综合以上结果,黑鲷群体的遗传变异主要来源于群体内。
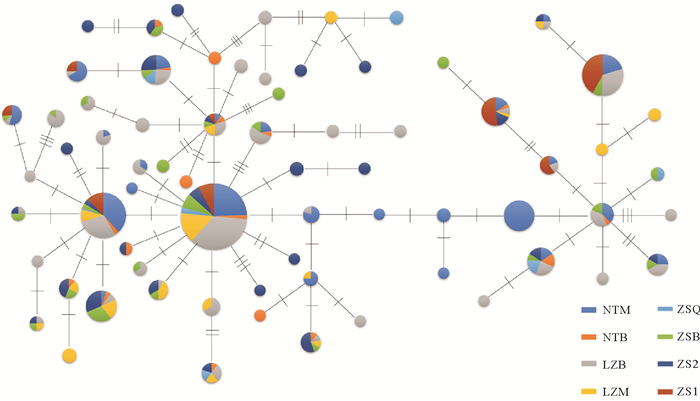
|
(连接单倍型的线上的垂直刻度线表示核苷酸替代的数量,圆圈的大小与单倍型频率成正比。The perpendicular tick marks on the lines joining haplotypes represent the number of nucleotide substitutions, and the sizes of circles were proportional to haplotypes frequency. ) 图 1 黑鲷群体的单倍型网络图 Fig. 1 Haplotype network diagram of Acanthopagrus schlegelii |
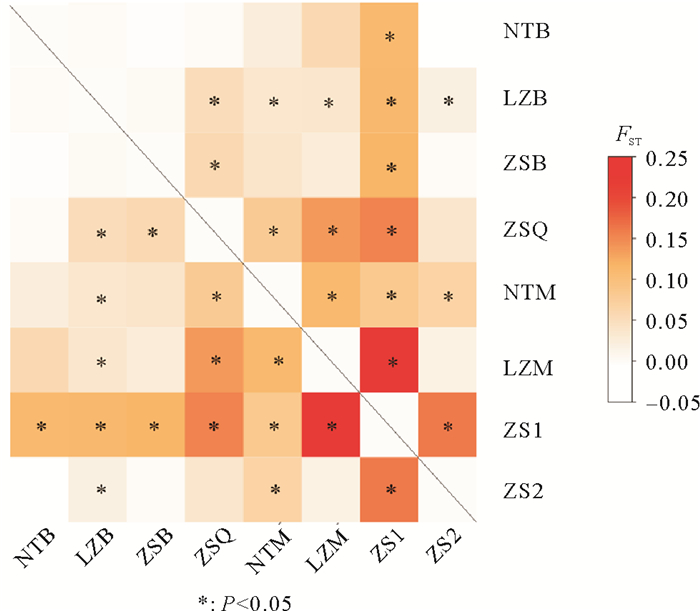
|
图 2 黑鲷群体间的遗传分化指数FST值 Fig. 2 Genetic differentiation FST among populations of Acanthopagrus schlegelii |
|
|
表 4 黑鲷群体的AMOVA分析 Table 4 AMOVA of Acanthopagrus schlegelii populations |
遗传变异水平高的物种会呈现出高的遗传多样性[20]。物种的遗传多样性越高,其进化潜力及环境适应性更强,越有利于保护物种多样性和生态系统的多样性,而遗传的均一性可能影响群体或物种的生存,应充分重视物种的群体遗传结构和遗传多样性的研究[21]。
在以往的研究中,大多是基于黑鲷野生群体的遗传多样性研究或养殖群体与野生群体的比较研究,而监测放流苗种对黑鲷野生种群影响的研究较少[11]。An等[22]利用6个微卫星位点对韩国济州岛的黑鲷养殖与野生群体进行遗传多样性分析,结果发现养殖群体有大量稀有等位基因丢失,且养殖与野生群体间存在显著的遗传异质性。Jeong等[23]采用微卫星技术对日韩海域6个黑鲷野生群体、1个养殖群体以及1个回捕群体进行了遗传多样性及遗传结构分析,结果显示8个黑鲷群体的遗传多样性水平均较高,回捕群体并未受到增殖放流活动的影响。在本研究中,海捕群体的单倍型多样度高于亲鱼群体的单倍型多样度,同时也高于除舟山放流场鱼苗以外的其他放流鱼苗的单倍型多样度。结合Jeong等[23]研究,本研究中黑鲷增殖放流活动或许尚未对海捕群体造成明显的遗传学影响。Wang等[24]在许氏平鲉(Sebastes schlegelii)的增殖放流活动研究中也发现类似的现象,结果表明,与野生种群相比,放流种群的遗传多样性没有明显降低,回捕群体的遗传多样性指数在短期内也没有明显变化,推测短期内的增殖放流活动不会对野生种群的遗传多样性造成影响。此外,本研究中的舟山放流场鱼苗来自两个不同的育苗场,亲本数量多且杂,所以相比较于其他放流苗种,遗传多样性较高。赵林林[25]对黑鲷养殖群体与野生群体的遗传多样性比较分析中也出现过养殖群体的遗传多样性指数(h:0.912 7,π:0.010 9)高于某些野生群体的遗传多样性指数(h:0.800 0~0.974 6,π:0.007 2~0.013 6)的情况,推测是亲本来源复杂导致的。大多数育苗场在进行育苗时未对亲本的数量及来源给予关注,导致苗种具有较低的遗传多样性[26]。且若亲本与野生群体的遗传多样性差异显著,那么苗种与野生群体的遗传差异会增大[27],一般表现为苗种的遗传多样性较低[28]。将育苗场孵化的苗种放流到自然海域中,有利于黑鲷的自然繁殖,使自然海区黑鲷资源量有所提升[4],但同时可能会干扰当地的基因库,对野生群体造成负面影响,例如等位基因或单倍型的损失等[29]。如Shan等[11]的研究结果显示开展黑鲷增殖放流活动的珠江口流域的黑鲷回捕群体的遗传多样性低于其他海域的黑鲷野生群体。
群体间遗传分化指数FST显示,黑鲷放流群体间、放流群体与亲鱼群体间以及放流群体与海捕群体间均产生显著的遗传分化。赵爽等[30]对我国黑鲷北方海域2个群体和南方海域3个群体的比较分析中发现北方海域群体之间、南方海域群体之间没有显著的遗传分化,但南北方群体之间存在中等程度的遗传分化,建议将中国近海黑鲷作为两个管理单元。Yamashita等[31]利用线粒体DNA控制区序列发现中国部分野生群体之间也存在南北方遗传分化现象,但日本近海的黑鲷野生群体间遗传分化较小。本研究中3个海捕群体同属于北方群体,群体间遗传分化较小,与这些研究结果一致。此外,我们发现放流群体与海捕群体的遗传分化较大,这可能是由于养殖场在繁育过程中使用较少的亲体导致的。Wang等[32]在对鞍带石斑鱼(Epinephelus lanceolatus)养殖群体与野生群体的比较分析中也发现了类似现象。当与本地群体遗传差异较大的苗种被放入到自然海域中时,可能会造成本地群体的特有基因丧失[33]。同时,黑鲷南北方群体间的遗传分化提示我们在进行增殖放流时应选择当地的苗种,以免外来苗种与当地自然海域的野生群体遗传分化过大而产生负面影响。
4 结语本研究中的黑鲷亲鱼群体、放流群体以及海捕群体遗传多样性均较高,但放流群体和海捕群体间存在较大的遗传分化。在今后的增殖放流活动中,应注意亲本群体的数量及来源,尽可能选择遗传多样性与野生群体相近的亲本群体,缩小放流苗种与野生群体间的遗传差距,减小放流群体对野生群体可能造成的遗传学影响,保护黑鲷种质资源,实现资源可持续利用。
| [1] |
伍汉霖, 邵广昭, 赖春福, 等. 拉汉世界鱼类系统名典[M]. 青岛: 中国海洋大学出版社, 2017: 238. Wu H L, Shao G Z, Lai C F, et al. Latin-Chinese Dictionary of Fish Names by Classification System[M]. Qingdao: China Ocean University Press, 2017: 238. (  0) 0) |
| [2] |
朱德芬. 黑鲷人工养殖技术讲座——第一讲黑鲷生物学特性及增养殖概况[J]. 水产养殖, 1996(1): 30-32. Zhu D F. Lecture on artificial culture technology of black sea bream—Lecture 1 biological characteristics and aquaculture of black sea bream[J]. Journal of Aquaculture, 1996(1): 30-32. (  0) 0) |
| [3] |
Law C S W, Sadovy de M Y. Reproductive biology of black seabream Acanthopagrus schlegelii, threadfin porgy Evynnis cardinalis and red pargo Pagrus major in the northern South China Sea with consideration of fishery status and management needs[J]. Journal of Fish Biology, 2017, 91(1): 101-125. DOI:10.1111/jfb.13331 (  0) 0) |
| [4] |
仲霞铭, 倪金弟, 汤建华, 等. 黑鲷全人工繁育及其增殖放流[J]. 水产养殖, 1998(5): 28-29. Zhong X M, Ni J D, Tang J H, et al. Full artificial breeding and proliferation and release of black sea bream[J]. Journal of Aquaculture, 1998(5): 28-29. (  0) 0) |
| [5] |
时金荣, 沈毓秀. 江苏南部沿海黑鲷的增殖放流[J]. 海洋渔业, 1994(4): 168. Shi J R, Shen Y X. Proliferation and release of black sea bream in the south coast of Jiangsu Province[J]. Marine Fisheries, 1994(4): 168. (  0) 0) |
| [6] |
郑亮, 杨振宇, 崔敬, 等. 山东省海阳市渔业资源增殖放流概况[J]. 渔业研究, 2021, 43(3): 322-329. Zheng L, Yang Z Y, Cui J, et al. A general situation of fishery resources enhancement in Haiyang City of Shandong Province[J]. Journal of Fisheries Research, 2021, 43(3): 322-329. (  0) 0) |
| [7] |
《中国水产》编辑部. 全国放鱼日, 增殖正当时, 农业农村部部长韩长赋出席增殖放流主会场活动[J]. 中国水产, 2018(7): 2-3. 《China Fisheries》Editorial Department. At the right time of national fish release day, Han Changfu, minister of agriculture and rural areas, attended the main venue of proliferation and release[J]. China Fisheries, 2018(7): 2-3. (  0) 0) |
| [8] |
曾诗淇. 2020年水生生物增殖放流活动在全国范围同步举行[J]. 农产品市场, 2020(12): 2. Zeng S Q. In 2020, aquatic biological proliferation and release activities will be held simultaneously throughout the country[J]. Agricultural Products Market, 2020(12): 2. (  0) 0) |
| [9] |
Hamasaki K, Toriya S, Shishidou H, et al. Genetic effects of hatchery fish on wild populations in red sea bream Pagrus major (Perciformes, Sparidae) inferred from a partial sequence of mitochondrial DNA[J]. Journal of Fish Biology, 2010, 77(9): 2123-2136. DOI:10.1111/j.1095-8649.2010.02826.x (  0) 0) |
| [10] |
Gonzalez E B, Nagasawa K, Umino T. Stock enhancement program for black sea bream (Acanthopagrus schlegelii) in Hiroshima Bay: Monitoring the genetic effects[J]. Aquaculture, 2008, 276(1-4): 36-43. DOI:10.1016/j.aquaculture.2008.02.004 (  0) 0) |
| [11] |
Shan B, Liu Y, Song N, et al. Genetic diversity and population structure of black sea bream (Acanthopagrus schlegelii) based on mitochondrial control region sequences: The genetic effect of stock enhancement[J]. Regional Studies in Marine Science, 2020, 35: 101188. DOI:10.1016/j.rsma.2020.101188 (  0) 0) |
| [12] |
Avise J C, Arnold J, Ball R M, et al. Intraspecific phylogeography: The mitochondrial DNA bridge between population genetics and systematics[J]. Annual Review of Ecology and Systematics, 1987, 18(1): 489-522. DOI:10.1146/annurev.es.18.110187.002421 (  0) 0) |
| [13] |
Sugaya T, Sato M, Yokoyama E, et al. Population genetic structure and variability of Pacific herring Clupea pallasiiin the stocking area along the Pacific coast of northern Japan[J]. Fisheries Science, 2008, 74: 579-588. (  0) 0) |
| [14] |
Guo E M, Liu Y, Cui Z X, et al. Genetic variation and population structure of swimming crab (Portunus trituberculatus) inferred from mitochondrial control region[J]. Molecular Biology Reports, 2012, 39(2): 1453-1463. (  0) 0) |
| [15] |
赵祥, 郑建, 高天翔, 等. 基于线粒体DNA控制区的黄姑鱼养殖群体与野生群体比较研究[J]. 中国海洋大学学报(自然科学版), 2021, 51(8): 11-19. Zhao X, Zheng J, Gao T X, et al. Comparative analysis of genetic variation in cultured and wild populations of Nibea albiflora based on mitochondrial DNA control region[J]. Periodical of Ocean University of China, 2021, 51(8): 11-19. (  0) 0) |
| [16] |
Iguchi K, Watanabe K, Nishida M. Reduced mitochondrial DNA variation in hatchery population of ayu (Plecoglossus altivelis) cultured for multiple generations[J]. Aquaculture, 1999, 178(3): 235-243. (  0) 0) |
| [17] |
Sambrook J, Fritsch E F, Maniatis T. Molecular Cloning: A Laboratory Manual[M]. New York: Cold Spring Harbor Laboratory Press, 1989.
(  0) 0) |
| [18] |
Lalitha S. Primer premier 5[J]. Biotech Software & Internet Report, 2000, 1(6): 270-272. (  0) 0) |
| [19] |
Excoffier L, Lischer H E. Arlequin suite ver 3.5: A new series of programs to perform population genetics analyses under Linux and Windows[J]. Molecular Ecology Resources, 2010, 10(3): 564-567. (  0) 0) |
| [20] |
Alarcón J A, Magoulas A, Georgakopoulos T, et al. Genetic comparison of wild and cultivated European populations of the gilthead sea bream (Sparus aurata)[J]. Aquaculture, 2004, 230(1-4): 65-80. (  0) 0) |
| [21] |
施立明. 遗传多样性及其保存[J]. 生物科学信息, 1990(4): 158-164. Shi L M. Genetic diversity and its preservation[J]. Chinese Bulletin of Life Science, 1990(4): 158-164. (  0) 0) |
| [22] |
An H S, Hong S W, Lee J U, et al. Genetic diversity of wild and farmed black sea bream populations in Jeju[J]. Animal Cells and Systems, 2010, 14(1): 37-44. (  0) 0) |
| [23] |
Jeong D S, Umino T, Kuroda K, et al. Genetic divergence and population structure of black sea bream Acanthopagrus schlegelii inferred from microsatellite analysis[J]. Fisheries Science, 2003, 69(5): 896-902. (  0) 0) |
| [24] |
Wang L, Wu Z, Wang Y, et al. Genetic assessment of a black rockfish, Sebastes schlegelii, stock enhancement program in Lidao Bay, China based on mitochondrial and nuclear DNA analysis[J]. Frontiers in Marine Science, 2020, 7: 94. (  0) 0) |
| [25] |
赵林林. 两种海洋鱼类的遗传多样性及基于线粒体基因组的石首鱼科系统发育研究[D]. 青岛: 中国海洋大学, 2015: 101-114. Zhao L L. Genetic Diversity of Two Marine Fish and Phylogeny of Sciaenidae Based on Mitochondrial Genome[D]. Qingdao: Ocean University of China, 2015: 101-114. (  0) 0) |
| [26] |
Jonsson B, Jonsson N. Cultured Atlantic salmon in nature: A review of their ecology and interaction with wild fish[J]. ICES Journal of Marine Science, 2006, 63(7): 1162-1181. (  0) 0) |
| [27] |
Bert T M, Crawford C R, Tringali M D, et al. Genetic management of hatchery-based stock enhancement[M]//Ecological and Genetic Implications of Aquaculture Activities. Dordrecht: Springer, 2007: 123-174.
(  0) 0) |
| [28] |
杨爽, 宋娜, 张秀梅, 等. 基于线粒体控制区序列的三疣梭子蟹增殖放流亲蟹遗传多样性研究[J]. 水产学报, 2014, 38(8): 1089-1096. Yang S, Song N, Zhang X M, et al. Genetic diversity of swimming crab (Portunus trituberculatus) from four broodstock populations in stock enhancement inferred from mitochondrial control region[J]. Journal of Fisheries of China, 2014, 38(8): 1089-1096. (  0) 0) |
| [29] |
Sekino M, Hara M, Taniguchi N. Loss of microsatellite and mitochondrial DNA variation in hatchery strains of Japanese flounder Paralichthys olivaceus[J]. Aquaculture, 2002, 213(1-4): 101-122. (  0) 0) |
| [30] |
赵爽, 章群, 乐小亮, 等. 中国近海5个黑鲷地理群体的遗传变异[J]. 海洋科学, 2010, 34(2): 75-79. Zhao S, Zhang Q, Yue X L, et al. Genetic variation among 5 stocks of Acanthopagrus schlegeli in China's coastal waters[J]. Marine Sciences, 2010, 34(2): 75-79. (  0) 0) |
| [31] |
Yamashita Y, Sanchez G, Kawai K, et al. The role of the isolation of the marginal seas during the Pleistocene in the genetic structure of black sea bream Acanthopagrus schlegelii(Bleeker, 1854) in the coastal waters of Japan[J]. PeerJ, 2021, 9: e11001. (  0) 0) |
| [32] |
Wang Q, Wang X, Xie Z, et al. Microsatellite analysis of the genetic relationships between wild and cultivated giant grouper in the South China Sea[J]. Journal of Genetics, 2016, 95(2): 369-376. (  0) 0) |
| [33] |
Einum S, Fleming I A. Implications of stocking: Ecological interactions between wild and released salmonids[J]. Nordic Journal of Freshwater Research, 2001, 75: 56-70. (  0) 0) |
2. East China Sea Fishery Research Institute, Chinese Academy of Fishery Sciences, Shanghai 200090, China;
3. Jiangsu Marine Fisheries Research Institute, Nantong 226007, China;
4. Fishery College, Zhejiang Ocean University, Zhoushan 316022, China
 2023, Vol. 53
2023, Vol. 53


