2. 唐山海都水产食品有限公司,河北 唐山 063000;
3. 青岛海洋科学与技术试点国家实验室,山东 青岛 266237
水产养殖系统中栖息着多种多样的微生物群落,这些微生物在生态系统物质循环和能量流动中起着关键作用[1-2]。同时,这些环境中的微生物对环境变化响应非常迅速,也是监测水质变化和病害爆发的重要生物指标[3-4]。而养殖生物肠道中的微生物则与其机体的健康状况直接相关。研究表明,鱼类肠道微生物在宿主肠道上皮细胞更新、营养物质消化吸收、介导免疫反应等方面均扮演着不可或缺的角色[5-8]。由于鱼类长期直接暴露在水中,其肠道微生物更易受到外界环境的影响。大量研究表明,鱼类肠道微生物与环境微生物之间存在密切的相互作用。例如,Giatsis等发现罗非鱼(Oreochromis niloticus)肠道菌群与养殖水体菌群共享大量OTUs,说明微生物可从水中成功转移到肠道[9],而Wu等则发现沉积物是异育银鲫(Carassius auratus gibelio)肠道菌群的重要来源[10]。这种肠道微生物与养殖环境微生物之间的紧密联系,使二者可被看作一个有机的整体。而环境压力引起的环境微生物群落的变化,会影响系统中微生物之间的网络关系,最终有可能导致宿主肠道菌群的失调并爆发疾病[11-12]。因此,了解水产养殖生态系统中微生物群落的结构与多样性以及环境微生物和宿主微生物之间的相互作用,对于监测养殖生物健康状态、防控水产病害发生、维持生态系统平衡等都具有重要的意义[13-14]。
近年来,在土地和水资源短缺的情况下,水产养殖系统呈现出典型的集约化发展趋势[15]。然而,简单的集约化发展是一把“双刃剑”,其负面影响不容忽视[15-16]。池塘养殖是中国水产养殖业的主要生产方式之一,其产量占到了中国水产养殖总产量的48.84%(中国渔业统计年鉴,2020)[17]。而在常规的池塘集约化养殖模式中,养殖水体既是养殖生物的生活区,也是残饵、粪便的分解区[18],这很容易造成养殖水体水质下降和水产病害爆发[19-20]。同时,养殖活动产生的污染物向邻近水域的无序排放,也进一步给养殖周边区域水体环境带来负面影响[21]。池塘跑道养殖系统(In-Pond Raceway System, IPRS)又称为池塘工程化循环水养殖系统,是由美国奥本大学和美国大豆出口协会设计并推广到我国的一种养殖模式,从原理上属于生态集约化养殖模式[22]。通常,一个典型的池塘跑道养殖系统主要由鱼类集约化养殖区、水质生态净化区和固体废弃物收集区等几个功能区构成。IPRS借助推水装置和功能分区在池塘中实现了循环微流水养殖,既可以通过局部集约化提高鱼类养殖产量,又可以通过固体废弃物的集中处理和养殖(种植)净水生物解决池塘养殖的自身污染问题,从而有效提高池塘养殖的经济、社会和生态效益。
据不完全统计,中国养殖跑道已经超过6 000条,养殖品种达到20余种,主要应用在淡水养殖环境,而海水IPRS的报道尚较少[23]。此外,目前中国不同地区、不同模式IPRS的养殖效果、生态和经济效益还存在较大差异,IPRS还有很多技术问题需要研究与优化。当前国内外对IPRS的研究多集中在技术推广应用方面,而对于其生态系统的组成、结构和功能特征等基础理论研究关注不足,阻碍了对IPRS的进一步优化与完善。本研究采用16S rRNA基因高通量测序技术首次对海水池塘跑道养殖系统中养殖鱼类肠道、水体和沉积物细菌群落的结构和多样性进行研究,以期查明不同细菌群落之间的差异,并进一步探究IPRS中养殖鱼类肠道菌群和环境菌群之间的相互作用关系。通过对海水池塘跑道养殖系统不同生境中细菌群落的表征和比较,不仅有助于从微生态角度了解海水池塘跑道养殖系统结构和功能特点,也可为IPRS养殖管理策略的优化及养殖技术的完善提供理论参考。
1 材料和方法 1.1 养殖系统本研究于2020年在河北省唐山市海都水产食品有限公司的养殖基地(39°03′32″N,118°17′24″E)进行,以中国花鲈(Lateolabrax maculatus)和红鳍东方鲀(Takifugu rubripes)为主养种类的海水池塘跑道养殖系统(IPRS)作为研究对象。该系统所在池塘为泥沙质底,面积约为4 hm2,养殖期间水深维持在为1.8~2.0 m。IPRS系统的平面结构如图 1所示,主要由鱼类集约化养殖区、固体废物收集区和水质净化区等构成。在池塘鱼类集约化养殖区修建了8个20 m×5 m×2 m的流水槽用于养殖花鲈,放养密度为5 000尾/槽,集约化养殖区的面积约占池塘总面积的2.1%。养殖区后部为水生植物浮床,种植具有净水作用的海马齿(Sesuvium portulacastrum),种植区约占池塘总面积的8.5%左右。沿着水流在种植区后投放硬壳蛤(Mercenaria mercenaria),投放区域约占池塘总面积的13.5%。此外,为了尽可能利用水体资源和增加池塘生产力,在净化区内养殖红鳍东方鲀,放养密度为6 750尾/hm2。
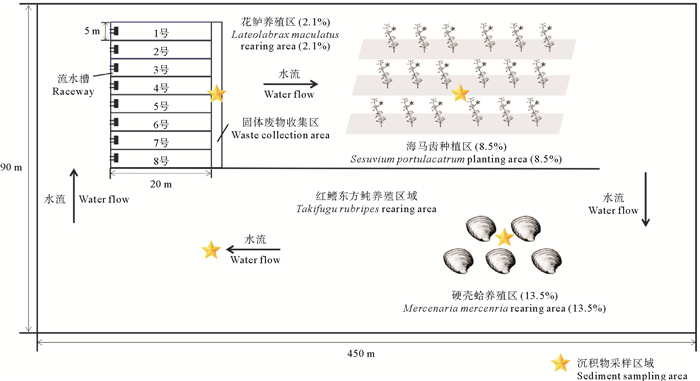
|
图 1 海水池塘跑道养殖系统平面示意图 Fig. 1 Sketch diagram of the marine in-pond raceway pond system |
养殖过程中不换水,投喂红鳍东方鲀和花鲈的饵料均为冰鲜的玉筋鱼(Ammodytes personatus),并根据生长状况、天气条件、水温变化和摄食情况等因素合理地调整投喂量和投喂频率。
1.2 样品采集在养殖中期,分别选取和采集池塘跑道养殖系统中花鲈肠道(BI)、河鲀肠道(PI)、水体(W)、固体废物收集区沉积物(S1)、海马齿种植区沉积物(S2)、硬壳蛤养殖区沉积物(S3)以及非养殖(种植)净水区沉积物(S4)在内的7种不同生境的微生物样品,每组样品分别采集5个重复。
对于水体样品,沿着循环水流方向均匀地设置5个采样点,在每个采样点用灭菌后的有机玻璃采水器从水面下0.5 m处采集1 L水样,然后立即放在冰上运回实验室。使用0.22 μm聚合纤维滤膜对水样进行过滤后,将滤膜置于2 mL无菌冻存管并保存在-80 ℃冰箱中。沉积物样品的具体采样区域如图 1所示,在每个采样区域中设置5个均匀分布的采样点,使用灭菌后的采泥器从沉积物表层(0~5 cm)采集样品,泥样采集后立即置于无菌冻存管并储存于液氮中。对于肠道样品,用MS-222(间氨基苯甲酸乙酯甲磺酸盐)将鱼麻醉后,在无菌条件下,先用75%的酒精擦拭鱼体表面,再用灭菌后的剪刀和镊子剖开腹部,取出肠道并挤出内容物,用少量的磷酸盐缓冲液(PBS)冲洗后放入无菌冻存管中,收集3条鱼的肠道作为1个独立的平行样品,随后立即储存在-80 ℃冰箱中等待下一步实验。
1.3 DNA提取与16S rRNA基因测序根据说明书的指示,使用MagPure Soil DNA LQ Kit试剂盒(美基生物,广州,中国)对样本中的DNA进行提取,利用琼脂糖凝胶电泳和NanoDrop2000仪器(NanoDrop Technologies, Wilmington, 美国)检测抽提DNA的质量和浓度。随后以提取的DNA为模板,根据选择的测序区域,使用带barcode的特异引物进行PCR扩增。在本研究中,为了研究细菌群落的多样性,使用通用引物343F(5′-TACGGRAGGCAGCAG-3′)和798R(5′-AGGGTATCTAATCCT-3′)对16S rRNA基因中的V3~V4高变区进行特异扩增。第一轮PCR体系组成以及具体反应流程如表 1所示。第一轮扩增产物通过琼脂糖凝胶电泳进行检测和分离,并使用Agencourt AMPure XP beads(Beckman Coulter Inc., Brea, CA, 美国)进行纯化。纯化后的扩增产物随即进行第二轮PCR,以添加测序接头,体系组成以及具体反应流程如表 2所示。
|
|
表 1 第一轮PCR反应体系与流程 Table 1 First round PCR reaction system and procedure |
|
|
表 2 第二轮PCR反应体系与流程 Table 2 Second round PCR reaction system and procedure |
二轮扩增产物经过再次纯化后,使用Qubit dsDNA assay kit(Life Technologies, Carlsbad, CA, 美国)检测浓度并构建等量的文库。高通量测序在欧易生物技术有限公司的Illumina Miseq平台上进行。最终,从测序平台上获得原始的双端序列数据(Paired-end sequence)。
1.4 生物信息分析原始数据通过barcode序列拆分后生成独立的Fastq格式文件,然后在QIIME2开源平台上进行菌群组成和多样性分析[24]。使用DADA2算法对原始的双端序列执行减除低质量碱基、降噪、合并以及去除嵌合体等过程以获得有效的特征表和代表序列[25]。在QI-IME2平台上,基于SILVA(132-release)数据库训练了一个专门针对扩增片段(343F/798R)的朴素贝叶斯分类器(Naive Bayes classifier)[26-29],并对代表序列进行物种分类注释。为了便于下游分析,根据最小的样品序列数作为稀释深度对特征表进行均一化处理。利用QIIME2的“phylogeny”插件构建有根系统发育树,用于后续微生物多样性分析。
使用QIIME2的“diversity”插件计算Shannon指数、Simpson指数、Chao1指数和Pielou’s均匀度指数等,以评估细菌群落的α多样性,α多样性数值通过Kruskal-Wallis检验进行组间比较,取P < 0.05为差异显著。通过基于Bray-Curtis距离的主坐标分析(PCoA)来评估细菌群落在整体结构上的差异,并在R(version 4.1.0)上使用“Vegan”包来执行PERMANOVA分析以检验群落结构差异的显著性[30]。构建基于Unweighted unifrac距离的层级样品聚类分析以探究不同样品间的系统发育关系。利用LEfSe(Linear discriminant analysis effect size)分析来鉴别群落间的差异分类群[31],|LDA score|>3.5且P < 0.05被设置为筛选的阈值。Venn分析在R语言上使用“VennDiagram”包进行,相似性分析在R语言上使用“Vegan”包进行[32]。
2 结果 2.1 细菌群落多样性分析经过QIIME2平台处理后,从35个微生物样品中共计获得1 676 433条高质量双端合并序列。这些序列被聚类到16 200个ASVs(Amplicon sequence variant)特征,平均每个样品约有867个ASVs。为了便于后续分析,对获得的ASV特征表进行均一化处理,以20 966作为稀释深度对所有样品进行重抽样。样品的稀释曲线如图 2所示,随着随机抽样测序深度的不断增加,仅能发现极少量的新特征,这表明测序深度已经饱和,样品具有代表性。
|
( BI、PI、S1、S2、S3、S4和W分别代表花鲈肠道、红鳍东方鲀肠道、固体废物收集区沉积物、海马齿种植区沉积物、硬壳蛤养殖区沉积物、非养殖(种植)净水区沉积物以及水体微生物样品。下同。BI、PI、S1、S2、S3、S4 and W are microbial samples from gut of spotted seabass, gut of tiger puffer, sediment in solid waste collection area, sediment in Sesuvium portulacastrum planting area, sediment in Mercenaria mercenaria rearing area, sediment in non-rearing (planting) area and water. The same as blow. ) 图 2 微生物样品的稀释曲线 Fig. 2 Rarefaction curves of microbial samples |
如图 3所示,IPRS不同生境中细菌群落在α多样性上存在显著差异(P < 0.05),其顺序从大到小依次为:沉积物>肠道>水体。两种养殖鱼类肠道菌群的α多样性也存在显著差异,其中花鲈的Pielou’s均匀度、Simpson指数和Shannon指数均显著高于红鳍东方鲀(P < 0.05)。然而,不同功能区中的沉积物菌群在α多样性上无显著差异(P>0.05)。
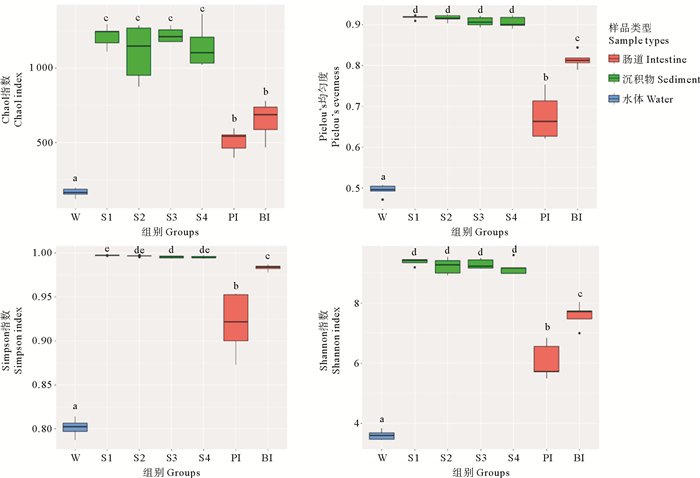
|
图 3 IPRS不同生境中细菌群落的α多样性比较分析 Fig. 3 Comparative analysis of alpha diversity of bacterial communities in different habitats of IPRS |
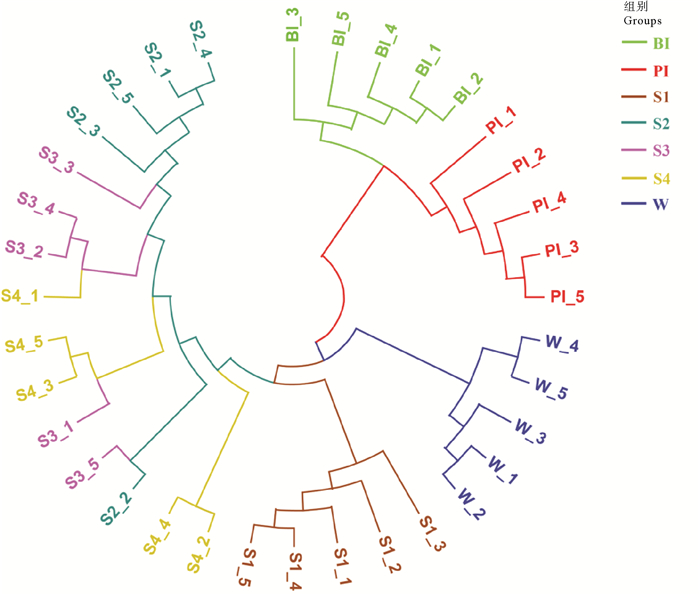
IPRS不同生境细菌群落PCoA分析结果如图 4(a)所示,前两个成分共解释了50.98%的差异。代表水体、沉积物、河鲀肠道和花鲈肠道样品的点在坐标轴中出现明显的分离,非参数多元方差分析(PERMANOVA)进一步表明这4个生境的菌群结构存在显著差异(P < 0.05)。基于Unweighted unifrac距离的层级聚类分析结果如图 5所示。可以看出,水体、沉积物和鱼类肠道样品分别隶属于不同分支,表明生活在相同类型栖息环境中的细菌群落具有更加相近的遗传发育关系。IPRS不同功能区中沉积物菌群的PCoA分析结果如4(b)所示,4组样品相互之间均存在显著的差异(P < 0.05)。其中,S1与净化区沉积物样品的差异较大。这种现象同样也在层级聚类分析中被发现(见图 5),固体废物收集区沉积物样品被单独聚为一簇,而净化区沉积物样品则被聚到另一簇。
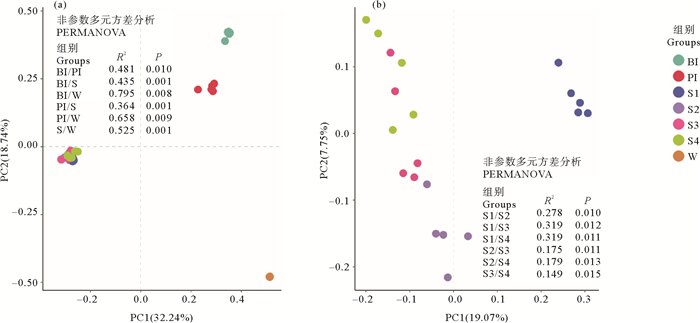
|
图 4 基于Bary-Curtis距离的所有样品(a)和沉积物样品(b)的PCoA分析 Fig. 4 PCoA analysis based on Bary-Curtis distance for all samples (a) and sediments samples (b) |
|
图 5 基于unweighted Unifrac距离的层级聚类分析 Fig. 5 Hierarchical clustering analysis based on unweighted Unifrac distance |
对特征表进行均一化处理后,保留了15 732个ASVs,平均每个样品约有847个ASVs,这些ASVs可被分类到52个有效菌门和1 221个有效菌属。
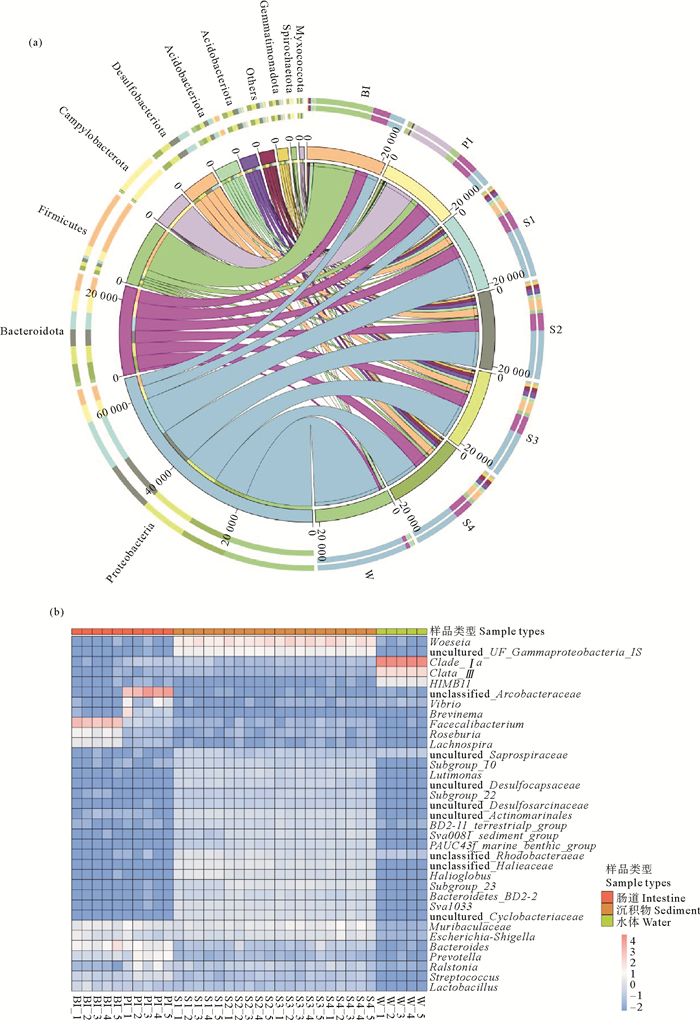
IPRS不同生境中细菌群落在门水平上的优势组成如图 6(a)所示。河鲀肠道中的主要优势菌门为弯曲杆菌门(Campylobacterota,41.17%)、拟杆菌门(Bacteroidota,20.92%)、变形菌门(Proteobacteria,18.25%)和厚壁菌门(Firmicutes,10.29%)等;花鲈肠道中的主要优势菌门为厚壁菌门(57.92%)、拟杆菌门(17.13%)、变形菌门(15.97%)和放线菌门(Acti-nobacteriota,4.77%)等;沉积物中的主要优势菌门为变形菌门(48.06%)、拟杆菌门(17.79%)、脱硫杆菌门(Desulfobacterota,10.53%)和酸杆菌门(Acidobacteria,5.07%)等;水体中的优势菌门为变形菌门(91.03%)和拟杆菌门(3.73%)。
|
图 6 IPRS不同生境中细菌群落在门(a)和属(b)水平的优势组成 Fig. 6 Dominant composition of bacterial communities in different habitats of IPRS at phylum (a) and genus (b) levels |
属水平上的优势组成如图 6(b)所示。通过热图可以看出,不同生境中细菌群落的优势菌属存在明显差异。花鲈肠道中相对丰度最高的优势菌属为栖粪杆菌属(Faecalibacterium,28.67%),其次为拟杆菌属(Bacteroides,7.79%)、罗氏菌属(Roseburia,5.20%)和Muribaculaceae(4.16%);在河鲀肠道中,一些来自弓形杆菌科的未知菌属unclassified_Arcobacteracea占据了最高丰度(41.10%),其次为普雷沃菌属(Prevotella,5.98%)、拟杆菌属(5.68%)和Muribaculaceae(4.22%);沉积物中的主要优势菌属有伍斯菌属Woeseia(14.75%)、Muribaculaceae(4.29%)、unclassified_Halieaceae(2.98%)和Subgroup_23(2.64%)等;水体中的主要优势菌属有Clade_Ⅰa(62.54%)、Clade_Ⅲ(17.39%)和HIMB11(4.49%)。
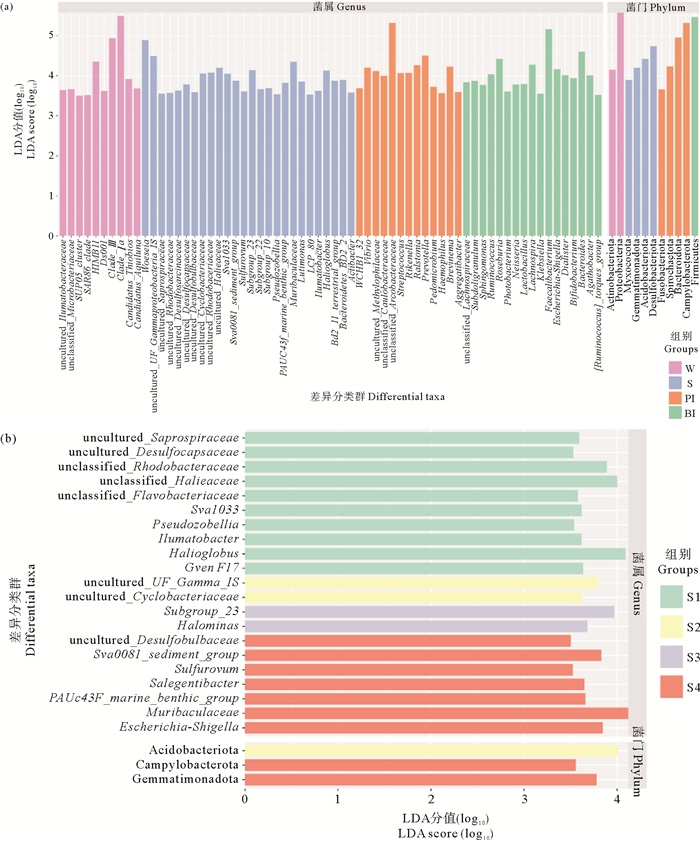
2.3 细菌群落组成差异分析IPRS不同生境中细菌群落LEfSe分析结果如图 7(a)所示。在门水平上,变形菌门和放线菌门在水体中显著富集;脱硫杆菌门、酸杆菌门、芽单胞菌门(Gemmatimonadota)和黏菌门(Myxomycota)在沉积物中显著富集;弯曲杆菌门、拟杆菌门、螺旋体门(Spirochaetota)和梭杆菌门(Fusobacteriota)在河鲀肠道中显著富集;而在花鲈肠道内,仅厚壁菌门出现显著的富集。在属水平上,26个差异菌属在沉积物中显著富集,主要包括伍斯菌属、Muribaculaceae、unclassified_Halieaceae、Subgroup_23和Halioglobus等;20个差异菌属在花鲈肠道中显著富集,主要包括栖粪杆菌属、拟杆菌属、罗氏菌属、毛螺菌属(Lachnospira)和埃希氏-志贺氏杆菌属(Escherichia-Shigella)等;12个差异菌属在河鲀肠道中显著富集,主要包括unclassified_Arcobacteraceae、普雷沃菌属、罗尔斯顿菌属(Ralstonia)、短螺旋体属(Brevinema)和弧菌属(Vibrio)等;10个差异菌属在养殖水体中显著富集,主要包括Clade_ Ⅰa、Clade_Ⅲ、HIMB11、Candidatus_Thiobios和Candidatus_Aquiluna等。
|
( (a) 基于IPRS鱼类肠道、沉积物和水体菌群;(b) 基于不同功能区沉积物菌群。LDA分值经过log10运算转换。(a) Based on fish gut, sediment and water bacterial communities in IPRS; (b) Based on sediment bacterial communities in different functional areas. LDA scores converted by log10. ) 图 7 LEfSe分析中差异类群(门和属水平)的LDA分值 Fig. 7 LDA scores of differential taxa (phylum and genus level) in LEfSe analysis |
IPRS不同功能区中沉积物菌群LEfSe分析结果如图 7(b)所示。在门水平上,酸杆菌门在S2中显著富集,弯曲杆菌门和芽单胞菌门在S4中显著富集,而在S1和S3中未出现显著富集的菌门。在属水平上,uncultured_Saprospiraceae、uncultured_Desulfocapsaceae、unclassified_Rhodobacteraceae、unclassified_Halieaceae、unclassified_Flavobacteriaceae、Sva1033、假佐贝尔氏菌属(Pseudozobellia)、Ilumatobacter、Halioglobus和Gven_F17在S1中显著富集;uncultured_Cyclobacteriaceae和一些来自于Gammaproteobacteria_Incertae_Sedis菌目的不可培养细菌在S2中显著富集;Subgroup_23和Halomonas在S3中显著富集;uncultured_Desulfobulbaceae、Sva0081_sediment_group、硫卵菌属(Sulfurovum)、需盐杆菌属(Salegentibacter)、Muribaculaceae、PAUC43f_marine_benthic_group和埃希氏-志贺氏杆菌属在S4中显著富集。
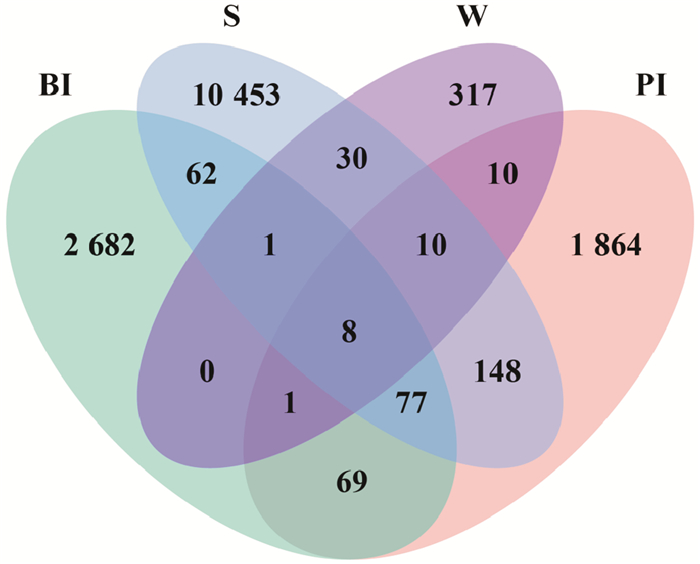
2.4 细菌群落相似性分析IPRS不同生境细菌群落Venn分析结果如图 8所示。可以看出,所有微生物样品共有8个ASVs。河鲀肠道样品与水体和沉积物样品共有29和243个ASVs,分别占河鲀肠道样品总ASVs数的1.33%和11.11%;花鲈肠道样品与水体和沉积物样品共有10和148个ASVs,分别占花鲈肠道样品总ASVs数的0.34%和5.10%。
|
图 8 IPRS不同生境细菌群落Venn分析 Fig. 8 Venn analysis of bacterial communities in different habitats of IPRS |
IPRS不同细菌群落间的相似性分析结果如表 3所示。可以看出,S1、S2、S3和S4均属于沉积物样品,故拥有最高的组间相似性(均在35%以上)。而水体样品较特殊,与其他样品的平均相似度小于1%。鱼类肠道样品与沉积物样品的平均相似度为3.43%,大于肠道样品与水体样品的0.48%。花鲈肠道样品与沉积物样品的平均相似度为2.85%,低于河鲀肠道样品与沉积物样品的平均相似度(4.00%)。
|
|
表 3 菌群相似性分析 Table 3 Similarity percentage analysis of bacterial communities |
近年来,IPRS作为一种高效的养殖系统,已经在斑点叉尾鮰(Ictalurus punctatus)[33]、杂交鲶鱼(Ictalurus punctatus ×I. furcatus)[34]、草鱼(Ctenopharyngodon idella)、尼罗罗非鱼(Oreochromis niloticus)[35]、大口黑鲈(Micropterus salmoides)[36]、宝石鲈鱼(Scortum barcoo)[37]和梭鱼(Liza haematocheila)[38]等鱼类的集约化养殖中进行较多的推广应用。这种模式对于改进传统池塘养殖技术以实现增产和生态可持续性之间的最佳平衡具有重要意义,对于传统池塘养殖技术的升级换代具有重要的参考价值。然而,目前国内外对IPRS的研究多集中在技术应用方面,而对于其生态系统的组成、结构和功能特征等基础理论研究关注尚不足,阻碍了对IPRS的进一步优化与完善。因此,研究IPRS细菌群落的结构和多样性将有助于从微生态角度进一步了解这一生态系统特征,为制定更好的养殖管理策略提供科学依据。
3.1 IPRS细菌群落组成和多样性在水产养殖系统中,微生物群落特征可以作为反映生态系统状态的指标[39]。因此,了解系统中微生物群落的组成和多样性对于维护生态系统平衡、防控水产病害发生具有重要意义。本研究利用16S rRNA基因高通量测序技术,比较了IPRS不同生境(水体、沉积物和鱼类肠道)中细菌群落的特征。可以看出,水体、沉积物和鱼类肠道菌群在多样性、结构与优势种组成上均存在显著差异。多样性分析结果表明,沉积物菌群的α多样性最高,显著高于水体和肠道,相似现象也存在于混养鲢(Hypophthalmichthys molitrix)鳙(H. nobilis)鱼[40]以及单养花鲈[41]、鲤鱼(Cyprinus carpio)[42]的常规池塘养殖系统中。而层级聚类和PCoA分析结果发现,栖息在同种生境中的细菌群落具有更加相近的系统发育关系,而不同生境的菌群在结构上则存在显著差异。研究表明,遗传距离相近的细菌类群具有更加相似的生态位[43],因此,在不同的环境压力选择下[44],不同生境中往往聚集了不同生态位的细菌群落,这一机制可以在一定程度上解释本研究中获得的结果。
IPRS不同生境比较发现,水体、沉积物和鱼类肠道菌群的优势种组成存在明显差异。在养殖水体中,来自于变形菌门的Clade_Ⅰa、Clade_Ⅲ和HIMB11占据了绝对的丰度优势,累积相对丰度达84.42%。其中,Clade_Ⅰa和Clade_Ⅲ均属于菌目SAR11_clade的分支,而SAR11_clade在海洋中大量存在,占到了海洋表层浮游细菌群落的三分之一[45]。在沉积物菌群中,最优势的类群是来自变形菌门的伍斯菌属。伍斯菌属是近些年被分离鉴定出来的新物种,是海洋沉积物细菌区系中的核心成员,广泛存在于我国北方沿海的沉积物环境中[46-48]。两种鱼类比较可见,在花鲈肠道菌群中,厚壁菌门、拟杆菌门和变形菌门占据了最高丰度,这与文献[49-50]的研究结果一致,这些细菌被普遍认为是花鲈肠道菌群中的优势类群。除此之外,栖粪杆菌属在花鲈肠道中也显著富集。栖粪杆菌是人和动物肠道中最具优势的细菌之一[51],这类细菌可以产生丁酸和具有生物活性的抗炎分子(如莽草酸和水杨酸)[52],对于维持肠道微生态平衡具有重要作用。与此同时,尽管拟杆菌属在两种养殖动物的肠道内均被大量检出,但在花鲈肠道内显著富集。这类细菌能在肠道内发酵植物来源的底物,并通过产生高水平的短链脂肪酸为宿主提供额外的能量[53-54]。与花鲈相比,河鲀肠道的弯曲杆菌门、拟杆菌门、变形菌门和厚壁菌门在丰度上占据绝对优势,累积相对丰度可达90.63%。在属水平上,部分来自弓形菌科的未知菌属unclassified_Arcobacteracea在河鲀肠道中显著富集。弓形菌科的成员广泛分布在水生环境中[55],这类细菌往往与人类和动物的疾病密切相关,例如Aliarcobacter butzleri和Aliarcobacter cryaerophilus已经被国际食品微生物标准委员会列为严重危害人类健康的细菌[56]。从以上两种养殖鱼类肠道菌群组成分析可以发现,花鲈和河鲀肠道菌群存在显著差异,部分潜在有益菌在花鲈肠道中大量富集,而一些潜在致病菌则在河鲀肠道中大量富集。然而,这一现象是鱼类种类(宿主遗传)差异造成的,还是两种鱼类被养殖在IPRS不同功能区生境(流水槽内、外)中造成的,尚需要进一步研究确定。
3.2 不同功能区沉积物细菌群落结构与组成池塘跑道养殖系统与常规池塘养殖系统最大的区别在于养殖区和净化区的划分[18, 34]。在本研究的IPRS中,构建了固体废物收集区、海马齿种植区和硬壳蛤养殖区,以期通过固体废物集中处理和生物净水的手段来实现养殖尾水的循环利用,最终达到生态集约化养殖的目的。本研究发现,IPRS不同功能区沉积物菌群在结构上存在显著差异,固体废物收集区和净化区的差异尤为明显,层级聚类分析也表明二者沉积物中的细菌具有相对较远的系统发育关系。从差异类群看,固体废物收集区沉积物中显著富集了一些能够降解有机物和去除过剩营养盐的细菌。例如,Sva1033和uncultured_Desulfocapsaceae具有还原硫酸盐的能力[57],而unclassified_Flavobacteriaceae和unclassified_Rhodobacteraceae则分别来自黄杆菌科和红杆菌科,这两个科的细菌已被广泛证实具有降解有机物和去除氮、磷营养盐的能力,经常在一些在污水处理系统中被大量发现[58-59],这一群落特征也与固体废物收集区的功能相匹配。不同净化区中沉积物菌群在组成上也存在一定的差异。其中,海马齿种植区沉积物中显著富集了uncultured_Cyclobacteriaceae和一些来自于Gammaproteobacteria_Incertae_Sedis菌目的不可培养细菌,而环杆菌科(Cyclobacteriaceae)种属在人工湿地沉积物[60]和红树林根际土壤[61]中也被大量发现。在硬壳蛤养殖区沉积物中,盐单胞菌属出现显著的富集,这是一种嗜盐反硝化细菌,对高盐废水具有良好的脱氮作用[62]。在未养(种)殖生物的其他净化区沉积物中则显著富集了一些与硫循环相关的细菌,如硫卵菌属、Sva0081_sediment_group和uncultured_Desulfobulbaceae等[63-65]。沉积物是物质交换和营养循环的重要场所[66],其中的微生物是碳、氮和磷等生物地球化学循环的重要参与者[67],与水体污染和富营养化直接相关[68]。因此,对不同功能区中沉积物细菌群落的研究,有助于从微生态角度了解IPRS生态系统的功能和状态。从本研究结果总体分析,池塘跑道养殖系统由于功能区的划分形成了许多小生境,而在这些小生境沉积物中生存的细菌群落也具有明显不同的组成与功能。
3.3 鱼类肠道菌群和环境菌群的关联了解水产养殖系统中鱼类肠道微生物和环境微生物间的相互作用关系,有助于制定有效的微生态调控策略以促进养殖鱼类的健康。本研究对养殖鱼类肠道菌群与环境菌群进行了比较,发现鱼类肠道样品与沉积物样品共有更多的ASVs,且鱼类肠道菌群与沉积物菌群的平均相似度为3.43%,高于与水体菌群的相似度(0.48%)。这一研究结果与Sun等的发现相似,在海水混养池塘中,鱼类肠道菌群与沉积物菌群共有更多的OTUs[69]。Zhang等也发现鲫鱼(Carassius auratus)肠道菌群中的优势类群梭杆菌门(Fusobacteria)主要来源于沉积物,而水体可能仅作为传播的媒介[42]。以上研究结果表明,沉积物菌群与养殖鱼类肠道菌群之间可能存在更加紧密的联系。
进一步比较发现,河鲀肠道样品与沉积物样品共有更多的ASVs,且河鲀肠道菌群与沉积物菌群的平均相似度为4.00%,高于花鲈肠道菌群与沉积物菌群的平均相似度(2.85%)。这表明河鲀肠道菌群与沉积物菌群在组成上具有更高的相似度,说明河鲀肠道菌群受到沉积物的影响相对更大。上述结果可能与两种鱼类的具体养殖方式及生活习性有关。在本研究中,花鲈被放养在IPRS的流水槽内,而红鳍东方鲀则被放养在相对开阔的流水槽外区域。红鳍东方鲀是一种中下层鱼类,夏季甚至可以潜入底泥中躲避高温,食性则主要以底栖或埋栖动物(如贝类、甲壳类)和底层鱼类为主[70]。因此,养殖在流水槽外区域的红鳍东方鲀会因为捕食行为及潜泥习性而与沉积物频繁接触,增加了微生物进入肠道并成功定植的机会[7]。而花鲈因为养殖在流水槽中,其活动受到限制,缺乏直接接触底泥或沉积物的机会,导致其肠道菌群组成受环境细菌的影响较小。与此类似,室内循环水养殖的相关研究也发现环境细菌对鱼类肠道菌群组成的影响总体较小[71-72]。
4 结语本研究采用16S rRNA基因高通量测序技术对池塘跑道养殖系统中鱼类肠道、水体和沉积物菌群组成和结构进行了比较研究。结果表明,IPRS不同生境中细菌群落多样性和结构均存在显著差异,细菌群落的优势组成也有所不同。养殖鱼类肠道菌群与水体菌群相似度较小,而与沉积物菌群相似度较大。与花鲈相比,河鲀肠道菌群受到沉积物的影响更大。不同功能区沉积物菌群结构也存在显著差异。其中,固体废物收集区与净化区差别较大,部分可降解有机物和去除氮、磷、硫等营养盐的细菌类群在固体废物收集区的沉积物中大量富集。
| [1] |
Abraham T J, Ghosh S, Nagesh T S, et al. Distribution of bacteria involved in nitrogen and sulphur cycles in shrimp culture systems of West Bengal, India[J]. Aquaculture, 2004, 239(1-4): 275-288. DOI:10.1016/j.aquaculture.2004.06.023 (  0) 0) |
| [2] |
Moriarty D J W. The role of microorganisms in aquaculture ponds[J]. Aquaculture, 1997, 151(1-4): 333-349. DOI:10.1016/S0044-8486(96)01487-1 (  0) 0) |
| [3] |
Guan X Y, Zhou W, Hu C M, et al. Bacterial community temporal dynamics and disease-related variations in the seawater of Pyropia (layer) seedling pools[J]. Journal of Applied Phycology, 2018, 30(2): 1217-1224. DOI:10.1007/s10811-017-1272-6 (  0) 0) |
| [4] |
Infante-Villamil S, Huerlimann R, Condon K, et al. Bacterial signatures of productivity decay in Penaeus monodon ponds infected with PirA toxin[J]. Aquaculture, 2019, 511: 734202. DOI:10.1016/j.aquaculture.2019.734202 (  0) 0) |
| [5] |
Rawls J F, Samuel B S, Gordon J I. Gnotobiotic zebrafish reveal evolutionarily conserved responses to the gut microbiota[J]. Proceedings of the National Academy of Sciences of the United States of America, 2004, 101(13): 4596-4601. DOI:10.1073/pnas.0400706101 (  0) 0) |
| [6] |
Round J L, Mazmanian S K. The gut microbiota shapes intestinal immune responses during health and disease[J]. Nature Reviews Immunology, 2009, 9(5): 313-323. DOI:10.1038/nri2515 (  0) 0) |
| [7] |
Talwar C, Nagar S, Lal R, et al. Fish gut microbiome: Current approaches and future perspectives[J]. Indian Journal of Microbiology, 2018, 58(4): 397-414. DOI:10.1007/s12088-018-0760-y (  0) 0) |
| [8] |
Wang A R, Ran C, Ringo E, et al. Progress in fish gastrointestinal microbiota research[J]. Reviews in Aquaculture, 2018, 10(3): 626-640. DOI:10.1111/raq.12191 (  0) 0) |
| [9] |
Giatsis C, Sipkema D, Smidt H, et al. The impact of rearing environment on the development of gut microbiota in tilapia larvae[J]. Scientific Reports, 2015, 5(1): 18206. DOI:10.1038/srep18206 (  0) 0) |
| [10] |
Wu S G, Tian J Y, Gatesoupe F J, et al. Intestinal microbiota of gibel carp (Carassius auratus gibelio) and its origin as revealed by 454 pyrosequencing[J]. World Journal of Microbiology & Biotechnology, 2013, 29(9): 1585-1595. (  0) 0) |
| [11] |
Xiong J B, Wang K, Wu J F, et al. Changes in intestinal bacterial communities are closely associated with shrimp disease severity[J]. Applied Microbiology and Biotechnology, 2015, 99(16): 6911-6919. DOI:10.1007/s00253-015-6632-z (  0) 0) |
| [12] |
Zhu J Y, Dai W F, Qiu Q F, et al. Contrasting ecological processes and functional compositions between intestinal bacterial community in healthy and diseased shrimp[J]. Microbial Ecology, 2016, 72(4): 975-985. DOI:10.1007/s00248-016-0831-8 (  0) 0) |
| [13] |
Bentzon-Tilia M, Sonnenschein E C, Gram L. Monitoring and managing microbes in aquaculture-towards a sustainable industry[J]. Microbial Biotechnology, 2016, 9(5): 576-584. DOI:10.1111/1751-7915.12392 (  0) 0) |
| [14] |
Dittmann K K, Rasmussen B B, Castex M, et al. The aquaculture microbiome at the centre of business creation[J]. Microbial Biotechnology, 2017, 10(6): 1279-1282. DOI:10.1111/1751-7915.12877 (  0) 0) |
| [15] |
Cressey D. Future fish[J]. Nature, 2009, 458(7237): 398-400. DOI:10.1038/458398a (  0) 0) |
| [16] |
Bostock J, Mcandrew B, Richards R, et al. Aquaculture: Global status and trends[J]. Philosophical Transactions of the Royal Society B-Biological Sciences, 2010, 365(1554): 2897-2912. DOI:10.1098/rstb.2010.0170 (  0) 0) |
| [17] |
农业农村部渔业渔政管理局, 全国水产技术推广总站, 中国水产学会. 中国渔业统计年鉴[M]. 北京: 中国农业出版社, 2020. Fisheries Administration of Ministry of Agriculture and Rural Affairs, National Fisheries Technology Extension Center, China Society of Fisheries. China Fishery Statistical Yearbook[M]. Beijing: China Agriculture Press, 2020. (  0) 0) |
| [18] |
Yoo K H, Masser M P, Hawcroft B A. An in-pond raceway system incorporating removal of fish wastes[J]. Aquacultural Engineering, 1995, 14(2): 175-187. DOI:10.1016/0144-8609(94)P4434-D (  0) 0) |
| [19] |
Farrelly J C, Chen Y S, Shrestha S. Occurrences of growth related target dissolved oxygen and ammonia in different catfish pond production systems in southeast Arkansas[J]. Aquacultural Engineering, 2015, 64: 68-77. DOI:10.1016/j.aquaeng.2014.10.002 (  0) 0) |
| [20] |
Li T T, Long M, Ji C, et al. Alterations of the gut microbiome of largemouth bronze gudgeon (Coreius guichenoti) suffering from furunculosis[J]. Scientific Reports, 2016, 6(1): 30606. DOI:10.1038/srep30606 (  0) 0) |
| [21] |
Cao L, Wang W, Yang Y, et al. Environmental impact of aquaculture and countermeasures to aquaculture pollution in China[J]. Environmental Science and Pollution Research, 2007, 14(7): 452-462. DOI:10.1065/espr2007.05.426 (  0) 0) |
| [22] |
Brown T W, Boyd C E, Chappell J A. Approximate water and chemical budgets for an experimental, in-pond raceway system[J]. Journal of the World Aquaculture Society, 2012, 43(4): 526-537. DOI:10.1111/j.1749-7345.2012.00576.x (  0) 0) |
| [23] |
董双林, 田相利, 高勤峰. 水产养殖生态学[M]. 第二版. 北京: 科学出版社, 2021. Dong S L, Tian X L, Gao Q F. Aquaculture Ecology[M]. Second Edition. Beijing: Science Press, 2021. (  0) 0) |
| [24] |
Bolyen E, Rideout J R, Dillon M R, et al. Reproducible, interactive, scalable and extensible microbiome data science using QIIME 2[J]. Nature Biotechnology, 2019, 37(8): 852-857. DOI:10.1038/s41587-019-0209-9 (  0) 0) |
| [25] |
Callahan B J, Mcmurdie P J, Rosen M J, et al. DADA2: High-resolution sample inference from Illumina amplicon data[J]. Nature Methods, 2016, 13(7): 581-583. DOI:10.1038/nmeth.3869 (  0) 0) |
| [26] |
Altschul S F, Gish W, Miller W, et al. Basic local alignment search tool[J]. Journal of Molecular Biology, 1990, 215(3): 403-410. DOI:10.1016/S0022-2836(05)80360-2 (  0) 0) |
| [27] |
Wang Q, Garrity G M, Tiedje J M, et al. Naive Bayesian classifier for rapid assignment of rRNA sequences into the new bacterial taxonomy[J]. Applied and Environmental Microbiology, 2007, 73(16): 5261-5267. DOI:10.1128/AEM.00062-07 (  0) 0) |
| [28] |
Werner J J, Koren O, Hugenholtz P, et al. Impact of training sets on classification of high-throughput bacterial 16s rRNA gene surveys[J]. Multidisciplinary Journal of Microbial Ecology, 2012, 6(1): 94-103. (  0) 0) |
| [29] |
Quast C, Pruesse E, Yilmaz P, et al. The SILVA ribosomal RNA gene database project: Improved data processing and web-based tools[J]. Nucleic Acids Research, 2013, 41(D1): 590-596. (  0) 0) |
| [30] |
Oksanen J, Blanchet F G, Kindt R, et al. Vegan: Community ecology package[CP/OL]. (2020-11-28)[2021-10-13]. https://cran.r-project.org/web/packages/vegan/index.html.
(  0) 0) |
| [31] |
Segata N, Izard J, Waldron L, et al. Metagenomic biomarker discovery and explanation[J]. Genome Biology, 2011, 12(6): 60. DOI:10.1186/gb-2011-12-6-r60 (  0) 0) |
| [32] |
Clarke K R. Non-parametric multivariate analyses of changes in community structure[J]. Australian Journal of Ecology, 1993, 18(1): 117-143. DOI:10.1111/j.1442-9993.1993.tb00438.x (  0) 0) |
| [33] |
Brown T W, Hanson T R, Chappell J A, et al. Economic feasibility of an in-pond raceway system for commercial catfish production in West Alabama[J]. North American Journal of Aquaculture, 2014, 76(1): 79-89. DOI:10.1080/15222055.2013.862195 (  0) 0) |
| [34] |
Brown T W, Chappell J A, Boyd C E. A commercial-scale, in-pond raceway system for Ictalurid catfish production[J]. Aquacultural Engineering, 2011, 44(3): 72-79. DOI:10.1016/j.aquaeng.2011.03.003 (  0) 0) |
| [35] |
Wang Y Y, Xu P, Nie Z J, et al. Growth, digestive enzymes activities, serum biochemical parameters and antioxidant status of juvenile genetically improved farmed tilapia (Oreochromis niloticus) reared at different stocking densities in in-pond raceway recirculating culture system[J]. Aquaculture Research, 2019, 50(4): 1338-1347. DOI:10.1111/are.14010 (  0) 0) |
| [36] |
Yuan J L, Ni M, Liu M, et al. Analysis of the growth performances, muscle quality, blood biochemistry and antioxidant status of Micropterus salmoides farmed in in-pond raceway systems versus usual-pond systems[J]. Aquaculture, 2019, 511: 734241. DOI:10.1016/j.aquaculture.2019.734241 (  0) 0) |
| [37] |
欧红霞, 王广军, 李希国, 等. 跑道式池塘循环水养殖对宝石鲈营养成分及血清生化指标的影响[J]. 渔业现代化, 2021, 48(2): 29-34. Ou H X, Wang G J, Li X G, et al. Effects of in-pond raceway aquaculture systems on nutritional components and serum biochemical parameters of Scortum barcoo[J]. Fishery Modernization, 2021, 48(2): 29-34. DOI:10.3969/j.issn.1007-9580.2021.02.005 (  0) 0) |
| [38] |
周凡, 丁理法, 彭建, 等. 池塘内循环"跑道"与传统土池模式养成梭鱼的营养成分对比研究[J]. 南方水产科学, 2021, 17(3): 70-76. Zhou F, Ding L F, Peng J, et al. Comparative analysis of muscle nutrient composition bewteen in-pond raceway and usual-pond system cultured Liza haematocheila[J]. South China Fisheries Science, 2021, 17(3): 70-76. (  0) 0) |
| [39] |
Zhao Z L, Jiang J W, Pan Y J, et al. Temporal dynamics of bacterial communities in the water and sediments of sea cucumber (Apostichopus japonicus) culture ponds[J]. Aquaculture, 2020, 528: 735498. DOI:10.1016/j.aquaculture.2020.735498 (  0) 0) |
| [40] |
Kuang T X, He A Y, Lin Y F, et al. Comparative analysis of microbial communities associated with the gill, gut, and habitat of two filter-feeding fish[J]. Aquaculture Reports, 2020, 18: 10051. (  0) 0) |
| [41] |
Jing X J, Su S Y, Zhang C F, et al. Dynamic changes in microbial community structure in farming pond water and their effect on the intestinal microbial community profile in juvenile common carp (Cyprinus carpio L.)[J]. Genomics, 2021, 113(4): 2547-2560. DOI:10.1016/j.ygeno.2021.05.024 (  0) 0) |
| [42] |
Zhang H X, Tang H S, Zang Y, et al. Microorganism's adaptation of Crucian carp may closely relate to its living environments[J]. Microbiologyopen, 2019, 8(3): 650. (  0) 0) |
| [43] |
Andersson A F, Riemann L, Bertilsson S. Pyrosequencing reveals contrasting seasonal dynamics of taxa within Baltic Sea bacterioplankton communities[J]. Multidisciplinary Journal of Microbial Ecology, 2010, 4(2): 171-181. (  0) 0) |
| [44] |
Vellend M. Conceptual synthesis in community ecology[J]. Quarterly Review of Biology, 2010, 85(2): 183-206. DOI:10.1086/652373 (  0) 0) |
| [45] |
Morris R M, Rappe M S, Connon S A, et al. SAR11 clade dominates ocean surface bacterioplankton communities[J]. Nature, 2002, 420(6917): 806-810. DOI:10.1038/nature01240 (  0) 0) |
| [46] |
Du Z J, Wang Z J, Zhao J X, et al. Woeseia oceani gen. nov., sp nov., a chemoheterotrophic member of the order Chromatiales, and proposal of Woeseiaceae fam. nov.[J]. International Journal of Systematic and Evolutionary Microbiology, 2016, 66: 107-112. DOI:10.1099/ijsem.0.000683 (  0) 0) |
| [47] |
Guo C C, Zhang X W, Luan S M, et al. Diversity and structure of soil bacterial community in intertidal zone of Daliao River estuary, Northeast China[J]. Marine Pollution Bulletin, 2021, 163: 111965. DOI:10.1016/j.marpolbul.2020.111965 (  0) 0) |
| [48] |
Zhang X W, Ji Z, Shao Y T, et al. Seasonal variations of soil bacterial communities in Suaeda wetland of Shuangtaizi River estuary, Northeast China[J]. Journal of Environmental Sciences, 2020, 97: 45-53. DOI:10.1016/j.jes.2020.04.012 (  0) 0) |
| [49] |
Duan Y F, Xiong D L, Li Y, et al. Changes in the microbial communities of the rearing water, sediment and gastrointestinal tract of Lateolabrax maculatus at two growth stages[J]. Aquaculture Reports, 2021, 20: 100742. DOI:10.1016/j.aqrep.2021.100742 (  0) 0) |
| [50] |
Duan Y F, Xiong D L, Li Y, et al. Changes in the gastrointestinal microbial community of Lateolabrax maculatus in a naturally occurring Microcystis aeruginosa bloom environment[J]. Aquaculture, 2020, 528: 735444. DOI:10.1016/j.aquaculture.2020.735444 (  0) 0) |
| [51] |
Cao Y, Shen J, Ran Z H. Association between Faecalibacterium prausnitzii reduction and inflammatory bowel disease: A meta-analysis and systematic review of the literature[J]. Gastroenterology Research and Practice, 2014, 2014: 872725. (  0) 0) |
| [52] |
Sokol H, Pigneur B, Watterlot L, et al. Faecalibacterium prausnitzii is an anti-inflammatory commensal bacterium identified by gut microbiota analysis of Crohn disease patients[J]. Proceedings of the National Academy of Sciences of the United States of America, 2008, 105(43): 16731-16736. DOI:10.1073/pnas.0804812105 (  0) 0) |
| [53] |
Van Kessel M A, Dutilh B E, Neveling K, et al. Pyrosequencing of 16S rRNA gene amplicons to study the microbiota in the gastrointestinal tract of carp (Cyprinus carpio L.)[J]. AMB Express, 2011, 1: 41. DOI:10.1186/2191-0855-1-41 (  0) 0) |
| [54] |
Nayak S K. Role of gastrointestinal microbiota in fish[J]. Aquaculture Research, 2010, 41(11): 1553-1573. DOI:10.1111/j.1365-2109.2010.02546.x (  0) 0) |
| [55] |
Hsu T T D, Lee J. Global distribution and prevalence of Arcobacter in food and water[J]. Zoonoses and Public Health, 2015, 62(8): 579-589. DOI:10.1111/zph.12215 (  0) 0) |
| [56] |
Venancio I, Luís Â, Domingues F, et al. The prevalence of Arcobacteraceae in aquatic environments: A systematic review and meta-analysis[J]. Pathogens, 2022, 11(2): 244. DOI:10.3390/pathogens11020244 (  0) 0) |
| [57] |
Song J, Hwang J, Kang I, et al. A sulfate-reducing bacterial genus, Desulfosediminicola gen. nov., comprising two novel species cultivated from tidal-flat sediments[J]. Scientific Reports, 2021, 11(1): 19978. DOI:10.1038/s41598-021-99469-5 (  0) 0) |
| [58] |
Cui Y W, Huang J L, Alam F. Fast granulation of halophilic activated sludge treating low-strength organic saline wastewater via addition of divalent cations[J]. Chemosphere, 2021, 264(1): 128396. (  0) 0) |
| [59] |
Gui X W, Li Z L, Wang Z J. Kitchen waste hydrolysate enhances sewage treatment efficiency with different biological process compared with glucose[J]. Bioresource Technology, 2021, 341: 125904. DOI:10.1016/j.biortech.2021.125904 (  0) 0) |
| [60] |
柳晓东, 朱海珍, 姜民志, 等. 北京龙形水系三种沉水植物根际及叶际微生物群落特征[J]. 生物工程学报, 2021, 37(10): 3663-3674. Liu X D, Zhu H Z, Jiang M Z, et al. Characteristics of the rhizosphere and phyllosphere microbial community of three submerged plants in the dragon-shaped water system of Beijing[J]. Chinese Journal of Biotechnology, 2021, 37(10): 3663-3674. (  0) 0) |
| [61] |
孙静, 王素英, 张德超. 海南红树林根系土壤中可培养细菌的多样性分析[J]. 海洋科学, 2014, 38(7): 27-33. Sun J, Wang S Y, Zhang D C. Diversity of culturable bacteria from the soil of root system of mangrove forest of Beigang island in Hainan Province[J]. Marine Sciences, 2014, 38(7): 27-33. (  0) 0) |
| [62] |
Pan Z L, Zhou J, Lin Z Y, et al. Effects of COD/TN ratio on nitrogen removal efficiency, microbial community for high saline wastewater treatment based on heterotrophic nitrification-aerobic denitrification process[J]. Bioresource Technology, 2020, 301: 122726. DOI:10.1016/j.biortech.2019.122726 (  0) 0) |
| [63] |
Sun Q L, Zhang J, Wang M X, et al. High-throughput sequencing reveals a potentially novel Sulfurovum species dominating the microbial communities of the seawater-sediment interface of a deep-sea cold seep in South China Sea[J]. Microorganisms, 2020, 8(5): 687. DOI:10.3390/microorganisms8050687 (  0) 0) |
| [64] |
Fan X F, Ding S M, Gong M D, et al. Different influences of bacterial communities on Fe (Ⅲ) reduction and phosphorus availability in sediments of the Cyanobacteria-and macrophyte-dominated zones[J]. Frontiers in Microbiology, 2018, 9: 2636. DOI:10.3389/fmicb.2018.02636 (  0) 0) |
| [65] |
Muller H, Marozava S, Probst A J, et al. Groundwater cable bacteria conserve energy by sulfur disproportionation[J]. Multidisciplinary Journal of Microbial Ecology, 2020, 14(2): 623-634. (  0) 0) |
| [66] |
Shen X X, Xu M, Li M, et al. Response of sediment bacterial communities to the drainage of wastewater from aquaculture ponds in different seasons[J]. Science of the Total Environment, 2020, 717: 137180. DOI:10.1016/j.scitotenv.2020.137180 (  0) 0) |
| [67] |
Yang Y Y, Dai Y, Wu Z, et al. Temporal and spatial dynamics of archaeal communities in two freshwater lakes at different trophic status[J]. Frontiers in Microbiology, 2016, 7: 451. (  0) 0) |
| [68] |
Bilotta G S, Brazier R E. Understanding the influence of suspended solids on water quality and aquatic biota[J]. Water Research, 2008, 42(12): 2849-2861. DOI:10.1016/j.watres.2008.03.018 (  0) 0) |
| [69] |
Sun F L, Wang Y S, Wang C Z, et al. Insights into the intestinal microbiota of several aquatic organisms and association with the surrounding environment[J]. Aquaculture, 2019, 507: 196-202. DOI:10.1016/j.aquaculture.2019.04.026 (  0) 0) |
| [70] |
Kato A, Doi H, Nakada T, et al. Takifugu obscurus is a euryhaline fugu species very close to Takifugu rubripes and suitable for studying osmoregulation[J]. BMC Physiology, 2005, 5(1): 18. DOI:10.1186/1472-6793-5-18 (  0) 0) |
| [71] |
Hossain M S, Dai J C, Qiu D R. European eel (Anguilla anguilla) GI tract conserves a unique metagenomics profile in the recirculation aquaculture system (RAS)[J]. Aquaculture International, 2021, 29(4): 1529-1544. DOI:10.1007/s10499-021-00692-8 (  0) 0) |
| [72] |
Zhao R X, Symonds J E, Walker S P, et al. Effects of feed ration and temperature on Chinook salmon (Oncorhynchus tshawytscha) microbiota in freshwater recirculating aquaculture systems[J]. Aquaculture, 2021, 543: 736965. DOI:10.1016/j.aquaculture.2021.736965 (  0) 0) |
2. Tangshan Haidu Seafood Company Limited, Tangshan 063000, China;
3. Pilot National Laboratory for Marine Science and Technology(Qingdao), Qingdao 266237, China
 2023, Vol. 53
2023, Vol. 53


