花鲈(Lateolabrax maculatus)隶属于鲈形目(Perciformes)鮨科(Serranidae)花鲈属(Lateolabrax),俗名海鲈、鲈鱼、七星鲈、寨花等,广泛分布于中国沿海以及朝鲜沿海,是中国重要的海产经济鱼类[1]。由于花鲈具有广温广盐性的特点,已经成为南北方网箱养殖的主要对象,养殖规模正逐步扩大。同时花鲈耐受能力强,是环境因子胁迫研究的理想对象。
溶解氧是影响水生动物生长、代谢等生命活动的重要环境因子,是维持水生动物生存的基本条件[2]。而当今人口增长、温室效应,鱼类高密度养殖等因素致使鱼类面临着低氧环境的威胁,低氧对鱼类的生长、存活、行为、生理生化以及发育和繁殖等产生影响[3]。生物体在适应低氧环境的长期进化中已经形成了一系列的基因表达调控机制。这些基因能够接受低氧信号的调节并参与低氧应答,是低氧应答机制中重要的分子基础[4]。低氧诱导因子(Hypoxia Inducible Factors,hifs)是其中的关键基因之一,在生物体的环境缺氧应答机制中起着非常重要的作用。
低氧诱导因子(hifs)最先由Semenza等[5]在低氧诱导的细胞核提取物中发现,是由α和β亚基组成的异源二聚体转录因子。HIFa亚基主要包括3种:HIF1α、HIF2α和HIF3α三者均受氧气浓度的调节与影响,并能够使HIF的活性发生变化[6]。HIF1b也称为芳香烃受体核转位蛋白(Aryl Hydrocarbon Receptor Nuclear Translocator,arnt),是一个重要的转录调控因子,对氧浓度的变化不敏感,其功能主要与其他转录因子形成异源二聚体参与调控过程,如低氧下与HIFa聚合成二聚体参与低氧基因的调控[7-10]以及参与器官形成、神经发育[18]等。hifs基因在催化低氧相关基因表达中发挥重要作用,研究表明hifs可通过与低氧反应元件(Hypoxia Response Element, HRE)相结合来调控相关靶基因的表达,从而参与低氧状态机体的生理响应,例如:促进红细胞生成、血管生成,碳水化合物转运和代谢,线粒体代谢和凋亡等[7-8, 11-12]。
目前,关于鱼类hifs的研究主要集中在hifα亚基[13-15]。有关花鲈低氧调控的研究也较少,其分子机制尚不清楚。本研究对花鲈4个hifs基因序列进行了全面的分析研究,同时利用实时荧光定量PCR手段检测了花鲈hifs在各组织中的表达模式,对hifs在低氧胁迫下的表达变化以及相关的分子机制进行了探讨。本研究结果为深入研究花鲈4个hifs基因在低氧调控中的功能提供了理论基础,同时为了解其它鱼类在低氧胁迫时机体的生理调控与分子调控机制提供参考。
1 材料与方法 1.1 实验用鱼实验于中国海洋大学鱼类繁殖生理与种子工程实验室进行。实验对象为体长(21.43±0.77)cm、体重(149.20±19.22)g的花鲈幼鱼。实验用鱼于400 L的塑料圆桶中暂养2周,暂养期间溶氧为7.5~8.0 mg/L,温度为(19±0.5)℃,盐度为30。每天09:00和17:00投饵2次,投饵后清除残饵并换水。实验开始前一天停止投喂。
1.2 低氧胁迫实验实验容器为3个400 L的塑料圆桶,实验开始前通过快速注入氮气来降低溶氧含量,利用溶氧仪(YSI DO200)来实时监测桶内溶氧水平。当溶氧量达到2 mg/L时,往3个实验圆桶内平均放入40尾花鲈幼鱼,适应10 min后开始计时,期间通过注入少量空气以及适量氮气来维持桶内溶解氧的相对稳定。实验分为低氧胁迫与常氧恢复两个阶段,低氧为(1.56±0.24)mg/L,低氧胁迫24 h。24 h后通过迅速注入纯氧使氧气水平达到8 mg/L,期间通过注入纯氧和适量氮气来维持溶解氧的相对稳定,常氧维持在(7.72±0.18)mg/L。低氧胁迫期间的取样时间点分别为3、6、12和24 h,复氧取样时间点分别为3和12 h。
1.3 样品采集与处理对低氧胁迫与常氧恢复的各时间点以及暂养桶中样品进行取样,其中暂养桶中样品作为对照组取样,每个桶每个时间点各取样3尾花鲈幼鱼,用MS-222(200 mg/L)进行麻醉,然后快速解剖进行肝、鳃、肌肉、脑、垂体、心脏、肠、胃、脾、肾组织的取样。样品迅速放于液氮中速冻后放于-80 ℃冰箱保存备用。
1.4 总RNA的提取与cDNA的合成总RNA的提取采用Trizol (Invitrogen, USA)提取法。cDNA按照PrimeScriptTM RT reagent Kit with gDNA Eraser (Perfect Real Time) (RR047A Takara)试剂盒说明书进行合成。合成的cDNA暂时存于-20 ℃备用。
1.5 花鲈4个hifs基因序列与进化分析花鲈4个hifs基因序列来自花鲈基因组(未公开)及转录组(SRR4409341 (LS)和SRR4409397 (HS)),通过NCBI-Blast(https://blast.ncbi.nlm.nih.gov/Blast.cgi)在线分析软件进行比对,初步确定各基因,利用ORFfinder(https://www.ncbi.nlm.nih.gov/orffinder/)在线分析软件,对确定的基因序列进行阅读框、氨基酸序列推定。同时通过DNAMAN Version 6.0软件进行氨基酸序列同源性的比较。利用MEGA7.0软件构建系统进化树进一步确定各基因,采用的方法为邻接法(Neighbor-joining, NJ),bootstrap数值为1 000。
1.6 共线性分析为进一步注释花鲈4个hifs基因,本研究进行了共线性分析。共线性分析是基于花鲈4个hifs基因与其它物种对应基因间临近基因进行对比建立起来的。对于hifs各基因临近基因的查找来自花鲈基因组,其它物种基因位置的确定来自Genomicus[16]和Ensembl基因组数据库(http://www.ensembl.org/)。
1.7 花鲈4个hifs基因实时荧光定量PCR(qRT-PCR)表达分析根据花鲈基因组4个hifs基因序列,设计相关qRT-PCR引物(见表 1)。20 μL qRT-PCR反应体系包括:10 μL SYBRⓇFAST qPCR Master Mix (2×),0.4 μL ROX Reference Dye (50×),2 μL cDNA模板,上下游引物各0.4 μL以及6.8 μL的ddH2O。使用Step One Plus Real-Time PCR system (Applied Biosystems) PCR仪进行定量实验。qRT-PCR的反应程序为:95 ℃预变性5 s,95 ℃,5 s,60 ℃退火30 s,共40个循环。18s RNA作为内参基因,基因的相对表达量采用2-ΔΔCT法进行计算。
|
|
表 1 实时定量PCR表达分析的引物序列 Table 1 Primer sequences for mRNA expression analysis |
所得数据均用平均值±标准差(Mean±SD)表示,用软件SPSS 19.0进行单因素方差分析(One-way ANOVA)及Duncan多重比较,以P<0.05作为差异显著水平,用Excel2010进行作图。
2 结果 2.1 花鲈4个hifs基因序列分析对花鲈hifs基因序列信息进行统计(见表 2)。如表 2所示,hifs基因CDS序列长度范围为2 043~2 676 bp,分别编码733~891个氨基酸。用于花鲈hifs基因同源性分析的物种包括人(Homo sapiens)、鼠(Mus musculus)、鸡(Gallus gallus)、斑马鱼(Danio rerio)。对花鲈hifs基因与以上物种同源性分析结果进行统计(见表 3)。如表 3所示,hif1a与其它物种同源性为59.8%~65.1%,hif2a为48.8%~61.6%,hif3a为39.9%~58.5%,hif1b为59.6%~82.0%。
|
|
表 2 hifs基因序列信息统计 Table 2 The summary of spotted sea bass hifs genes |
|
|
表 3 花鲈与其他物种hifs基因间氨基酸序列比对 Table 3 Comparison of the amino acid sequences of the hifs genes between the spotted sea bass and other species |
对花鲈和不同物种的4个hifs基因进行系统进化分析,构建进化树(见图 1)。如图 1所示,选择的物种主要包括人、鼠、鸡、斑马鱼、虹鳟(Oncorhynchus mykiss)、斑点叉尾鮰(Ictalurus punctatus)、青鳉(Oryzias latipes)等。进化树共分为两大分支:hif1α、hif2α和hif3α共同聚为其中一个较大的分支;同时又分为两个分支,其中hif1α和hif2α聚为一支,说明hif1α和hif2α同源关系相对较近,hif3α单独聚为一支。而hif1β单独聚为另一分支。说明hif1β在进化关系上与hif1α、hif2α和hif3α同源性相对较远。
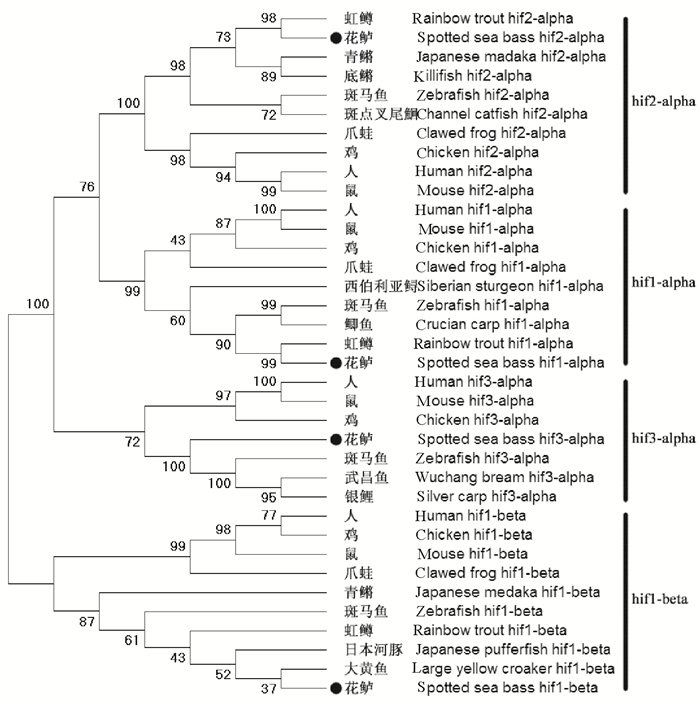
|
(黑色圆点表示花鲈hifs基因。The black dots in the chart indicate the positions of the hifs genes. ) 图 1 花鲈和其它物种hifs基因的邻接系统发育树 Fig. 1 The neighbor-joining phylogenetic tree for hifs genes in spotted sea bass and other species |
对花鲈和不同物种的4个hifs基因进行共线性分析(见图 2)。如图 2所示,用于共线性分析的物种为人、鼠、虹鳟、斑点叉尾鮰。对于hif1α基因(见图 2(A)),花鲈与虹鳟具有相同的临近基因trmt5,花鲈与人也具有相同的临近基因trmt5,但排列顺序相反,其它相同基因的排列顺序不一致。花鲈和人、鼠hif2α基因临近的pigf、msh2基因相同(见图 2(B))。花鲈hif3a基因与人、鼠有相同临近基因pak4(见图 2(C))。对于hif1β基因(见图 2(D)),花鲈与斑点叉尾鮰具有相同的临近基因rps9和cnot3,花鲈与人临近基因相同但排列顺序不同。因此,各hif基因的共线性相对保守,进一步证明本研究对花鲈hif基因注释的正确性。
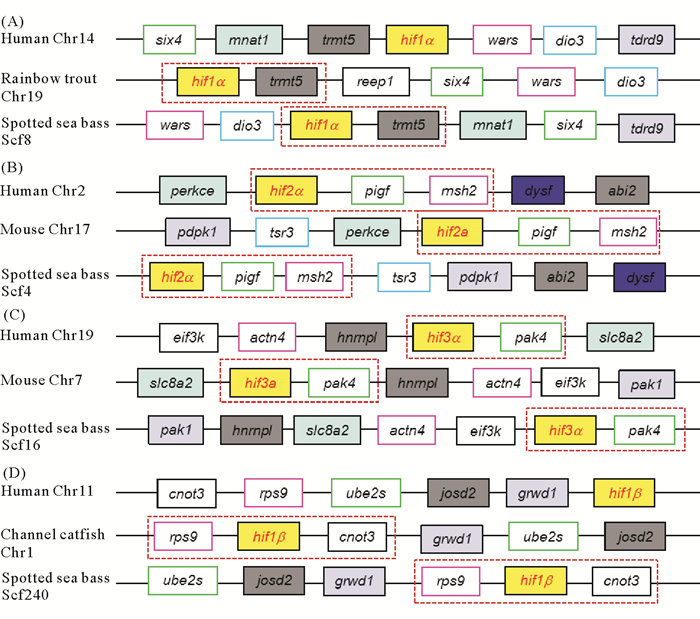
|
((A)表示hif1α基因;(B)表示hif2α基因;(C)表示hif3α基因;(D)表示hif1β基因。(A) indicated hif1α gene; (B) indicated hif2α gene; (C) indicated hif3α gene; (D) indicated hif1β gene. ) 图 2 花鲈和其它物种hifs基因共线性分析 Fig. 2 Syntenic analysis for hifs genes in spotted sea bass and other species |
花鲈10个组织分别为肝、鳃、肌、脑、垂、心、肠、胃、脾、肾。其中以表达量较小的肌肉组织作为参照,进行4个hifs基因相对表达量的计算(见图 3)。由图 3可知,hifs基因在10个组织中的表达情况不同,其中hif1α在花鲈脾中表达量最高,其表达量是肌肉16.59倍,在肌肉中表达量最低,其它组织依次为心脏,鳃,肾,肠,肝,胃,垂体,脑;hif2α在肌肉、脑中的表达量较低,而在鳃中的表达量最高,约为肌肉中的24.76倍;hif3α在肠和胃中表达相对较高,其次为脾脏、心脏和肾,其它组织的表达量相对较低;hif1β在鳃组织中的表达量最高,其表达量约为肌肉的22.33倍。
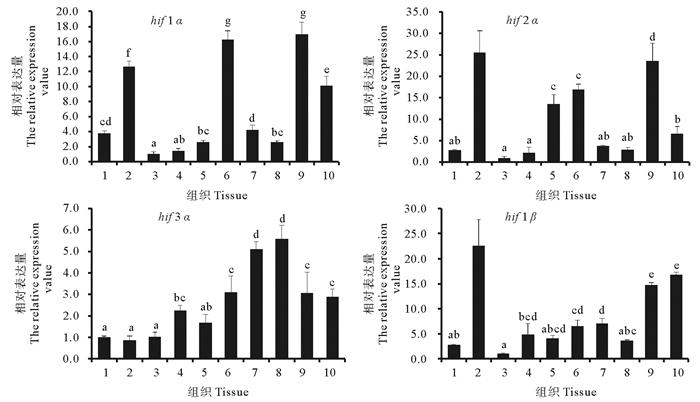
|
(10个组织从左至右分别为:1:肝,2:鳃,3:肌肉,4:脑,5:垂体,6:心脏,7:肠,8:胃,9:脾,10:肾。The order of ten tissues from left to right: 1:liver, 2:gill, 3:muscle, 4:brain, 5:pituitary, 6:heart, 7:intestine, 8:stomach, 9:spleen, 10:kidney. ) 图 3 花鲈hifs基因在不同组织中的表达情况 Fig. 3 The expression of hifs genes of spotted sea bass in different tissues |
实验结果以对照组0 h基因的表达量为参照,测定了低氧胁迫与常氧恢复阶段花鲈肝组织和鳃组织hifs基因的表达情况(见图 4和5)。由图 4和5可知,hifs各个基因对低氧的响应时间和响应程度存在显著差异。如图 4所示,肝组织中,低氧胁迫3 h后,hifs基因相对表达量均出现了显著升高(P<0.05),其中hif1α、hif2α、hif3α分别升高了3.76,3.81,3.82倍,hif1β升高2.51倍;低氧胁迫12 h后,仅有hif1α表达量仍显著高于对照组(P<0.05);低氧胁迫24 h以及恢复正常溶氧3和12 h时,各基因的表达量与对照组相比均没有显著差异。如图 5所示,鳃组织中,低氧胁迫3 h后,hif1α、hif2α与hif3α相对表达量显著升高(P<0.05),其中hif1α升高2.43倍,hif2α升高1.76倍,hif3α升高6.89倍;低氧胁迫6h后,仅hif3α、hif1β基因相对表达量显著高于对照组(P<0.05),hif3α相对表达量升高6.01倍,hif1β则升高2.11倍;而其它时间点各基因相对表达量均无显著差异。
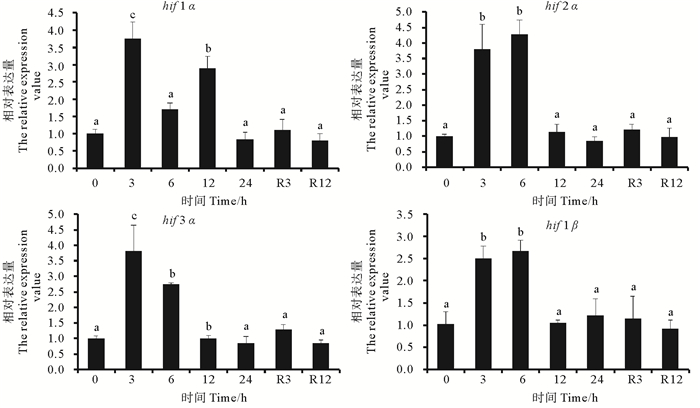
|
(0~24 h为低氧胁迫期间的取样时间点,R3h和R12h为复氧取样时间点。0~24 h is the sampling time point during hypoxic stress, R3h and R12h is the sampling time point during reoxygenation.The same as below. ) 图 4 低氧胁迫下hifs基因在花鲈肝脏中的表达情况 Fig. 4 The expression profile of hifs genes of spotted sea bass in liver tissue under hypoxia challenge |
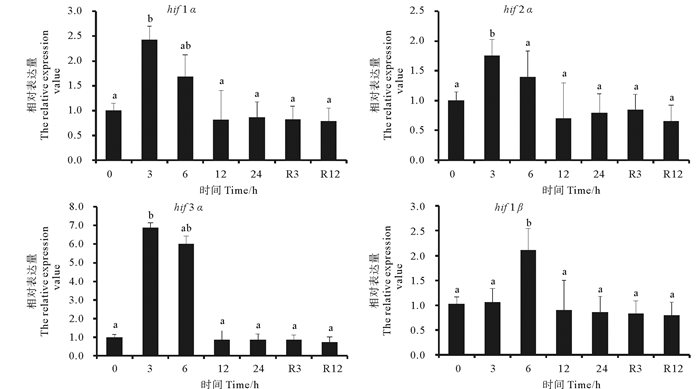
|
图 5 低氧胁迫下hifs基因在花鲈鳃中的表达情况 Fig. 5 The expression profile of hifs genes of spotted sea bass in gilltissue under hypoxiachallenge |
温室效应等导致的全球变暖,以及鱼类高密度养殖,经常会使养殖水体出现短期的急性缺氧状况,严重的急性缺氧会造成“翻塘”,引起鱼类的瞬间大量死亡,对养殖生产造成大量的损失。因此,研究与低氧胁迫相关的基因在急性低氧中的表达情况,对鱼类低氧应答研究具有一定的实际指导意义。在本实验中,我们从花鲈4个hifs基因鉴定、基因组织表达和低氧诱导hifs表达三个方面探索了急性低氧对花鲈hifs基因的影响。
3.1 花鲈hifs基因序列分析本文4个hifs基因序列均来自花鲈基因组,其氨基酸长度分别为818、891、680和733 aa,各基因长度与其他物种相似,说明各基因在进化过程中保持着较强的保守性,这可能与其生物学功能相关。基于4个hifs基因氨基酸序列构建的进化树中,各基因首先与鱼类聚为一支,然后在与哺乳类聚在一起,这与Mu等[15]和Shen等[17]对hif1α和hif2α的研究结果相一致。共线性结果表明hifs各基因与相应物种具有相同的临近基因,进一步证明了本研究对花鲈4个hifs基因命名的准确性。
3.2 hifs基因在花鲈10个组织中的差异表达分析花鲈hifs基因在10个组织中的表达分析实验结果表明:hifs基因在不同组织的表达丰度不同,如hif1α在花鲈的脾脏中表达最高,肌肉中表达最低,hif2α在鳃中表达最高,肌肉中表达最低,hif3α在胃中表达最高,鳃中表达最低;hif1β表达量在鳃组织中最高,肌肉组织中最低。总体而言,花鲈hifs表达量在鳃、脾脏、胃中相对较高,而在肌肉中相对较低,表明hifs表达具有组织特异性。姜华鹏等[18]对高原软刺裸鲤(Gymnocypris dobula)hif1B和hif2A的研究得到了与本实验相似的结果,其在鳃中表达量最高,而在脾脏、脑和皮肤中的表达量较低。而张慧[19]对黄颡鱼(Pelteonagrus fulvidsco)hif1α的研究结果与本实验不同,其在肝脏中相对表达量最高,肾脏次之,鳃中最少。相似的Mohindra V等[20]对印度鲶(Clarias batrachus)hifα的研究与本研究结果也不一致,其表达量均在脑中最高。以上结果表明,hifs的表达同时具有物种差异性。
3.3 低氧诱导hifs在花鲈肝、鳃组织中的表达分析鱼类鳃器官是鱼体的主要呼吸器官并且与外界水环境直接接触,最先感知水体溶解氧的变化,同时鳃表面积大,具有调节渗透压、酸碱平衡,排泄废物等功能,相比于其他器官更容易受到低氧胁迫的影响,对鱼类的生长造成威胁[21-22]。同时肝脏是动物在低氧胁迫时进行代谢调整的关键器官[23],并且低氧会诱导肝细胞的凋亡[24-25]。研究花鲈hifs在低氧胁迫下鳃、肝组织中的表达情况,对研究花鲈的低氧应答机制具有理论意义。实验结果表明,hifs基因对低氧的响应时间和响应程度存在一些差异。肝组织中,低氧胁迫3 h后,hifs基因表达量均出现了显著升高,说明在低氧条件下肝器官能够迅速做出应对;而低氧胁迫12 h后,仅hif1α表达量显著高于对照组,其余基因与对照组相比差异不显著,由此可以看出hif1α基因在肝组织低氧调控12 h内持续起作用,而其它hifs基因仅在前6 h起作用。在鳃组织中,与肝组织中的变化规律相似,低氧胁迫3 h后hif1α、hif2α、hif3α显著升高,hif1β基因表达无显著差异。说明前3 h的低氧胁迫中,在鳃组织仅hifα基因起调控作用。而低氧胁迫6 h后,hif1β基因与hif3α基因表达量显著升高,其他基因表达无显著变化,说明6 h后,hif3α和hif1β基因共同调控低氧胁迫。关于其他鱼类hifα的研究结果与本实验相似,如印度鲶[20]、胭脂鱼(Myxocyprinus asiaticus)[26]、河鲈(Perca fluviatilis)[27]低氧胁迫后,肝中hifa基因的表达量显著升高,斑点叉尾鮰[14]受低氧胁迫5 h后,hifα各基因在鳃、肝中表达量均显著升高,许氏鲆鮋(Sebastes schlegeli)[15]、团头鲂(Megalobrama amblycephala)[17]低氧胁迫后,分别在肝、鳃组织中hif2α表达量显著升高。在hifs中,常氧条件下α亚基没有活性,当处于低氧环境时,α亚基会迅速被激活并与相应的低氧靶基因反应元件结合,促使基因转录,进而引发低氧应答[28]。β亚基则较为稳定,需与α亚基构成二聚体后响应低氧胁迫。在本实验中,肝组织中hif1β基因在低氧3和6 h显著升高,说明花鲈肝组织中hif1β基因可能是配合hifα基因的升高,相互作用共同响应低氧应答。相似的,鳃组织中在低氧胁迫6 h时hif1β、hif3α显著升高,此时可能是其相互作用响应低氧胁迫。hifs基因在对低氧的响应时间以及强度上存在显著差异,反映出各个基因在低氧应答中所起的作用以及表达模式不同,其具体的调控模式与分子机制还需进一步研究。
4 结语低氧诱导因子hifs是低氧应激的关键调节因子,在鱼类的环境缺氧应答中起着非常重要的作用。本实验鉴定、分析了花鲈4个hifs基因:hif1α、hif2α、hif3α、hif1β,并检测了各个基因的组织表达情况。结果表明花鲈4个hifs基因存在组织表达特异性,说明hifs参与了不同的生物学过程。进一步对hifs基因进行低氧胁迫后肝、鳃组织中的表达分析,结果表明不同基因在不同组织中对低氧的响应时间和响应程度存在显著差异。本实验对花鲈hifs基因对低氧的响应模式进行了初步的研究,其具体的生理与分子机制还需要进一步研究。
| [1] |
温海深, 张美昭, 李吉方, 等. 我国花鲈养殖产业现状与种子工程研究进展[J]. 渔业信息与战略, 2016, 31(2): 105-111. Wen H S, Zhang M Z, Li J F, et al. Research progress of aquaculture industry and its seed engineering in spotted sea bass (Lateolabrax maculatus) of China[J]. Fish Information & Strategy, 2016, 31(2): 105-111. (  0) 0) |
| [2] |
Solstorm D, Oldham T, Solstorm F, et al. Dissolved oxygen variability in a commercial sea-cage exposes farmed Atlantic salmon to growth limiting conditions[J]. Aquaculture, 2018, 486: 122-129. DOI:10.1016/j.aquaculture.2017.12.008
(  0) 0) |
| [3] |
徐贺, 陈秀梅, 王桂芹, 等. 低氧胁迫在水产养殖中的研究进展[J]. 饲料工业, 2016, 37(2): 33-37. Xu H, Chen X M, Wang G Q, et al. Research progress of hypoxia on aquaculture[J]. Feed Industry, 2016, 37(2): 33-37. (  0) 0) |
| [4] |
张思源, 柴志欣, 钟金城. 牦牛高原低氧适应研究进展[J]. 江苏农业科学, 2016, 44(3): 13-17. Zhang S Y, Chai Z X, Zhong J C. Research progress of hypoxia adaptation on plateau yak[J]. Jiangsu Agricultural Science, 2016, 44(3): 13-17. (  0) 0) |
| [5] |
Semenza G L. HIF-1: Mediator of physiological and pathophysiological responses to hypoxia[J]. Journal of Applied Physiology, 2000, 88(4): 1474-1480. DOI:10.1152/jappl.2000.88.4.1474
(  0) 0) |
| [6] |
Palazon A, Goldrath A, Nizet V, et al. HIF transcription factors, inflammation and immunity[J]. Immunity, 2014, 41(4): 518. DOI:10.1016/j.immuni.2014.09.008
(  0) 0) |
| [7] |
Jr K W, Ratcliffe P J. Oxygen sensing by metazoans: The central role of the HIF hydroxylase pathway[J]. Molecular Cell, 2008, 30(4): 393-402. DOI:10.1016/j.molcel.2008.04.009
(  0) 0) |
| [8] |
Prabhakar N R, Semenza G L. Adaptive and maladaptive cardiorespiratory responses to continuous and intermittent hypoxia mediated by hypoxia-inducible factors 1 and 2[J]. Physiol Reviews, 2012, 92(3): 967-1003. DOI:10.1152/physrev.00030.2011
(  0) 0) |
| [9] |
王卓, 张万起. 芳香烃受体核转位蛋白的结构及相关功能[J]. 生命科学, 2007, 19(1): 73-77. Wang Z, Zhang W Q. Constitutive function of the aryl hydrocarbon receptor nuclear translocator (ARNT)[J]. Chinese Bulletin of Life Sciences, 2007, 19(1): 73-77. DOI:10.3969/j.issn.1004-0374.2007.01.016 (  0) 0) |
| [10] |
Shimoda L A, Semenza G L. HIF and the lung: Role of hypoxia-inducible factors in pulmonary development and disease[J]. American Journal of Respiratory & Critical Care Medicine, 2011, 183(2): 152-156.
(  0) 0) |
| [11] |
Duan C. Hypoxia-inducible factor 3 biology: Complexities and emerging themes[J]. Am J Physiol Cell Physiol, 2015, 310(4): 260-269.
(  0) 0) |
| [12] |
Keith B, Johnson R S, Simon M C. HIF1a and HIF2a: Sibling rivalry in hypoxic tumour growth and progression[J]. Nature Reviews Cancer, 2011, 12(1): 9-22.
(  0) 0) |
| [13] |
Townley I K, Karchner S I, Skripnikova E, et al. Sequence and functional characterization of hypoxia-inducible factors, HIF1a, HIF2aa, and HIF3a, from the estuarine fish, Fundulus heteroclitus[J]. American Journal of Physiology Regulatory Integrative & Comparative Physiology, 2017, 312(3): 412.
(  0) 0) |
| [14] |
Geng X, Feng J, Liu S, et al. Transcriptional regulation of hypoxia inducible factors alpha (HIF-a) and their inhibiting factor (FIH-1) of channel catfish (Ictalurus punctatus) under hypoxia[J]. Comparative Biochemistry & Physiology Part B Biochemistry & Molecular Biology, 2014, 169(1): 38-50.
(  0) 0) |
| [15] |
Mu W, Wen H, Li J, et al. HIFs genes expression and hematology indices responses to different oxygen treatments in an ovoviviparous teleost species Sebastes schlegelii[J]. Marine Environmental Research, 2015, 110: 142-151. DOI:10.1016/j.marenvres.2015.04.008
(  0) 0) |
| [16] |
Louis A, Nguyen N T, Muffato M, et al. Genomicus update 2015: KaryoView and MatrixView provide a genome-wide perspective to multispecies comparative genomics[J]. Nucleic Acids Research, 2015, 43: 682-689. DOI:10.1093/nar/gku1312
(  0) 0) |
| [17] |
Shen R J, Jiang X Y, Pu J W, et al. HIF-1a and -2a genes in a hypoxia-sensitive teleost species Megalobrama amblycephala: cDNA cloning, expression and different responses to hypoxia[J]. Comparative Biochemistry & Physiology Part B Biochemistry & Molecular Biology, 2010, 157(3): 273-280.
(  0) 0) |
| [18] |
姜华鹏, 张驰, 王丛丛, 等. 软刺裸鲤和齐口裂鳆鱼HIF1B和HIF2A的克隆及低氧适应性的表达分析[J]. 淡水渔业, 2015, 45(5): 11-18. Jiang H P, Zhang C, Wang C C, et al. Cloning of HIF1B and HIF2A genes and their expressions response to hypoxia in Gymnocypris dobula and Schizothorax prenanti[J]. Freshwater Fisheries, 2015, 45(5): 11-18. DOI:10.3969/j.issn.1000-6907.2015.05.002 (  0) 0) |
| [19] |
张慧.黄颡鱼HIF-1a基因克隆与表达特征分析[D].保定: 河北大学, 2010. Zhang H. Cloning of the Hypoxia Inducible Factor-1a and Gene Expression Analysis of Pelteonagrus fulvidsco[D]. Baoding: Hebei University, 2010. http://cdmd.cnki.com.cn/Article/CDMD-10075-2010208677.htm (  0) 0) |
| [20] |
Mohindra V, Tripathi R K, Singh R K, et al. Molecular characterization and expression analysis of three hypoxia-inducible factor alpha subunits, HIF-1a, -2a and -3a in hypoxia-tolerant Indian catfish, Clarias batrachus (Linnaeus, 1758)[J]. Molecular Biology Reports, 2013, 40(10): 5805-5815. DOI:10.1007/s11033-013-2685-1
(  0) 0) |
| [21] |
陈世喜, 王鹏飞, 区又君, 等. 急性和慢性低氧胁迫对卵形鲳鲹鳃器官的影响[J]. 南方水产科学, 2017, 13(1): 124-130. Chen S X, Wang P F, Qu Y J, et al. Acute and chronic hypoxia effect on gills of golden pompano (Trachinotus ovatus)[J]. South China Fisheries Science, 2017, 13(1): 124-130. DOI:10.3969/j.issn.2095-0780.2017.01.016 (  0) 0) |
| [22] |
Nilsson G E. Gill remodeling in fish--a new fashion or an ancient secret?[J]. Journal of Experimental Biology, 2007, 210(14): 2403-2409. DOI:10.1242/jeb.000281
(  0) 0) |
| [23] |
Aluru N, Vijayan M M. Stress transcriptomics in fish: A role for genomic cortisol signaling[J]. General and Comparative Endocrinology, 2009, 164(2): 142-150.
(  0) 0) |
| [24] |
黎一鸣, 徐心, 吉鸿, 等. 肝动脉缺血对肝细胞凋亡的影响及其机制初探[J]. 西安交通大学学报(医学版), 2002, 23(4): 393-394. Li Y M, Xu X, Ji M, et al. Effects of hepatic artery ischemia on hepatocytic apoptosis and its mechanism[J]. Journal of Xi'an Jiaotong University (Med Sci), 2002, 23(4): 393-394. DOI:10.3969/j.issn.1671-8259.2002.04.021 (  0) 0) |
| [25] |
赵金坤, 梁宏伟, 邹桂伟, 等. 低氧胁迫对鲢肝和脑细胞凋亡的影响[J]. 西北农林科技大学学报(自然科学版), 2016, 44(7): 34-38. Zhao J K, Liang H W, Zou G W, et al. Influence of hypoxia stress on apoptosis of hepatocyte and brain cells of liver carp (Hypophthalmichthys molitrix)[J]. Joutnal of Northwest A&F University, 2016, 44(7): 34-38. (  0) 0) |
| [26] |
Nan Chen, Li Ping Chen, Jie Zhang, et al. Molecular characterization and expression analysis of three hypoxia-inducible factor alpha subunits, HIF-1a/2a/3a of the hypoxia-sensitive freshwater species, Chinese sucker[J]. Gene, 2012, 498(1): 81-90.
(  0) 0) |
| [27] |
Rimoldi S, Terova G, Ceccuzzi P, et al. HIF-1a mRNA levels in Eurasian perch (Perca fluviatilis) exposed to acute and chronic hypoxia[J]. Molecular Biology Reports, 2012, 39(4): 4009-4015. DOI:10.1007/s11033-011-1181-8
(  0) 0) |
| [28] |
肖武汉. 低氧信号传导途径与鱼类低氧适应[J]. 中国科学:生命科学, 2014, 44(12): 1227-1235. Xiao W H. Hypoxic signal transduction pathway and hypoxia adaptation in fish[J]. Chinese Science: Life Science, 2014, 44(12): 1227-1235. (  0) 0) |
 2020, Vol. 50
2020, Vol. 50


