磷脂酶D(Phospholipase D, PLD),也叫做磷脂酰胆碱磷脂水解酶(EC 3.1.4.4),属于磷酸二酯酶超家族中的一员[1],其主要作用于脂类物质的磷氧键,能够催化两类反应:磷脂的水解和转磷脂化反应,它既能够催化磷酸二酯键的水解,如水解磷脂酰胆碱生成胆碱和磷脂酸,又能催化碱基交换反应的进行,进而合成新型磷脂物质。通常可以利用PLD的转磷脂化反应进行磷脂的改性,从而合成新型的功能性磷脂,譬如磷脂酰甘油(Phosphatidylglycerol, PG)[2],磷脂酰乙醇胺(Phosphatidylethanolamine, PE)[3],富含二十二碳六烯酸的磷脂酰丝氨酸(Docosahexaenoic acid-containing phosphatidylserine, DHA-PS)[4],以及磷脂酰葡萄糖(Phosphatidyl-glucose)[5]等。
迄今为止,PLD在植物(集中在叶子、根、种子等部位)、动物(主要与细胞膜结合,分布在肝脏和脑等器官内)、微生物中均有发现,其中微生物来源较为广泛,已经报道的含有PLD的微生物有链霉菌(Streptomyces)、大肠杆菌(Escherichia coli)、沙门氏杆菌(Bacillus salmonelle)、棒状杆菌(Corynebacterium)、假单胞菌(Pseudomonas)、苍白杆菌(Ochrobactrum)、放线菌(Actinomycetes)、抗辐射不动杆菌(Acinetobacter radioresistens)等[6-8]。
微生物来源的PLD及其在不同宿主中的异源表达,更是当前的一大研究热点,其中使用率最高的就是大肠杆菌[9-11]表达系统,本实验室[12]曾利用大肠杆菌表达系统,对抗辐射不动杆菌(A. radioresistens a2)来源的PLD基因进行异源表达,并进行固定化研究。Chiaki Ogino等[13]利用变铅青链霉菌(Streptomyces lividans)对肉桂链霉菌(Streptoverticillium cinnamoneum)来源的PLD进行了异源表达,使PLD的表达量大大提高。张莹等[14]第一次使用毕赤酵母(Pichia pastoris)和解脂耶氏酵母(Yarrowia lipolytica)表达系统,实现了链霉菌属PLD的异源表达。Liu等[15]也曾用毕赤酵母表达系统对色褐链霉菌(Streptomyces chromofuscus)来源的PLD基因进行异源表达,并用于催化磷脂酰胆碱(PC)合成磷脂酰丝氨酸(PS)。路福平等[16]报道可以使用枯草芽孢杆菌表达系统对PLD进行异源表达。在本研究中,我们选用了重要的原核表达宿主之一——大肠杆菌进行PLD的异源表达。
海洋中富含生物资源,许多具有重要功能活性的酶均来源于海洋,而海洋来源的PLD却鲜少报道[17-18]。本研究对海洋来源的嗜麦芽窄食单胞菌(Stenotrophomonas maltophilia) OUC_Est10进行了基因组测序[19],经过对测序结果的分析,从中找到1个可能具有磷脂酶D活性的假定基因,并设计全长引物,对此基因进行扩增,与表达载体pET-28a(+)连接,构建重组质粒,导入到E. coli BL21(DE3)并进行表达。通过对S. maltophilia OUC_Est10中的PLD基因进行大肠杆菌异源表达,得到了重组的磷脂酶D-PLDEst10,通过酶联比色法[20]对PLDEst10的水解活力进行测定,并研究了该酶的米氏常数Km和最大反应速率Vmax及pH、温度等对酶活的影响;在水-乙醚双相体系下分别催化PC和DHA-PC转酯合成PS和DHA-PS,并通过薄层层析法和高效液相法对PLDEst10的转酯化酶活进行测定[21]。
1 材料与方法 1.1 菌株与载体S. maltophilia OUC_Est10由本实验室从海洋中筛选并保存[19]。
E. coli DH5α及E. coli BL21(DE3)购自天根生化科技有限公司。
大肠杆菌pET-28a(+)载体,购自全式金公司。
1.2 酶与试剂Phanta Max Super-Fidelity DNA Polymerase,购自诺唯赞生物科技有限公司;DNA分子量标准Marker,购自Takara公司;细菌基因组提取试剂盒,快速质粒小提试剂盒,购自天根生化科技有限公司;胶回收试剂盒,购自OMEGA公司;His标签蛋白纯化试剂盒,购自康为世纪生物科技有限公司;其他试剂均为分析纯级别。
1.3 主要溶液配置LB培养基:酵母粉5 g/L,蛋白胨10 g/L,NaCl 10 g/L,pH=7.4,121 ℃,灭菌20 min。
ZYP-5052培养基:每25 mL培养基中,加入5 g/L的酵母粉,10 g/L的蛋白胨,50 μL的1 mol/L MgSO4,1.25 mL的20×P (1 mol/L, Na2HPO4,1 mol/L KH2PO4,0.5 mol/L (NH4)2SO4),121 ℃灭菌20 min,使用时加入0.5 mL的50×5052母液(25%甘油,2.5%葡萄糖,10% α-乳糖)。
水解反应底物:取0.1 g 95% PC溶解在1 mL的乙醚中,再加入9 mL超纯水,在匀浆机作用下1 min形成乳浊状,4 ℃放置保藏,保质期为2周。
转酯反应底物:称取一定量的大豆来源的95% PC,利用乙醚溶解,终浓度为10 mg/mL;称取一定量的鱿鱼中提取的DHA-PC,利用乙醚溶解,终浓度为10 mg/mL。
1.4 实验方法 1.4.1 磷脂酶D基因的PCR扩增及产物纯化利用NCBI数据库与S. maltophilia OUC_Est10基因组序列进行比对分析,发现一段假定的PLD编码序列。按照预测得到的PLD基因开放阅读框(ORF)序列,设计引物Primer F (CCGGAATTCATGAGCCTGCCCAAGCCCCT)与Primer R (CCCAAGCTTCTACAGCTGCGATTCCACCG),含有EcoRI和HindⅢ两个限制性酶切位点,以提取的S. maltophilia OUC_Est10的基因组DNA为模板,使用高保真的DNA聚合酶,对PLD基因进行PCR扩增。反应体系(50 μL)如下:DNA模板1 μL(终浓度为50 ng),Phanta Max Super-Fidelity DNA Polymerase 1 μL(1 U),2×Phanta Max Buffer 25 μL(终浓度为2 mmol/L),前后引物分别为2 μL(终浓度为0.4 μmol/L),dNTP Mix 1 μL(终浓度为0.2 mmol/L),ddH2O 18 μL。反应条件为:95 ℃预变性3 min,95 ℃变性15 s,55 ℃退火15 s,72 ℃延伸2 min,重复变性到延伸步骤30个循环,75 ℃最终延伸5 min,10 ℃保温。PCR扩增结束后,根据目的产物的扩增效果,采用跑琼脂糖凝胶电泳的方法或者利用PCR产物纯化试剂盒进行产物回收。
1.4.2 PLDEst10-pET-28a(+)重组质粒的构建将PCR扩增得到的目的基因序列和pET-28a(+)质粒,分别用EcoRI和HindⅢ限制性内切酶进行双酶切,连接后转化E. coli DH5α,并通过卡那霉素抗性平板获得重组体的单菌落。经过阳性克隆验证,将阳性克隆子的质粒再转入到大肠杆菌菌株BL21中进行蛋白表达。
1.4.3 酶液制备及纯化将构建的产PLDEst10的E. coli BL21(DE3)接种到ZYP-5052培养基中,20 ℃发酵诱导表达48 h,收集菌体超声破碎,冷冻离心后得到PLDEst10粗酶液,经过0.45 μm滤膜过滤备用。
使用康为世纪生物科技有限公司的His标签蛋白纯化试剂盒,进行PLDEst10粗酶液的纯化,具体操作如下:首先收集菌体后,加入细菌裂解液,利用超声波破碎,冷冻离心,收集上清中的可溶性蛋白,过滤备用。然后组装层析柱,柱平衡结束后即可上蛋白样,控制液体加入速度来控制流速,收集穿透峰。纯化过程使用15倍柱体积的柱平衡缓冲液冲洗柱子,洗去杂蛋白。随后使用咪唑浓度为80 mmol/L的洗脱缓冲液进行洗脱,收集洗脱峰。洗脱结束后,依次使用10倍柱体积的去离子水洗涤柱子,再用3倍柱体积的20%乙醇平衡,封柱后2~8 ℃保存。纯化结束后,利用SDS-PAGE蛋白电泳进行表达纯化的检测。
1.4.4 酶学性质研究 1.4.4.1 PLDEst10的Km和Vmax测定采用双倒数法测定PLDEst10的Km和Vmax值。底物PC浓度分别为0.5、0.6、0.7、0.8、0.9、1.0、1.5、2.0、2.5、3.0和4.0 mmol/L,在37 ℃恒温水浴锅内反应10 min,进行显色反应,根据PLD水解活力-酶联比色法测定相应酶活[20],进一步计算Km和Vmax值。
1.4.4.2 PLDEst10水解活力最适pH和pH稳定性为考察pH对PLDEst10水解活力的影响,分别在pH为4.0~11.0的缓冲液中进行反应,反应结束后进行水解酶活力检测。为考察PLDEst10对pH的稳定性,将PLDEst10分别置于上述pH的缓冲液中,并在4 ℃冰箱内过夜放置,然后按照酶联比色法测定水解酶活,对残余酶活进行计算。
1.4.4.3 PLDEst10水解活力最适温度和温度稳定性为考察温度对PLDEst10水解活力的影响,分别将PLDEst10置于30、35、40、45、50、55和65 ℃条件下反应,并按照酶联比色法进行水解酶活的测定。为考察PLDEst10对温度的稳定性,将PLDEst10分别置于40、45和50 ℃下放置0、0.5、1、2、3和4 h,然后按照酶联比色法测定水解酶活,对残余酶活进行计算。
1.4.4.4 PLDEst10催化转酯反应转酯活力的测定采用粗酶粉,每组反应中冷冻酶粉用量均为60 mg,在两相反应体系[22]中利用PLDEst10催化合成PS和DHA-PS的反应体系为:1 mL 10 mg/mL乙醚溶解的大豆来源的磷脂PC/鱿鱼来源的磷脂DHA-PC与1 mL 1 mol/L丝氨酸溶液(溶于柠檬酸-柠檬酸钠缓冲液,20 mmol/L,pH 6.0),混匀后在40 ℃震荡反应12 h。反应产物利用毛细管点样吹干后,将硅胶板在展开剂(三氯甲烷:甲醇:水=65:25:4(v/v/v))中展开,然后利用碘蒸气显色。
2 结果与讨论 2.1 PLDEst10-pET-28a(+)重组质粒的构建如图 1A所示,根据假定PLD基因设计引物扩增出的片段与目的片段大小一致,其分子量大约为1 500 bp。图 1B显示,连接有PLDEst10编码基因的pET-28a(+)构建成功。将阳性克隆子导入到E. coli BL21(DE3)感受态细胞中。随后进行了质粒提取并送去测序,经验证符合预期结果,表明用于表达PLDEst10的E. coli BL21(DE3)工程菌株构建成功。
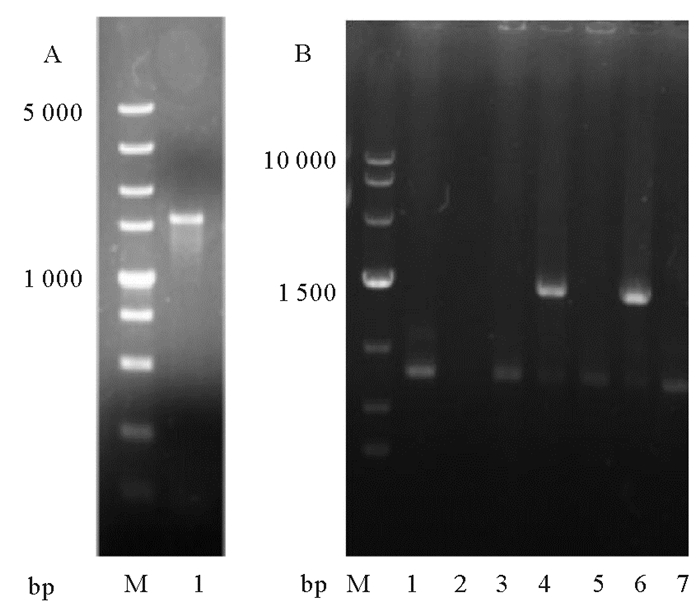
|
图 1 PCR扩增及阳性克隆验证结果 Fig. 1 Results of PCR amplification and positive clones verificatioin |
将构建成功的PLDEst10的表达工程菌株进行诱导培养,得到的PLDEst10粗酶液,用His标签蛋白纯化试剂盒对其进行纯化,将蛋白样品进行SDS-PAGE电泳检测,结果如图 2所示,与含pET-28a(+)空质粒的E. coli BL21(DE3)发酵上清液相比,表达PLDEst10的E. coli BL21(DE3)发酵上清液在63~75 kDa之间有一条明显的条带,预测的PLDEst10分子量约为60 kDa,与电泳结果相一致。已报道的磷脂酶D的分子量大小不等[23-24],分布在16~120 kDa之间,以嗜麦芽窄食单孢菌为PLD的基因来源,得到了新的磷脂酶D-PLDEst10,丰富了磷脂酶D的来源。
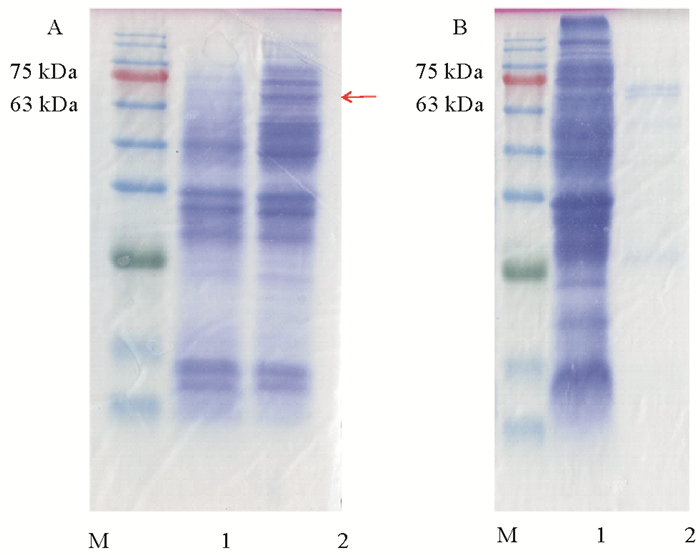
|
(A. M为Marker,1为含pET-28a(+)空质粒菌株上清,2为表达PLDEst10的E. coli BL21(DE3)上清;B. M为Marker,1为表达PLDEst10的E. coli BL21(DE3)上清,2为纯化的PLDEst10。A. M: Marker, 1: Supernatant of E. coli BL21(DE3) containing pET-28a(+),2: Supernatant of E. coli BL21(DE3) expressing PLDEst10.B. M: Marker, 1: Supernatant of E. coli BL21(DE3) expressing PLDEst10,2: Purified PLDEst10.) 图 2 重组PLDEst10的异源表达和纯化 Fig. 2 Heterologous expression and purification of recombinant PLDEst10 |
测定PLDEst10的Km和Vmax值时,底物反应速度随底物浓度而变化,当底物PC浓度在0.5~1.0 mmol/L增加的过程中,反应速度基本上成线性增加,当底物PC浓度超过1.0 mmol/L时,反应速度增加缓慢,当底物PC浓度达到2 mmol/L时,反应速度趋于稳定。根据双倒数法计算,得到PLDEst10的Km和Vmax值分别为Km=0.77 mmol/L,Vmax=1.33 mmol·(L·min)-1。
2.3.2 PLDEst10水解活力最适pH和pH稳定性为考察pH对PLDEst10的影响,将酶液在不同pH条件下催化底物PC水解,由图 3显示的结果可知,PLDEst10在pH=9.0的Tris-HCl缓冲液中酶活最高,以该条件下对应的酶活为100%计算,可以发现PLDEst10在pH为6.0~10.0之间相对酶活均高于80%,可见其pH适应范围广泛,已报道的大多数磷脂酶D在碱性条件下水解活力较好[25],与本研究结果相一致。不同缓冲液之间存在pH的交叉,经过对比可以发现不同的盐离子对PLDEst10的酶活有一定影响,在弱酸和强碱条件下,酶活差异更突出,pH同为6.0时,磷酸缓冲液中PLDEst10的酶活明显高于柠檬酸-柠檬酸钠缓冲液中的酶活,即使pH同为10.0,但不同缓冲液的碱性条件下,碳酸钠-碳酸氢钠缓冲液中PLDEst10的酶活比甘氨酸-氢氧化钠缓冲液的酶活低很多。
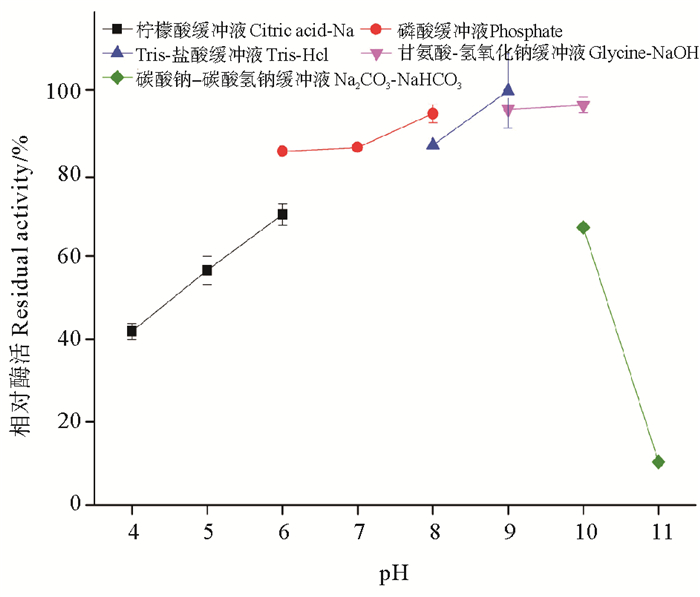
|
图 3 pH对PLDEst10酶活的影响 Fig. 3 Effect of pH on the relative activity of enzyme |
PLDEst10对于pH稳定性的研究中,将PLDEst10在不同pH环境下4 ℃保存过夜后,进行酶活的测定,结果显示PLDEst10在pH=9.0的Tris-HCl缓冲液中酶活保留最高,残存酶活为66.2%,将该酶活计为100%,由图 4可以看出,PLDEst10在pH为6.0~10.0的条件下相对残留酶活能在50%以上,表明PLDEst10对pH耐受性较好。而在pH为4.0~5.0的柠檬酸-柠檬酸钠缓冲液和pH为10.0~11.0的碳酸钠-碳酸氢钠缓冲液中,相对残留酶活均较低,说明弱酸和强碱对PLDEst10的酶活影响较大。相对来说,PLDEst10在酸性环境下的稳定性比在碱性环境中的保持得更好。
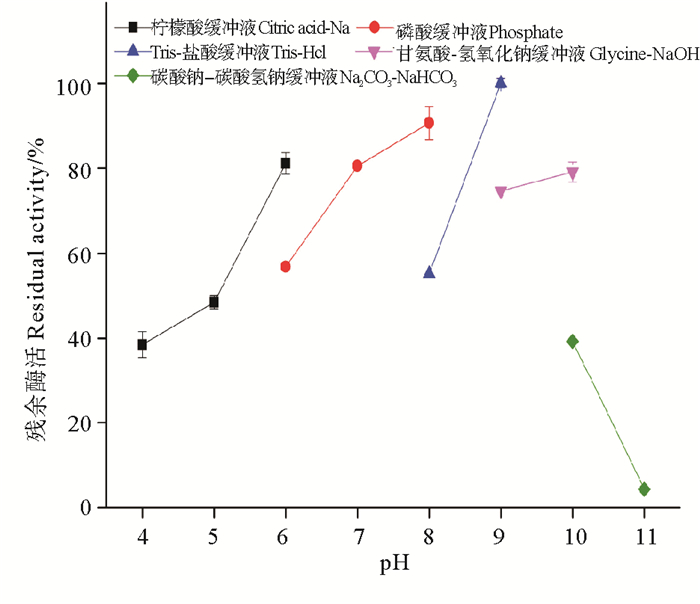
|
图 4 pH对PLDEst10稳定性的影响 Fig. 4 Effect of pH on the stability of PLDEst10 |
温度对PLDEst10酶活具有较大的影响,由图 5显示结果可知,反应温度是40 ℃时,PLDEst10的水解酶活力达到最高值,即40 ℃为最适温度,与抗辐射不动杆菌(A. radioresistens a2)为基因来源的PLDa2的最适反应温度相近[26],将该温度条件下测定的水解活力记为100%,在30~45 ℃范围内,相对酶活力仍能高于60%。而当温度高于45 ℃时,PLDEst10酶活出现显著降低,高温达到55~60 ℃时,酶活力下降至20%左右,高温会引起蛋白变性而影响酶活。
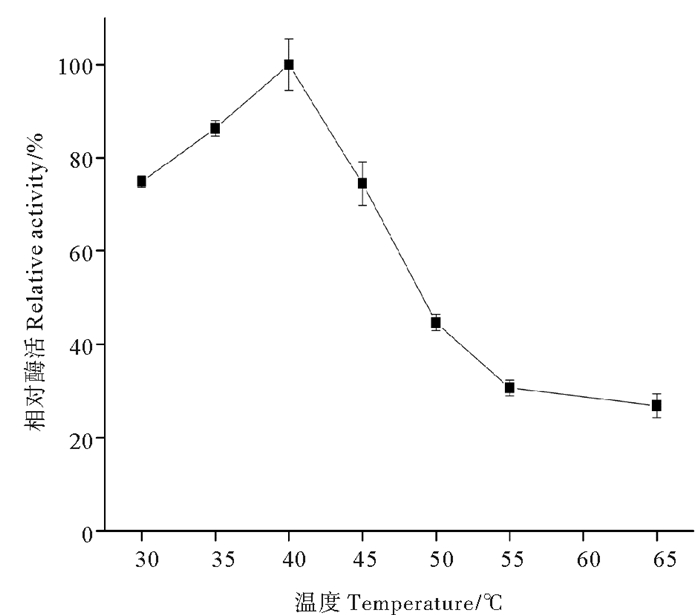
|
图 5 温度对PLDEst10酶活的影响 Fig. 5 Effect of temperature on the relative activity of PLDEst10 |
在温度稳定性方面,将40 ℃下的初始酶活记作100%,由图 6可知,40、45和50 ℃高温对PLDEst10酶活的影响都较大,当PLDEst10在40和45 ℃下放置30 min时,其酶活均出现明显下降,降低了50%左右,随后一直放置到4 h期间,酶活变化已经不太明显,呈现缓慢下降趋势。50 ℃对PLDEst10酶活的影响趋势,整体下降缓慢,应该是50 ℃条件下反应时,PLDEst10的蛋白结构本身已受到破坏,故在此高温放置更长时间,未产生更明显的影响。

|
图 6 温度对PLDEst10稳定性的而影响 Fig. 6 Effect of temperature on the stability of PLDEst10 |
根据文献[3, 8]报道,酸性条件更利于磷脂酶D催化转酯化反应,本研究中采用pH=6.0的柠檬酸-柠檬酸钠缓冲液,在两相体系下进行催化反应,利用反应产物中各组分的极性大小不同,在硅胶板上展开时迁移速率不同,从而实现磷脂各组分的分离,薄板层析结果如图 7所示,可以判断克隆表达出来的目的蛋白PLDEst10具有磷脂酶D的催化活性,能够催化PC和DHA-PC相应生成PS和DHA-PS。
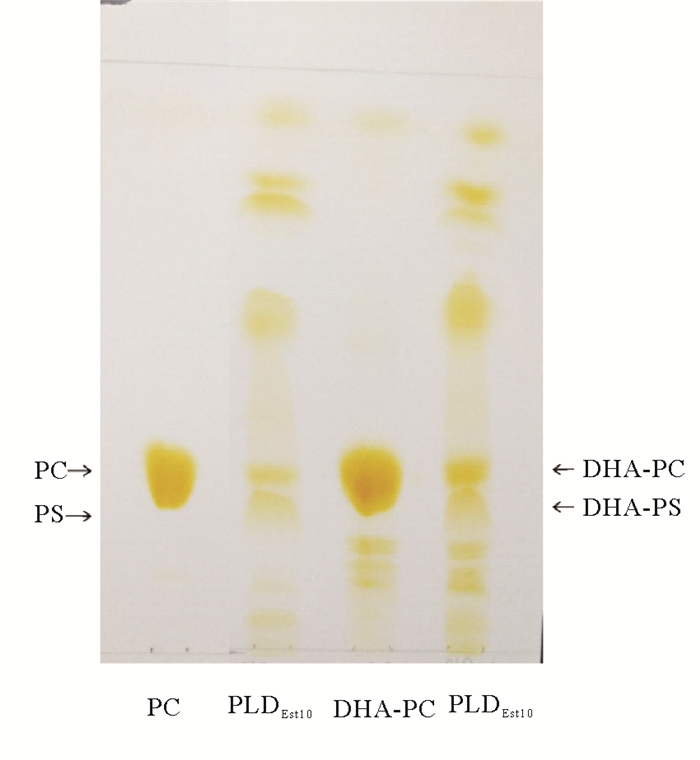
|
图 7 PLDEst10催化转酯产物的TLC图 Fig. 7 TLC analysis of transesterification product catalysed by PLDEst10 |
使用高效液相测定[26]也分别检测到了PS与DHA-PS的生成(见图 8),这进一步印证了TLC的结果。如图 8A所示,大部分底物PC转化为产物PS,说明PLDEst10具有较强的转酯活性。但另一方面,当以DHA-PC为底物时,仍有大量底物剩余,仅有少量DHA-PS生成(见图 8B),该结果显示PLDEst10对鱿鱼磷脂中DHA-PC的催化活性不及大豆磷脂PC,因此在将来的工作中需要进一步揭示PLDEst10的底物选择性机制,并在此基础上进行理性设计改造以获具有更高DHA-PS得率的人工设计PLD突变酶。
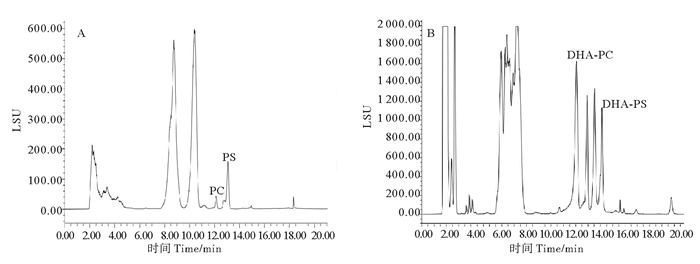
|
(A. PLDEst10催化PC合成PS;B. PLDEst10催化DHA-PC合成DHA-PS。A. PLDEst10 catalysed PC synthesis PS. B. PLDEst10 catalysed DHA-PC synthesis DHA-PS.) 图 8 PLDEst10催化转酯产物的HPLC图 Fig. 8 HPLC analysis of transesterification product catalysed by PLDEst10 |
(1) 从海洋来源嗜麦芽窄食单胞菌OUC_Est10中克隆并表达了PLDEst10,丰富了磷脂酶D基因来源。
(2) 采用酶联比色法测定了PLDEst10的酶学性质,其米氏常数和最大反应速率分别为Km=0.77 mmol/L,Vmax=1.33 mmol·(L·min)-1;最适反应温度是40 ℃;最适反应pH=9.0,且在中性和碱性条件下酶活较稳定。
(3) 采用水-乙醚双相体系进行催化转酯反应,经过薄层层析法和高效液相法测定,证实PLDEst10可以催化PC和DHA-PC分别生成功能性磷脂PS和DHA-PS,这为新型DHA磷脂的制备提供了重要的工具酶。
| [1] |
Waite M. The PLD superfamily: Insights into catalysis[J]. Biochimica et Biophysica Acta, 1999, 1439(2): 187-197. DOI:10.1016/S1388-1981(99)00094-3
(  0) 0) |
| [2] |
Simkhada J R, Lee H J, Jang S Y, et al. A novel low molecular weight phospholipase D from Streptomyces sp. CS684[J]. Bioresource Technology, 2009, 100(3): 1388-1393. DOI:10.1016/j.biortech.2008.09.008
(  0) 0) |
| [3] |
邓杨敏, 李冰麟, 董文博, 等. 磷脂酰乙醇胺的酶促合成及底物抑制动力学[J]. 化工进展, 2017, 36(7): 2601-2606. DENG Yang-min, LI Bing-lin, DONG Wen-bo, et al. Synthesis of phosphatidylethanolamine by enzymatic catalysis and its substrate inhibition kinetics[J]. Chemical Industry and Engineering Progress, 2017, 36(7): 2601-2606. (  0) 0) |
| [4] |
Masashi H, Tatuya S, Teruyuki K, et al. Conversion to docosahexaenoic acid-containing phosphatidylserine from squid skin lecithin by phospholipase D-mediated transphosphatidylation[J]. Journal of Agricultural and Food Chemistry, 2000, 48(10): 4550-4554. DOI:10.1021/jf991186s
(  0) 0) |
| [5] |
Song S, Cheong L Z, Guo Z, et al. Phospholipase D (PLD) catalyzed synthesis of phosphatidyl-glucose in biphasic reaction system[J]. Food Chemistry, 2012, 135(2): 373-379. DOI:10.1016/j.foodchem.2012.05.020
(  0) 0) |
| [6] |
Nakazawa Y, Uchino M, Sagane Y, et al. Isolation and characterization of actinomycetes strains that produce phospholipase D having high transphosphatidylation activity[J]. Microbiological Research, 2009, 164(1): 43-48. DOI:10.1016/j.micres.2006.11.003
(  0) 0) |
| [7] |
钱娟, 庞洋, 王昕. 磷脂酶D的制备及催化磷脂酰胆碱合成磷脂酰丝氨酸[J]. 中国油脂, 2017, 42(9): 66-71. QIAN Juan, PANG Yang, WANG Xin. Preparation of phospholipase D and catalytic synthesis of phosphatidylserine from phosphatidylcholine[J]. China Oils and Fats, 2017, 42(9): 66-71. DOI:10.3969/j.issn.1003-7969.2017.09.013 (  0) 0) |
| [8] |
Mao X Z, Liu Q Q, Qiu Y Q, et al. Identification of a novel phospholipase D with high transphosphatidylation activity and its application in synthesis of phosphatidylserine and DHA-phosphatidylserine[J]. Journal of Biotechnology, 2017, 249: 51-58. DOI:10.1016/j.jbiotec.2017.03.029
(  0) 0) |
| [9] |
Lee J S, Bat-Ochir M, Demirev A V, et al. Molecular cloning of the phospholipase D gene from Streptomyces sp. YU100 and its expression in Escherichia coli[J]. Journal of Microbiology, 2009, 47(1): 116-122. DOI:10.1007/s12275-008-0161-8
(  0) 0) |
| [10] |
Zambonelli C, Morandi P, Vanoni M A, et al. The gene encoding Streptomyces PMF PLD, a phospholipase D with high transphosphatidylation activity[J]. Enzyme and Microbial Technology, 2003, 33(5): 676-688. DOI:10.1016/S0141-0229(03)00190-X
(  0) 0) |
| [11] |
Yang L, Xu Y, Chen Y, et al. Efficient extracellular expression of Phospholipase D in Escherichia coli with an optimized signal peptide[J]. IOP Conf. Series: Materials Science and Engineering, 2018, 301(1): 012105.
(  0) 0) |
| [12] |
刘倩倩, 邱永乾, 刘炯钦, 等. 大孔树脂固定化磷脂酶D[J]. 工业微生物, 2016, 46(3): 25-30. LIU Qian-Qian, QIU Yong-Qian, LIU Jiong-Qin, et al. Immobilization of phospholipase D by Macroporous resin[J]. Industrial Microbiology, 2016, 46(3): 25-30. DOI:10.3969/j.issn.1001-6678.2016.03.005 (  0) 0) |
| [13] |
Ogino C, Kubo T, Yoshino K, et al. Over-expression system for secretory phospholipase D by Streptomyces lividans[J]. Applied Microbiology and Biotechnology, 2004, 64(6): 823-828. DOI:10.1007/s00253-003-1552-8
(  0) 0) |
| [14] |
张莹.在不同宿主中表达链霉菌磷脂酶D的研究[D].上海: 华东理工大学, 2013. ZHANG Ying. Phospholipase D Safe and Efficient Expression in Different Hosts[D]. Shanghai: East China University of Science and Technology, 2013. http://www.wanfangdata.com.cn/details/detail.do?_type=degree&id=Y2509983 (  0) 0) |
| [15] |
Liu Y, Zhang T, Qiao J, et al. High-yield phosphatidylserine production via yeast surface display of phospholipase D from Streptomyces chromofuscus on Pichia pastoris[J]. Journal of Agricultural and Food Chemistry, 2014, 62(23): 5354-5360. DOI:10.1021/jf405836x
(  0) 0) |
| [16] |
张业尼, 路福平, 李玉, 等. 磷脂酰丝氨酸合成酶基因的克隆及在枯草芽孢杆菌中的表达[J]. 中国生物工程杂志, 2008, 28(9): 56-60. ZHANG Ye-Ni, LU Fu-Ping, LI Yu, et al. Cloning of the phosphatidylserine synthese gene and its secretive expression in Bacillus subtilis[J]. China Biotechnology, 2008, 28(9): 56-60. (  0) 0) |
| [17] |
Lu P, Gao X W, Dong H, et al. Identification of a novel esterase from marine environmental genomic DNA libraries and its application in production of free all-trans-astaxanthin[J]. Journal of Agricultural and Food Chemistry, 2018, 66(11): 2812-2821. DOI:10.1021/acs.jafc.7b06062
(  0) 0) |
| [18] |
Liang Y X, Ma X Q, Zhang L J, et al. Biochemical characterization and substrate degradation mode of a novel exotype beta-agarase from Agarivorans gilvus WH0801[J]. Journal of Agricultural and Food Chemistry, 2017, 65(36): 7982-7988. DOI:10.1021/acs.jafc.7b01533
(  0) 0) |
| [19] |
Dong H, Rui J, Sun J A, et al. Complete genome sequencing and diversity analysis of lipolytic enzymes in Stenotrophomonas maltophilia OUC_Est10[J]. Acta Microbiologica Sinica, 2017, 57(11): 1-6.
(  0) 0) |
| [20] |
邱永乾.微生物脂肪酶库构建及磷脂酶D的筛选与应用[D].青岛: 中国海洋大学, 2015. QIU Yong-qian. Constructions of Lipase Producing Strains Library and Screeing of PLD Producing Strains[D]. Qingdao: Ocean University of China, 2015. http://www.wanfangdata.com.cn/details/detail.do?_type=degree&id=Y2900216 (  0) 0) |
| [21] |
汪勇, 王兴国, 胡学烟. 磷脂含量及组成的分析检测方法[J]. 中国油脂, 2002, 27(4): 64-67. WANG Yong, WANG Xing-Guo, HU Xue-Yan. Methods for determination and analysis of phospholipids composition and content[J]. China Oils and Fats, 2002, 27(4): 64-67. DOI:10.3321/j.issn:1003-7969.2002.04.023 (  0) 0) |
| [22] |
Hagishita T, Nishikawa M, Hatanaka T. Isolation of phospholipase D producing microorganisms with high transphosphatidylation activity[J]. Biotechnology Letters, 2000, 22(20): 1587-1590. DOI:10.1023/A:1005644032415
(  0) 0) |
| [23] |
Simkhada J R, Lee H J, Jang S Y, et al. A novel low molecular weight phospholipase D from Streptomyces sp. CS684[J]. Bioresource Technology, 2009, 100(3): 1388-1393.
(  0) 0) |
| [24] |
Ulbrich-Hofmann R, Lerchner A, Oblozinsky M, et al. Phospholipase D and its application in biocatalysis[J]. Biotechnology Letters, 2005, 27(8): 535-544. DOI:10.1007/s10529-005-3251-2
(  0) 0) |
| [25] |
Li B L, Wang J, Zhang X L, et al. An enzyme net coating the surface of nanoparticles: A simple and efficient method for the immobilization of phospholipase D[J]. Industrial and Engineering Chemistry Research, 2016, 55(40): 10555-10565. DOI:10.1021/acs.iecr.6b02192
(  0) 0) |
| [26] |
刘倩倩.磷脂酶D克隆表达和固定化合成磷脂酰丝氨酸工艺研究[D].青岛: 中国海洋大学, 2016. LIU Qian-qian. Study of Cloning Expression Phospholipase D and Immobilized Synthetic Phosphatidylserine[D]. Qingdao: Ocean University of China, 2016. (  0) 0) |
 2018, Vol. 48
2018, Vol. 48


